Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Boletín de la Sociedad Botánica de México
Print version ISSN 0366-2128
Bol. Soc. Bot. Méx n.88 México Jun. 2011
Genética
Diversidad genética de Oreopanax xalapensis (Araliaceae) en Los Altos de Chiapas
Genetic diversity of Oreopanax xalapensis (Araliaceae) in the Highlands of Chiapas
Lorena Ruiz–Montoya1, Verenyzee Correa–Vera, Fanny Carely Alfaro–González, Neptalí Ramírez–Marcial y Rodrigo Verónica–Vallejo
Departamento de Ecología y Sistemática Terrestres, El Colegio de la Frontera Sur, Carretera Panamericana y Periférico Sur S/N, C.P. 29290, Barrio Ma. Auxiliadora, San Cristóbal de Las Casas, Chiapas. 1Autora para correspondencia: lruiz@ecosur.mx
Recibido: 19 de julio del 2010.
Aceptado: 12 de abril de 2011.
Resumen
La diversidad genética de una especie determina su capacidad de respuesta a los cambios ambientales, por lo que su conocimiento puede ser de importancia para el diseño de estrategias de restauración de bosques. En este estudio se analiza la diversidad y estructura genética de cinco poblaciones de Oreopanax xalapensis en Los Altos de Chiapas. Esta especie se está utilizando en prácticas de restauración del bosque mesófilo de montaña en la región, pero se desconocen los niveles de variación genética de las poblaciones naturales de las que se obtienen las semillas. Para la descripción genética se usaron 18 loci enzimáticos evidenciados en acetato de celulosa, y con base en las frecuencias genotípicas se determinaron las frecuencia alélicas y se obtuvieron los siguientes estimadores de diversidad genética: polimorfismo, heterocigosidad observada (HO) y esperada (HE), y el número de alelos promedio. El estadístico FST se utilizó para estimar el grado de diferenciación entre subpoblaciones. El número de alelos promedio fue de 1.8–2.0 por locus; se registró un polimorfismo de 66 al 100%. El promedio de HO fue de 0.362, y HE de 0.379. El grado de estructuración fue significativamente alto (FST = 0.214), la estimación indirecta de flujo genético entre las subpoblaciones fue de Nm = 5.0 individuos por generación. Las subpoblaciones de O. xalapensis son genéticamente diversas y se encuentran estructuradas; lo anterior implica que para una práctica de restauración efectiva debiera considerarse la colecta de semillas de todas las subpoblaciones para tener una mejor representatividad de su diversidad genética.
Palabras clave: bosque mesófilo de montaña, diferenciación genética, flujo genético, Oreopanax xalapensis, restauración de bosques.
Abstract
The genetic diversity of a species determines its ability to respond to environmental changes, thus their knowledge can be important for designing strategies for forest restoration. This study analyzes the diversity and genetic structure of five populations of Oreopanax xalapensis in the Highlands of Chiapas. This species is being used for restoration of the montane cloud forests into the region, but unknown levels of genetic variation in natural populations where seeds are obtained. We used 18 enzymatic loci screened by electrophoresis in cellulose acetate. Genotype frequencies were used to calculate the allelic frequency and obtained the following estimates of genetic diversity: proportion of polymorphism, observed (HO) and expected heterocigosity (HE); and average number of alleles. The FST statistic was used to estimate the level of genetic differentiation among subpopulations. The average number of alleles per locus was between 1.8 and 2.0; the proportion of loci polymorphic ranged from 66 to 100%. Average HO was 0.362, and HE = 0.379. The degree of structure was significantly high (FST = 0.214), the indirect estimate of gene flow among subpopulations was Nm = 5.0 individuals per generation. Subpopulations of O. xalapensis are genetically diverse and are structured; this implies that for an effective restoration practice should be considered the collection of seeds of all populations for a better representation of genetic diversity.
Key words: forest restoration, genetic differentiation, genetic flow, montane cloud forest, Oreopanax xalapensis.
La fragmentación forestal es el proceso de reducción y separación de áreas o superficies boscosas. Los efectos de la fragmentación se reflejan directamente en una reducción de los tamaños poblacionales de árboles, especialmente en especies asociadas a comunidades sucesionales tardías (Cayuela et al., 2006b; Golicher et al., 2008); en casos extremos este proceso puede llevar a la extinción local (Dobson, 1999; Lande, 1999; Frankham et al., 2002; Paschke et al., 2002), debido a que las poblaciones pequeñas son más vulnerables a las fluctuaciones y/o catástrofes ambientales como incendios, inundaciones o sequías (Keller y Waller, 2002).
La conservación de especies incluye el mantenimiento de la diversidad genética, albergada dentro o entre poblaciones, de manera que las especies se conserven como entidades dinámicas capaces de responder ecológica y genéticamente a los cambios ambientales, producto de las actividades humanas o eventos naturales, de manera que la evolución de las especies continúe (Caro y Laureson, 1994; Dobson, 1999; Lande, 1999; Frankham et al., 2002; Stockwell et al., 2003).
Un sistema ecológico ideal para evaluar los efectos de la fragmentación forestal sobre la diversidad genética de árboles es el bosque mesófilo de montaña (BMM), debido a su propia condición "insular", por distribuirse de manera discontinua en condiciones ambientales específicas y por ser uno de los tipos de vegetación con mayor diversidad en México (Rzedowski, 1992). En este tipo de bosque se han registrado al menos 2,500 especies de plantas vasculares que crecen exclusiva o preferentemente ahí (Rzedowski, 1996). Por su alto valor estratégico para la conservación este tipo de ecosistemas se considera uno de los más amenazados del mundo, pues las tendencias sugieren una reducción de su superficie y empobrecimiento florístico (Bubb et al., 2004; Toledo–Aceves et al., 2011).
En Chiapas, el BMM se distribuye fundamentalmente en dos áreas: en la vertiente septentrional de la Altiplanicie Central, en las Montañas del Norte y en ambas declives de la Sierra Madre (Ramírez–Marcial et al., 2010a). En estas áreas se ha reconocido que el cambio de uso del suelo ha sido uno de los factores principales del empobrecimiento florístico (González–Espinosa et al., 2007a, b; 2008). La agricultura de milpa y producción de café, la extracción de leña y madera, la ganadería de ladera y el establecimiento de nuevos centros de población humana, han reducido y fragmentado notablemente el BMM (González–Espinosa et al., 1995; Ochoa–Gaona y González–Espinosa, 2000; Cayuela et al., 2006a).
Durante las últimas dos décadas, se han realizado distintos esfuerzos para la conservación y restauración del BMM dentro de territorios predominantemente indígenas de Chiapas (González–Espinosa et al., 2007a, b, 2008). Una estrategia viable de conservación y recuperación de especies del BMM en Chiapas ha sido la reintroducción de individuos de especies nativas con diferente afinidad sucesional (Quintana–Ascencio et al., 2004; Ramírez–Marcial et al., 2005, 2008, 2010b). El uso de especies nativas no sólo contribuye a la recuperación de áreas boscosas, sino que puede ayudar a emular la estructura, función, diversidad y dinámica del bosque original. Esta visión de la restauración de un bosque requiere la aplicación de conocimientos de sucesión ecológica, biología de poblaciones y genética evolutiva (Hufford y Mazer, 2003).
La introducción de plantas en áreas deterioradas o perturbadas es un procedimiento común y relativamente efectivo para la conservación in situ del BMM; sin embargo, a largo plazo las poblaciones pueden declinar, o no crecer, debido a factores genéticos que no se consideraron durante el establecimiento inicial (Li et al., 2005). Algunas experiencias de restauración con poblaciones de herbáceas indican que pueden ocurrir fenómenos genéticos como el cuello de botella, el efecto fundador, depresión por endogamia, exogamia y barrido genético (genetic swamping, Montalvo et al., 1997; Robichaux et al., 1997; Lessica y Allendorf, 1999; Montalvo y Ellstrand, 2000; Hufford y Mazer, 2003; Glaubitz et al., 2003), pero con árboles se conoce aún poco (Glaubitz et al., 2003; Li et al., 2005). Una baja diversidad genética de las poblaciones introducidas puede conducir a procesos de endogamia, que a su vez incrementa las tasas de mortalidad porque se expresan genes recesivos que impactan negativamente en componentes de adecuación de las plantas (Hamrick et al., 2006; Keller y Waller, 2002; Futuyma, 2009).
Una especie típica del BMM en México es Oreopanax xalapensis (Kunth) Decne. & Planch., cuya estructura genética microespacial ya ha sido estudiada en Chiapas (Vera–Maloof, 2009). En dicho trabajo se analizó la estructura genética de subpoblaciones de O. xalapensis considerando plántulas, juveniles y adultos en tres condiciones sucesionales del BMM (bosque incipiente, intermedio y maduro). El estudio registró variación en polimorfismo, en la diversidad genética [medida como heterocigosidad esperada HE] entre fases sucesionales y estados de desarrollo, cuyo promedio fue de HE = 0.31 y una diferenciación entre las subpoblaciones del 23% (FST). Este es un estudio a escala local y se desconoce si los parámetros genéticos son constantes a una escala espacial mayor (regional). Por otro lado, la descripción de la estructura genética de las poblaciones de especies de árboles que componen el BMM de Chiapas, en este caso O. xalapensis, puede ser importante para avanzar en el conocimiento de los efectos del movimiento deliberado de individuos (semillas o plántulas) durante las prácticas de restauración sobre la estructura genética poblacional.
Por lo anterior, consideramos que una primera aportación a este componente lo constituye la descripción de la estructura genética de las poblaciones naturales remanentes de algunas de las especies típicas del BMM. Los objetivos de este estudio fueron conocer: (1) la magnitud de la diversidad genética que contiene O. xalapensis en Los Altos de Chiapas, y cómo se distribuye dentro y entre subpoblaciones, (2) cuál es el flujo genético y (3) explorar cualitativamente si la diversidad genética está asociada al grado de intervención humana. Debido a que O. xalapensis es una de las especies utilizadas en los programas de restauración del BMM de Chiapas (Ramírez–Marcial et al., 2005, 2010b), se espera que los resultados derivados del presente estudio sean de utilidad para establecer mejores prácticas de restauración considerando atributos de diversidad genética.
Materiales y método
Descripción de la especie. Oreopanax xalapensis (Kunth) Decne. & Planch. (Araliaceae) es un árbol de 8 a 10 m de altura y de hasta 60 cm de diámetro. Se desarrolla preferentemente en sitios húmedos y protegidos de la insolación directa, es conocida en Chiapas como yich'acmut (pata de pájaro) o mazorca (Ramírez–Marcial et al., 2010a). En México se distribuye a lo largo de la vertiente del Golfo desde el norte de Veracruz hasta el norte de Chiapas, y en la vertiente del Pacífico desde Jalisco hasta Chiapas en altitudes entre 800 a 2,700 m, y se le encuentra también en Guatemala, Honduras, El Salvador, Nicaragua, Costa Rica hasta Panamá (Rzedowski y McVaugh, 1996; Pennington y Sarukhán, 1998). Esta especie es un elemento común del interior de los bosques de pino–encino, pino–encino–liquidámbar, de encino y mesófilo de montaña y frecuentemente se asocia con bosques secundarios de diversa composición y edad sucesio–nal (Meave et al., 1992). En Chiapas es común encontrarla en asociación con Myrsine juergensenii, Rhamnus sharpii, Styrax magnus y Ternstroemia lineata (Quintana–Ascencio et al., 2004). Es una especie morfológicamente plástica que puede presentar el tronco recto y la copa densa o ramificada a partir de pocos metros de altura. La floración ocurre entre septiembre y octubre, y la fructificación entre abril y mayo (Ramírez–Marcial et al., 2010a). La multiplicación de los individuos puede ser de manera vegetativa o sexual (Ruiz–Ruvalcaba, 2004). Las inflorescencias son extremadamente variables en tamaño y número de cabezuelas, se presentan en racimos de capítulos hasta de 50 cm de largo. Los frutos cambian de coloración durante su maduración en una secuencia que va del verde, seguido por tonos blanquecinos y finalmente maduran con colores pardos o violáceos (Lundell, 1966; Sosa, 1979). Los frutos son dispersados por algunas especies de aves y pequeños roedores (Ruíz–Ruvalcaba, 2004, J.L. Rangel, com. pers.).
Selección de sitios. Con base en las similitudes florísticas y estructurales se eligieron los siguientes sitios dentro de la región de Los Altos de Chiapas:
(1) Estación Biológica Cerro Huitepec (Huitepec), se ubica a 4.5 km al oeste de la ciudad de San Cristóbal de Las Casas, Chiapas. Geográficamente se localiza entre 16° 44' 38" Norte y 92° 40'15" Oeste, con altitudes que oscilan desde los 2,230 hasta los 2,710 m. Forma parte de un cono cinerítico cuyo origen geológico data del terciario y está constituido por una serie de laderas con pendientes pronunciadas (40–60%). Las especies del dosel predominantes son Clethra chiapensis, Cleyera theoides, Persea americana, Quercus laurina, Q. crassifolia y Q. rugosa (Ramírez–Marcial et al., 1998).
(2) Rancho Merced–Bazom (Bazom), se localiza a 16 km al este de San Cristóbal de Las Casas, en el municipio de Huixtán (16°44'50" N y 92° 29'45" O) entre 2,4002,500 m de altitud. La vegetación incluye un mosaico heterogéneo de comunidades sucesionales del bosque de pino–encino. Las especies predominantes a nivel del dosel son Pinus montezumae, P. pseudostrobus, P. tecunumanii, Quercus laurina, Q. crispipilis, Q. crassifolia y Q. rugosa (González–Espinosa et al., 1991, 2007b).
(3) Mitzitón (Mitzitón), se localiza a 13 km en línea recta al sureste de San Cristóbal de Las Casas entre los 16°39' 52" N y 92°32' 44" O, con una variación altitudinal de 2,380–2,500 m. Aunque comparte similitudes florísticas con Bazom, la dominancia de Pinus spp. es mayor en Mitzitón (Galindo–Jaimes et al., 2002).
(4) Cerro Tzontehuitz (Tzontehuitz), es la cima más elevada (2,910 m) dentro de la región, se localiza a los 16° 48' 49.5'' N, 92° 34' 48'' O; junto con Huitepec se consideran los últimos dos fragmentos extensos de vegetación de BMM en la región (Ramírez–Marcial et al., 2010a).
(5) La Yerbabuena (Yerbabuena), se localiza en el municipio de Pueblo Nuevo Solixtahuacán a los 17° 10' 28''N, 92° 53' 57''O con una variación altitudinal entre los 1,900 a 2,200 m y pendientes de medias a altas (hasta 60°). La vegetación dominante corresponde al bosque de pino–encino–liquidámbar, destacando como especies dominantes del dosel Cleyera theoides, Quercus benthamii y Podo–carpus matudae (Ramírez–Marcial et al., 2001).
Grado de perturbación. Todos los sitios elegidos se consideran que tienen una historia evolutiva común, pero que las diferencias actuales se deben al distinto régimen de perturbación humana. En todos ellos ha ocurrido algún tipo de perturbación humana como extracción de leña, madera y otros productos forestales (González–Espinosa et al., 2007a). La composición florística y la dominancia de algunas especies son indicadoras del tiempo que han sido intervenidos (Ramírez–Marcial et al., 2001; Galindo–Jaimes et al., 2002). Por ejemplo, la dominancia en el dosel de especies como Clethra chiapensis, Cleyera theoides, Persea americana, Quercus laurina, Q. crassifolia y Q. rugosa indican una fase sucesional avanzada o de baja intervención humana (Ramírez–Marcial et al., 1998; Quintana–Ascencio et al., 2004). En cambio, los bosques con mayor dominancia de Pinus spp. indican que han estado sometidos a una perturbación de mayor frecuencia y/o intensidad y por tanto se consideran en estados sucesionales tempranos (Galindo–Jaimes et al., 2002). Estas perturbaciones de baja intensidad pero frecuentes promueven un incremento en la temperatura y reducción de la humedad del aire y del suelo que se refleja en un bajo reclutamiento de plántulas de árboles tolerantes a la sombra (Ramírez–Marcial et al., 2001; Galindo–Jaimes et al., 2002). En cambio, en los otros sitios más conservados, se han registrado una continuidad natural del bosque y la regeneración de numerosas especies de árboles tolerantes, como Oreopanax xalapensis (Vera–Maloof, 2009). Con base en la información disponible sobre la composición florística y estructura de los sitios (González–Espinosa et al., 1991; Ramírez–Marcial et al., 1998, 2001, 2010a; Galindo–Jaimes et al., 2002), se estableció el siguiente orden de mayor a menor grado de perturbación: Mitzitón > Bazom > Tzontehuitz > Yerbabuena > Huitepec.
Análisis genético. Se colectó material vegetativo (hojas tiernas) de 30 individuos adultos por sitio. Los individuos colectados en cada sitio constituyeron una subpoblación y la unión de todos ellos representó a la población. Los individuos se eligieron al azar y cuidando que la distancia entre uno y otro fuera mayor a 10 m. Las muestras se conservaron en nitrógeno líquido y posteriormente en un ultracongelador a –70 °C hasta realizar la electroforesis de enzimas en acetato de celulosa (Herbet y Beaton, 1993). Se maceró una muestra de un 1 cm2 de hoja en 250 μl de una solución de 3:1 partes de YO y VEG II respectivamente (Yeh y O'Malley, 1980; Cheliak y Pitel, 1984). Posteriormente se centrifugó a 13,000 rpm durante dos minutos, el sobrenadante se usó para la separación de 12 enzimas (Cuadro 1). Los corrimientos se realizaron en condiciones de temperatura ambiente, a 120 V y 50 mA durante 120 minutos. Las bandas o variantes enzimáticas se notaron mediante los protocolos de tinción de Hebert y Beaton (1993). Se identificaron 18 loci enzimáticos (la mayoría con dos alelos), con base en la distancia de desplazamiento y separación entre las variantes enzimáticas. Se tomaron en cuenta aquellas bandas que fueron claramente visibles y consistentes con la estructura y loci esperados para las distintas enzimas (Weeden y Wendel, 1989a, b).
Análisis. Con las frecuencias genotípicas se calcularon las frecuencias alélicas y a partir de ellas se obtuvieron cinco parámetros para estimar la diversidad genética de las poblaciones de Oreopanax xalapensis: (1) la proporción de loci polimórficos, (2) la heterocigosidad promedio observada (HO), y la heterocigosidad esperada (HE) sin sesgo (Nei, 1978), (3) el índice de fijación como f = 1– HO / HE, (4) el número de alelos promedio por locus (Hartl y Clark, 1997) y (5) el índice de diversidad de Shannon (I). También se evaluó el equilibrio de Hardy–Weinberg (EHW) mediante una prueba de χ2. Todos los parámetros genéticos previos se obtuvieron con el programa GenAlex ver 6.41 (Peakall y Smouse, 2006). La heterogeneidad de las frecuencias alélicas entre las poblaciones se valoró mediante una prueba Ji–Cuadrada 
donde Nj es el tamaño de muestra en la j–ésima subpoblación, y N es la muestra total, pj y  es la frecuencia alélica en la j–ésima subpoblación y la media de la frecuencia alélica respectivamente (Hedrick, 2000).
es la frecuencia alélica en la j–ésima subpoblación y la media de la frecuencia alélica respectivamente (Hedrick, 2000).
La distancia genética se evaluó calculando el índice de distancia genética de Nei (1978) y a partir de la matriz se realizó una agrupación por el método de UPGMA en el programa TFPGA (Miller, 1997). Se calcularon los estadísticos o coeficientes F de Wright en GenAlex ver 6.41 (Peakall y Smouse, 2006); mediante estos estadísticos es posible estimar la reducción de la heterocigocidad individual promedio con relación a toda la población (FIT), con las subpoblaciones (FIS) y de la heterocigosidad de las subpoblaciones con respecto al total (FST). Se evaluó si FIT y FIS diferían de cero con una prueba de χ2 = F2N(k–1), con gl = (k(k–1), donde k es el número de alelos observados; y para FST se usó χ2 = 2NFST (k–1) con gl = (k–1) (s–1) donde N es el tamaño de la población, k es el número de alelos y s es el número de subpoblaciones (Workman y Niswander, 1970). Adicionalmente se utilizaron métodos de re–muestreo, Bootstrap y Jacknife, para establecer intervalos de confianza y el error estándar de la estimación promedio de los estadísticos.
Para explorar si las diferencias de diversidad genética entre subpoblaciones eran estadísticamente significativas, se realizó una análisis de varianza de HE previa transformación de los valores con arcoseno (HE)1/2.
El patrón de flujo genético entre las subpoblaciones puede ser de especial interés para la restauración ecológica, ya que un procedimiento común es incrementar el movimiento deliberado de individuos (y sus genes) entre diferentes sitios, es necesario conocer cuál es el flujo natural de genes para definir la dirección de transferencia con mayor apego al patrón natural. Un método indirecto para estimar el flujo genético (Nm) es con base en el grado de diferenciación de las subpoblaciones mediante la fórmula: Nm = 1/4 (1/ FST–1); donde N es el tamaño de la población, m la fracción de inmigrantes a N (Hartl y Clark, 1997). Los valores de Nm menores que 1 señalan un flujo genético bajo, mientras que valores igual o mayores a uno, indican un flujo genético suficiente para contrarrestar la diferenciación genética producto de la selección o de la deriva genética (Slatkin, 1994; Hartl y Clark, 1997; Templeton, 2006; Magallán–Hernández et al., 2009). Sin embargo, el flujo genético estimado de esta manera debe tomarse con precaución ya que se asumen condiciones de equilibrio entre deriva y flujo genético que biológicamente puede ser improbable (Whitlock y Mc–Cauley, 1999). Por ello se realizó un análisis de asignación multilocus con el programa IMMANC (Rannala y Mountain, 1997). Este análisis asume que diferentes loci son segregados de forma independiente y que están en equilibrio de ligamiento. El valor crítico de asignación es logaritmo natural del cociente de la probabilidad de que un genotipo multilocus se forme en la subpoblación de donde se colectó y la probabilidad de que se haya formado en una población distinta: lnΛ = lnPc–lnPi, donde Pc es la probabilidad de que el genotipo multilocus se formó en la subpoblación de colecta y Pi en otra subpoblación. Un valor lnΛ negativo indica que es más probable que el genotipo multilocus se produzca en una población distinta a la de colecta, lo que se interpreta como que es un inmigrante o descendiente de inmigrates recientes (Rannala y Mountain, 1997).
Resultados
Diversidad genética. Se analizó un total de 237 individuos de Oreopanax xalapensis, y el promedio por subpoblación fue de 27.8. Para seis enzimas se revelaron dos loci, y en total se identificaron 18 loci. Dos loci fueron monomórficos y para los otros 16 loci se reconocieron dos alelos (Cuadro 1) . El número de alelos promedio fue entre 1.8 y 2 (Cuadro 2) , cuya frecuencia fue significativamente heterogénea entre las poblaciones (Cuadro 3). El alelo de desplazamiento lento del locus G6PH–1 tuvo una frecuencia > 0.7 en Bazom, Tzontehuitz y Yerbabuena. El locus G3PDH–1 se observó cercano a la fijación en todas las poblaciones (Cuadro 3).


Se registró 100% de polimorfismo para tres poblaciones (Huitepec, Bazom y Mitzitón), y 66% y 78% para Yerbabuena y Tzontehuitz, respectivamente (Cuadro 2). Los valores más bajos de heterocigosidad observada (HO) se registraron en Tzontehitz (0.282), seguido de Yerbabuena (0.319), Huitepec (0.402), Mitzitón (0.416) y Bazom (0.473) (Cuadro 2). La heterocigocidad esperada (HE) estuvo por lo general por arriba de la observada, la más alta se registró en Bazom y la más baja en Tzontehuitz (Cuadro 2). El valor más alto del índice de diversidad de Shannon (I) se encontró en Bazom, seguido en orden descendente por Mitzitón, Huitepec, Yerbabuena y Tzontehuitz (Cuadro 2). Las diferencias entre las subpoblaciones de HE fueron estadísticamente significativas (F = 2.59, g.l. = 4/80, P = 0.04), pero no se observó una clara relación entre el estado de perturbación y la diversidad genética (HE) (Figura 1). Por otro lado, el índice de fijación (f) de cada locus y sobre los loci fue positivo para cuatro subpoblaciones y próximos a cero (Cuadro 3).

Todos los loci estuvieron fuera del EHW en al menos una subpoblación, destacan los loci EST, EM y FUM–2, que estuvieron en desequilibrio en cuatro poblaciones (contando la situación monomórfica) (Cuadro 4). La subpoblación con el menor número de loci en desequilibrio fue Huitepec (seis loci); en Mitzitón y Tzontehuitz se observaron nueve y siete loci en desequilibrio respectivamente, y en la Yerbabuena siete y Bazom diez (Cuadro 4).
Estructura genética. Sólo para el locus EST–1 se registró un valor de FIS positivo y significativamente distinto de cero, lo que indica una correlación alta de alelos idénticos (0.63) al nivel de subpoblaciones (Cuadro 5). Los valores de FIT de los loci EST–1, FA, SOD–1, GOT–1, GOT–2, y EM difieren significativamente de cero con valores positivos (Cuadro 5). Los valores de FST para EST–1, IDH–1, SOD–1, GOT–1, GOT–2, EM y FUM son distintos de cero lo que indica una diferenciación genética significativa entre las subpoblaciones (Cuadro 5).

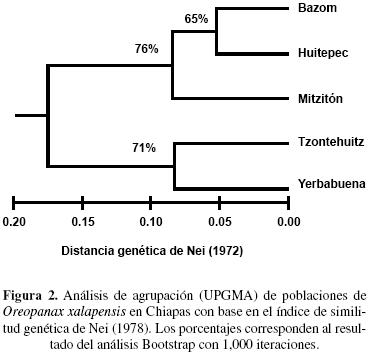
El análisis de agrupación muestra dos grupos de subpoblaciones, uno lo constituyeron las subpoblaciones de Bazom, Huitepec y Mtizitón, y el otro grupo estuvo formado por Tzontehuitz y Yerbabuena (Figura 2).
Con base en el valor de FST se obtuvo un flujo genético entre las subpoblaciones de Nm = 5.0 individuos por generación (Cuadro 5). Del análisis de asignación IMMANC resultó que 43% (26 individuos) de la subpoblación de Bazom se asignaron a una subpoblación distinta, principalmente de Huitepec (Cuadro 6), mientras que 29 (48%) individuos de Huitepec se asignaron a Bazom, Mitziton y Tzontehuitz. En Mitzitón 26 individuos (46%) se asignaron a Bazom, Huitepec y Tzontehuitz. En Tzontehuitz diez (34%) individuos se asignaron a Bazom, Huitepec y Mtizitón. Únicamente tres individuos (10%) de Yerbabuena se asignaron a Tzontehuitz (Cuadro 6).

Discusión
La diversidad genética promedio (HE = 0.379) de Oreopanax xalapensis en Los Altos de Chiapas fue dos veces mayor a la registrada en especies de angiospermas usando isoenzimas (HE = 0.131 en Hamrick et al., 1992, HE = 0.113 en Xie et al., 2002), o marcadores genéticos basados en ADNmt (HE = 0.133 Rowden et al., 2004), pero menos a lo que se ha registrado para otras especies del BMM de Chiapas como son Magnolia sharpii y M. schiedeana, para las cuales se ha registrado una diversidad promedio de HE = 0.4 (Newton et al., 2008). Por otra parte Vera–Maloof (2009) estudió la diversidad genética de plántulas, juveniles y adultos de O. xalapensis de bosques en estado sucesional diferente en Huitepec, y la diversidad promedio fue de HE= 0.32, valor cercano al registrada en este estudio. El presente estudio sugiere que O. xalapensis en Los Altos de Chiapas es genéticamente diversa y esta diversidad está contenida en las subpoblaciones, ya que al menos tres poblaciones (Bazom, Huitpec, Mitzitón) tuvieron una diversidad mayor al promedio.
Se considera que Oreopanax xalapensis es una especie de fase sucesional intermedia a madura del BMM (Quintana–Ascencio y González–Espinosa, 1993; González–Espinosa et al., 2007b), es tolerante a moderados niveles de perturbación y resiste radiación solar directa en estado adulto (Meave et al., 1992; Ramírez–Marcial, 2003; Quintana–Ascencio et al., 2004); el reclutamiento de plántulas puede ser alto (Ruiz–Ruvalcaba, 2004; Mejía–Domínguez, 2006) en especial en boques incipientes o fases temprana de la sucesión (Vera–Maloof, 2009), pude llegar a reproducirse sincrónicamente a temprana edad (a alturas inferiores a los 3 m y diámetros < 3 cm; N. Ramírez–Marcial, obs. pers.). Cambios ambientales específicos (p. ej., incrementos de temperatura) promueve la sincronía de algunos eventos fenológicos en árboles del Japón (Doi et al., 2010). La posible reproducción temprana y el alto reclutamiento de plántulas puede favorecer la formación continua de genotipos heterocigotos con lo que se mantiene una diversidad relativamente alta.
Por otra parte, es probable que ocurra una incorporación constante de individuos, con genotipos distintos, mediante la reproducción in situ pero posiblemente es a través de la dispersión de semillas promovida por diversas especies de aves que conjuntamente contribuyen a un aporte constante de variantes genéticas como se ha observado en otras plantas tropicales (Hamrick y Loveless, 1986). Las semillas de O. xalapensis son dispersadas por aves como Turdus rufitorques (primavera de montaña), Myadestes occidentalis (clarín o jilguero) (Ruiz–Ruvalcaba, 2004) y Trogon mexicanus (trogon de montaña) (J. Rangel–Salazar, com. pers.), y roedores como Peromyscus spp. y Reithrodontomys spp. (López–Barrera et al., 2005), por lo que además de HE alta se encontró un flujo genético considerable (Nm) entre las subpoblaciones. El análisis de asignación resultó que entre 50 y 60% de los individuos de una subpoblación son producidos en la subpoblación de colecta, el resto tiene alta posibilidad de ser inmigrante o descendiente de inmigrantes recientes (Rannala y Mountain, 1997), lo que también sugiere un considerable movimiento de genes entre las poblaciones de O. xalapensis.
No obstante el posible el flujo genético entre las subpoblaciones hay una diferenciación moderada entre ellas, revelada por el estadístico FST (0.25) y por la heterogeneidad de las frecuencias alélicas, lo cual puede ser reflejo de la influencia de factores que moldean la diversidad genética (Neigel, 2002). Estos factores pueden estar asociados a eventos de selección o deriva genética y estos a su vez con la historia de uso del bosque (Lande, 1999; Vornam et al., 2004; Baucom et al., 2005). Sin embargo, no se encontró evidencia de una relación entre el grado de perturbación con la diversidad genética. Se esperaba una relación positiva entre la diversidad genética (HE) y el grado de perturbación, bajo la lógica de que en los sitios conservados están las condiciones de temperatura y humedad del suelo propicias para el establecimiento y desarrollo de O. xalapensis (Quintana–Ascencio et al., 2004) y por lo tanto más variantes genética se podrían establecer; y porque hay el antecedente de que O. xalapensis tuvo la mayor diversidad en el bosque con la fase sucesional más avanzanda en Huitepec (Vera–Maloof, 2009). Es necesario tener estimaciones cuantitativas del grado perturbación y de factores ambientales (por ejemplo radiación solar, humedad) que incidan en la emergencia, establecimiento y crecimiento de plántulas, para poder conocer mejor la relación entre la diversidad genética y la perturbación del bosque que habita O. xalapensis.
Los valores de FIT y FIS fueron positivos para la mayoría de los loci y pocos de ellos fueron distintos de cero significativamente. La endogamia, revelada por los loci EST–1 al nivel de las subpoblaciones (FIS) y la detectada por EST–1, FA, GOT–1, GOT–2 y EM (FIT) al de la población, se reflejará en la adecuación en la medida en que los genes que codifican para estas enzimas tengan contribución en la expresión de componentes de la adecuación de O. xalpaensis. Vera–Maloof (2009) encontró un mayor coeficiente de endogamia en el bosque maduro (FIT= 0.328) y en promedio para los tres bosques sucesionales que estudió de 0.27, mientras que en el presente estudio se observó un coeficiente de endogamia promedio de 0.253 que no difiere significativamente de cero. Lo que indica que la correlación de genes idénticos es aleatoria (Chaves et al., 2011).
Por otro lado el índice de fijación (f) fue más bajo en Mitzitón el cual es un sitio con mayor perturbación y el más alto en Tzontehuitz con perturbación intermedia con respecto a los otros sitios. Un bajo índice de fijación sugiere que el número de heterocigotos observados es cercano al número esperado en apareamiento aleatorio (Hartl y Clark, 1997). La correlación del coeficiente de endogamia o índice de fijación con la depresión por endogamia es desconocido para Oreopanax xalapensis, pero se sabe que en Eugenia dysenterica DC (Myrtaceae) se correlaciona positivamente con la emergencia de la plántula y con el crecimiento de la planta en estados iniciales de desarrollo (Chaves et al., 2011). Por lo que se puede sugerir nuevamente que el alto reclutamiento de plántulas en bosques incipientes o intervenidos (Vera–Maloof, 2009), incrementa la frecuencia de heterocigotos, y así de diversidad genética, que permitirá a la especie responder genéticamente a los cambios ambientales producidos durante el proceso de sucesión o intervención humana.
En conclusión, la población de Oreopanax xalapensis de Los Altos de Chiapas es genéticamente diversa (HE > 0.25), las frecuencias alélicas son heterogéneas entre las subpoblaciones, con apareamiento aleatorio y las subpoblaciones están genéticamente diferenciadas de forma moderada (FST), con niveles de flujo genético altos y con un posible patrón asimétrico. No se encontró evidencia de relación entre la diversidad genética y el estado de perturbación de los bosques.
Este estudio indica que una buena práctica de restauración deberá considerar la colecta de semillas en todos los sitios para asegurar que la diversidad genética de Oreopanax xalapensis esté debidamente representada, de manera que se favorezca la permanencia de la especie y de los procesos evolutivos de la misma (Vucetich y Waite, 2001; Frankham et al., 2002; Stockwell et al., 2003; Rice y Emery, 2003).
Agradecimientos
Agradecemos a M. Martínez–Icó, H.E. Castañeda Ocaña, A. Luna Gómez, M. Gutiérrez–Gómez, M. Girón–Intzin por su apoyo durante las etapas de campo. Este trabajo fue apoyado por Fondo Mixto de fomento a la investigación Científica y tecnológica (CONACYT)–Gobierno del estado de Chiapas (FOMIX Chiapas) a través del proyecto CHIS–2006–C06–44064, y apoyo complementario del Fondo Institucional de Fomento Regional para el Desarrollo Científico, Tecnológico y de Innovación (FORDECYT–CONACyT) a través del Convenio 116306: Innovación socioambiental para el desarrollo en áreas de alta pobreza y biodiversidad de la frontera sur de México.
Literatura citada
Baucom R.S., Estill J.C. y Cruza M.B. 2005. The effect of deforestation on the genetic diversity and structure in Acer saccharum (Marsh): Evidence for the loss and restructuring of genetic variation in a natural system. Conservation Genetics 6:39–50. [ Links ]
Bubb P., May I., Miles L., y Sayer J. 2004. Cloud Forest Agenda. UNEP–WCMC, Cambridge. [ Links ]
Caro T.M. y Laureson M.K. 1994. Ecological and genetic factors in conservation: a cautionary tale. Science 263:485–486. [ Links ]
Cayuela L., Golicher D. y Rey–Benayas J.M. 2006a. The extent, distribution, and fragmentation of vanishing montane cloud forest in the Highlands of Chiapas, Mexico. Biotropica 38:544-554. [ Links ]
Cayuela L., Golicher D.J., Rey–Benayas J.M., González–Espinosa M. y Ramírez–Marcial N. 2006b. Fragmentation, disturbance and tree diversity conservation in tropical montane forests. Journal of Applied Ecology 43:1172–1181. [ Links ]
Chaves L.J., Vencovsky R., Silva R.S.M., Telles M.P.C., Zucchi M.I. y Coelho A.S.G. 2011. Estimating inbreeding depression in natural plant populations using quantitative and molecular data. Conservation Genetics 12:569–576. [ Links ]
Cheliak W.M. y Pitel J.A. 1984. Techniques for starch gel electrophoresis of enzymes from forest trees species. Reporte PI–X–42. Petawa National Forestry Institute, Chalk River, Ontario. [ Links ]
Dobson A.P. 1999. Introduction: Genetics and conservation biology. En: Landweber, L.F. y Dobson A.P. Eds. Genetic and The Extinction of Species, pp.xiii–xix, Princeton University Press, Princeton. [ Links ]
Doi H., Takahashi M. y Katano I. 2010. Genetic diversity increases regional variation in phenological dates in response to climate change. Global Change Biology 16:373–379. [ Links ]
Frankham R., Ballou J.D. y Briscoe D.A. 2002. Introduction to Conservation Genetics. Cambridge University Press, Cambridge. [ Links ]
Futuyma D.J. 2009. Evolution. Sinauer, Sunderland. [ Links ]
Galindo–Jaimes L., González–Espinosa M., Quintana–Ascencio P. y García–Barrios L. 2002. Tree composition and structure in disturbed stands with varying dominance by Pinus spp. in the highlands of Chiapas, México. Plant Ecology 162:259–272. [ Links ]
Glaubitz J.C., Wu H.X. y Moran G.F. 2003. Impacts of silviculture on genetic diversity in the native forest species Eucalyptus sieberi. Conservation Genetics 4:275–287. [ Links ]
Golicher D.J., Cayuela L., Alkemade J.R.M., González–Espinosa M. y Ramírez–Marcial N. 2008. Applying climatically associated species pools to the modeling of compositional change in tropical montane forests. Global Ecology and Biogeography 17:262–273. [ Links ]
González–Espinosa M., Quintana–Ascencio P.F., Ramírez–Marcial N. y Gaytán–Guzmán P. 1991. Secondary succession in disturbed Pinus–Quercus forests in the highlands of Chiapas, Mexico. Journal of Vegetation Science 2:351–360. [ Links ]
González–Espinosa M., Ochoa–Gaona S., Ramírez–Marcial N. y Quintana–Ascencio P.F. 1995. Current land–use trends and conservation of old–growth forest habitats in the highlands of Chiapas, Mexico. En: Wilson M.M. y Sader S.A. Eds. Conservation of Neotropical Migrant Birds in Mexico, pp. 190–198, Maine Agriculture and Forest Experiment Station, Miscellaneous Publication 727, Orono. [ Links ]
González–Espinosa M., Ramírez–Marcial N., Camacho–Cruz A. y Rey–Benayas J.M. 2008. Restauración de bosques en montañas tropicales de territorios indígenas de Chiapas, México. En: González–Espinosa M., Rey–Benayas J.M. y Ramírez–Marcial N. Eds. Restauración de Bosques en América Latina, pp. 137-162, Mundi–Prensa y Fundación Internacional para la Restauración de Ecosistemas, México, D.F. [ Links ]
González–Espinosa M., Ramírez–Marcial N., Camacho–Cruz A., Holz S.C., Rey–Benayas J.M. y Parra–Vázquez M.R. 2007a. Restauración de bosques en territorios indígenas de Chiapas: modelos ecológicos y estrategias de acción. Boletín de la Sociedad Botánica de México 80 (Suplemento):11–23. [ Links ]
González–Espinosa M., Ramírez–Marcial N., Newton A.C., Rey–Benayas J.M., Camacho–Cruz A., Armesto J.J., Lara A., Premoli A.C., Williams–Linera G., Altamirano A., Alvarez–Aquino C., Cortés M., Echeverría C., Galindo–Jaimes L., Muñiz–Castro M.A., Núñez–Avila M.C., Pedraza R.A., Rovere A.E., Smith–Ramírez C., Thiers O. y Zamorano C. 2007b. Restoration of forest ecosystems in fragmented landscapes of temperate and montane tropical Latin America. En: Newton A.C. Ed. Biodiversity Loss and Conservation in Fragmented Forest Landscapes: The Forests of Montane Mexico and Temperate South America, pp. 335–369, CAB International, Wallingford. [ Links ]
Hamrick J.L. y Loveless M.D. 1986. The influence of seed dispersal mechanisms on the genetic structure of plant populations. En: Estrada A. y Fleming T.H. Eds. Frugivores and Seed Dispersal, pp. 212–223. Dr. W. Junk Plublishers, Dordrecht. [ Links ]
Hamrick J.L., Godt M.J.W. y Gonzales E. 2006. Conservation of genetic diversity in old–growth forest communities of the southeastern United States. Applied Vegetation Science 9:51–58. [ Links ]
Hamrick J.L., Godt M.J.W. y Sherman–Broyles S.L. 1992. Factors influencing levels of genetic diversity in woody plant species. New Forest 6:95–124. [ Links ]
Hartl D.L. y Clark A.G. 1997. Principles of Population Genetics. Sinauer, Sunderland. [ Links ]
Hedrick P.W. 2000. Genetics of Populations. Jones and Bartlett Publishers, Sudbury. [ Links ]
Herbet P.D.N. y Beaton M.J.1993. Methodologies for allozyme analysis using cellulose acetate electrophoresis. Helena Laboratories, Beaumont. [ Links ]
Hufford K.M. y Mazer. S.J. 2003. Plant ecotypes: genetic differentiation in the age of ecological restoration. Trends in Ecology and Evolution 18:147–155. [ Links ]
Keller L.F. y Waller D.M. 2002. Inbreeding effects in wild populations. Trends in Ecology & Evolution 17:230–241. [ Links ]
Lande R. 1999. Extinction risks from anthropogenic ecological and genetic factors. En: Landweber L.F. y Dobson A.P. Eds. Genetic and the Extinction of Species, pp. 1–22, Princeton University Press, Princeton. [ Links ]
Lessica P. y Allendorf F.W. 1999. Ecological genetics and the restoration of plant communities: mix or match? Restoration Ecology 7:42–50. [ Links ]
Li Y.Y., Chen X.Y., Zhang X., Wu T.Y., Lu H.P. y Cai Y.W. 2005. Genetic differences between wild and artificial populations of Metasequoia glyptostroboides: Implications for species recovery. Conservation Biology 19:224–231. [ Links ]
López–Barrera F., Newton A. y Manson R. 2005. Edge effects in a tropical montane forest mosaic: experimental tests of post–dispersal acorn removal. Ecological Research 20:31–40. [ Links ]
Lundell C.L. 1966. Myrsinaceae. En: Standley P.C. y Williams L.O. Eds. Flora of Guatemala, Volume 24, Part 8 Nos. 1 and 2, Araliaceae to Plumbaginaceae, pp. 135–200. Chicago Natural History Museum, Chicago. [ Links ]
Magallán–Hernández F., Martínez M., Hernández–Sandoval L. y Oyama K. 2009. Estructura genética de poblaciones de Eriocaulon bilobatum (Eriocaulaceae): una especie amenazada de humedales temporales. Boletín de la Sociedad Botánica de México 85:81–88. [ Links ]
Meave J., Soto M., Calvo L., Paz H. y Valencia S. 1992. Análisis sinecológico del bosque mesófilo de montaña de Omiltemi, Guerrero. Boletín de la Sociedad Botánica de México 52:31–77. [ Links ]
Mejía–Domínguez N.R. 2006. Dinámica de la comunidad de árboles de un bosque mesófilo de montaña en la Sierra Madre Sur (Oaxaca), México. Tesis de Maestría, Facultad de Ciencias, Universidad Nacional Autónoma de México, México, D.F. 82 pp. [ Links ]
Miller M.P. 1997. Tools for population genetics analysis (TFPGA) v1.3: A Windows program for the analysis of allozymes and molecular population genetic data. Northen Arizona University, Flagstaff. [ Links ]
Montalvo A.M. y Ellstrand N.C. 2000. Transplantation of the subshrub Lotus scoparius testing the home site advantage hypothesis. Conservation Biology 14:1034–1045. [ Links ]
Montalvo A.M., William S.L., Rice K.J., Buchmann S.L., Cory C., Handel S.N., Nabhan G.P., Primack R. y Robichaux R.H. 1997. Restoration biology: A population biology perspective. Restoration Ecology 5:277– 290. [ Links ]
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89:583-590. [ Links ]
Neigel J.E. 2002. Is FST obsolete? Conservation Genetics 3:167-173. [ Links ]
Newton A.C., Gow J., Robertson A., Williams–Linera G., Ramírez–Marcial N., González–Espinosa M., Allnutt T.R. y Ennos R. 2008. Genetic variation in two rare endemic Mexican trees, Magnolia sharpii and Magnolia schiedeana. Silvae Genetica 57:348–356. [ Links ]
Ochoa–Gaona S. y González–Espinosa M. 2000. Land–use and deforestation in the highlands of Chiapas, Mexico. Applied Geography 20:17–42. [ Links ]
Paschke M., Abs C. y Schmid B. 2002. Relationship between population size, allozyme variation, and plant performance in the narrow endemic Cochlearia bavarica. Conservation Genetics 3:131–144. [ Links ]
Peakall R. y Smouse P.E. 2006. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes 6:288–295. [ Links ]
Pennington T.D. y Sarukhán J. 1998. Arboles Tropicales de México. Universidad Nacional Autónoma de México y Fondo de Cultura Económica, México, D.F. [ Links ]
Quintana–Ascencio P.F. y González–Espinosa M. 1993. Afinidad fitogeográfica y papel sucesional de la flora leñosa de los bosques de pino–encino de Los Altos de Chiapas, México. Acta Botanica Mexicana 21:43–57. [ Links ]
Quintana–Ascencio P.F., Ramírez–Marcial N., González–Espinosa M. y Martínez–Icó M. 2004. Sapling survival and growth of conifer and broad–leaved trees in successional highland habitats in Mexico. Applied Vegetation Science 7:81–88. [ Links ]
Ramírez–Marcial N. 2003. Survival and growth of tree seedlings in antropogenically distubed Mexican montane rain forest. Journal of Vegetation Science 14:881–890. [ Links ]
Ramírez–Marcial N., Camacho–Cruz A. y González–Espinosa M. 2005. Potencial florístico para la restauración de bosques en Los Altos y Montañas del Norte de Chiapas. En: González–Espinosa M., Ramírez–Marcial N. y Ruiz–Montoya L. Eds. Diversidad Biológica de Chiapas, pp. 325–370. Plaza y Valdez y Consejo de Ciencia y Tecnología del Estado de Chiapas, México, D.F. [ Links ]
Ramírez–Marcial N., Camacho–Cruz A. y González–Espinosa M. 2008. Clasificación de grupos funcionales vegetales para la restauración del Bosque Mesófilo de Montaña. En: Sánchez–Velázquez L.R., Galindo–González J. y Díaz–Fleischer F. Eds. Ecología, Manejo y Conservación de los Ecosistemas de Montaña en México, pp. 51–72, Comisión Nacional Para el Conocimiento y Uso de la Biodiversidad, Universidad Veracruzana, Mundi Prensa México, México, D.F. [ Links ]
Ramírez–Marcial N., Camacho–Cruz A., Martínez–Icó M., Luna–Gómez A., Golicher D. y González–Espinosa M. 2010a. Árboles y Arbustos de los Bosques de Montaña en Chiapas. El Colegio de la Frontera Sur, San Cristóbal de Las Casas, Chiapas. [ Links ]
Ramírez–Marcial N., González–Espinosa M. y Williams–Linera G. 2001. Anthropogenic disturbance and tree diversity in Montane Rain Forests in Chiapas, Mexico. Forest Ecology and Management 154:311–326. [ Links ]
Ramírez–Marcial N., González–Espinosa M., Camacho–Cruz A. y Ortiz–Aguilar D. 2010b. Forest restoration in Lagunas de Montebello National Park, Chiapas, Mexico. Ecological Restoration 28:354–360. [ Links ]
Ramírez–Marcial N., Ochoa–Gaona S., González–Espinosa M. y Quintana–Ascencio P.F. 1998. Análisis florístico y sucesional en la estación biológica Cerro Huitepec, Chiapas, México. Acta Botánica Mexicana 44:59–85 [ Links ]
Rannala B. y Mountain J.L. 1997. Detecting immigration by using multilocus genotypes. Proceedings of National Academy of Sciences 94:9197–9201. [ Links ]
Rice K.J. y Emery N.C. 2003. Managing microevolution: restoration in the face of global change. Frontiers in Ecology and Environment 1:469–478. [ Links ]
Robichaux R.H., Friar E.A. y Mount D.W. 1997. Molecular genetic consequences of a population bottleneck associated with reintroduction of the Mauna Kea silversword (Argyroxiphium sandwicense ssp. sandwicense [Asteraceae]). Conservation Biology 11:1140–1146. [ Links ]
Rowden, A. Robetson A., Allnut T., Heredia S., Williams–Linera G., Newton A.C. 2004. Conservation genetics of Mexican beech, Fagus grandifolia var. mexicana. Conservation Genetics 5:475–484. [ Links ]
Ruiz–Ruvalcaba S.E. 2004. Variación demográfica de Oreopanax xalapensis en comunidades sucesionales de Los Altos de Chiapas, Mexico. Tesis de Licenciatura, Universidad Nacional Autónoma de México, Facultad de Estudios Superiores Iztacala, Tlalnepantla, estado de México, 91 pp. [ Links ]
Rzedowski J. 1992. Diversidad del universo vegetal de México: Perspectivas de un conocimiento sólido. En: Sarukhan J. y Dirzo R. Eds. México Ante los Retos de la Biodiversidad, pp. 251–258, Comisión Nacional para el Conocimiento y Uso de la Biodiversidad, México, D.F. [ Links ]
Rzedowski J. 1996. Análisis preliminar de la flora vascular de los bosques mesófilos de montaña de México. Acta Botanica Mexicana 35:25–44. [ Links ]
Rzedowski J. y McVaugh R. 1996. La Vegetación de Nueva Galicia. University of Michigan Herbarium, Ann Arbor. [ Links ]
Slatkin M. 1994. Gene flow and population structure. En: Real L.A. Ed. Ecological Genetics pp. 3–17. Princeton University Press, Princeton. [ Links ]
Sosa V. 1979. Araliaceae. Flora de Veracruz Fascículo 8. Instituto Nacional de Investigaciones sobre Recursos Bióticos, Xalapa. [ Links ]
Stockwell C.A, Hendry A.P. y Kinnison M.T. 2003. Contemporary evolution meets conservation biology. Trends in Ecology and Evolution 18:94–101. [ Links ]
Templeton A.R. 2006. Population Genetics and Microevolutionary Theory. John Wiley & Sons, Hoboken. [ Links ]
Toledo–Aceves T., Meave J.A., González–Espinosa M. y Ramírez–Marcial N. 2011. Tropical montane cloud forests: current threats and opportunities for their conservation and sustainable management in Mexico. Journal of Environmental Management 92:974–981. [ Links ]
Vera–Maloof F.Z. 2009. Diversidad genética y variación demográfica de Oreopanax xalapensis (Araliaceae) sobre un gradiente sucesional del bosque mesófilo de montaña de Chiapas. Tesis de Maestría, EL Colegio de la Frontera Sur, San Cristóbal de Las Casas, Chiapas, 106 pp. [ Links ]
Vornam B., Decarli N. y Gailing O. 2004. Spatial distribution of genetic variation in a natural beech stand (Fagus sylvatica L.) based on microsatellite markers. Conservation Genetics 5:561–570. [ Links ]
Vucetich, J.A. y Waite T.A. 2001. Migration and inbreeding: the importance of recipient population size for genetic management. Conservation Genetics 2:167–171. [ Links ]
Workman P.L. y Niswander J.D. 1970. Population studies on southwestern Indian tribes. II. Local genetic differentiation in the Papago. American Journal of Human Genetics 22:24–49. [ Links ]
Whitlock M.C. y McCauley D.E. 1999. Indirect measure of gene flow and migration FST≠1/(4Nm+1). Heredity 82:117–125. [ Links ]
Weeden J.F. y Wendel N.F. 1989a, Visualization and interpretation of plant isozymes. En: Soltis, D.E. y Soltis S.P. Eds. Isozymes in Plant Biology, pp. 5–45. Chapman and Hall, Londres. [ Links ]
Weeden J.F. y Wendel N.F. 1989b. Genetic of plant isozymes. En: Soltis, D.E. y Soltis S.P. Eds. Isozymes in Plant Biology, pp. 46–72. Chapman and Hall, Londres. [ Links ]
Xie C.Y., El–Kassabay Y.A. y Ying C.C. 2002. Genetics of red alder (Alnus rubra Bong.) populations in British Columbia and its implications for gene resources management. New Forests 24:97–112. [ Links ]
Yeh F.C.H. y O'Malley D. 1980. Enzyme variation in natural populations of Douglas–fir, Pseudotsuga menziesii (Mirb.) Franco, from British Columbia. 1. Genetics variation patterns in coastal populations. Silvae Genetica 29:83–92. [ Links ]














