Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Boletín de la Sociedad Botánica de México
versión impresa ISSN 0366-2128
Bol. Soc. Bot. Méx no.85 México dic. 2009
Genética
Estructura Genética de poblaciones de Eriocaulon bilobatum (Eriocaulaceae): una especie amenazada de humedades temporales
Genetic estructure of populations of Eriocaulon bilobatum (Eriocaulaceae): an endangered species of temporary wetlands
Fabiola Magallán Hernández1,3, Mahinda Martínez1, Luis Hernández Sandoval1 y Ken Oyama2
1 Universidad Autónoma de Querétaro (UAQ). Av. de las Ciencias s/n Juriquilla, Querétaro, 76230, México. 3 Autor para la correspondencia. Correo electrónico: carfabios@yahoo.com.mx
2 Centro de Investigaciones en Ecosistemas, Universidad Nacional Autónoma de México. Antigua carretera a Pátzcuaro No. 8701 Col. Ex–Hacienda de San José La Huerta, Morelia Michoacán, 58190, México.
Recibido: 1 de junio de 2009.
Aceptado: 23 de septiembre de 2009.
Resumen
Eriocaulon bilobatum es una especie acuática que vive en humedales temporales del centro de México. Es una herbácea anual, enraizada emergente, con reproducción sexual y asexual, monoica y polinizada por insectos. Es una especie rara y vulnerable a la extinción debido a que sus hábitats se encuentran amenazados. Los objetivos de este estudio fueron determinar la estructura y diversidad genética de poblaciones de E. bilobatum a nivel regional, y conocer si éstas se relacionan con sus características ecológicas y de historia de vida en forma similar a plantas terrestres. Se analizaron 160 individuos en cuatro poblaciones con la técnica de electroforesis de isoenzimas en geles de almidón. E. bilobatum presenta mayor diversidad genética (A=2.32, Ae=1.31, P=69.65, Ho=0.134, He=0.197, HT=0.221) que especies con características ecológicas y de historia de vida similares, endogamia moderada (FIS = 0.312) y baja diferenciación genética entre sus poblaciones (FST = 0.053 y GST = 0.048). Su diversidad y estructura genética está determinada por el sistema de reproducción y su historia de vida más que el hecho de vivir en ambientes acuáticos.
Palabras clave: diversidad genética, características ecológicas, hábitats aislados, historias de vida, plantas acuáticas.
Abstract
Eriocaulon bilobatum is an aquatic species that inhabits temporary wetlands in central Mexico. It is annual, herbaceous, emergent, with sexual and asexual reproduction, monoecious and insect pollinated. It is a rare and vulnerable species due to its endangered habitats. The objectives of this study were to determine the diversity and genetic structure of E. bilobatum and to know if there is a correlation with genetic diversity and its ecological and life history traits. Using horizontal starch–gel electrophoresis, we screened 160 individuals from four populations. E. bilobatum has a higher genetic diversity (A=2.32, Ae=1.31, P=69.65, Ho=0.134, He=0.197, HT=0.221) than species with similar ecological and life history traits, moderate levels of inbreeding (FIS = 0.312) and low genetic differentiation among populations (FST = 0.053 y GST = 0.048). Its diversity and genetic structure are determined by the mating system and life history traits, more than by inhabiting aquatic environments.
Key words: aquatic plants, ecological traits, genetic diversity, life history traits, isolated habitat.
Las características de historia de vida de las plantas determinan los niveles de diversidad y estructura genética de sus poblaciones (Hamrick et al., 1979; Loveless y Hamrick, 1984; Hamrick y Godt, 1990). Por ejemplo, se ha sugerido que especies con distribución amplia, reproducción cruzada, polinización por viento y tiempo generacional largo presenten mayor variación genética que aquellas con características opuestas. Estas generalizaciones se han hecho con base en estudios de plantas terrestres ya que existen relativamente pocos estudios sobre la diversidad y estructura genética de plantas acuáticas (Barrett et al., 1993; Elam, 1998). Las angiospermas acuáticas son generalmente más polimórficas que las plantas terrestres a nivel de especie, mientras que, a nivel de cada población las plantas acuáticas presentan menos diversidad genética que las plantas terrestres (Barrett et al., 1993; Laushman, 1993). La alta diversidad genética a nivel de especie se puede deber a los altos niveles de diferenciación genética entre poblaciones (Edwards y Sharitz, 2000), lo cual está relacionado con la distribución de sus hábitats, que se encuentran como "islas" dentro de paisajes terrestres (Elam, 1998). Esta distribución disminuye la probabilidad de flujo génico entre las poblaciones y aumenta la diferenciación a través de la deriva génica (Barrett et al., 1993; Elam, 1998). Además, la destrucción de los hábitats naturales ha sido determinante para que se manifiesten algunos procesos genéticos como la erosión genética y la endogamia, como consecuencia de la disminución de los tamaños poblacionales (Edwards y Sharitz, 2000; Rocha y Gasca, 2007). Para este trabajo, se eligió a Eriocaulon bilobatum, una planta acuática que presenta una forma de vida enraizada emergente y vive en sitios fuertemente amenazados. Debido a sus características florales y la distribución de sus poblaciones aisladas y escasas, consideramos que E. bilobatum tendría una diversidad genética moderada o baja dentro de sus poblaciones, bajo o nulo flujo génico y alta diferenciación genética entre sus poblaciones. Los objetivos de este estudio fueron determinar la diversidad y estructura genética de E. bilobatum y conocer cómo se relacionan con sus características ecológicas y de historia de vida.
Materiales y métodos
Especie de estudio. Eriocaulon bilobatum Morong. (Eriocaulaceae) es una especie acuática de amplia distribución que vive en humedales temporales aislados. Se eligió como sitio de estudio la parte centro de México en los estados de Guanajuato y Querétaro. Es una monocotiledónea herbácea anual, con forma de vida enraizada emergente, que mide hasta 15 cm de largo incluyendo la inflorescencia (Calderón, 1996). Es una especie rara y amenazada ya que sus hábitats han sido alterados por la actividad humana. Localmente puede ser abundante y se reproduce tanto sexual como asexualmente por medio de rizomas. E. bilobatum es monoica, con el desarrollo de las flores femeninas antes que el de las masculinas, por lo que no es probable que exista autogamia (Magallán, 2004). una especie de mosca del género Notiphila Fallén visita sus inflorescencias, estos dípteros se posan en la inflorescencia y se mueven por toda su superficie; el tiempo que permanecen es variable, caminan por el pedúnculo de la inflorescencia hacia abajo y vuelan hacia las inflorescencias de otros individuos de la especie. Ni el polen, ni los óvulos están en contacto directo con el agua, ya que las flores maduran hasta que emergen del agua. El polen presenta granos circulares, sincolpados, de 20–30 μm en ambos diámetros (polar y ecuatorial) y ornamentación escabrada (Magallán, 2004), asociada a dispersión por insectos (Moore et al., 1991). Estas observaciones permiten suponer que E. bilobatum tiene polinización entomófila. Características de los ecosistemas de estudio. Los humedales estudiados presentan una estacionalidad marcada, que tiene un período de inundación de a seis a ocho meses y uno de sequía de cuatro a seis meses. Se forman en depresiones poco profundas de suelos impermeables que se llenan únicamente por agua de lluvia, sin entradas de agua por drenaje de ríos o arroyos; el período de inundación es cuando la temperatura es ideal para el crecimiento de las plantas y el nivel de agua disminuye hasta secarse completamente entre diciembre y marzo dependiendo de la precipitación anual. Las depresiones o cuencas en las que se establecen los humedales de estudio tuvieron su origen gracias a la actividad volcánica, por lo que se distribuyen principalmente en el Eje Volcánico Transversal, se trata de ecosistemas poco comunes y en exploraciones de campo se registraron mayormente en los estados de Querétaro y Guanjuato, México (Magallán, 2004). Los humedales en los que habita E. bilobatum son ecosistemas muy importantes por la alta biodiversidad de especies, formas de vida y procesos ecológicos, además de contener varias especies raras o endémicas de México (Novelo, 2000). Sin embargo, están siendo fuertemente amenazados por actividades humanas (Magallán, 2004) razón por la que es importante conocer la diversidad y estructura genética de especies de plantas representativas de estos ecosistemas para sentar las bases que permitan proponer estrategias de conservación.
Muestro de las poblaciones y procedimiento de laboratorio. En la zona de estudio (Querétaro y Guanajuato) se tienen registrados (Magallán, 2004) doce humedales temporales, sin embargo E. bilobatum únicamente se encuentra en cuatro de éstos. Se colectaron 160 individuos (40 por población) en los cuatro humedales donde se distribuye la especie (Figura 1). Las colectas se llevaron a cabo durante los meses de agosto y septiembre, las plantas se colectaron de forma aleatoria, separadas entre sí al menos un metro, para evitar colectar propágulos por rizomas. Se transportaron a 4°C y en el laboratorio se almacenaron en bolsas de plástico a –70 °C. Se usó la técnica de electroforesis horizontal de isoenzimas en geles de almidón (Cheliak y Pitel, 1984; Werth, 1985; Wendel y Weeden, 1989). Para la extracción de enzimas se usó el buffer "YO:VEG" (Yeh y O'Malley, 1980; Cheliak y Pitel, 1984), el procedimiento consistió en macerar las hojas con dos gotas del buffer de extracción y una pizca de arena esterilizada, con el extracto obtenido, se impregnaron los rectángulos de 3 mm de ancho por 14 mm de largo (wicks) hechos con papel de cromatografía Whatman 3MM. Los wicks se almacenaron a –70°C para ser utilizados al siguiente día. Los geles se prepararon a concentraciones del 12 % de almidón (Wendel y Weeden, 1989). Se usaron nueve enzimas en tres sistemas: C (CPX, PGD y PGI), D (GDH, PGM, SDH) y Morfolina (IDH, ME, MNR). El Sistema C se corrió a 60 mA durante siete horas, el sistema D se corrió a 30 mA por ocho horas y el sistema Morfolina a 60 mA durante seis horas. Los geles se tiñeron a 37°C. La nomenclatura de los loci y de los alelos se estableció de la siguiente manera: a los loci que estuvieran más cerca del ánodo se les asignó el número 1, los que seguían el número 2 y así sucesivamente conforme se acercaran al cátodo, se usó el mismo criterio para los alelos, asignando letras. Al momento de introducir los wicks en el gel, también se colocaba un wick testigo de un genotipo ya conocido, con el objetivo de asignar correctamente la nomenclatura de los loci y alelos.
Se usaron wicks impregnados con amido black para determinar el frente de migración. La información acerca de la estructura cuaternaria de las enzimas, que sirvió para llevar a cabo la adecuada interpretación de las bandas, se obtuvo de Wendel y Weeden (1989). Se llevó a cabo una primera lectura de los patrones de bandeo con los geles en fresco, éstos se fijaron en alcohol al 50 % o en una solución 5:5:1 de metanol, agua y ácido acético.
Análisis estadístico. Se obtuvieron los siguientes estadísticos: Variación genética dentro de las poblaciones locales (Hartl y Clark, 1997; Hedrick, 2000): 1. Número de alelos por locus (A). 2. Número efectivo de alelos por locus (Ae). 3. Porcentaje de loci polimórficos (P), 4. Hetorocigosidad observada (Ho). 5. Heterocigosidad esperada (He). Diversidad genética dentro y entre poblaciones locales (Hedrick, 2000; Culley et al., 2002): 1. Diversidad genética total (HT). 2. Diversidad genética dentro de las subpoblaciones (HS). 3. Diversidad genética entre las poblaciones (DST). 4. Coeficiente de diferenciación genética (GST). Índice de endogamia y estructura genética (Hartl y Clark, 1997; Slatkin, 1987): 1. Índice de Fijación (F). 2. Estadísticos F de Wright: Coeficiente de endogamia entre individuos de una población local (FIS), coeficiente de endogamia entre individuos en relación a la población total (FIT) y diferenciación genética entre poblaciones (FST). Para evaluar si F, FIS y FIT eran significativamente diferentes de cero, se llevó a cabo una prueba de χ2 = F2N(k–1) con gl = [k (k–1)] / 2, donde F es el coeficiente de endogamia, N es el tamaño de la muestra y k es el número de alelos (Li y Horvitz, 1953). Para saber si los valores de FST eran significativamente diferentes de cero se hizo una prueba de χ2 = 2NFST(k–1) con gl = [(k–1) (s–1)], donde N es el tamaño de la muestra, k es el número de alelos y s es el número de subpoblaciones (Workman y Niswander, 1970). Flujo génico (Slatkin, 1987) (Nm). Identidades genéticas (Nei, 1978; Hedrick, 2000) (I).
Para calcular los estadísticos, primeramente se obtuvieron los genotipos individuales para cada población y se conformaron matrices de datos. El programa Biosys–1 (Swofford y Selander, 1981) se usó para obtener las frecuencias alélicas, los índices de variación genética dentro de las poblaciones (excepto Ae), el índice de endogamia (F), los estadísticos F de Wright y la matriz de identidad (I). Los demás índices se calcularon con las frecuencias alélicas y sus respectivas fórmulas.
Resultados
Se analizaron 14 loci de nueve enzimas en cuatro poblaciones de E. bilobatum: CPX–1, CPX–2, GDH–1, IDH–1, IDH–2, IDH–3, ME–1, MNR–1, PGD–1, PGD–2, PGI–1, PGI–2, PGM–1 y SDH–1. De los 14 loci definidos, 13 fueron polimórficos al menos en alguna de las poblaciones estudiadas y un locus (IDH–3) resultó monomórfico en todas. Las frecuencias alélicas de los 13 loci polimórficos para cada una de las cuatro poblaciones estudiadas se muestran en el Cuadro 1.

Variación genética dentro de las poblaciones. Se registraron un total de 48 alelos en los 14 loci definidos. El número de alelos por locus (A) fue de 2.3 con un intervalo de 1.9 a 2.9, mientras que el número efectivo de alelos por locus (Ae) fue de 1.3 con un intervalo de 1.2 a 1.4 (Cuadro 2). Se observa que en las cuatro poblaciones locales el índice A fue mayor que Ae, lo cual indica la presencia de alelos raros. El número promedio de individuos analizados para las cuatro poblaciones locales fue de 37.7. El porcentaje de loci polimórficos (P) fue de 69.7 con un intervalo de 42.9 a 85.7. El promedio de heterocigosidad observada (Ho) fue de 0.134 con un intervalo de 0.084 a 0.138 y el de heterocigosidad esperada (He) de acuerdo al equilibrio de Hardy–Weinberg fue de 0.197 con un intervalo de 0.129 a 0.239. En todas las poblaciones la heterocigosidad observada fue menor que la esperada, indicando deficiencia de individuos heterocigotos. El tamaño de muestra por población así como los valores de A, Ae, P, Ho y He se muestran en el Cuadro 2. Diversidad genética dentro y entre poblaciones. La población Huimilpan 1 presenta la menor diversidad genética intrapoblacional (He= 0.129), mientras que la población J. Rosas 4 presenta mayor diversidad genética (He= 0.239), las poblaciones San Juan 2 (He= 0.222) y Amealco 3 (He= 0.130) presentan diversidad genética intermedias. La diversidad genética total (HT) es moderada, siendo el promedio igual a 0.221, con un intervalo de 0.045 en el locus PGD–2 a 0.465 en el locus PGI–2. La mayor parte de la diversidad genética se encuentra dentro de las poblaciones (HS = 0.210) y poca entre las poblaciones (DST = 0.012), lo cual indica que las cuatro poblaciones locales comparten una gran proporción de la diversidad total. El coeficiente de diferenciación genética (GST = 0.048) indica que el 4.8 % de la diversidad genética se encuentra entre poblaciones y el 95.2 % dentro de las poblaciones. Los valores de HT, HS, DST y GST se muestran en el Cuadro 3.

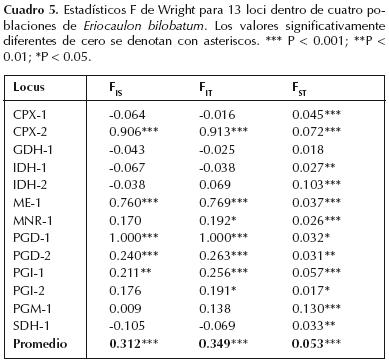
Índice de endogamia y estructura genética. Se obtuvieron 45 valores de índice de fijación o endogamia (F) en los 13 loci polimórficos de las cuatro poblaciones estudiadas (Cuadro 4). Se observó exceso de homocigotos en 15 loci (33.3 %) cuyos valores fueron positivos y significativamente diferentes de cero. De los valores restantes ninguno fue significativamente diferente de cero, lo cual indica que el 66.7 % de los loci están en equilibrio de Hardy–Weinberg. Las poblaciones Huimilpan 1 y San Juan 2 no presentan valores significativamente diferentes de cero, indicando que se encuentran en equilibrio de Hardy–Weinberg, mientras que las poblaciones Amealco 3 y Juventino Rosas 4 presentan valores positivos y significativamente diferentes de cero, lo que señala que estas dos poblaciones tienen exceso de individuos homocigotos (endogamia) (Cuadro 4). Los estadísticos F de Wright: FIS, FIT y FST se muestran en el Cuadro 5. Las estimaciones de FIS fueron significativamente diferentes de cero en cinco loci (38.5 %), de los ocho valores restantes, ninguno fue significativamente diferentes de cero, por lo que 61.5 % de los loci se encuentran en equilibrio de Hardy–Weinberg. El promedio de FIS fue de 0.312 y fue significativamente diferente de cero, indicando que existe endogamia entre individuos dentro de las poblaciones locales. Las estimaciones de FIT fueron significativamente diferentes de cero en siete loci (53.8 %), indicando que un poco más de la mitad de los loci presentan exceso de homocigotos, de los seis valores restantes, ninguno fue significativamente diferente de cero, por lo que 46.2 % de los loci están en equilibrio de Hardy–Weinberg. El promedio de FIT presentó un valor de 0.349 y fue significativamente diferente de cero, señalando que la diferenciación de las poblaciones locales con respecto a la población total se debe a la endogamia. Los valores de FST fueron significativamente diferentes de cero en doce loci (92.3 %) y solamente un locus (GDH–1) no fue significativamente diferente de cero. El promedio de FST tiene un valor de 0.053 y tuvo diferencias significativas de cero, lo cual indica diferenciación genética moderada entre poblaciones locales (Cuadro 5). Este valor concuerda con el índice GST, sugiriendo que la mayoría de la diversidad genética de E. bilobatum se encuentra dentro de las poblaciones.

Flujo génico. El flujo génico entre poblaciones presenta valores de 4.925 (calculado con el promedio de GST) y de 4.467 (calculado con el promedio de FST obtenido de biosys) (Cuadro 6). El valor de Nm mayor a uno indica que existe alto flujo génico entre las poblaciones locales estudiadas de E. bilobatum (Slatkin, 1987).

Identidades genéticas. Las identidades genéticas de Nei (1978) tienen un intervalo de 0.976 a 0.993 (Cuadro 7), con un promedio de I = 0.986, indicando que las cuatro poblaciones locales estudiadas son muy parecidas genéticamente. Las poblaciones más parecidas genéticamente son la Huimilpan 1 y la Amealco 3, que no son las más cercanas geográficamente. La población con mayor grado de diferenciación es la San Juan 2, la cual no es la más alejada geográficamente (Figura 1).
Discusión
Al comparar la diversidad genética total de Eriocaulon bilobatum (HT= 0.221), con los promedios registrados por Hamrick y Godt (1990) para especies terrestres con historias de vida similares, tales como especies anuales (HT= 0.330), con distribución restringida (HT= 0.300), con entrecruzamiento por animales (HT= 0.310) y con reproducción sexual y asexual (HT= 0.305), se observa que sus niveles de diversidad total son mas bajos que en especies terrestres. Estos resultados contradicen la generalización de que, a nivel de especie, las angiospermas acuáticas presentan mayor diversidad genética que las terrestres (Barrett et al., 1993; Laushman, 1993). Por otro lado, al comparar la diversidad genética a nivel de población de E. bilobatum (A=2.32, P=69.65, He=0.197) con el promedio registrados por Hamrick y Godt (1990) para especies terrestres con historias de vida similares, tales como especies anuales (A=1.48, P=30.20, He=0.105), con distribución restringida (A=1.45, P=30.60, He=0.105), con entrecruzamiento por animales (A=1.54, P=35.90, He=0.124) y con reproducción sexual y asexual (A=1.47, P=29.40, He=0.103), se observa que E. bilobatum presenta niveles más altos de variación genética. Estos resultados contradicen la generalización de que, a nivel de población, las angiospermas acuáticas presentan menor diversidad genética que las terrestres (Barrett et al., 1993; Laushman, 1993). Los resultados de este estudio muestran, que en comparación con especies terrestres, E. bilobatum presenta baja diversidad genética total y alta diversidad genética a nivel población, es posible que esto se deba a que las cuatro poblaciones locales estudiadas comparten una gran proporción de la diversidad total, es decir, la mayor parte de la diversidad genética se encuentra dentro de las poblaciones (HS = 0.210) y poca entre las poblaciones (DST = 0.012).
Por otra parte, al comparar los niveles de variación genética dentro de las poblaciones de Eriocaulon bilobatum con los registrados en los pocos estudios hechos en especies acuáticas con historias de vida similares, se observa que E. bilobatum (A=2.32, Ae=1.31, P=69.65, Ho=0.134, He=0.197) presenta valores parecidos a las especies acuáticas entomófilas tales como Limnanthes alba (A=2.04, P=63.00), Sagittaria isoetiformis (A=2.33, P=67.61, He=0.218) y Sagittaria teres (A=2.19, P=48.21, He=0.101) (Elam, 1998; Edwards y Sharitz, 2000) y niveles más altos que los de especies acuáticas autógamas tales como Limnanthes bakeri (A=1.20, P=22.00, He=0.114) y L. floccosa (A=1.43, P=23.00, He=0.003) (Hickman, 1993; Elam, 1998). Los resultados muestran que existe endogamia moderada dentro de las poblaciones de E. bilobatum (FIS = 0.312), lo cual sin duda está relacionado con su sistema de reproducción y puede explicarse por el comportamiento del polinizador que visita flores contiguas que pueden ser del mismo individuo. Sin embargo, los niveles de endogamia también pueden ser el resultado de la falta de flujo génico (transporte de polen, semillas, frutos y/o propágulos clonales) entre poblaciones (Elam, 1998). Aunque los valores de Nm indican que existe alto flujo génico (Nm = 4.92 y 4.46), no hay que pasar por alto que este estadístico se calcula a partir de los valores de FST o GST , asumiendo que la diferenciación o similitud entre poblaciones es el resultado directo del flujo génico. Sin embargo, el hecho de que las poblaciones estudiadas son muy parecidas genéticamente (FST = 0.053 y GST = 0.048), también puede deberse a cuestiones históricas (polimorfismos ancestrales), es decir, existe la posibilidad de que los genotipos colonizadores provengan de una misma fuente.
Al analizar las frecuencias alélicas (Cuadro 1) se observa que no existen loci exclusivos en ninguna población, por lo que no existen indicios de una divergencia inicial. Se observa que las poblaciones con mayor grado de aislamiento geográfico (Amealco 3 y Juventino Rosas 4) (Figura 1) son las que presentan valores significativos de endogamia, lo cual puede indicar que en realidad existe poco flujo génico entre poblaciones. Si Eriocaulon bilobatum se dispersara por medio de propágulos clonales, además de la baja diferenciación genética, existiría baja diversidad genética dentro de las poblaciones, situación que no se presenta en este estudio. Tampoco es probable que el flujo de genes a larga distancia sea por polen, ya que, por cuestiones energéticas, generalmente los insectos pequeños tienen patrones de movimiento restringido (Loveless y Hamrick, 1984). En cuanto a los mecanismos de dispersión, se descartan el agua y las actividades humanas, ya que no existen corrientes de agua que unan a los humedales temporales y no se conoce algún uso humano para esta especie. Por lo que es muy probable que el flujo génico sea por medio de las semillas y que éstas sean dispersadas a través del viento y aves. Se esperaba que la deriva génica fuera un proceso determinante en la estructura genética, ya que muchas plantas acuáticas anuales que están adaptadas a ambientes efímeros (como los humedales de estudio), pasan regularmente por "cuellos de botella" y ciclos de colonización – extinción (Barrett et al., 1993). Sin embargo, no se encontró evidencia de la acción de la deriva génica en la estructura poblacional de E. bilobatum, ya que no existe alta diferenciación ni baja diversidad genética, que son dos consecuencias importantes de esta fuerza evolutiva. Es probable que la presencia de un banco de semillas esté jugando un papel importante en el reclutamiento de nuevos individuos, debido a que los humedales temporales de estudio pasan por períodos de sequía – inundación, es necesario que las plantas tengan mecanismos que les permiten sobrevivir de un periodo a otro (banco de semillas). La persistencia del banco de semillas amortigua los "cuellos de botella" y permite que la diversidad genética alta se mantenga en las poblaciones (Edwards y Sharitz, 2000).
Al confrontar los resultados con la hipótesis inicial, se observa que la diversidad genética dentro de las poblaciones de Eriocaulon bilobatum es mayor a la que esperada, probablemente su sistema de reproducción sea el factor más importante en el incremento de su diversidad genética. Debido a la presencia de alelos raros, también es posible que la mutación sea un proceso evolutivo que esté incrementando la diversidad genética. Los resultados en cuanto al flujo génico son contrarios a los planteados en la hipótesis, sin embargo, debido a los niveles de endogamia dentro de éstas, consideramos que en realidad existe bajo flujo génico entre poblaciones y que probablemente la similitud genética se debe a factores históricos.
La diversidad y estructura genética de Eriocaulon bilobatum está determinada por el sistema de reproducción y su historia de vida más que el hecho de vivir en ambientes acuáticos. Los resultados obtenidos en este estudio son contrarios a los resultados de la mayoría de los estudios en plantas acuáticas, en los que se ha encontrado un patrón general de baja diversidad genética a nivel poblacional, alta diversidad a nivel de especies y alta diferenciación poblacional (Barrett et al., 1993; Laushman, 1993). Por el contrario, E. bilobatum presenta alta diversidad genética a nivel de población, menor diversidad a nivel de especie y baja diferenciación poblacional.
Agradecimientos
Los autores agradecen a P. Balderas por su ayuda en el trabajo de campo, a A. Beltrán por su ayuda en el laboratorio, a R. Luna–Reyes y N. Pérez–Naser por sus enseñanzas en la técnica de electroforesis, a K. Foote por la determinación taxonómica del díptero y a C. Rojas por la elaboración del mapa de distribución. Esta investigación fue financiada por el Consejo Nacional de Ciencia y Tecnología (CONACYT) (29104N) y por el Consejo de Ciencia y Tecnología de Querétaro (CONCYTEQ).
Literatura citada
Barrett S., Eckert C. y Husband B. 1993. Evolutionary processes in aquatic plant populations. Aquatic Botany 44:105–145. [ Links ]
Calderón G. 1996. Familia Eriocaulaceae. Flora del Bajío y de regiones adyacentes. Fascículo 46. Instituto de Ecología A.C. Centro Regional del Bajío. Pátzcuaro Michoacán, México. [ Links ]
Cheliak W. y Pitel J. 1984. Techniques for starch gel electrophoresis of enzymes from forest tree species. information Report PI–X–42. Petawawa National Forestry Institute. Chalk River, Ontario. [ Links ]
Culley T.M., Wallace L.E., Gengler–Nowak K.M. y Crawford D.J. 2002. A comparison of two methods of calculating GST, a genetic measure of population differentiation. American Journal of Botany 89:460–465. [ Links ]
Edwards A.L. y Sharitz R.R. 2000. Population genetics of two rare perennials in isolated wetlands: Sagittaria isoetiformis and S. teres (Alismataceae). American Journal of Botany 87:1147–1158. [ Links ]
Elam D. 1998. Population genetics of vernal pool plants: Theory, data and conservation implications. En: Witham C.W., Bauder E., Belk D., Ferren W. y Ornduff R. Eds. Ecology, Conservation and Management of Vernal Pool Ecosystems, pp.180–189, California Native Plants Society. Sacramento, CA. [ Links ]
Hamrick J.L., Linhart Y.B. y Mitton J.B. 1979. Relationships between life history characteristics and electrophoretically detectable genetic variation in plants. Annual Review of Ecology and Systematics 10:173–200. [ Links ]
Hamrick J.L. y Godt M.J. 1990. Allozyme diversity in plant species. En: Brown A.H.D., Clegg M.T., Kahler A.L. y Weir B.S. Eds. Plant Population Genetics, Breeding, and Genetic Resources, pp.43–63, Sinauer Associates, Inc Publ. Sunderland, MA. [ Links ]
Hartl D.L. y Clark A.G.1997. Principles of Population Genetics. Sinauer Associates, Inc Publ. Sunderland, MA. [ Links ]
Hedrick PW. 2000. Genetics of Populations. Jones & Barlettl Publ. Sudbury MA. [ Links ]
Hickman J.C. 1993. The Jepson Manual, higher plants of California. University of California Press. CA. [ Links ]
Laushman R.H. 1993. Population genetics of hydrophilous angiosperms. Aquatic Botany 44:147–158. [ Links ]
Li C.C. y Horvitz D.G. 1953. Some methods of estimating the inbreeding coefficient. American Journal of Human Genetics 5:107–117. [ Links ]
Loveless M.D. y Hamrick J.L. 1984. Ecological determinants of genetic structure in plant populations. Annual Review of Ecology and Systematics 15:65–95. [ Links ]
Magallán F. 2004. Diversidad genética de plantas acuáticas en los charcos temporales de Amealco y Huimilpan, Querétaro. Tesis de Doctorado. Universidad Autónoma de Querétaro. 121 p. [ Links ]
Moore P.D., Webb J.A. y Collins M.E. 1991. Pollen Analysis. Blackwell Scientific, Oxford, UK. [ Links ]
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics 89:583–590. [ Links ]
Novelo A. 2000. Informe final del proyecto S133. Inventario de la vegetación acuática vascular de cuatro regiones hidrológicas prioritarias del centro de México. CONACYT. México. [ Links ]
Rocha M. y Gasca J. 2007. Ecología molecular de la conservación. En: Eguiarte L., Souza V. y Aguirre X. Eds. Ecología Molecular, pp.251–272. SEMARNAT–INE–UNAM–CONABIO, México. [ Links ]
Slatkin M. 1987. Gene flow and the geographic structure of natural populations. Science 236:787–792. [ Links ]
Swofford D. y Selander R. 1981. Biosys–1: A computer program for the analysis of allelic variation in population genetics and biochemical systematics. User's manual. Illinois Natural History Survey, Chicago IL. [ Links ]
Wendel J. y Weeden N. 1989. Visualization and interpretation of plant isozymes. En Soltis D. y Soltis P. Eds. Isozymes in Plant Biology, pp.5–45. Dioscorides Press. Portland, Oregon. [ Links ]
Werth C. 1985. Implementing an isozyme laboratory at a field station. Virginia Journal of Science 36:53–76. [ Links ]
Workman P. y Niswander D. 1970. Population studies on Southwestern Indian tribes. II. Local genetic differentiation in the Pa–pago. American Journal of Human Genetics 22:24–29. [ Links ]
Yeh F.C.H. y O'Malley D. 1980. Enzyme variations in natural populations of Douglas fir, Pseudotsuga menziesii (Mub.) Franco, from British Columbia. 1. Genetic variation patterns in coastal populations. Silvae Genetica 29:83–92. [ Links ]














