Services on Demand
Journal
Article
Indicators
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Boletín de la Sociedad Botánica de México
Print version ISSN 0366-2128
Bol. Soc. Bot. Méx n.83 México Dec. 2008
Filogeografía
Fragmentación y expansión demográfica en las poblaciones mexicanas de Pinus ayacahuite var. ayacahuite
Range expansion and fragmentation in Mexican populations of Pinus ayacahuite
Alejandra Ortíz–Medrano1,2, Alejandra Moreno–Letelier1 y Daniel Piñero1
1 Departamento de Ecología Evolutiva, Instituto de Ecología, Universidad Nacional Autónoma de México, Apdo. Postal 70–275, C.P. 04510, México D.F. 2 Autor para la correspondencia. Correo–e: aortiz@ecologia.unam.mx
Recibido: 8 de febrero de 2008.
Aceptado: 18 de agosto de 2008.
Resumen
Se determinó la estructura filogeográfica y demografía histórica de las poblaciones mexicanas de Pinus ayacahuite var. ayacahuite usando tres microsatélites de cloroplasto en 198 individuos de 14 poblaciones. Se encontraron 12 haplotipos y niveles medios de diversidad genética (He = 0.705). Se definieron dos grupos máximamente diferenciados con un análisis espacial de varianza molecular. Entre estos grupos se detecta correlación significativa entre las distancias genéticas y las geográficas, pero no dentro de ellos. Se encontró estructura filogeográfica significativa en la muestra producida por la existencia de los dos grupos. Además, se detectaron dos expansiones demográficas, la primera en toda la especie, y la segunda sólo en las poblaciones más sureñas. Finalmente se realizó un análisis de clados anidados para complementar nuestras observaciones. Los resultados muestran que el Istmo de Tehuantepec actuó como corredor para las especies de afinidades templadas en algún periodo frío de inicios del Pleistoceno, y posteriormente como una barrera.
Palabras clave: demografía histórica, filogeografía, Pinaceae, Pleistoceno.
Abstract
This study explores the phylogeographic structure and the demographic history of the Mexican populations of Pinus ayacahuite var. ayacahuite. Three chloroplast microsatellites were amplified in 198 individuals from 14 populations. Twelve haplotypes were found and an average genetic diversity (He) of 0.705. Two maximally differentiated groups were determined with a spatial analysis of molecular variance. A significant correlation was detected between the genetic and geographic distances between these two groups, but not within them. A significant phylogeographic structure was found, produced by the existence of the two groups. Two demographic expansions were detected, the first in the entire species, the second only in the southernmost populations. Additionally, a nested clade analysis was performed to complement our observations. The results showed that the Tehuantepec Isthmus was a corridor for species of temperate affinities in a cold period in early Pleistocene, and later acted as a barrier.
Key words: historical demography, phylogeography, Pinaceae, Pleistocene.
El género Pinus incluye aproximadamente 110 especies, de las cuales alrededor del 50% habitan en México, haciendo de este país el de mayor diversidad de este género en el mundo. Las palinofloras del Terciario en México registran la presencia más antigua de pinos en el norte de este país en el Oligoceno. Durante el Mioceno se encuentra a los géneros Pinus y Picea en lo que actualmente es el estado de Veracruz, aunque con una representación muy pobre. Es durante el Plioceno que tanto la abundancia como la diversidad de los elementos de flora neártica aumentan hacia el sur de México y Centroamérica (Graham, 1999). En este registro de elementos de climas templados en Latinoamérica se observa un patrón que incluye al tiempo (temprano a tardío) y a la dirección (norte a sur), y coincide con los cambios climáticos que sucedieron en esos periodos.
Pinus ayacahuite Ehrenberg (subgénero Strobus), es una especie que se distribuye desde el centro de México hasta Centroamérica; se encuentra desde la latitud 15°15'N hasta 20°55'N, y en la longitud de 92°10'W a 103°55'W (Eguiluz Piedra, 1978). Crece entre los 2200 y los 3000 msnm y se localiza principalmente en lomas y cañadas. Tiene preferencia por lugares sombríos y húmedos, el clima donde se desarrolla es templado caliente con temperaturas medias anuales de 13°C, y con una precipitación de 700 a 1200 mm anuales (Eguiluz Piedra, 1978). Se le conocen tres variedades: la variedad brachyptera del norte del país, la variedad veitchii del centro de éste, y la variedad típica, que es la que se considera en este trabajo. En todo caso, aún existe controversia sobre si la variedad brachyptera deba ser reconocida como una especie diferente, como Pinus strobiformis (Perry, 1991; Farjon y Styles 1997; Musálem y Ramírez, 2003; Moreno–Letelier y Piñero, en prensa).
Los patrones geográficos de la variación genética tanto en animales como en plantas han mostrado ser el resultado de procesos contemporáneos, históricos, y/o una combinación de ambos (Templeton et al., 1995; Cuenca et al., 2003). El impacto de las glaciaciones en diferentes especies de coniferas ha sido evaluado con métodos filogeográficos en diferentes estudios (Echt et al., 1998; Vendramin et al., 1999; Marshall et al., 2002; Hwang et al., 2003), y en particular existen varios que se refieren al género Pinus (Mitton et al., 2000; Burban y Petit, 2003; Afzal–Rafii y Dodd, 2007; Bucci et al., 2007; Moreno–Letelier y Piñero, en prensa). Sin embargo, éstos se concentran en especies de Europa y Norteamérica, donde en general los periodos fríos obligaron a las especies a migrar hacia el sur y restringieron sus áreas de distribución a los refugios pleistocénicos, desde donde después, en los periodos postglaciales, pudieron recolonizar hacia el norte. A pesar de que el sureste mexicano es considerado como un área de gran endemismo (Marshall y Liebherr, 2000), no existen muchos estudios filogeográficos sobre las especies de afinidades templadas en esta zona, los cuales podrían contribuir al entendimiento de los procesos históricos que dieron forma a la estructura genética actual de las especies. En este sentido, Pinus ayacahuite var. ayacahuite puede ser un buen modelo para entender el impacto de los cambios climáticos históricos en los patrones de distribución de especies de afinidades templadas en climas subtropicales, particularmente del sur de México.
Los microsatélites son secuencias simples y cortas (de una a seis bases) que se repiten en segmentos de menos de un kilobase y se encuentran en el genoma de todos los organismos eucariontes y organelos (Schlötterer, 2000). Se conoce que las secuencias de microsatélites pueden ser diferentes en el número de repeticiones de individuo a individuo, en general son muy polimórficos y se consideran selectivamente neutros, por lo que se han convertido en un marcador molecular muy usado en los estudios de genética de poblaciones (Chambers y MacAvoy, 2000; Schlöterer, 2000). En particular, los microsatélites de cloroplasto resultan muy útiles para el análisis filogeográfico ya que el cromosoma del cloroplasto es una molécula no recombinante y por lo tanto todos sus loci están ligados (Navascués y Emerson, 2005). Estos marcadores se han utilizado exitosamente en el estudio de estructura genética de poblaciones y filogeografía de coníferas (Petit et al., 2002; Burban y Petit, 2003; Vaxevanidou et al., 2006; Afzal–Raffi y Dodd, 2007), y en algunos casos se ha encontrado que son de mayor utilidad que otros marcadores para este tipo de estudios debido a sus altos niveles de variación (Escalante, 2001).
El propósito de este trabajo fue estudiar la estructura genética de Pinus ayacahuite var. ayacahuite en México, determinar si existe estructura filogeográfica y explorar las posibles causas históricas de esto, así como inferir los procesos demográficos que han operado en las poblaciones de esta especie.
Materiales y Métodos
Poblaciones. Las poblaciones fueron elegidas tratando de cubrir la distribución de Pinus ayacahuite var. ayacahuite en México, y están basadas en las poblaciones referidas por Farjon y Styles (1997), Perry (1991), y en las colecciones depositadas en el Herbario Nacional (MEXU), del Instituto de Biología, UNAM y en el herbario IEB del Instituto de Ecología, A.C. (Apéndice 1). En cada población se colectaron acículas de individuos elegidos al azar. En total se colectaron 198 individuos en 14 poblaciones, con un número variable de individuos en cada población (Apéndice 1). Extracción de ADN y amplificación de microsatélites. Se extrajo el ADN total de cada individuo con el protocolo modificado de CTAB 2% (Vázquez–Lobo, 1996). Se amplificaron tres loci de microsatélites de cloroplasto basados en los polimórficos para P. strobiformis (Pt30204, Pt71936 y Pt63718, Vendramin et al., 1996; Moreno–Letelier y Piñero, en prensa) con las siguientes condiciones para un volumen final de 25μl: 10mM de buffer Tris–HCL (Promega), 2.5mM de MgCl2, 0.2μM de cada primer, 150μM de cada dNTP, 25ng de ADN y 1U de polimerasa Taq (Promega). La amplificación se llevó a cabo en un termociclador Applied Biosystems 2720 con los siguientes parámetros: (1) calentamiento inicial a 95°C por 5 minutos, (2) 25 ciclos de un minuto de desnaturalización a 95°C, alineación a 55°C por 1 minuto, y extensión de 1 minuto a 72°C, (3) extensión final de 1 minuto a 72°C. Posteriormente se corrieron estos productos en geles de acrilamida 6%, urea 7M, a 60W, con diferentes tiempos dependiendo del tamaño del microsatélite. Los geles se tiñeron con nitrato de plata, y la determinación del tamaño de los alelos se hizo visualmente, tomando como referencia a un individuo previamente secuenciado cuyo tamaño para cada microsatélite era conocido; este individuo se incluyó en cada gel.
Variación y estructura genética. Se asignaron como haplotipos a cada combinación diferente de los tamaños de las variantes en los tres loci utilizados. Con estos datos se estimó la diversidad genética de Nei (1987). La estructura genética de las poblaciones fue estimada con los valores pareados de Rst. La diversidad genética y los valores pareados de RST y FST fueron obtenidos con el software Arlequin 3.1 (Excoffier et al., 2005). Para definir la estructura genética de poblaciones se usó el software SAMOVA 1.0 (Dupanloup et al., 2002). Este programa define grupos de poblaciones que están máximamente diferenciados entre ellos (aquellos para los cuales la proporción de varianza genética total debida a diferencias entre grupos es máxima, es decir ΦCT), con el fin de encontrar barreras genéticas y geográficas, además de realizar un análisis jerárquico de varianzas moleculares (AMOVA).
Filogeografía y demografía histórica. Prueba de permutaciones alélicas.– Se efectuó la prueba de comparación entre FST y RST desarrollada por Hardy et al. (2003) con el programa SPAGeDI (Hardy y Vekemans, 2002). La prueba compara el valor observado de RST con el valor esperado cuando las diferencias en los tamaños alélicos no contribuyen a la diferenciación genética (pRsT). Bajo la hipótesis nula, el procedimiento no debe afectar las medidas de diferenciación como Rst, pero si los tamaños alélicos contribuyen a la diferenciación genética, pRsT dependerá sólo de la identidad y tendrá un valor menor a Rst. Es importante notar que pRsT tiene un valor igual al FST observado de los datos. Esta comparación puede responder a la pregunta de si los alelos más relacionados están dentro de cada población o en poblaciones distintas, es decir, si existe alguna señal filogeográfica dentro de las poblaciones (Hardy y Vekemans, 2002).
Prueba de Mantel.– Para verificar si los distintos alelos más relacionados están entre poblaciones cercanas o entre poblaciones distantes se realizó una prueba de Mantel con el programa XLSTAT para Microsoft Excel, con la que se evaluó la relación entre una matriz de distancias geográficas y una matriz de distancias genéticas. El coeficiente de correlación r se estimó con 10000 permutaciones. Las distancias geográficas fueron obtenidas con un calculador de distancias entre localidades geográficas (Bogan, http://www.go.ednet.ns.ca/~larry/bsc/jslatlng.html). Para la matriz de distancias genéticas se usaron los valores pareados de RST.
Distribución mismatch.– La distribución mismatch se basa en el hecho de que la información genética molecular, en particular la distribución de diferencias pareadas entre sitios nucleotídicos o de restricción, contiene información sobre los tamaños poblacionales pasados (Rogers, 2004). Un crecimiento poblacional súbito produce distribuciones unimodales, mientras que una población con tamaño estable o que está declinando produce distribuciones multimodales. Para el análisis se utilizó el software Arlequín 3.1 (Excoffier et al., 2005), donde se codificaron los datos de micro satélites binariamente: el número de repeticiones se codificó con "1", y los alelos más cortos se codificaron llenando las diferencias de repeticiones con "0" (Navascués et al., 2006; Moreno–Letelier y Piñero, en prensa). El análisis se llevó a cabo con toda la muestra de tal manera que los patrones encontrados se refieren a los procesos por los que ha pasado la especie completa, y también para dos grupos diferenciados genéticamente, para encontrar posibles procesos posteriores dentro de estos grupos. El poder del estimado se evaluó con la distribución de la suma de diferencias cuadradas (SSD, por sus siglas en inglés) obtenida de un bootstrap de 10,000 réplicas. Con el bootstrap también se estimó el intervalo de confianza de 95% del parámetro τ. La fecha de la expansión poblacional se calculó con la fórmula τ = 2Lμt, donde L es el número de loci de microsatélites, μ es la tasa de mutación por locus, y t es el tiempo en generaciones (Navascués et al., 2006). Debido a que no existe un estudio demográfico completo de P. ayacahuite, se utilizó como tiempo de generación la edad a la primera reproducción de P. strobiformis, 15 años (Krugman y Jenkinson, 1974), la cual podría ser tomada como un estimado mínimo del tiempo de generación.
Análisis de clados anidados.– La red de haplotipos se generó con el programa TCS 1.21 (Clement et al., 2000) que utiliza la parsimonia estadística para hacer un algoritmo que comienza estimando el número máximo de diferencias entre haplotipos como resultado de sustituciones simples con un 95% de confianza. Posteriormente se realizó un análisis de clados anidados con el programa GeoDis 2.5 (Posada et al., 2000), y se determinaron eventos demográficos con la clave de inferencia para este programa (noviembre 11 de 2005, http://darwin.uvigo.es/software/geodis.html).
Resultados
Diversidad genética. Las combinaciones de los tres loci dieron en total 12 haplotipos. La diversidad genética para toda la muestra fue de He = 0.705 (0.027), Osmm= 5.260, Oam= 1.833 (0.247).
Estructuración genética. El valor de Φct más alto definido por SAMOVA se encontró con dos grupos de poblaciones: el primero (grupo A) formado por las poblaciones de El Porvenir, Rancho Nuevo y Arcotete, el segundo (grupo B) por las poblaciones de Omiltemi, Trinidad, Ixtlán, Suchixtepec, San Rafael, Pingüica, Agua Blanca, Chichicaxtla, Ciénega Larga, Pueblo Nuevo y Zempoala (figura 1). El AMOVA para estos grupos muestra que 83.19% de la variación encontrada es entre grupos, 2.53% entre poblaciones dentro de los grupos, y 14.28% entre poblaciones. El AMOVA para el grupo A muestra que el 99.06% de la variación encontrada está dentro de las poblaciones, mientras que el 0.94% está entre poblaciones. Para el grupo B, 75.11% de la variación está dentro de las poblaciones y 24.89% entre ellas.
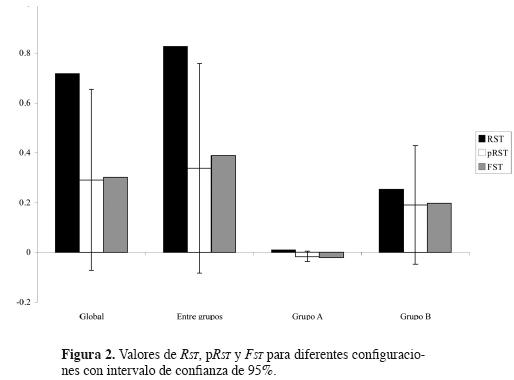
Contribución de las mutaciones paso a paso – Prueba de permutaciones alélicas. Con el programa SPAGeDi se encontró que RST = 0.8277 > FST = 0.3883, p < 0.05. En este caso, así como en la comparación con todas las poblaciones, el valor de RST no se encuentra dentro del intervalo de confianza del 95% de pRsT promedio (figura 2).
Prueba de Mantel. La correlación entre la matriz de distancias geográficas y la matriz de distancias genéticas resultó significativa (r= 0.649, p<0.05, figura 3). Al hacer esta misma prueba dentro de cada grupo, los resultados son diferentes, ya que en ninguno de ellos se encontró correlación significativa entre las distancias geográficas y las genéticas (p = 0.85 y 0.35 para el grupo A y el grupo B, respectivamente).

Distribución mismatch o de diferencias pareadas. Los resultados para detectar expansiones poblacionales se reportan en el cuadro 1. A partir de τ se calcularon las fechas de expansión; para toda la muestra fue de 398,750 años, mientras que para el grupo A fue de 249,000 años (figuras 4 y 5).



Análisis de clados anidados. El clado 1–1 y el 1–2, que conforman el clado 2–1, están formados por haplotipos que se encuentran casi exclusivamente en el grupo B, excepto por el H3, el haplotipo más común en la muestra y cuya representación en el grupo A es mínima (< 6%, apéndice 2). Los clados 1–3 y 1–4, que juntos forman al clado 2–2, tienen a la mayoría de sus haplotipos representados exclusivamente en el grupo A (figura 6). A pesar de estas marcadas diferencias entre los grupos, la red de haplotipos no muestra grandes discontinuidades genéticas, ningún haplotipo se distingue de sus más cercanos por más de un paso mutacional, lo cual indica que divergieron recientemente (Templeton, 2006).
Los resultados del análisis de clados anidados según la clave de inferencia son en su mayoría inconclusos ya que en la mayoría se obtuvo que el muestreo fue inadecuado para poder distinguir entre varios fenómenos (cuadro 2); a pesar de que tratamos de cubrir la distribución completa en México de la especie, es probable que existan poblaciones que no incluimos en este estudio, así como hibridización con Pinus ayacahuite var. veitchii cuyo rango de distribución incluye espacios intermedios entre los grupos A y B definidos aquí.

Discusión
Los resultados del análisis del ADN de cloroplasto de Pinus ayacahuite var. ayacahuite muestran que existen dos pozas genéticas diferenciadas; los dos grupos diferenciados por SAMOVA están también claramente identificados en la prueba de permutaciones, en la prueba de Mantel y en la red de haplotipos (figuras 2, 3 y 6). Los valores de RST más altos que los de FST –p(RsT)– indican que la tasa de mutación no es despreciable en comparación con la tasa de migración, y que por lo tanto es probable que las mutaciones paso a paso hayan contribuido a la diferenciación genética. En este caso, esto se cumple en la comparación de todas las poblaciones y especialmente en la comparación entre grupos, lo cual responde afirmativamente a la pregunta de si los distintos haplotipos están más relacionados dentro de las poblaciones que entre ellas. Esto significa que entre grupos la tasa de mutación es un aspecto importante en la diferenciación. Sin embargo, este último supuesto no se cumple cuando se analiza cada grupo independientemente, es decir, dentro de los dos grupos los haplotipos están más relacionados entre poblaciones que dentro de cada una de ellas, por lo que la tasa de mutación es despreciable en comparación con la de migración. La prueba de Mantel mostró un patrón similar, pues el resultado fue significativo sólo en la comparación de todas las poblaciones y no en las comparaciones dentro de cada grupo. Este análisis indica que los diferentes haplotipos están más relacionados entre poblaciones cercanas que entre poblaciones más distantes, lo cual señala que las poblaciones de cada grupo están más relacionadas entre sí que con las poblaciones del otro grupo, pero que dentro de cada grupo la distancia geográfica entre poblaciones no interviene en la diferenciación genética. Sin embargo, el análisis de clados anidados indica que dentro del grupo B existe aislamiento por distancia en todas las poblaciones y a diferentes niveles; un patrón de aislamiento por distancia detectado en una prueba de Mantel debe ser tratado con cuidado ya que puede deberse a efectos históricos, en vez de ser el reflejo de un balance entre la deriva génica y el flujo génico mediado por la geografía en escalas de tiempos menores, es por esta razón que se recomienda realizar pruebas de Mantel parciales (Pires de Campos Telles y Diniz–Filho, 2005); de forma paralela, la correlación entre la matriz de distancias genéticas y geográficas puede no resultar significativa, siendo esto resultado de efectos de otras variables involucradas en la estructura de las distancias genéticas, sin que se excluya totalmente el aislamiento por distancia. En otros estudios donde se utilizó la prueba de permutaciones con microsatélites se han encontrado resultados similares, donde RST es significativamente mayor a pRsT en escalas geográficas grandes, y cercano a FST en escalas menores (para una revisión ver Hardy et al., 2003). Particularmente, Afzal–Raffi y Dodd (2007) con microsatélites de una especie de pino, Pinus nigra, llevaron a cabo un análisis entre seis regiones y dentro de ellas; en todas las comparaciones entre regiones encontraron que RST era significativamente mayor a pRsT, mas no así dentro de cada región. Estos resultados son consistentes con un modelo donde, en escalas pequeñas, la tasa de migración ha determinado la baja diferenciación, y donde, en escalas grandes, la tasa de migración es similar o inferior a la tasa de mutación y/o donde los dos grupos han divergido por un largo tiempo. De ser esto cierto, el modelo de mutación (y por lo tanto el tamaño de los alelos) no tendría relevancia en escalas pequeñas, por lo cual FST sería el parámetro elegido en este nivel de análisis. Por el contrario, RST sería el indicado en escalas grandes, pues es en estos niveles donde el modelo de mutación y los tamaños alélicos son informativos sobre la historia evolutiva (Hardy et al., 2003).
Estructuración entre grupos. Cuando se encuentra una estructura genética y geográfica, el patrón puede ser resultado de dos procesos distintos. El primero es un evento de vicarianza que restringe el flujo génico entre juegos de poblaciones por aislamiento geográfico. El segundo es el aislamiento por distancia, donde las poblaciones intercambian más migrantes con las poblaciones vecinas que con las distantes. En teoría, el aislamiento por distancia resulta en una relación directa entre las distancias genéticas y geográficas, pero en la realidad estas dos alternativas no pueden ser distinguidas fácilmente debido a que la dispersión de los puntos en la regresión de distancias es por lo general muy grande (Bossart y Prowell, 1998). Para distinguir entre estas dos posibilidades, Bossart y Prowell (1998) recomiendan estudiar los efectos de las poblaciones por separado o en conjunto en la correlación de distancias geográficas y genéticas, para evaluar si ciertas poblaciones o grupos de poblaciones son responsables de la correlación del aislamiento por distancia. Cuando los valores significativos de la correlación están asociados a poblaciones solas o a conjuntos de ellas, sugieren a la vicarianza como la explicación más viable, mientras que si la significancia está dispersa a través de los pares de poblaciones, apoyan al aislamiento por distancia. Como se mencionó antes, las pruebas de Mantel realizadas mostraron que la correlación entre distancias genéticas y geográficas resultó significativa al comparar a todas las poblaciones, pero al comparar cada grupo no se encontraron pruebas de aislamiento por distancia, por lo que proponemos que entre los grupos ocurrió un evento de vicarianza. Este resultado no excluye que haya habido eventos de flujo génico restringido por aislamiento por distancia, aunque éstos tuvieron que haber sido posteriores a la fragmentación, según el orden de anidamiento (Templeton, 2006).
Vicarianza: el Istmo de Tehuantepec. El Istmo de Tehuantepec es considerado como una barrera biogeográfica ya que durante mucho tiempo estuvo sumergido bajo el agua, y aún al emerger, debido a su baja elevación, los hábitats ecológicos que presenta son distintos a los de las elevaciones que separa (Peterson et al., 1999; Marshall y Liebherr, 2000). Actualmente forma una barrera parcial entre los pinos de las montañas de Oaxaca y los pinos de las tierras altas de Chiapas (Perry, 1991). La prueba de mismatch detectó expansión poblacional cuando todas las poblaciones juntas fueron analizadas y en el grupo A.
Esto quiere decir que ocurrió una primera expansión poblacional antes de la divergencia de los dos grupos, y posteriormente otra en el sur de México. Determinar la fecha exacta de las expansiones demográficas es difícil, dado que ni la tasa de mutación ni el tiempo de generación se conocen con certeza en esta especie. Sin embargo, la historia geológica en relación con los tiempos de expansión puede proporcionar las pistas necesarias para calcular esta fecha. Esta aproximación se ha llevado a cabo anteriormente con una expansión poblacional de pinos revelada por microsatélites de cloroplasto (Navascués et al., 2006). El Istmo de Tehuantepec emergió durante el Plioceno (Perry, 1991), lo cual coincide con un enfriamiento general y drástico de la Tierra durante el Mioceno tardío y el Plioceno, probablemente reflejo de las etapas tempranas de las glaciaciones (Savin, 1977). Las composiciones palinológicas de México y Centroamérica de estas épocas indican también un patrón de baja de temperatura. Más aún, existe evidencia de la presencia de un bosque frío en estas regiones, así como de que estas condiciones frías fueron progresivamente en descenso después del Plioceno, cuando las condiciones cálidas y tropicales comenzaron a dominar (Graham, 1987; 1999). Durante el último período glaciar se estima que el decremento de temperatura fue de 5 a 9°C (Vázquez–Selem y Heine, 2004). Existe evidencia de que en regiones tropicales de Centroamérica, donde en el presente no existen glaciares, durante el Pleistoceno fueron suficientemente frías y húmedas como para albergarlos a alturas mayores de 3400 m (Lachniet, 2004). En otros taxa de clima templado se han detectado eventos de vicarianza donde el Istmo de Tehuan–tepec juega también un papel importante (Sullivan et al., 1997; Peterson et al., 1999). Estos eventos se relacionan con las glaciaciones del Pleistoceno temprano y medio, donde el Istmo parece haber sido una fuerte barrera durante las fluctuaciones climáticas. Toledo (1982) sugirió que el efecto de estas fluctuaciones en la distribución de las zonas fitogeográficas en América fue muy dramático, ya que las tierras bajas de México estaban ocupadas por bosques de pino–encino y bosques de niebla durante los periodos fríos. La actual fragmentación de los bosques templados en los diferentes sistemas montañosos aparentemente experimentó muchos ciclos de expansión y contracción durante el Pleistoceno con al menos una expansión que resultó en una biota templada continua y efímera (Sullivan et al., 1997). Esto hubiera propiciado las condiciones necesarias para que Pinus ayacahuite var. ayacahuite expandiera su rango de las montañas de Oaxaca a las tierras altas de Chiapas, dado que los pinos colonizaron México de norte a sur (Perry, 1991).
Los resultados de este trabajo sugieren que el Istmo de Tehuantepec ha jugado dos papeles diferentes en la historia de los bosques templados de México, uno como corredor para éstos y otro como barrera. La estructura filogeográfica e historia demográfica de Pinus ayacahuite var. ayacahuite encontradas en este estudio concuerdan con la hipótesis de que el impacto de los cambios climáticos del Pleistoceno llegó hasta el sur de México y Centroamérica, permitiendo primero a las especies expandirse hacia el sur y después restringiendo el flujo a través del Istmo. Las glaciaciones del periodo Cuaternario tuvieron consecuencias en la distribución de las especies, las cuales se expresan de diferente manera en las zonas boreales, templadas y tropicales (Hewitt, 2000). Se mencionan los refugios pleistocénicos como las áreas geográficas donde las especies sobrevivieron durante las glaciaciones; se han identificado varios de estos lugares especialmente en Europa y Norteamérica (para una revisión ver Hewitt, 2000). En especies de árboles existen varios estudios sobre este tema (para varios ejemplos ver Newton et al., 1999 y Hewitt, 2000). Sin embargo, se conoce poco sobre las consecuencias de las glaciaciones en especies con afinidades ecológicas templadas en latitudes tropicales, como es el caso de Pinus ayacahuite. En estas regiones los períodos fríos habrían sido benéficos para las especies templadas, y durante los períodos interglaciares estas especies habrían sufrido los mismos efectos que las de regiones boreales durante las glaciaciones. De esta forma el concepto de refugio pleistocénico tendría que ser modificado para las especies templadas en regiones tropicales y subtropicales, aunque se requieren aún muchos más estudios filogeográficos para estar en posibilidades de establecer patrones generales.
Agradecimientos
Agradecemos el apoyo de los proyectos PAPIIT (IN215406) y SEMARNAT_CONACYT (C01–0201/A–1) así como la colaboración de Rodolfo Salas, Nadia Santini, Lev Jardón, Alejandra Vázquez y Dieter Wimberger en la colecta de las poblaciones y ayuda en el laboratorio.
Literatura citada
Afzal–Rafii Z. y Dodd R.S. 2007. Chloroplast DNA supports a hypothesis of glacial refugia over postglacial recolonization in disjunct populations of black pine (Pinus nigra) in Western Europe. Molecular Ecology 16: 723–736 [ Links ]
Bogan L., http://www.go.ednet.ns.ca/~larry/bsc/jslatlng.html [ Links ]
Bossart J.L. y Prowell D.P. 1998. Genetic estimates of population structure and gene flow: limitations, lessons and new directions. Trends in Ecology and Evolution 13: 202–206 [ Links ]
Bucci G., González–Martínez S.C, Le Provost G., Plomion C., Ribeiro M.M., Sebastiani F., Alía R. y Vendramin G.G. 2007. Range–wide phylogeography and gene zones in Pinus pinaster Ait. revealed by chloroplast microsatellite markers. Molecular Ecology 16: 2137–2153 [ Links ]
Burban C. y Petit R.J. 2003. Phylogeography of maritime pine inferred with organelle markers having contrasted inheritance. Molecular Ecology 12: 1487–1495 [ Links ]
Chambers G.K. y MacAvoy E.S. 2000. Microsatellites: consensus and controversy. Comparative Biochemistry and Phisiology Part B 126: 455–476 [ Links ]
Clement M., Posada D. y Crandall K. 2000. TCS: a computer program to estimate gene genealogies. Molecular Ecology 9: 1657–1660 [ Links ]
Cuenca A., Escalante A.E. y Piñero D. 2003. Long–distance colonization, isolation by distance, and historical demography in a relictual Mexican pinyon pine (Pinus nelsonii Shaw) as revealed by paternally inherited genetic markers (cpSSRs). Molecular Ecology 12: 2087–2097 [ Links ]
Dupanloup I., Schneider S. y Excoffier L. 2002. A simulated annealing approach to define the genetic structure of populations. Molecular Ecology 11: 2571–2581 [ Links ]
Echt C.S., deVerno L.L., Anzidei M. y Vendramin G.G. 1998. Chloroplast microsatellites reveal population genetic diversity in red pine, Pinus resinosa Ait. Molecular Ecology 7: 307–316 [ Links ]
Eguiluz Piedra T. 1978. Ensayo de integración de los conocimientos sobre el género Pinus en México. Tesis de ingeniero agrónomo especialista en bosques. Departamento de Enseñanza, Investigación y Servicio en Bosques. Escuela Nacional de Agricultura, Chapingo, México. 623 pp. [ Links ]
Escalante A.E. 2001. Estructura genética de poblaciones de Pinus pinceana G. Gordon & Glendinning usando como marcadores moleculares microsatélites de cloroplasto (cpSSR's). Tesis de Licenciatura, Facultad de Ciencias, Universidad Nacional Autónoma de México, Ciudad de México, México. 95 pp. [ Links ]
Excoffier L., Laval G. y Schneider S. 2005. Arlequin ver. 3.0: An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online 1: 47–80 [ Links ]
Farjon A. y Styles B. 1997. Flora Neotropica: Pinus (Pinaceae). New York Botanical Garden. Nueva York, EUA. 293 pp [ Links ]
Graham A. 1987. Tropical American Tertiary floras and paleoenvironments: Mexico, Costa Rica and Panama. American Journal of Botany 74: 1519–1531 [ Links ]
Graham A. 1999. The Tertiary history of the northern temperate element in the northern Latin American biota. American Journal of Botany 86: 32–38 [ Links ]
Hardy O.J., Charbonnel N., Fréville H. y Heuertz M. 2003. Microsatellite allele sizes: a simple test to assess their significance on genetic differentiation. Genetics 163: 1467–1482 [ Links ]
Hardy O. y Vekemans X. 2002. SPAGeDi: a versatile computer program to analyse spatial genetic structure at the individual or population levels. Molecular Ecology Notes 2: 618–620 [ Links ]
Hewitt G. 2000. The genetic legacy of the Quaternary ice ages. Nature 405: 907–913 [ Links ]
Hwang S.–Y., Lin T.–P, Ma C.–S., Lin C.–L., Chung J.–D. y Yang J.–C. 2003. Postglacial population growth of Cunninghamia konishii (Cupressaceae) inferred from phylogeographical and mismatch analysis of chloroplast DNA variation. Molecular Ecology 12: 2689–2695 [ Links ]
Krugman S.L. y Jenkinson J.L. 1974. Pinus L. En: Nisley R.G. Ed. Woody Plant Seed Manual. Disponible en la red http://www.nsl.fs.fed.us/wpsm [ Links ]
Lachniet M.S. 2004. Late quaternary glaciation of Costa Rica and Guatemala, Central America. En: Ehlers y Gibbard Eds. Quaternary glaciations – extent and chronology, Part III, pp. 135138, Elsevier, Amsterdam. [ Links ]
Marshall C.J. y Liebherr J.K. 2000. Cladistic biogeography of the Mexican transition zone. Journal of Biogeography 27: 203216 [ Links ]
Marshall H.D., Newton C. y Ritland K. 2002. Chloroplast phylogeography and evolution of highly polymorphic microsatellites in lodgepole pine (Pinus contorta). Theoretical and Applied Genetics 104: 367–378 [ Links ]
Mitton J., Kreiser B. y Latta R. 2000. Glacial refugia of limber pine (Pinus flexilis James) inferred from the population structure of mitochondrial DNA. Molecular Ecology 9: 91–97 [ Links ]
Moreno–Letelier A. y Piñero D. En prensa. Phylogeographic structure of Pinus strobiformis Engelm. across the Chihuahuan Desert filter–barrier. Journal of Biogeography [ Links ]
Musálem M. y Ramírez A. 2003. Monografía de Pinus ayacahuite. INIFAP. México, D.F. [ Links ]
Navascués M. y Emerson B. 2005. Chloroplast microsatellites: measures of genetic diversity and the effect of homoplasy. Molecular Ecology 14: 1333–1341 [ Links ]
Navascués M., Vaxevanidou Z., González–Martínez S.C., Climent J., Gil L. y Emerson B. 2006. Chloroplast microsatellites reveal colonization and metapopulation dynamics in the Canary Island pine. Molecular Ecology 15: 2691–2698 [ Links ]
Nei M. 1987. Molecular evolutionary genetics. Columbia University Press. Nueva York. [ Links ]
Newton A.C., Allnutt T.R., Gillies A.C.M., Lowe A.J. y Ennos R.A. 1999. Molecular phylogeography, intraspecific variation and the conservation of tree species. Trends in Ecology and Evolution 14: 140–145 [ Links ]
Perry J.P 1991. The pines of Mexico and Central America. Timber Press. EUA. [ Links ]
Peterson A.T., Soberón J. y Sánchez–Cordero V. 1999. Conservatism of ecological niches in evolutionary time. Science 285: 1265–1267 [ Links ]
Petit R.J., Csaikl U.M., Bordács S., Burg K., Coart E., Cottrell J., van Dam B., Deans J.D., Dumolin–Lapègue S., Fineschi S., Finkeldey R., Gillies A., Glaz I., Goicoechea P.G., Jensen J.S., König A.O., Lowe A.J., Madsen S.F., Mátyás G., Munro R.C., Olalde M., Pemonge M., Popescu F., Slade D., Tabbener H., Taurchini D., de Vries S.G.M., Ziegenhagen B., Kremer A. 2002. Chloroplast DNA variation in European white oaks – Phylogeography and patterns of diversity based on data from over 2600 populations. Forest Ecology and Managment 156: 5–26 [ Links ]
Pires de Campos Telles M. y Diniz–Filho J.A.F. 2005. Multiple Mantel tests and isolation–by–distance, taking into account long–term historical divergence. Genetics and Molecular Research 4: 742–748 [ Links ]
Posada D., Crandall K.A. y Templeton A.R. 2000. GeoDis: A program for the Cladistic Nested Analysis of the Geographical Distribution of Genetic Haplotypes. Molecular Ecology 9: 487–488 [ Links ]
Rogers A. 2004. Lecture notes on gene genealogies. www.anthro.utah.edu/~rogers/bio5410/Lectures/a_alu.pdf [ Links ]
Savin S.M. 1977. The history of the earth's surface temperature during the past 100 million years. Annual Review of Earth and Planetary Sciences 5: 319–355 [ Links ]
Schlötterer, C. 2000. Evolutionary dynamics of microsatellite DNA. Chromosoma 109: 365–371 [ Links ]
Sullivan J., Markert J.A. y Kilpatrick C.W. 1997. Phylogeography and molecular systematics of the Peromyscus aztecus species group (Rodentia: Muridae) inferred using parsimony and likelihood. Systematic Biology 46: 426–440 [ Links ]
Templeton A.R. 2006. Population genetics and microevolutionary theory. John Wiley and Sons Inc. Hoboken, New Jersey. 705 pp. [ Links ]
Templeton A.R., Routman R. y Phillips C.A. 1995. Separating population structure from population history: a cladistic analysis of the geographical distribution of mitochondrial DNA hap–lotypes in the tiger salamander, Ambystoma tigrinum. Genetics 149: 767–782 [ Links ]
Toledo V.M. 1982. Pleistocene changes in vegetation in tropical Mexico. En: Prance G.T. Ed. Biological diversification in the tropics, pp 93–111, Columbia University Press, Nueva York. [ Links ]
Vaxevanidou Z., González–Martínez S.C., Climent J. y Gil L. 2006. Tree populations bordering on extinction: A case study in the endemic Canary Island pine. Biological Conservation 129: 451–460 [ Links ]
Vázquez–Lobo A. 1996. Filogenia de hongos endófitos del género Pinus: Implementación de técnicas moleculares y resultados preliminares. Tesis de licenciatura, Facultad de Ciencias, Universidad Nacional Autónoma de México, México, D.F. 66 pp. [ Links ]
Vázquez–Selem L. y Heine K. 2004. Late quaternary glaciation of Mexico. En: Ehlers y Gibbard Eds. Quaternary glaciations – extent and chronology, Part III, pp. 233–242, Elsevier, Amsterdam. [ Links ]
Vendramin G.G., Lelli L., Rossi P y Morgane M. 1996. A set of primers for the amplification of 20 chloroplast microsatellites in Pinaceae. Molecular Ecology 5: 595–598 [ Links ]
Vendramin G.G., Degen B., Petit R.J., Anzidei M., Madaghiele A. y Ziegenhagen B. 1999. High level of variation at Abies alba chloroplast microsatellite loci in Europe. Molecular Ecology 8: 1117–1126 [ Links ]














