Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Veterinaria México
versión impresa ISSN 0301-5092
Vet. Méx vol.43 no.2 Ciudad de México abr./jun. 2012
Artículos científicos
Potencial uso de espectroscopia de reflectancia en el infrarrojo cercano (NIRS) para identificación de charqui de bovino, llama y caballo
Potential use of near infrared reflectance spectroscopy (NIRS) for the identification of beef, llama and horse jerky
Willy Mamani–Linares*, Daniel Alomar**, Carmen Gallo***
* Becario MECESUP2 AUS 0601, Programa de Doctorado en Ciencias Veterinarias, Facultad de Ciencias Veterinarias, Universidad Austral de Chile, Valdivia, Chile.
** Instituto de Producción Animal, Facultad de Ciencias Agrarias, Universidad Austral de Chile, Valdivia.
*** Instituto de Ciencia Animal, Facultad de Ciencias Veterinarias, Universidad Austral de Chile, Valdivia.
Responsable de correspondencia:
Willy Mamani Linares,
teléfono: +56 63 221548,
correo electrónico: willymlmvzupea_2@hotmail.com
Recibido el 25 de febrero de 2011
aceptado el 18 de octubre de 2011
Abstract
Visible and near infrared reflectance spectroscopy (VIS/NIRS) was evaluated as a tool to discrimínate jerky from different species. Spectra were taken by reflectance in a NIRSystems 6500 monochromator and the software NIRS 3.0 and WinIsi II Version 1.02A were used. Twenty samples of jerky corresponding to beef, llama and horses, respectively, were ground, homogenized and analyzed spectrally. The regression equations (PLS) were developed testing different mathematical treatments. The results for jerky show that NIRS can successfully discriminate 100% of llama, 95% of horses and 80% of beef samples, probably as a consequence of differences in intramuscular fat, protein and water contents of the different species. Thus, NIRS is a fast, inexpensive and non–destructive method that can be used to discriminate jerky from these species.
Key words: discrimination of jerky, beef, llama, horses, NIRS.
Resumen
Se usó espectroscopía visible y de reflectancia en el infrarrojo cercano (VIS/NIRS) como herramienta para discriminar charqui de diferentes especies. Los espectros se tomaron por reflectancia en un equipo monocromador NIRSystems modelo 6500, con un software NIRS 3.0, y WinIsi II Versión 1.02 A. Se molieron, homogenizaron y analizaron espectralmente 20 muestras de charqui correspondientes a bovino, llama y caballo. Se desarrollaron ecuaciones de regresión (PLS) probando diferentes tratamientos matemáticos. Los resultados para charqui muestran que NIRS puede discriminar satisfactoriamente 100% de las muestras de llama, 95% de caballos y 80% de bovino, probablemente como consecuencia de diferencias en el contenido de grasa intramuscular, proteína y agua de las diferentes especies. Así, la técnica NIRS muestra ser un método rápido, económico y no destructivo que puede usarse para discriminar charqui de diferentes especies.
Palabras clave: discriminación de charqui, bovino, llama, caballo, NIRS.
Introducción
La determinación de la autenticidad de los alimentos y la detección de la adulteración son problemas importantes en la industria alimentaria, y cada vez están llamando más la atención.1,2 La principal preocupación en la autentificación de carne y productos de la carne es la sustitución de materias primas de alto valor con materiales más baratos, como lo son cortes menos costosos, vísceras, sangre, agua, huevo, gluten u otras proteínas de origen animal o vegetal.3,4 El molido o procesado de la carne elimina las características morfológicas de los músculos, lo que hace difícil identificar un tipo de músculo de otro. Por esta razón, la sustitución de carne de una especie por otra de menor calidad, es una forma económica de adulteración en la industria de la carne y constituye un acto fraudulento que podría tener repercusiones económicas y de salud.5
La intensificación de la agricultura y la urbanización en las últimas décadas ha creado una gran preocupación en muchos consumidores sobre la autenticidad y la inocuidad de la carne.4,6,7 Por lo tanto, los métodos de análisis se han centrado en la identificación de las especies de carne en crudo, cocida y productos elaborados.
La identificación de carne de diferentes especies, así como la diferenciación entre carne fresca y congelada, se han llevado a cabo por métodos inmunológicos8,9 procedimientos enzimáticos,10 técnicas de electroforesis,11 PCR y Real–time PCR.12,13 Estos métodos permiten detectar una amplia gama y bajos niveles de adulteración. En general, los métodos que usan DNA y RNA para la identificación de los productos son suficientemente confiables y su fiabilidad es más alta que la de otros métodos.14 Sin embargo, estas técnicas son destructivas, toman mucho tiempo, la metodología es tediosa y de alto costo, lo que las hace poco aptas para la aplicación en línea.15–17
Es necesario contar con métodos rápidos y confiables para la detección de la adulteración de la carne, a fin de aplicar los reglamentos y garantizar el control de calidad de los productos. Los métodos para lograrlo deberán ser específicos, sensibles, rápidos, económicos y capaces de analizar los productos procesados, cocidos, así como las carnes crudas, y proporcionar resultados cuantitativos.5 La espectroscopía de reflectancia en el infrarrojo cercano (NIRS) es una técnica versátil, rápida, de bajo costo y no tediosa, por no tener que preparar laboriosamente las muestras; éstas son las ventajas más importantes de esta tecnología, en comparación con los procedimientos antes mencionados.18
En el área de productos cárnicos la mayoría de los métodos de evaluación relacionados con NIRS han involucrado el desarrollo de calibraciones para la predicción cuantitativa de la composición química, física y sensorial de la carne.19–21
La NIRS se ha usado para determinar la idoneidad de productos como: sustitutos de la grasa en embutidos,22 identificación de carne procedente de diferentes categorías de animales,19–23 identificación de carne procedente de diferentes razas de animales,19–24 diferenciación de carne fresca y congelada,25–26 discriminación de carne de animales engordados con diferentes sistemas de alimentación.27–30
En la actualidad, existe información sobre cómo la NIRS puede también usarse para la detección y cuantificación de carne de diferentes especies, como la de canguro y bovino,31 cordero y ternera,32 vacuno, cerdo, pollo y cordero12,33–35 y detección de mezclas de carnes.36 Sin embargo, no se conocen trabajos sobre la identificación y autentificación de carnes no tradicionales, especialmente de carnes exóticas.37 Además de ser poca la información existente respecto a características cuantitativas y cualitativas de carne de especies como llamas y caballos, también lo son los estudios que se han realizado en la utilización de la técnica NIRS como herramienta para el análisis químico y como herramienta discriminante de calidad de carne y subproductos cárnicos como charqui de estas especies.
El presente estudio analiza la exactitud de la espectroscopia visible (VIS) y cercano infrarrojo (NIR) para identificar charqui de bovinos, llamas y caballos, a través del análisis discriminante de los espectros obtenidos a partir de muestras de charqui de estas especies.
Material y Métodos
Muestras
Se tomaron muestras de charqui de bovino (n = 20), llama (n = 20) y caballo (n = 20) comprada en diferentes supermercados y carnicerías. Para garantizar la diversidad de las muestras, se compraron no más de dos muestras de la misma marca y de la misma fecha de envasado. Las muestras de bovinos y caballos se adquirieron en el mercado local de Valdivia y Temuco, y las de llama se compraron en el mercado local de Arica. No se intentó identificar el origen anatómico, la edad o el tratamiento preliminar de almacenamiento de las muestras. Todas las muestras fueron almacenadas a temperatura ambiente en los días previos al análisis espectroscópico. Las muestras se prepararon eliminando los bordes y alguna evidencia de grasa intermuscular, y posteriormente se molieron en un procesador de alimentos,* se homogeneizaron completamente a mano, y se guardaron en bolsas de plástico selladas hasta su procesamiento.
Espectro
Cada muestra se homogeneizó a temperatura ambiente y se dividió en tres submuestras, con las cuales se llenaron cuidadosamente tres cubetas circulares con ventana de cuarzo de 35 mm de diámetro y 10 mm de espesor. Los espectros fueron tomados en el rango de 400 a 2500 nm a intervalos de 2 nm. En la lectura de los espectros se utilizó un equipo monocromador NIRSystems modelo 6500,** con detector de relectancia y un módulo de rotación de muestras. Se utilizó un software WinISI 1.0438 para manejar el equipo, procesar los datos ópticos y desarrollar las calibraciones. Los valores de absorbancia [log (1/R)] se almacenaron como promedio de las tres submuestras.
Análisis discriminante
Con el fin de identificar el charqui de bovino, llama y equino, se realizó un análisis discriminante mediante ecuaciones de regresión usando el método multivariado de los cuadrados mínimos parciales (PLS 2).38 Se separaron los archivos espectrales para cada una de las especies, y con el programa WinISI II versión 1.02 A, se generaron las ecuaciones discriminantes. En este método de análisis se establece una matriz de calibración con todas las muestras, asignándoles variables dummy con un valor 2 si el espectro pertenece a un grupo en particular (de acuerdo con el archivo), o un valor 1 si no pertenece a ese grupo. La calibración se realizó con los datos espectrales de las muestras utilizando los valores de referencia (uno o dos) asignados como variables dummy. La validación cruzada se utilizó para comprobar la precisión de la calibración y lograr un mínimo valor de error estándar en la validación.19 En el desarrollo de las ecuaciones se probaron diferentes tratamientos matemáticos, intentando las mejores combinaciones. De acuerdo con esta ecuación, la muestra se clasificó según una categoría específica (bovino, llama o caballo) si el valor de predicción de dummy fue de ± 0.5. Para visualizar la posición relativa de las muestras de diferentes especies se presentan gráficamente en tres planos por medio del PCAs.
Resultados
Caracterización espectral
En la Figura 1 se muestra el espectro promedio en las regiones del visible y el infrarrojo cercano (400–2500 nm) para el promedio de todas las muestras de charqui de bovino, llama y caballo. Se observan diferencias visibles entre los espectros de absorción de las especies en la región visible (pigmentos respiratorios y musculares) y en la región del cercano infrarrojo (contenido de grasa, proteína y agua).
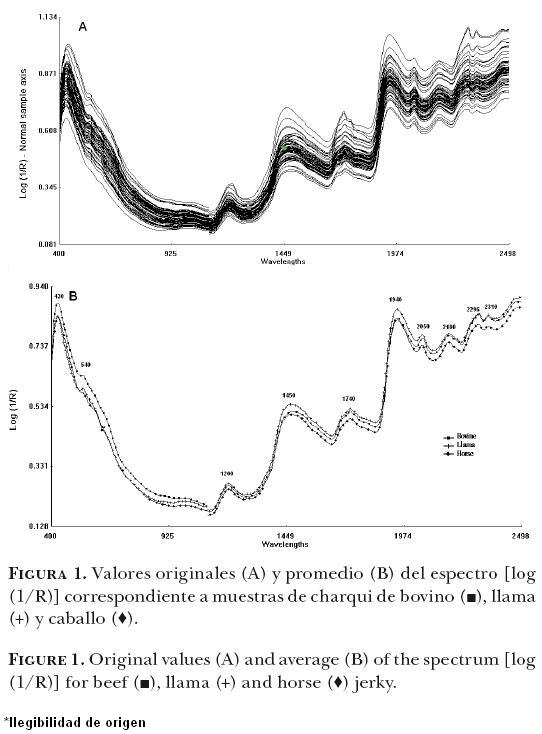
En la región visible (400–700nm), se observan bandas de absorción en 430 y 540 nm. Las muestras de bovino presentan bandas de absorción más altas que las de llama y caballo.
En la región NIRS se observaron bandas de absorción en 984–996 (segundo sobretono de OH), 14501460 (primer sobretono de OH) y 1936–1946 nm (tono combinado) relacionadas con contenido de agua, en 1014–1032 (segundo sobretono de NH2), 1470–1472, 1480–1482 (primer sobre tono de CON–HR), 2046–2066, 2176–2186, 2296–2306 nm (combinación de NH2, CONH2, CONHR) relacionados con contenido de proteína y en 1166–1176 (segundo sobre–tono de CH), 1190–1200 (segundo sobretono de CH3), 1720–1730 (primer sobre tono CH2), 2304–2308, 23442348 (combinación de tono de CH3, CH2, CH) nm relacionados con contenido de grasa. En general, en la región NIRS las muestras de llama y bovino tienen banda de absorción de mayor intensidad y variabilidad comparada con las muestras de caballo.
La derivada de segundo orden (2D) se calcula para acentuar los picos de absorbancia del promedio de las muestras (B, L y C) y reducir los efectos de los factores que causan los cambios de base (tamaño de las partículas, contenido de agua, etc.) y, para permitir la resolución de la superposición de picos se aplicó a los datos espectrales un tratamiento matemático de 2–5–4–1, haciendo hincapié en la información espectral útil. Es decir, primero se realizó una sustracción y después otra en los datos de absorbancia a una diferencia de 5 puntos de datos en todo el espectro, después de aplanar los segmentos de 5 puntos de los datos. Esta modificación se presenta en la Figura 2, cubriendo el rango NIR, desde 400 a 2500 nm. Los cambios de línea de base han sido casi eliminados, se ha resuelto cierto grado de solapamiento; y las diferencias entre especies en la absorción se limitan a algunas longitudes de onda significativas. Por lo tanto, la Figura 2 muestra diferencias importantes entre los grupos B, L y E en las bandas relacionadas con la absorción, por los enlaces OH, CH y NH, que podría ser útil para diferenciar grupos de productos cárnicos.
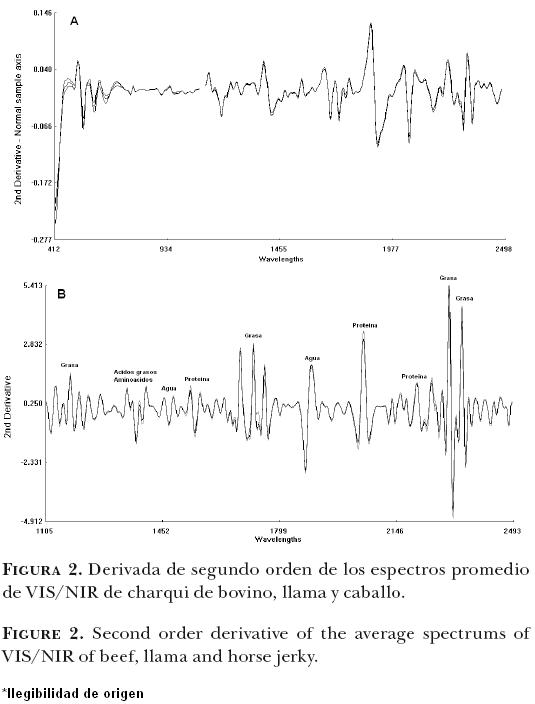
Aunque se observan diferencias entre los grupos espectrales promedio de los charquis de bovino, llama y caballo, se consideró necesario recurrir a un análisis discriminante que permitiera obtener una mayor información asociada con los tipos de muestras que se estaban utilizando.
Análisis discriminante
En el desarrollo de las calibraciones se aplicaron diferentes tratamientos matemáticos, generando sendas ecuaciones con diferentes resultados en la clasificación de las muestras espectrales. En el Cuadro 1 se muestra el resultado del mejor tratamiento matemático (2–12–8–1) para las diferentes especies, de acuerdo con el porcentaje de muestras espectrales clasificadas correctamente.
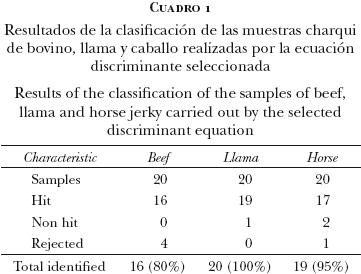
En dicho cuadro se muestran los porcentajes de reconocimiento en la optimización del modelo PLS. Se obtuvo una tasa de 100% de reconocimiento para el grupo de llama, y 95% para el grupo de caballo, lo que significa que las 20 muestras de cada grupo se clasificaron de manera adecuada (más de 95%). Por otro lado, de las 20 muestras del grupo bovino, 16 fueron clasificadas adecuadamente y 4 fueron rechazadas.
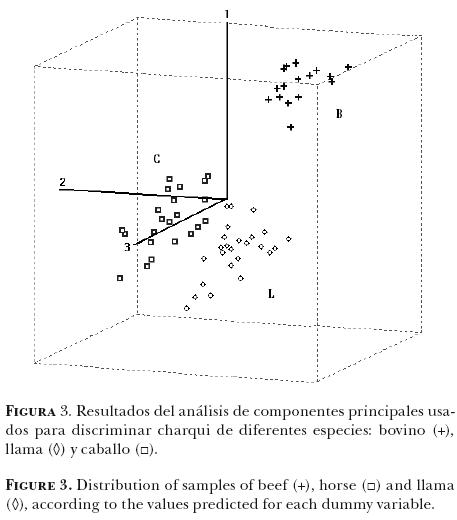
A continuación, la matriz de los datos de absorbancia (2D) se redujo a un sistema de coordenadas del eje, por lo que cada muestra se definió por la puntuación correspondiente a cada componente PLS. Cuando el conjunto de toda la muestra estuvo representada en un plano XYZ de acuerdo con las calificaciones de componente PLS 1, componente PLS 2 y componente PLS 3, fue posible observar tres grupos diferentes (Figura 3): el grupo C, relacionado con los caballos, el grupo B relacionado con los bovinos y el grupo L relacionado con las llamas. Por tanto, las muestras que se encuentran juntas presentan características espectrales similares.
Discusión
En la región visible, los espectros de absorción para hemoglobina se observan a 430 nm,39 mientras que mioglobina, oximioglobina, metamioglobina, y proteínas responsables del color del músculo a menudo tienen una fuerte absorción a 515–700 nm.19,34,36
En la región NIRS se muestran espectros de absorción de 980, de 1440–1460 y de 1932–1960 nm, relacionados con contenido de agua de las muestras y con tercero, segundo y primer sobretono de OH respectivamente.19,23,34,40 Alrededor de 1174, 1200–1204 nm, las bandas de absorción están relacionadas con el segundo sobretono de CH.23,34,36 En 1724, 1738–1740, 17601760 nm con primer sobretono de CH2,19,34 ambos relacionados con grasa y ácidos grasos y en 2310 nm con combinación de CH asociado con contenido de grasa, ácidos grasos saturados y no saturados.34 Varias bandas de absorción aparecen también para proteínas en 1010–1020 (segundo sobretono), 1510 (primer sobretono), 1980, 2050 y 2180 nm (combinación de tonos).19,40 Estas características espectrales concuerdan con los resultados aquí obtenidos.
Resultados de diferentes trabajos muestran que la NIRS permite discriminar correctamente carne de diferentes especies, razas y categorías de animales usando modelos matemáticos como regresión de cuadrados mínimos parciales (PLS), análisis de componentes principales (PCA), análisis multivariado (MANOVA) y SIMCA (Soft Independent Modeling Class Analogy). Prieto et al.23 clasificaron correctamente 100% de las muestras de carne de los grupos de bovinos (animales jóvenes y toros castrados), Cozzolino y Murray,34 usaron PCA y PLS, con el cual identificaron correctamente más del 80% de muestras de carne de diferentes especies (bovino, cerdo, pollos y ovinos), Ding y Xu31 identificaron correctamente 100% de las muestras de carne de diferentes especies (bovinos y kanguro), Alomar et al.18 identificaron correctamente 78% de las muestras de carne de diferentes razas de bovinos (Friesian y Hereford) usando PLS, Ripoll et al.24 identificaron 48.9% de las muestras de carne procedente de diferentes razas de caprinos, y Ortiz–Somovilla et al.36 clasificaron correctamente 92% de las salchichas de cerdo frescas de los curados, usando PLS y Mamani–Linares et al.37 identificaron correctamente las muestras de todas las especies (bovino, llama y caballo) por el espectro de absorción de su carne (reflectancia) o jugo de su carne (transflectancia).
Se concluye que la espectroscopia visible y del cercano infrarrojo (NIRS–VIS), combinada con el análisis de regresión PLS basado en variables "dummy" puede ser utilizada como una herramienta para discriminar charqui de bovino, llama y caballo, a través del análisis de los datos espectrales.
Agradecimientos
Los autores desean agradecen al proyecto MECESUP2 AUS 0601 del gobierno de Chile por proporcionar la beca WML y financiar este estudio. Asimismo, el agradecimiento es extensivo a la escuela de graduados de la Facultad de Ciencias Veterinarias por el apoyo y la asistencia técnica prestada por el personal del Laboratorio de Nutrición Animal de la Facultad de Ciencias Agrarias de la Universidad Austral de Chile para el desarrollo de este trabajo.
References
1. MONIN G. Recent methods for predicting quality of whole meat. Meat Sci 1998; 49: S231–S243. [ Links ]
2. CORDELLA CH, MOUSSA I, MARTEL AC, SBIRRAZZUOLI N, LIZZANI–CUVELIER L. Recent developments in food characterization and adulteration detection: Technique–oriented perspectives. J Agric Food Chem 2002; 50: 1751–1764. [ Links ]
3. HARGIN KD. Authenticity issues in meat and meat products. Meat Sci 1996; 43: 277. [ Links ]
4. AL JOWDER O, KEMSLEY K, WILSON RH. Detection of adulteration in cooked meat products by mid–infrared spectroscopy. J Agric Food Chem 2002; 50: 1325–1329. [ Links ]
5. MEZA–MARQUEZ OG, GALLARDO–VELAZQUEZ T, OSORIO–REVILLA G. Application of mid–infrared spectroscopy with multivariate analysis and soft independent modeling of class analogies (SIMCA) for the detection of adulterants in minced beef. Meat Sci 2010; 86: 511–519. [ Links ]
6. ALJOWDER O, DEFERNEZ M, KEMSLEY K, WILSON RH. Mid–infrared spectroscopy and chemometrics for the authentication of meat products. J Agric Food Chem 1999; 47: 3210–3218. [ Links ]
7. MURRAY I, AUCOTT L, PIKE IH. Use of discriminant analysis on visible and near infrared reflectance spectra to detect adulteration of fishmeal with meat and bone meal. J Near Infrared Spectrosc 2001; 9: 297–311. [ Links ]
8. PATTERSON RLS, JONES SJ. Review of current techniques for the verification of the species origin of meat. Analyst 1990; 115: 501–505. [ Links ]
9. SMITH CJ. Applications of immunoassay to the detection of food adulteration. An overview. In: MORGAN MRA, SMITH CJ, WILLIAMS PA, editors. Food safety and quality assurance–applications of immunoassay Systems. London: Elsevier Applied Science, 1991: 13–32. [ Links ]
10. SHARMA NK, SRIVATAVA VA, GILL JPS, JOSHI DV. Differentiation of meat from food animals by enzyme assay. Food Control 1994; 5: 219–221. [ Links ]
11. SIEBERTE S, BENEKE B, BENTLER W. Beef, pork, and sheep meat–detecting previous frozen treatment by isoelectric–focusing in polyamide acryl gel (PAGIF) during routine diagnosis. Fleischwirtschaft 1994; 74: 417–420. [ Links ]
12. FAJARDO V, GONZALEZ I, LOPEZ–CALLEJA I, MARTIN M, ROJAS T, GARCIA PE et al. PCR identification of meats from chamois (Rupicapra rupicapra), pyrenean ibex (Capra pyrenaica), and mouflon (Ovis ammon) targeting specific sequences from the mitochondrial D–loop region. Meat Sci 2007; 76: 644–652. [ Links ]
13. KESMEN Z, GULLUCE A, SAHIN F, YETIM H. Identification of meat species by TaqMan–based realtime PCR assay. Meat Sci 2009; 82: 444–449. [ Links ]
14. RAAMSDONK LWD, HOLST C, BAETEN V, BERBEN G, BOIX A, JONG J. New developments in the detection and identification of processed animal proteins in feeds. Anim Feed Sci Tech 2007; 133: 63–83. [ Links ]
15. AHMED FE. Detection of genetically modified organisms in foods, Trends Biotechnol 2002; 20: 215–223. [ Links ]
16. LIU Y, LYON BG, WINDHAM WR, LYON CE, SAVAGE EM. Prediction of physical, color, and sensory characteristics of broiler breasts by visible/near infrared reflectance spectroscopy. Poult Sci 2004; 83: 1467–1474. [ Links ]
17. PRIETO N, ROSS DW, NAVAJAS EA, NUTE GR, RICHARDSON RI, HYSLOP JJ et al. On–line application of visible and near infrared reflectance spectroscopy to predict chemical–physical and sensory characteristics of beef quality. Meat Sci 2009; 83: 96–103. [ Links ]
18. ALISHAHI A, FARAHMAND H, PRIETO N, COZZOLINO D. Identification of transgenic foods using NIR spectroscopy: A review. Spectrochim Acta A Mol Biomol Spectrosc 2010; 75: 1–7. [ Links ]
19. ALOMAR D, GALLO C, CASTAÑEDA M, FUCHSLOCHER R. Chemical and discriminant analysis of beef meat by near infrared reflectance spectroscopy (NIRS). Meat Sci 2003; 63: 441–450. [ Links ]
20. VILJOEN M, HOFFMANB LC, BRAND TS. Prediction of the chemical composition of mutton with near infrared reflectance spectroscopy. Small Rum Res 2007; 69: 88–94. [ Links ]
21. RIPOLL G, ALBERTÍ P, PANEA B, OLLETA JL, SAÑUDO C. Near–infrared reflectance spectroscopy for predicting chemical, instrumental. and sensory quality of beef. Meat Sci 2008; 80: 697–702. [ Links ]
22. ELLEKJAER MR, NAES T, ISAKSSON T, SOLHEIM R. Identification of sausages with fat–substitutes using near infrared spectroscopy. In: HILDRUM KI, ISAKSSON T, NAES T, TANDBERG A, editors. Near infrared spectroscopy: Bridging the gap between data analysis and NIR applications. London: Ellis Horwood, 1992: 320–326. [ Links ]
23. PRIETO N, ANDRES S, GIRALDEZ FJ, MANTECON AR, LAVIN P. Discrimination of adult steers (oxen) and young cattle ground meat samples by near infrared reflectance spectroscopy (NIRS). Meat Sci 2008; 79: 198–201. [ Links ]
24. RIPOLL G, ALCALDE MJ, HORCADA A, PANEA B. Suckling kid breed and slaughter weight discrimination using muscle colour and visible reflectance. Meat Sci 2011; 87: 151–156. [ Links ]
25. DOWNEY G, BEAUCHENE D. Discrimination between fresh and frozen then thawed beef M longissimus dorsi by combined visible and near infrared reflectance spectroscopy: A feasibility study. Meat Sci 1997; 45: 353–363. [ Links ]
26. THYOLDT K, ISAKSSON T. Differentiation of frozen and unfrozen beef using near infrared spectroscopy. J Sci Food Agric 1997; 73: 525–532. [ Links ]
27. OSORIO MT, ZUMALACARREGUI JM, PRIETO N, GIRALDEZ FJ, ANDRES S, MATEO J. Differentiation between carcasses from suckling lambs reared with ewe milk or milk replacers by fatty acid composition and near infrared reflectance spectroscopy of perirenal fat. Small Rum Res 2007; 72: 221–226. [ Links ]
28. PLA M, HERNANDEZ P, ARIÑO B, RAMIREZ JA, DIAZ I. Prediction of fatty acid content in rabbit meat and discrimination between conventional and organic production systems by NIRS methodology. Food Chem 2007; 100: 165–170. [ Links ]
29. DIAN PHM, ANDUEZA D, JESTIN M, PRADO IN, PRACHE S. Comparison of visible and near infrared reflectance spectroscopy to discriminate between pasture–fed and concentrate–fed lamb carcasses. Meat Sci 2008; 80: 1157–1164. [ Links ]
30. TEJERINA D, LOPEZ–PARRA MM, GARCIA–TORRES S. Potential used of near infrared reflectance spectroscopy to predict meat physicochemical composition of guinea fowl (Numida meleagris) reared under different production systems. Food Chem 2009; 113: 1290–1296. [ Links ]
31. DING HB, XU RJ. Differentiation of beef and kangaroo meat by visible and near infrared reflectance spectroscopy. J Food Sci 1999; 64: 814–817. [ Links ]
32. MCELHINNEY J, DOWNEY G. Chemometric processing of visible and near infrared reflectance spectra for species identification in selected raw homogenized meat. J Near Infrared Spectrosc 1999; 7: 145–154. [ Links ]
33. DOWNEY G. Discrimination strategies in food authentication: What is the correct question to ask? NIR News 2000; 11: 8–9. [ Links ]
34. COZZOLINO D, MURRAY I. Identification of animal meat muscles by visible and near infrared reflectance spectroscopy. Lebensm Wissenschaft Technol 2004; 37: 447–452. [ Links ]
35. GAYO J, SCOTT AH. Detection and Quantification of Species Authenticity and Adulteration in Crabmeat Using Visible and Near–Infrared Spectroscopy. J Agric Food Chem 2007; 55: 585–592. [ Links ]
36. ORTIZ–SOMOVILLA V, ESPAÑA–ESPAÑA F, DE PEDRO–SANZ EJ, GAITAN–JURADO AJ. Meat mixture detection in Iberian pork sausages. Meat Sci 2005; 71; 490–497. [ Links ]
37. MAMANI–LINARES LW, GALLOC, ALOMAR D. Identification of cattle, llama and horse meat by near infrared reflectance o transflectance spectroscopy. Meat Sci 2012, 90, 378–385 [ Links ]
38. ISI (INFRASOFT INTERNATIONAL, LLC). ISI windows near infrared software, WinISI II, version 1.02A, Foss NIRSystems, Silver Spring, MD, 1999: 236. [ Links ]
39. STRYER L. Biochemistry. 4th ed. New York: Stanford University, WH Freeman and Co., 1995. [ Links ]
40. CEN H, HE Y. Theory and application of near infrared reflectance spectroscopy in determination of food quality. Trends Food Sci Technol 2007; 18: 72–83. [ Links ]
*Moulinex 123, China.
** Foss NIRSystems, Slver Spring, MD, Estados Unidos de América.














