Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Veterinaria México
versión impresa ISSN 0301-5092
Vet. Méx vol.43 no.1 Ciudad de México ene./mar. 2012
Artículos científicos
Los productos alternativos de LYT1 de Trypanosoma cruzi tienen un patrón de localización diferencial
The alternative products of Trypanosoma cruzi LYT1 have different localization patterns
Gilberto Ballesteros-Rodea*,** Moisés Santillán*** Marisa Cruz-Aguilar* Claudia Márquez-Dueñas* César Israel Lugo-Caballero* Santiago Martínez-Calvillo† John Swindle‡ Rebeca Georgina Manning-Cela*
* Departamento de Biomedicina Molecular, Centro de Investigación y Estudios avanzados del IPN, Av. Instituto Politécnico Nacional 2508, col. San Pedro Zacatenco, Delegación Gustavo A. Madero, 07360, México, D.F. Correos electrónicos: gibaro90@hotmail.com, mari_cruz77@yahoo.com, caya_tobi@yahoo.com.mx, cesarl@mexico.com, rmanning@cinvestav.mx
** Facultad de Medicina Veterinaria y Zootecnia, Universidad Nacional Autónoma de México, Circuito Exterior, Ciudad Universitaria, Delegación Coyoacán, 04510, México, D.F. Correo electrónico: gibaro90@hotmail.com
*** Centro de Investigación y de Estudios Avanzados del IPN, Unidad Monterrey, Vía del Conocimiento 201, Parque de Investigación e Innovación Tecnológica, km 9.5 de la autopista nueva al aeropuerto, Apodaca, Nuevo León, 66600, México. Correo electrónico:msantillan@cinvestav.mx
† UBIMED, Facultad de Estudios Superiores Iztacala, Universidad Nacional Autónoma de México, Av. de los Barrios 1, col. Los Reyes Iztacala, Tlalnepantla, Estado de México, 54090, México. correo electrónico:scalv@campus.iztacala.unam.mx
‡ Comple Gen, Inc, Seattle, WA 98104, USA. Correo electronico: jtswindle@complegen.com
Responsable de correspondencia:
Rebeca G. Manning Cela,
Tel.: 57473322, Fax: 57473938,
correo electrónico: rmanning@cinvestav.mx
Recibido el 15 de octubre de 2010
Aceptado el 13 de mayo de 2011
Abstract
LYT1 is a molecule with lytic activity under acidic conditions that, as genetically demonstrated, participates in the infection and stage transition of T. cruzi. The differing functions of this protein result from alternative trans-splicing, resulting in proteins that contain either a secretion and nuclear sequence (LYT1s) or the nuclear sequence alone (LYT1n). To determine the localization of different LYT1 products, transgenic parasites expressing LYT1s or LYT1n fused to the enhanced green fluorescence sequence were analyzed. LYT1s-EGFP localized to the flagellum, vacuoles, membrane and regions of the nucleus and kinetoplast; LYT1n-EGFP localized to the nucleus and kinetoplast, and occasionally in vacuoles. These results show that even though different LYT1 products localize to the same sites, they are also found in different intracellular organelles and microenvironments, which could influence their multifunctional behavior.
Key words: flagellum, transfection, parasites, infectivity, alternative trans-splicing.
Resumen
LYT1 es una molécula con actividad lítica en condiciones ácidas, que según se demostró genéticamente, participa en el proceso de infección y transición de estadio de T. cruzi. Su diferente funcionalidad es resultado de la producción de dos proteínas, obtenidas por trans-empalme alternativo, que contienen una secuencia de secreción y una nuclear (LYT1s) o únicamente la secuencia nuclear (LYT1n). Para evaluar la localización de los diferentes productos de LYT1, se analizaron parásitos transgénicos que expresan la secuencia de LYT1s o LYT1n fusionada con la secuencia de la verde fluorescente. LYT1s-EGFP se localiza en flagelo, vacuolas, membrana y región del núcleo y cinetoplasto; mientras que, LYT1n-EGFP se localiza en la región del núcleo y cinetoplasto, y ocasionalmente en vesículas. Estos resultados muestran que aún cuando los distintos productos de LYT1 comparten algunos sitios de localización, también se encuentran en distintos organelos y microambientes intracelulares que podrían influir en su comportamiento multifuncional.
Palabras clave: flagelo, transfección, parásitos, infectividad, trans-empalme alternativo.
Introducción
La tripanosomiasis americana es una enfermedad parasitaria ocasionada por el protozoario flagelado Trypanosoma cruzi que afecta a varias especies de mamíferos y es considerada una zoonosis importante. El parásito presenta un ciclo de vida bifásico en el que se alternan cuatro estadios de desarrollo diferentes, dos en insectos hemípteros de la familia Reduviidae (epimastigote y tripomastigote metacíclico) y dos en el hospedero mamífero (amastigote y tripomastigote sanguíneo) en donde se lleva a cabo el ciclo intracelular del parásito.1
El ciclo intracelular es un proceso complejo que involucra diversos pasos consecutivos, que inicia con el reconocimiento y adhesión a la célula blanco por parte de moléculas específicas de la membrana del parásito. Después de su internalización mediante una vacuola parasitófora, el parásito es liberado al citoplasma de la célula hospedera en donde se diferencia a amastigote, replica y diferencia nuevamente a tripomastigote sanguíneo, el cual lisa a la célula infectada y propaga la infección.2
T. cruzi, al igual que muchos microorganismos intracelulares, requiere de proteínas líticas para completar su ciclo de vida, ya que depende de éstas para poder escapar de la vacuola fagocítica o del interior de la célula hospedera. Sin embargo, muy pocas han sido caracterizadas a la fecha.2
Hasta el momento se ha demostrado genéticamente la participación de dos moléculas en el proceso de infección de T. cruzi.3,4 Una de estas es LYT1, que es una molécula que presenta actividad lítica en condiciones ácidas y que participa en el proceso de infección y de transición de estadio del parásito. El gen LYT1 se clonó a partir de una biblioteca de expresión de T. cruzi de la cepa Y, aprovechando la reacción cruzada que presentó con el anticuerpo contra el componente C9 del complemento. La caracterización molecular indicó que LYT1 es un gen de copia única, con un marco de lectura abierto de 1,653 pb presente en dos alelos que codifica para una proteína de 552 aa, cuya secuencia no mostró homología con proteínas con función conocida.3 Por lo tanto, para dilucidar su función se obtuvieron y caracterizaron parásitos nulos, encontrando que la expresión de LYT1 no es esencial en epimastigotes y que estos parásitos nulos mostraban 3 fenotipos sobresalientes: una capacidad infectiva disminuida, un desarrollo acelerado in vitro y una actividad hemolítica disminuida.3
La diferente función de esta molécula se explica mediante experimentos de extensión 5' y RT-PCR que demostraron la presencia de tres transcritos obtenidos por trans-empalme alternativo. Dos codifican para la proteína completa que contiene una secuencia señal de secreción y una secuencia nuclear, relacionada con el proceso de infectividad. Y el tercero, que codifica para la proteína truncada (-28 aa) sin la secuencia señal de secreción y con la secuencia nuclear, relacionada con el proceso de diferenciación.5 También se demostró que el trans-empalme alternativo de LYT1 es regulado diferencialmente en los distintos estadio de desarrollo del parásito, en donde los transcritos que codifican para la proteína completa se expresan preferencialmente en tripomastigotes y amastigotes, mientras que el transcrito que codifica para la proteína truncada se expresa preferencialmente en epimastigotes.5 El análisis de una mutante dominante negativa de LYT1, mostró que presenta los tres fenotipos obtenidos en los mutantes nulos, sugiriendo que posiblemente LYT1 es parte de un complejo proteínico.6 Esta posibilidad se evaluó por ensayos de co-inmunoprecipitación y co-precipitación por afinidad a GST por centrifugación. El resultado fue que LYT1 interacciona con diversas proteínas cuyas funciones están relacionadas con el fenotipo de infección, de transición de estadio y de motilidad del parásito (resultados no publicados).
Estos antecedentes sugieren que la presencia de las diferentes secuencias de localización en LYT1, así como su posible interacción con distintas proteínas, pudiera ser resultado de una localización diferencial que expondría a la proteína a un microambiente diferente, lo que impactaría en su multifuncionalidad. Por lo tanto, en el presente trabajo se continuó con su caracterización, evaluando la localización de los distintos productos de LYT1.
Material y métodos
Parásitos
Los parásitos de T. cruzi cepa CL-Brener tipo silvestre (WT-Wild Type) y transgénicos se cultivaron en medio infusión de hígado y triptosa LIT,7 complementado con 10% de suero fetal bovino (SFB), 0.5% de penicilina (10,000 UI) / estreptomicina (10,000 µg) y 1% de hemina (5 mg/ml), a una temperatura de 28°C.
Construcción de pTREXn-EGFP, pTREXn-LYT1s-EGFP y pTREXn-LYT1n-EGFP
Las secuencias de LYT1 completa (LYT1s) y truncada (LYT1n), obtenidas por PCR fueron clonadas en el vector pEGFP-N1* en los sitios de restricción HindIII y KpnI. 1) LYT1s: contiene la secuencia completa de LYT1 alelo b (Genebank AF320626) a partir del primer ATG (+1) y una mutación en el segundo ATG (+85), obtenida en dos reacciones de PCR consecutivas empleando los oligos: LYT13 S y LYT17 AS (Cuadro 1) y como templado, ADN de PBS4.3 Kb-LYT1 alelo b (Genebank AF320626) y posteriormente los oligos LYT14 S y LYT17 AS y el producto de PCR anterior como templado. Como el plásmido pTREXn-LYT1s-EGFP contiene dos sitios 3' aceptores del trans-empalme, uno en la secuencia HXI del vector8 y otro en la posición +10 de la secuencia codificante de LYT1, fue necesario incorporar una mutación puntual conservativa en la posición +87 (C por G) de LYT1 para eliminar el sitio de inicio de traducción de la posición +85, y que se produzca únicamente la proteína completa de LYT1s-EGFP. 2) LYT1n: contiene la secuencia de LYT1 truncada que inicia a partir del segundo ATG de la posición +85 obtenida utilizando los oligos: LYT12 S y LYT17 AS (Cuadro 1) y ADN de PBS4.3 Kb-LYT1 alelo b (GeneBank AF320626) como templado. Los plásmidos obtenidos fueron digeridos con las enzimas HindIII y NotI para obtener los fragmentos LYT1s-EGFP y LYT1n-EGFP y sub-clonarlos en el vector pTREXn8 digerido con las mismas enzimas de restricción, para obtener los plásmidos pTREXn-LYT1s-EGFP y pTREXn-LYT1n-EGFP (Figura 1). Como testigo se sub-clonó la secuencia codificante para la proteína verde fluorescente aumentada (EGFP), obtenida de pEGFP-N1 en pTREXn en los sitios HindIII y NotI para obtener pTREXn-EGFP (Figura 1). Los plásmidos fueron secuenciados para verificar su correcta construcción.
Obtención de los parásitos transgénicos de LYT1s-EGFP y LYT1n-EGFP
Los epimastigotes WT (3 × 108) se lavaron con medio LIT frío no complementado, centrifugando a 800 g por 10 min. Los parásitos se re-suspendieron en 350 µl de medio LIT sin complementar y se colocaron en cubetas de electroporación (BTX 2 mm) a 4ºC. A la muestra se le adicionó 100 μg del ADN/40 μl de agua y se electroporó a 300 V y 12 ms con un equipo BTX ECM 830. Después de 5 min a temperatura ambiente, los epimastigotes se resuspendieron en 10 ml de medio LIT7 complementado y se incubaron a 28°C durante 24 h. Posteriormente los parásitos se seleccionaron con G418 a una concentración de 500 μg/ml. Una vez obtenidos los parásitos resistentes, estos se cultivaron nuevamente en ausencia del selector durante una semana. En estas condiciones los parásitos que contienen la secuencia transfectada de manera extracromosomal la eliminaron y murieron cuando se sometieron a una segunda ronda de selección. Los parásitos fueron clonados por dilución limitante sin presencia del selector. Durante la clonación y en los sub-cultivos posteriores, las células permanecieron fluorescentes lo que demostró la obtención de parásitos transfectados de manera estable.
Los plásmidos pTREXn-EGFP, pTREXn-LYT1s-EGFP y pTREXn-LYT1n-EGFP fueron utilizados para la obtención de los parásitos transgénicos EGFP, LYT1s-EGFP y LYT1n-EGFP, respectivamente.
Obtención de extractos proteínicos totales (EPT) de T. cruzi
Los epimastigotes de la cepa CL-Brener tipo silvestre (WT-wilde type) y los parásitos transfectados establemente (1 × 108) se cosecharon y fueron lavados 3 veces con 1 ml de PBS a pH 7.2 a 3,200 g a 4°C por 5 min. La pastilla obtenida fue resuspendida en 500 µl de amortiguador de lisis A (Tris-HCl 50 mM pH 7.8, Nonidet 40 1% (Np40), EDTA 5 mM, SDS 1%), 100 µl de la mezcla de inhibidores de proteasas** y 15 µl de ZnCl2 100 mM. La muestra fue incubada a 4°C durante 20 min y sonicada 3 veces por 1 min con intervalos de 10 seg a una amplitud de 40% (8 watts) en un sonicador.*** La muestra se centrifugó a 14,000 g por 10 minutos; al final se recuperó el sobrenadante. La concentración de proteínas fue cuantificada por el método de Lowry y su integridad se verificó en geles de acrilamida SDS-PAGE al 12%, teñidos con azul de Coomasie.
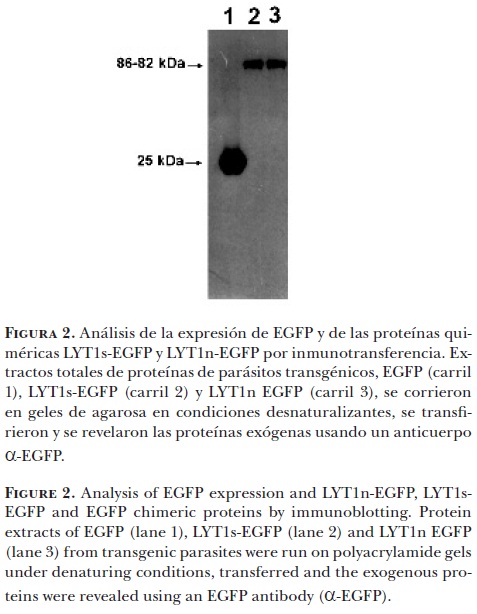
Análisis de la expresión de EGFP y de las proteínas quiméricas LYT1s-EGFP y LYT1n-EGFP en los parásitos transgénicos por inmunotransferencia
Los EPT (10 µg) se re-suspendieron en amortiguante Laemli 1x (62.5 mM Tris, pH 6.8, 10% glicerol, 3% SDS, 5% β-mercaptoetanol),9 fueron hervidos 5 min y cargados en geles SDS-PAGE al 4%. Las muestras se electrotransfirieron a membranas de nitrocelulosa a 100 V por una hora. Después de verificar la eficacia de transferencia con rojo Ponceau, las membranas se destiñeron con PBS 1x (NaCl 0.14 M, KCL 2.7 mM, Na2HPO4 10 mM, KH2PO4 1.8 mM) y se bloquearon con PBS1x-leche**** al 6% durante 1 h. Se incubó la membrana con el anticuerpo primario α-GFP IgG2a producido en ratón***** a una dilución 1:1000 en PBS1x-leche**** al 6% durante 1 h. Después de lavar la membrana con PBS 1x durante 10 min, PBS 1x-Tween-20 0.05% 10 min y PBS 1x por 10 min, se incubó 1 h con el anticuerpo secundario α-mouse IgG (H+L) conjugado con peroxidasa producido en cabra****** a una dilución 1:5000 en PBS1x-leche**** al 6%. La membrana se lavó nuevamente usando las mismas condiciones que para el anticuerpo primario y se reveló el ensayo por quimioluminiscencia utilizando el reactivo ECL (Amersham cat: RPN2106) como sustrato de la peroxidasa de acuerdo con las especificaciones del proveedor.
Determinación de la localización de las proteínas quiméricas por epifluorescencia
Los parásitos (4 × 106) se lavaron con PBS centrifugando a 800 g durante 6 min y se resuspendieron en 500 µl de paraformaldehido al 4%. Se tomaron 10 µl de muestra y se goteó en áreas delimitadas con lápiz graso en la superficie de laminillas siliconizadas.† La muestra se dejó secar en condiciones húmedas a temperatura ambiente durante 30 min, se lavó con PBS pre-enfriado en agitación y se permeabilizó con metanol absoluto durante 10 min. Después de lavar con PBS, se adicionaron 15 µl de DAPI (4',6-diamidino-2-phenylindole dihydrochloride)‡ a una dilución de 1:1000 durante 5 min, se lavó nuevamente y después de secar a temperatura ambiente, se montó la preparación en Airvol (16.8% de arvitol en una solución de NaCl 100 mM, KH2PO4 5 mM, Na2HPO4 10 mM). Los parásitos se observaron al microscopio de epifluorescencia******* equipado con un objetivo 60X/1.25******** y las imágenes se capturaron con una cámara********* adaptada al microscopio y se analizaron con el programa Image-Pro Plus 6.0. Para el análisis por fluorescencia confocal se utilizó un microscopio Leica,†† filtros de excitación: multifotónico 400 nm para DAPI y argón 488 nm para EGFP, y un objetivo 63X 1.4 con aceite de inmersión. Las imágenes fueron capturadas con el programa LAS AF (Leica Application Sui Advanced Fluorescence Lite) 1.7.0 build 1240.‡‡
Análisis del patrón de movimiento de los epimastigotes
Se capturaron imágenes y videos de las diferentes líneas de parásitos, utilizando la técnica de gota suspendida, con un microscopio de fluorescencia confocal Leica,† un filtro de excitación de 488 nm para EGFP y el programa LAS AF. Las trayectorias de los parásitos se analizaron a partir de los videos obtenidos utilizando el programa Image Pro Plus 6.0.
Resultados
Clonación y obtención de parásitos transfectados establemente con pTREXn-EGFP, pTREXn-LYT1s-EGFP y pTREXn-LYT1n-EGFP
A fin de expresar las diferentes variantes de LYT1 (completa y truncada) en los parásitos transgénicos, se clonaron sus secuencias codificantes en un vector de expresión para T. cruzi, como se describe en material y métodos. En cinetoplástidos no es posible utilizar promotores heterólogos, por lo que se utilizó el vector de expresión pTREXn que contiene un promotor ribosomal homólogo y la secuencia HXI que le confiere el sitio aceptador del trans-empalme al gen a expresar; de este modo se obtuvieron altos niveles de expresión. Las construcciones se usaron para transfectar de manera estable epimastigotes WT, y se caracterizó la expresión de LYT1s-EGFP, LYT1n-EGFP y EGFP (testigo), por inmunotransferencia, usando el anticuerpo α-GFP.
En la Figura 2 se muestra el reconocimiento del anticuerpo en bandas con los pesos moleculares esperados de 26 kDa para los parásitos EGFP (carril 1) y aproximadamente 82 kDa para LYT1ps-EGFP (carril 2) y 86 kDa para LYT1pn-EGFP (carril 3). No fue posible detectar la separación de las bandas de 82 kDa y 86 kDa debido a que no lo permite la resolución del sistema. Como era de esperarse, no hubo reconocimiento del anticuerpo en parásitos WT (dato no mostrado). Estos resultados indican que los parásitos LYT1s-EGFP, LYT1n-EGFP y EGFP expresan las proteínas exógenas y demuestran la generación exitosa de las líneas estables.
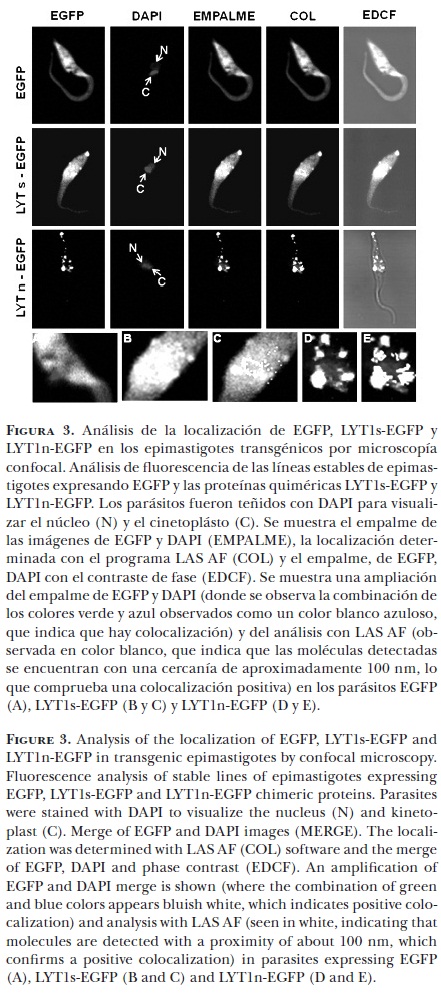
Las proteínas de LYT1, completa y truncada, presentan un patrón de localización diferente
Para determinar la localización de los diferentes productos de LYT1 en los epimastigotes transfectados, se evaluó la localización de EGFP como una medición indirecta de la presencia de LYT1s y LYT1n fusionados a EGFP, por microscopía confocal.
Como se observa en la Figura 3, el análisis de microscopía de fluorescencia de los epimastigotes EGFP mostró que la proteína verde fluorescente exógena se encuentra distribuida en todo el parásito. Como se esperaba, cuando se analizó el empalme de la fluorescencia de EGFP y de la tinción del núcleo y cinetoplasto con DAPI, no se observó colocalización. Por otro lado, mientras que los parásitos LYT1s-EGFP presentaron la fluorescencia en núcleo, cinetoplasto, flagelo, vesículas y membrana, los parásitos LYT1n-EGFP mostraron la fluorescencia preferentemente en núcleo y cinetoplasto, aunque en algunas ocasiones también se observó en vesículas. Estos resultados mostraron que ambos productos comparten la misma localización en núcleo, cinetoplasto y vesículas, lo que no es sorprendente, ya que comparten la secuencia de localización nuclear y se espera que sean transportados a su destino por medio de vesículas. Sin embargo, únicamente la forma completa de LYT1s-EGFP se localizó en membrana, ya que ésta es la única forma de la proteína que contiene una secuencia señal de secreción. Estos resultados mostraron que los diferentes productos de LYT1 presentan un patrón de localización diferencial.
La expresión exógena de LYT1s produce un defecto en la motilidad de los epimastigotes
Durante el cultivo de los parásitos LYT1s-EGFP y LYT1n-EGFP, se observaron cambios en su movimiento comparado con los parásitos testigo EGFP y WT. Por lo tanto, se analizó el patrón de movimiento y la trayectoria de los parásitos por video-microscopía de fluorescencia confocal, utilizando la técnica de gota suspendida, como se describe en la sección de métodos.
Los parásitos EGFP no mostraron ninguna diferencia en crecimiento y motilidad con respecto a los parásitos WT, lo que indicó que la expresión de la proteína fluorescente no tiene un efecto deletéreo y permitió utilizar a los parásitos EGFP como testigo en los subsiguientes experimentos. Como se muestra en la Figura 4, los parásitos EGFP mostraron un movimiento vigoroso y se desplazaron tanto distancias cortas como largas de manera direccional (panel A). Por el contrario, los parásitos que expresan a la proteína exógena LYT1s-EGFP presentaron una deficiencia dramática en su motilidad, desplazándose únicamente a distancias cortas (panel B). La observación y análisis de los videos mostró que esta deficiencia se caracterizó por un movimiento lento y descoordinado, dando una apariencia temblorosa a los epimastigotes. Además, presentaron incapacidad para coordinar su movimiento flagelar, girando de manera descontrolada sobre el mismo sitio, lo que resultó en una notoria dificultad en la motilidad direccional del parásito, aunque no perdieron su capacidad de moverse. En el caso de los parásitos LYT1n-EGFP, se observó una mezcla de comportamientos (panel C), ya que algunos parásitos se movían vigorosamente y recorrieron distancias grandes de manera comparable a lo observado en los epimastigotes testigo EGFP, mientras que otros mostraron un fenotipo similar a los parásitos LYT1s-EGFP.
Ya que la expresión exógena de EGFP no produjo ningún efecto deletéreo, estos resultados sugieren que la expresión exógena, al menos de la forma completa de LYT1 en los parásitos LYT1s-EGFP, es responsable de la falla observada en la motilidad. Será necesario realizar estudios futuros, tanto cuantitativos como de análisis vectorial y estadístico, para determinar si el defecto observado en los parásitos LYT1n-EGFP es significativamente diferente con respecto a los parásitos testigo, y determinar las alteraciones en la velocidad y ángulo de rotación que pudieran estarse dando en los parásitos LYT1s-EGFP y LYT1n-EGFP.
Discusión
Los tripanosomatidos, como T. cruzi, tienen la característica particular de producir transcritos policistrónicos que requieren de un procesamiento de trans-empalme 5' y poliadenilación 3' para su procesamiento y obtención de transcritos maduros. Esto resulta en que el principal nivel de regulación de su expresión sea pos-transcripcional y que sea posible la obtención de productos alternativos con diferente funcionalidad a partir de un mismo gen.10
Este comportamiento ha sido reportado para LYT1, que es la molécula en estudio en este trabajo de investigación, la cual es una proteína con actividad lítica en condiciones ácidas, que participa en el proceso de infección y transición de estadio de T. cruzi.3 Esta diferente funcionalidad es resultado de la producción de tres transcritos distintos obtenidos por trans-empalme alternativo. Dos de los cuales codifican para una proteína completa que contiene tanto una secuencia señal amino-terminal como una secuencia nuclear y que difieren únicamente en su secuencia no traducida 5'. Y otro que codifica para una proteína truncada con únicamente la secuencia nuclear.5 Estos transcritos son regulados en los distintos estadios de desarrollo de T. cruzi, expresándose mayoritariamente los que codifican para la proteína completa en tripomastigote y amastigote y para la proteína truncada en epimastigotes, lo que sugiere que posiblemente su expresión diferencial pudiera tener implicaciones en la biología de cada uno de los estadios de desarrollo del parásito.5
Ya que los diferentes transcritos de LYT1, codifican para proteínas con distintas secuencias de localización, en este trabajo se evaluó la posible localización diferencial de la proteína completa y truncada de LYT1. Para ello se obtuvieron y caracterizaron parcialmente parásitos transgénicos que expresan de manera exógena los diferentes productos de LYT1 fusionados a la proteína verde fluorescente mejorada (EGFP) y, de manera indirecta, su expresión (Figura 2) y localización (Figura 3) en epimastigotes de T. cruzi. El análisis de microscopía de fluorescencia de los epimastigotes que expresan a la proteína completa (LYT1s-EGFP) demostró su presencia en núcleo, cinetoplasto, vesículas y membrana, mientras que los que expresan a la proteína truncada (LYT1n-EGFP) mostraron la fluorescencia preferentemente en núcleo y cinetoplasto y ocasionalmente en vesículas. Estos resultados indican que los diferentes productos de LYT1 tienen patrones de localización distintos. Otro grupo de investigación obtuvo resultados similares, expresados de manera exógena en la proteína completa o truncada de los parásitos nulos de LYT1 y demostrados en la reconstitución diferencial de los distintos fenotipos.11 La proteína completa se localizó en membrana, fue secretada y en algunas ocasiones se observó en la región cineto-flagelar, y reconstituyó el fenotipo lítico y de infectividad. Por otro lado, la proteína truncada se localizó en la región adyacente cineto-flagelar y reconstituyó el fenotipo de diferenciación de estadio, demostrando que cada producto de LYT1 está implicado en funciones distintas.11 Los resultados del presente trabajo concuerdan con lo anterior, aunque además de la localización en membrana y cinetoplasto de LYT1s y en cinetoplasto de LYT1n, se observaron ambas formas de la proteína en núcleo y vesículas. Este resultado no es sorprendente, ya que tanto la proteína completa como truncada contienen una secuencia de localización nuclear y podrían ser llevadas a sus destinos finales por medio de vesículas de transporte.
Una posible explicación a estas diferencias es que la estrecha cercanía del núcleo y cinetoplasto en el epimastigote no permite distinguir claramente la localización en ambos organelos. La posibilidad de que la fusión de LYT1 con EGFP pueda, de alguna manera, deslocalizar a LYT1 es otra posibilidad. Sin embargo, se piensa que ello es poco probable, ya que se ha informado que este gen reportero por lo regular no modifica la localización de las proteínas a las cuales se fusiona.12,13 Además, la localización que se observó corresponde a la que se registró para la proteína endógena, siendo poco probable que se obtuviera una localización aberrante adicional a la localización de LYT1 endógena, aunque serán necesarios futuros experimentos para comprobar estas posibilidades.
Un fenotipo interesante e inesperado fue que la expresión exógena de LYT1-EGFP afectó el movimiento de los parásitos. La motilidad del epimastigote depende esencialmente del flagelo, el cual está conformado principalmente de microtúbulos.14-17 A pesar de que se conocen algunas moléculas involucradas en el movimiento del flagelo en células eucariotas, es muy poco lo que se sabe sobre el mecanismo que regula o coordina este movimiento flagelar.18 Los microtúbulos tienen un papel importante en el mantenimiento de la estructura de T. cruzi, participa en la separación de los cuerpos basales, en el crecimiento del nuevo flagelo, en la mitosis, en la citocinesis y por supuesto en la motilidad del parásito.14,16,19 También éstos conforman el saco flagelar que es por donde el parásito endocita y exocita sus proteínas, siendo un organelo importante en la regulación del tráfico vesicular que lleva las moléculas necesarias para el crecimiento y funcionamiento del flagelo.20-22
Los parásitos nulos de LYT13 y la mutante dominante negativa de LYT16 aparentemente no presentan defectos en la motilidad del parásito. Por lo tanto, estos resultados sugieren que la proteína LYT1 tiene posiblemente una participación indirecta en el movimiento flagelar de los epimastigotes, probablemente a través de su interacción con otras proteínas que participan en el proceso de motilidad del parásito.
Esta hipótesis se sustenta en el hecho de que nuestros resultados muestran que los parásitos transfectados LYT1-EGFP se comportan como mutantes dominantes negativas de alguna(s) proteína(s) que participa(n) en la motilidad del parásito y que se une a LYT1. Esta propuesta se basa en el hecho de que cuando se analizaron las moléculas que interactúan con LYT1 se encontró que se une a tubulina, cinesina y dineina, que son moléculas involucradas en el tráfico vesicular del parásito.23 Ya que este mecanismo es esencial para el transporte de las moléculas encargadas del crecimiento y buen funcionamiento flagelar, podría ser alterada la motilidad celular. Por lo tanto, si la expresión exógena de LYT1-EGFP y su interacción con tubulina, dineina y cinesina, están afectando la eficiencia del transporte intra-flagelar y por lo tanto el funcionamiento correcto de cualquiera de las moléculas necesarias para el movimiento del flagelo, sería posible obtener un fenotipo deletéreo como el observado en los parásitos transfectados.24,25
La expresión alternativa, cambio de localización y función diferencial de los productos de LYT1, podrían reflejar la optimización de los sistemas biológicos para cumplir con diversas funciones a partir de un único gen, como se ha reportado para otras moléculas en diferentes sistemas biológicos como tripanosomatidos,11,26,27 plantas11,28,29 y eucariontes superiores.30
Este estudio permite entender de los mecanismos moleculares involucrados en la regulación de la expresión e infección de T. cruzi y proporciona información que ayuda a entender los procesos biológicos del parásito, necesarios para el establecimiento de la enfermedad de Chagas.
Agradecimientos
Este trabajo fue realizado con el apoyo de los proyectos de CONACyT No. 42862 y 60152 otorgado a Rebeca G. Manning-Cela, y de Fogarty International Center of NIH No. R01 TW007255-01, CONACyT No. 47543 y PAPIIT IN203606-3 otorgado a Santiago Martínez-Calvillo. Durante el desarrollo de este trabajo, Gilberto Ballesteros-Rodea recibió la beca doctoral de CONACyT No. 116226. Se agradece a Iván Galván el apoyo técnico en el uso del microscopio confocal y a Víctor Rosales en el uso del FACS.
Referencias
1. BARRET MP, BURCHMORE RJ, STICH A, LAZZARI JO, FRASH AC, CAZZULO JJ et al. The trypanosomiases. Lancet 2003; 362: 1469-1480. [ Links ]
2. ESPINOZA GB, MANNING-CELA RG. An overview of mammalian cell infection immunology of parasitic diseases. Kerala, India 2007; 291-311. [ Links ]
3. MANNING-CELA R, CORTES A, GONZALEZ-REY E, VAN VOORHIS WC, SWINDLE J, GONZALEZ A. LYT1 protein is required for efficient in vitro infection by Trypanosoma cruzi. Infect Immun 2001; 69: 3916-3923. [ Links ]
4. CALER EV, VAENA DE AVALOS S, HAYNES PA, ANDREWS NW, BURLEIGH BA. Oligopeptidase B-dependent signaling mediates host cell invasion by Trypanosoma cruzi. EMBO J 1998; 17: 4975-4986. [ Links ]
5. MANNING-CELA R, GONZALEZ A, SWINDLE J. Alternative splicing of LYT1 transcripts in Trypanosoma cruzi. Infect Immun 2002; 70: 4726-4728. [ Links ]
6. MANNING-CELA R, SWINDLE J. Obtención y análisis de una mutante dominante negativa de LYT1 de Trypanosoma cruzi. Siicsalud. [Serie en línea: 2003 agosto 2003] [Citado: 2003 agosto 6]; Disponible en: www.siicsalud.com/des/des032/03805024.htm. [ Links ]
7. CAMARGO EP. Growth and differentiation in Trypanosoma cruzi. I. Origin of metacyclic trypanosomes in liquid media. Rev Instit Med Trop Sao Paulo 1964; 12: 93-100. [ Links ]
8. VAZQUEZ MP, LEVIN MJ. Functional analysis of the intergenic regions of TcP2beta gene loci allowed the construction of an improved Trypanosoma cruzi expression vector. Gene 1999; 239: 217-225. [ Links ]
9. LAEMMLI UK. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature 1970; 227: 680-685. [ Links ]
10. MARTINEZ-CALVILLO S, VIZUET-DE-RUEDA JC, FLORENCIO-MARTINEZ LE, MANNING-CELA RG, FIGUEROA-ANGULO EE. Gene expression in trypanosomatid parasites. J Biomed Biotechnol 2010; 2010: 525241. [ Links ]
11. BENABDELLAH K, GONZALEZ-REY E, GONZALEZ A. Alternative trans-splicing of the Trypanosoma cruzi LYT1 gene transcript results in compartmental and functional switch for the encoded protein. Mol Microbiol 2007; 65: 1559-1567. [ Links ]
12. TSIEN RY. The green fluorescent protein. Annu Rev Biochem 1998; 67: 509-544. [ Links ]
13. YANG F, MOSS LG, PHILLIPS GN, JR. The molecular structure of green fluorescent protein. Nat Biotechnol 1996; 14: 1246-1251. [ Links ]
14. GULL K. The cytoskeleton of trypanosomatid parasites. Annu Rev Microbiol 1999; 53: 629-655. [ Links ]
15. HUTCHINGS NR, DONELSON JE, HILL KL. Trypanin is a cytoskeletal linker protein and is required for cell motility in African trypanosomes. J Cell Biol 2002; 156: 867-877. [ Links ]
16. KOHL L, GULL K. Molecular architecture of the trypanosome cytoskeleton. Mol Biochem Parasitol 1998; 93: 1-9. [ Links ]
17. KOHL L, BASTIN P. The flagellum of trypanosomes. Int Rev Cytol 2005; 244: 227-285. [ Links ]
18. COSSON MP, COSSON J, ANDRE F, BILLARD R. cAMP/ATP relationship in the activation of trout sperm motility: their interaction in membrane-deprived models and in live spermatozoa. Cell Motil Cytoskeleton 1995; 31: 159-176. [ Links ]
19. ROBINSON DR, SHERWIN T, PLOUBIDOU A, BYARD EH, GULL K. Microtubule polarity and dynamics in the control of organelle positioning, segregation, and cytokinesis in the trypanosome cell cycle. J Cell Biol 1995; 128: 1163-1172. [ Links ]
20. BROOKS DR, TETLEY L, COOMBS GH, MOTTRAM JC. Processing and trafficking of cysteine proteases in Leishmania mexicana. J Cell Sci 2000; 113: 4035-4041. [ Links ]
21. KOZMINSKI KG, JOHNSON KA, FORSCHER P, ROSENBAUM JL. A motility in the eukaryotic flagellum unrelated to flagellar beating. Proc Natl Acad Sci USA 1993; 90: 5519-5523 [ Links ]
22. SOUTO-PADRON T, CAMPETELLA OE, CAZZULO JJ, DE SOUZA W. Cysteine proteinase in Trypanosoma cruzi: immunocytochemical localization and involvement in parasite-host cell interaction. J Cell Sci 1990; 96: 485-490. [ Links ]
23. LUGO-CABALLERO CI. Identificación de complejos proteicos de Trypanosoma cruzi asociados a LYT1p (tesis de maestría). Departamento de Biomedicina. México DF: Centro de Investigación y de Estudios Avanzados del Instituto Politécnico Nacional, 2008. [ Links ]
24. ROSENBAUM JL, WITMAN GB. Intraflagellar transport. Nat Rev Mol Cell Biol 2002; 3: 813-825. [ Links ]
25. KOHL L, ROBINSON D, BASTIN P. Novel roles for the flagellum in cell morphogenesis and cytokinesis of trypanosomes. EMBO J 2003; 22: 5336-5346. [ Links ]
26. CHAMOND N, GREGOIRE C, COATNOAN N, ROUGEOT C, FREITAS-JUNIOR LH, DA SILVEIRA JF et al. Biochemical characterization of proline racemases from the human protozoan parasite Trypanosoma cruzi and definition of putative protein signatures. J Biol Chem 2003; 278: 15484-15494. [ Links ]
27. ENGEL ML, HINES JC, RAY DS. The Crithidia fasciculata RNH1 gene encodes both nuclear and mitochondrial isoforms of RNase H. Nucleic Acids Res 2001; 29: 725-731. [ Links ]
28. WAMBOLDT Y, MOHAMMED S, ELOWSKY C, WITTGREN C, DE PAULA WB, MACKENZIE SA. Participation of leaky ribosome scanning in protein dual targeting by alternative translation initiation in higher plants. Plant Cell 2009; 21: 157-167. [ Links ]
29. SILVA-FILHO MC. One ticket for multiple destinations: dual targeting of proteins to distinct subcellular locations. Curr Opin Plant Biol 2003; 6: 589-595. [ Links ]
30. CHEN HH, LUCHE R, WEI B, TONKS NK. Characterization of two distinct dual specificity phosphatases encoded in alternative open reading frames of a single gene located on human chromosome 10q22.2. J Biol Chem 2004; 279: 41404-41413. [ Links ]
*Clontech, EUA.
**Complete®(Roche), Suiza.
***Sonics and Materials, Inc., EUA.
****Svelty (Nestlé S.A.), Suiza.
*****Santa Cruz Biotechnology # cat. GFP B-2 sc-9996, EUA.
******Zymed Laboratories # cat. 81-6520, EUA.
†Silane-pep™ slides sigma Diagnostics, EUA.
‡Molecular Probes™ Invitrogen detection technologies, EUA.
*******Olympus BX41, Japón.
********Oil Iris Ph3 UPlan FL N, EUA.
*********Evolution VF Fast Cooled Color Media Cibernetics, EUA.
††Microsystems TSC SP5, DM 1600, Mo, Alemania.
‡‡Leica Microsystems, Alemania.














