Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Veterinaria México
versión impresa ISSN 0301-5092
Vet. Méx vol.42 no.4 Ciudad de México oct./dic. 2011
Artículos científicos
Detección de Leptospira santarosai y L. kirschneri en bovinos: nuevos aislados con potencial impacto en producción bovina y salud pública
Detection of Leptospira santarosai and L. kirschneri in cattle: new isolates with potential impact in bovine production and public health
Carlos Alfredo Carmona–Gasca* Lemuel León Lara** Luz Olivia Castillo–Sánchez* José Manuel Ramírez–Ortega* Albert Ko*** Carlos Luna Palomera† Alejandro de la Peña–Moctezuma*
* Grupo de Investigación en Leptospira y Leptospirosis, Centro de Enseñanza Investigación y Extensión en Producción Animal en Altiplano, Facultad de Medicina Veterinaria y Zootecnia, Universidad Nacional Autónoma de México, 04510, México, DF.
** Centro de Investigación y Estudios Avanzados en Salud Animal, Facultad de Medicina Veterinaria y Zootecnia, Universidad Autónoma del Estado de México.
*** Weill Cornell Medical College and Oswaldo Cruz Foundation/Brazilian Ministry of Health, Rua Waldemar Falcao, 121–Brotas 40.295–001–Salvador, Brazil, BA.
† Facultad de Medicina Veterinaria y Zootecnia, Universidad Juárez Autónoma de Tabasco, Carretera Villahermosa–Teapa, km 25, La Huasteca 2a. sección, Villahermosa, Tabasco, México.
Responsable de correspondencia:
Dr. Alejandro de la Peña Moctezuma
Correo electrónico: delapema@unam.mx
Recibido el 7 de octubre de 2010.
Aceptado el 31 de mayo de 2011.
Abstract
Bovine leptospirosis causes high economic losses in cattle mainly due to reproductive failure, as well as representing public health risk. Since the last century, antibody titers against several Leptospira serovars have been detected by the microscopic agglutination test (MAT) in Mexico. With the exception of very few cases, the presence of serovars causing leptospirosis in cattle and other animal species has not been demonstrated by isolation in Mexico, and in such cases characterization had to be done abroad by complex and slow immunological approaches, by comparison with a number of reference strains. The present study was conducted to perform the molecular characterization of Leptospira isolates by multiple locus sequencing typing (MLST). A hundred and ninety seven sera and kidneys samples were collected immediately after slaughter, from grazing cattle coming from the south–eastern states of Mexico. Anti–Leptospira antibodies were detected by the MAT and kidneys were inoculated into EMJH and Fletcher's specific medium. A seropositivity of 60.4% (119 out of 197), with titers from 1:100 up to 1:3 200 was detected. Four isolates (2.03%), referred as CAL4, CAL6, CAL7 and MOCA45, were characterized by serology, ribotyping and MLST as L. kirschneri serovar Grippotyphosa; L. interrogans serovar Hardjo; L. santarosai serovar Mini and L. santarosai serovar Tarassovi, respectively. With the exception of serovar Hardjo, the three other isolates belong to serovars and species not previously isolated in Mexico. These findings make it necessary to evaluate the potential distribution of such serovars among cattle and their role on animal production and public health.
Key words: Leptospira, Tarassovi, mini, grippotyphosa, cattle, isolation, MLST.
Resumen
La leptospirosis bovina causa grandes pérdidas económicas a la ganadería por problemas reproductivos y también es un riesgo de salud pública. En México, desde el siglo pasado se ha registrado la presencia de anticuerpos contra serovariedades de Leptospira por la técnica de aglutinación microscópica (AM), en bovinos y otras especies animales. En muy pocos casos, la enfermedad fue demostrada por el aislamiento de Leptospira, y en tales casos, su caracterización se basó en métodos inmunológicos lentos que requirieron la comparación con cepas de referencia y que fueron realizados fuera de México. En el presente trabajo se realizó la caracterización molecular mediante la secuenciación de locus múltiples (MLST), de aislados de Leptospira obtenidos de riñones de bovinos recolectados en rastro, procedentes de las zonas Golfo y sur de México. Se obtuvieron muestras de suero y riñones de 197 bovinos para realizar la AM, y el cultivo en medios específicos EMJH y Fletcher. Se detectó una seropositividad del 60.4% (119 de 197), con títulos desde 1:100 hasta 1:3,200 y se obtuvieron cuatro aislados de Leptospira (2.03%), denominados CAL4, CAL6, CAL7 y MOCA45. Los aislados fueron caracterizados por serología, ribotipificación y MLST, como L. kirschneri serovariedad Grippotyphposa; L. interrogans serovariedad Hardjo; L. santarosai serovariedad Mini y L. santarosai serovariedad Tarassovi, respectivamente. A excepción de la serovariedad Hardjo, los aislados pertenecen a especies y serovariedades no aisladas anteriormente en la República Mexicana, esto sugiere la necesidad de evaluar su diseminación entre bovinos y su potencial efecto en la producción animal y en la salud pública.
Palabras clave: Leptospira, Tarassovi, mini, grippotyphosa, bovinos, aislamiento, MLST.
Introducción
La leptospirosis es una enfermedad zoonótica causada por especies y serovariedades patógenas del género Leptospira.1 En el ganado bovino, es una enfermedad que afecta a las unidades de producción tanto lecheras como de carne.2 La infección suele ser subclínica cuando es causada por serovariedades adaptadas al bovino como la serovariedad Hardjo, o bien por serovariedades no adaptadas, se pueden manifestar signos como fiebre, hematuria, hemoglobinuria, ictericia y muerte en los animales jóvenes; mientras que en hembras gestantes se pueden presentar abortos en cualquier estadio de la gestación, mortinatos, nacimiento de animales débiles, decremento en la producción láctea, agalactia transitoria, así como infertilidad.3 Resulta difícil estimar las pérdidas económicas causadas por la leptospirosis, en gran parte por la dificultad para realizar el diagnóstico preciso y también debido a la presencia de otras enfermedades bacterianas, virales y parasitarias que pueden cursar con los mismos signos.4
Los primeros estudios de esta enfermedad en bovinos fueron realizados por Mikhin y Azinov en Rusia en 1935;5 en Australia, en 1943 por Johnson,6 y en Estados Unidos, en 1944 por Jungherr;7 posteriormente se fueron identificando casos en prácticamente todo el mundo, donde las serovariedades más comúnmente registradas han sido Grippotyphosa, Pomona,8 Icterohaemorrhagiae,9 Hebdomadis,10 Sejroe11 y Hardjo.12 Esta última serovariedad ha sido reconocida como de distribución mundial y la más importante en el ganado bovino y en otros rumiantes.13 Además, en la serovariedad Hardjo se han reconocido dos subtipos clasificados en dos especies distintas: el subtipo Hardjobovis de L. borgpetersenii tiene importancia en Europa, América del Norte y Oceanía y el subtipo Hardjoprajitno de L. interrogans se encontró principalmente en el continente americano.14
En México, a la fecha sólo existe evidencia documentada de la presencia de L. interrogans serovariedad Hardjo (Hardjoprajitno), la cual fue inicialmente aislada a partir de un feto de bovino.15 Debido a que el aislamiento de Leptospira es un proceso lento que puede consumir de semanas hasta meses, el diagnóstico rutinario se basa en la prueba serológica de aglutinación microscópica (AM), donde se detectan anticuerpos contra cepas de referencia, representantes de diferentes serogrupos.16 En diferentes estudios serológicos en bovinos se han encontrado anticuerpos principalmente contra la serovariedad Hardjo, seguida de otras serovariedades, como Grippotyphosa, Pomona, Canicola, Icterohaemorrhagiae y Bratislava17,18 Lo anterior sólo refleja que los anticuerpos vacunales o de campo reaccionan contra las serovariedades utilizadas para el diagnóstico, pero existe un vacío de información sobre las serovariedades de Leptospira que son endémicas y que verdaderamente infectan a los bovinos en México, lo que requiere de su demostración mediante el aislamiento y caracterización. Se presume que otras especies y serovariedades de Leptospira pudieran estar causando enfermedad en los bovinos, pero que a la fecha no han sido identificadas y pudieran llegar a ser un problema económico en las unidades de producción, así como para la salud pública. Es conveniente conocer la distribución de las serovariedades que se encuentran en un área geográfica determinada, por lo que en el presente trabajo se realizó inicialmente un escrutinio serológico y bacteriológico, y posteriormente la caracterización inmunológica y molecular de los aislados para identificar otras especies y serovariedades de Leptospira presentes en las zonas del Golfo y sur de México.
Material y métodos
Recolección y procesado de muestras
Se recolectaron muestras de riñón y sangre de 197 bovinos sacrificados para el abasto. Los riñones libres de la cápsula se colocaron en una solución de cloruro de benzalconio al 0.025% de 5 a 10 minutos, para descontaminar su superficie. Posteriormente, se secaron con toallas de papel estériles y se les practicó de forma aséptica un corte longitudinal; se obtuvo entonces un macerado del interior del órgano con un rallador estéril, el cual se inoculó en los medios EMJH (Ellinghausen, McCullogh, Johnson y Harris) y Fletcher semisólidos, realizando tres diluciones décuples. Los cultivos se incubaron a 30°C durante tres meses y se revisaron semanalmente por microscopía de campo oscuro, para detectar formas bacterianas compatibles con leptospiras. Los sueros fueron clarificados por centrifugación a 903 g/5 min y se mantuvieron a –20°C hasta realizar la prueba de AM.
Detección de anticuerpos anti–Leptospira
La prueba se realizó usando una dilución inicial del suero de 1:50 en solución amortiguadora de fosfatos (SAF, pH 7.2 a 7.4) y tomando como punto de corte para reacciones positivas la dilución de 1:100.16 Se utilizaron 12 cepas de referencia de las serovariedades Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippotyphosa, Hardjo, Icterohaemorrhagiae, Mini, Pomona, Pyrogenes y Tarassovi cultivadas en medio EMJH líquido de 7 a 14 días, las cuales fueron donadas por el WHO/FAO/OIE–Collaborating Centre for Reference and Research on Leptospirosis, Australia and Western Pacific Region, de Brisbane, Queensland, Australia.
Identificación serológica de los aislados
Se produjeron sueros inmunes en conejos de 1.5 kg de peso a partir de los aislados cultivados en medio de Fletcher de 7 a 10 días a 30°C. Los cultivos se recolectaron del anillo de desarrollo conocido como zona de Dinger, se inactivaron a 56°C durante media hora y posteriormente, se inocularon por vía intravenosa en dosis de 1, 2, 4, y 6 ml, con una semana de diferencia entre cada inóculo.16 Los conejos que alcanzaron títulos séricos mínimos de 1:12,800 contra la cepa homóloga con la que fueron inoculados, fueron anestesiados con ketamina (60 mg/kg) y sangrados en blanco por punción cardiaca. Los sueros se obtuvieron por centrifugación a 903 g/5 min, distribuidos en volúmenes de 1.5 ml y congelados a –20°C hasta su análisis. La prueba de AM se realizó utilizando el protocolo previamente descrito por Myers,16 con los sueros inmunes y las cepas de referencia.
Extracción de ADN y reacción en cadena de la polimerasa
Los aislados adaptados a condiciones de laboratorio fueron cultivados en 100 ml de medio líquido EMJH y centrifugados a 10,000 g/30 min. La extracción de ADN se realizó por el método de tiocianato de guanidina y cloroformo–alcohol isoamílico descrito por Boom et al.19 Con la finalidad de confirmar el origen de los aislados, se realizó la prueba de reacción en cadena de la polimerasa (PCR), con los iniciadores G1: G2 que amplifican ADN de serovariedades de diferentes especies patógenas de Leptospira, y con B64–I: B64–II, iniciadores que amplifican serovariedades de L. kirschneri.20 También se utilizaron los iniciadores MILL1801 y MILL1802, diseñados especialmente para amplificar un fragmento de ADN de 1,289 pb, de una región del loci rfb codificante de enzimas para la biosíntesis del lipopolisacárido (LPS), de serovariedades no patógenas.21 Los amplicones resultantes de la PCR fueron visualizados en geles de agarosa teñidos con bromuro de etidio, para obtener un registro fotográfico mediante un digitalizador de imágenes (Gel Logic 200, Kodak®).**
Identificación genética por secuenciación del gen rrs2 (16S ribosomal)
El gen 16S rRNA de los aislados fue secuenciado en los laboratorios del instituto Fio–Cruz, Salvador, Brasil; las secuencias fueron remitidas electrónicamente a nuestro laboratorio donde fueron editadas, ordenadas y analizadas con el programa Sequencher®,***versión 4.6. Se identificaron las secuencias relacionadas en las bases de datos utilizando el Basic Local Alignment Search Tool (BLASTn), disponible en la red.22 El alineamiento de las secuencias y el árbol filogenético resultante fueron construidos utilizando el método de Neighbor–Joining con 2,000 réplicas con el modelo de Kimura de 2–parámetros, en el programa Molecular Evolutinary Genetics Analysis (MEGA), versión 4.0.23,24
Por otro lado, se utilizó el primer esquema de MLST, descrito por Ahmed et al.,25 diseñado para la tipificación de cepas de 6 especies patógenas de Leptospira, el cual está fundamentado en el análisis de las secuencias de alelos de los genes adk, secY, icdA, lipL32,lipL41 y rrs2. Las PCR se realizaron de acuerdo con las siguientes condiciones: 200 μM de dNTP's, 0.2 U de Taq Polimerasa, 5 μl de amortiguador 10X (Roche®),**** 10 pmol de iniciadores (sintetizados en el Instituto de Biotecnología, UNAM) y 30 a 60 ng de ADN, en un volumen total de reacción de 50 μl. Para el proceso se utilizó un termociclador (Perkin Elmer 2400 PCR System®,,***** con los parámetros de: desnaturalización inicial a 95°C/5 min, seguida por 35 ciclos de desnaturalización (94°C/30 seg), alineación (58°C/45 seg) y extensión (72°C/45 seg), y una extensión final de 7 min/72°C. Para visualizar los productos se realizaron electroforesis en geles de agarosa al 1% teñidos con bromuro de etidio. La purificación de los amplicones antes de su secuenciación, se realizó utilizando columnas para purificar productos de PCR Montagé®,† Las secuencias remitidas electrónicamente fueron editadas y analizadas con el programa Sequencer®, versión 4.6. Para designar el número de alelos encontrados por gen, las secuencias obtenidas de cada aislado fueron comparadas con una base de datos proporcionada por Niyaz Ahmed et al.,25 con el programa DAMBE (Data Analysis in Molecular Biology and Evolution).26 Para realizar el análisis filogenético por alelos se utilizó el programa START2 (Sequence Type Analysis and Recombinational Tests, Versión 2). Finalmente, el análisis filogenético se corroboró con las secuencias de cada gen concatenadas en una sola, con el programa MEGA (Molecular Evolutionary Genetics Analysis) versión 4.2.24
Resultados
Aglutinación microscópica de los sueros de bovino
Se detectó una frecuencia de sueros positivos de 60.40% en los bovinos muestreados (119 de 197) mediante AM, donde las serovariedades de Leptospira detectadas con mayor frecuencia fueron: Hardjo 39.90% (61/197), Tarassovi 7.10% (14/197), Pomona 3.04% (6/197), Grippotyphosa 1.52% (3/197), Icterohaemorrhagiae 1.52% (2/197), Bratislava 1.52% (2/197) y Canicola 0.50% (1/197).
Aislamiento e identificación serológica de los aislados
Se obtuvieron cuatro aislados (2.3%), a partir de los riñones de bovino, entre las semanas 12 y 13 de incubación en medio EMJH. Los aislados se denominaron CAL4, CAL6, CAL7 y MOCA45. Al realizar la identificación con el suero inmune homólogo, el suero del aislado CAL4 reaccionó con un título de 1:25,600 contra la serovariedad Grippotyphosa cepa Moskva V; el aislado CAL6 reaccionó con un título de 1:25,600 contra la serovariedad Hardjo subtipo Hardjoprajitno; el aislado CAL7 reaccionó con un título de 1:12,800 contra la serovariedad Mini cepa Sari y el aislado MOCA45 reaccionó con un título de 1:12,800 contra la serovariedad Tarassovi cepa Perepelitsin (Cuadro 1).
Identificación molecular de los aislados
La PCR mostró a los cuatro aislados como leptospiras patógenas. CAL4 amplificó el fragmento de ADN de 565 pb con los iniciadores B64–I : B64–II, mientras que los aislados CAL6, CAL7 y MOCA45 amplificaron el fragmento de ADN de 283 pb con los iniciadores G–1 : G–2. Ninguno de los aislados amplificó fragmentos de ADN con los iniciadores MILL1801 y MILL1802, específicos para identificar leptospiras saprófitas (Figura 1).
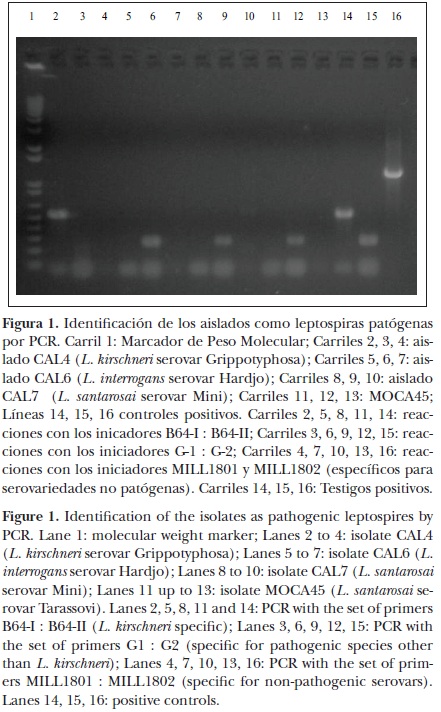
De acuerdo con el análisis filogenético del gen rrs2, los aislados fueron identificados como tres diferentes especies de Leptospira. Comparando las secuencias con mayor similitud con el programa Megablast (www.megablast.ncbi.org), se encontró que CAL4 tiene 99% de identidad con otras secuencias pertenecientes a L. kirschneri; CAL6 tiene 99% de identidad con cepas de L interrogans; CAL7 y MOCA45 tienen 99% de identidad con cepas de L. santarosai. La relación filogenética entre éstas se muestra en la Figura 2.
La identificación por MLST se realizó mediante la amplificación por PCR, de fragmentos internos de los seis genes, y comparando con el esquema previamente propuesto todos los amplificados fueron secuenciados en ambas direcciones. CAL6 mostró el tipo de secuencia 254 que lo identifica como L. interrogans serovariedad Hardjo subtipo Hardjoprajitno. Por el contrario, las cepas CAL4, CAL7 y MOCA45 tienen tipos de secuencias no registradas anteriormente en ninguna parte del mundo (Cuadro 2).
Discusión
La presentación clínica de leptospirosis en bovinos está influida por la serovariedad y la especie infectante. Existen serovariedades adaptadas al bovino, que causan enfermedad esporádicamente, serovariedades no adaptadas que comúnmente causan brotes de presentación aguda, y otras serovariedades de presentación rara en los bovinos en las cuales se desconoce el curso de la enfermedad.27 Por lo tanto, el control de la leptospirosis depende, en parte, de la detección serológica de las serovariedades prevalentes en la región y mejor aún, de su detección mediante el aislamiento. En el presente estudio se realiza la primera descripción de la caracterización molecular de cuatro aislados de Leptospira de las especies L. interrogans, L. kirschneri y L. santarosai (dos), aisladas a partir de bovinos en México. La clasificación genética de estos aislados se llevó a cabo mediante dos métodos, por secuenciación del gen rrs2 (16S ribosomal)28 y por la tipificación mediante la secuenciación de locus múltiples (MLST).25
Es sabido que Hardjo es una serovariedad adaptada al bovino, por lo que es posible que infecte a esta especie animal sin producir la enfermedad, sólo en ciertas condiciones de estrés causa enfermedad reproductiva en forma esporádica.29 La serovariedad Hardjo incluye dos subtipos que se encuentran en dos especies diferentes, pero que son serológicamente indistinguibles:30 L. borgpetersenii serovariedad Hardjo (subtipo Hardjobovis), ha sido aislada y registrada como la serovariedad más común entre las poblaciones de ganado bovino en todo el mundo. Por esta razón y a pesar de no haber sido aún aislada en México, se intuye que está presente en el país puesto que se ha identificado su presencia en países exportadores de ganado a México (http://www.siap.gob.mx).31–33 En el presente trabajo, se ha confirmado por medio del aislamiento la presencia de L. interrogans, serovariedad Hardjo (subtipo Hardjoprajitno) (aislado CAL6), la cual es común en el continente americano27 y ha sido previamente aislada en México a partir de un feto bovino.15
Estudios del locus rfb para la biosíntesis del LPS sugieren que el subtipo Hardjoprajitno evolucionó a partir de un ancestro de la serovariedad Copenhageni, el cual a lo largo del tiempo adquirió genes del locus rfb a partir del subtipo Hardjobovis; esto le permitió a dicho ancestro de alguna manera adaptarse al huésped bovino y dar origen así al subtipo Hardjoprajitno.34
Algunas otras serovariedades como Pomona, Grippotyphosa, Bratislava e Icterohaemorrhagiae, son serovariedades no adaptadas al bovino, que causan leptospirosis en su forma aguda.29 En México se ha registrado ampliamente la presencia de anticuerpos contra estas serovariedades, sin embargo, no existen a la fecha datos de su aislamiento a partir de muestras clínicas de bovinos.17,18 Por ello, es necesario identificar las serovariedades, adaptadas o no adaptadas al bovino, que pudieran asociarse con enfermedad aguda. En el presente trabajo, de acuerdo con los análisis serológico (AM) y moleculares (secuenciación de rrs2 y MLST), se identificó al aislado CAL4 como L. kirschneri serovariedad Grippotyphosa. La tipificación por MLST de CAL4 mostró un nuevo tipo de secuencia: ST259 (Cuadro 2), no registrado anteriormente en ninguna otra serovariedad de Leptospira y que filogenéticamente evolucionó a partir de la serovariedad Grippotyphosa cepa Moskva V.
En diferentes países del mundo se ha registrado la infección en bovinos con serovariedades atípicas, que se presentan en forma endémica en una región, pero no se conoce el impacto de dichas serovariedades en la producción y en la salud de los animales y del hombre, así como la probable fuente de infección, tal es el caso de las serovariedades Kennewicki en Chile,35 Zanoni en Australia,36 Fugis y Zimbabwe en Zimbabwe,37 y Guaricura en Brasil.38 En el presente trabajo se informa sobre el aislamiento de serovariedades que no son registradas comúnmente en bovinos, como son los casos de los aislados CAL7 de la serovariedad Mini (ST260) y MOCA45 de la serovariedad Tarassovi (ST261), ambos aislados de la especie L. santarosai. La serovariedad Mini ha sido registrada en las especies L. interrogans39 y L. borgpetersenii,40 pero Mini de L. santarosai no se había encontrado en bovinos; por otro lado, la serovariedad Tarassovi de la misma especie, L. santarosai, ha sido aislada previamente de bovinos en Centro y Sudamérica, pero no en México, hasta este informe.
De acuerdo con los resultados de este trabajo, no existe una relación absoluta entre la serología y el aislamiento; de los cuatro bovinos de los que se obtuvieron los aislados, sólo en el que se aisló CAL6 se observaron títulos de anticuerpos de 1:200 hacia la misma serovariedad, mientras que los otros tres bovinos (CAL4, CAL7, MOCA45) no mostraron títulos de anticuerpos contra ninguna serovariedad. En cuanto a la frecuencia de positivos por serología, la serovariedad Hardjo fue la más frecuentemente detectada en el presente estudio, pero se obtuvieron aislados de las serovariedades Tarassovi y Mini que no mostraron tener importancia por serología utilizando cepas de referencia. Es generalmente conocido que la sensibilidad de la prueba de AM aumenta cuando se incluyen las cepas aisladas (autóctonas) de la región geográfica.16 Al utilizar los aislados autóctonos aumenta la frecuencia de positivos por AM.17,41 Por lo anterior y con base en los resultados mostrados en este estudio, se sugiere incluir los aislados CAL4, CAL6, CAL7 y MOCA45 en posteriores estudios serológicos de bovinos, además de realizar la búsqueda de éstas y otras serovariedades que prevalecen en la región, mediante el aislamiento del agente etiológico, y no sólo con base en los resultados de serología.
Agradecimientos
Este trabajo fue financiado parcialmente por los proyectos UNAM/PAPIIT IN222806 e IN221409; SEP–CONACyT 83123 y el MACROPROYECTO No. 7 de la Universidad Nacional Autónoma de México. Se agradece al Consejo Nacional de Ciencia y Tecnología (CONACyT) la beca No. 165530 para estudios de posgrado de Carlos Alfredo Carmona Gasca.
Los autores agradecen profundamente la ayuda del Dr. Niyaz Ahmed de la Universidad de Hyderabad, India; así como del Dr. Rudy Hartskeerl, del Royal Tropical Institute, Países Bajos, por permitirnos el acceso a la base de datos que hizo posible el análisis de los aislados de Leptospira, mediante el esquema MLST por ellos propuesto.
References
1. ADLER B, DE LA PEÑA–MOCTEZUMA A. Leptospira and leptospirosis. Vet Microbiol 2010; 140: 287–296. [ Links ]
2. AMATREDJO A, CAMPBELL RSF. Bovine leptospirosis. Vet Bull 1975; 43: 875–891. [ Links ]
3. ELLIS WA. Leptospirosis as a cause of reproductive failure. Vet Clin North Am Food Anim Pract 1994; 10: 463–478. [ Links ]
4. ELLIS WA, LITTLE TWA. The present state of leptospirosis diagnosis and control. In: ELLIS WA, LITTLE TWA, editors. Current topics in veterinary medicine and animal science. Dordrecht Netherlands: Martinus Nijhoff Publishers, 1986: 13–21. [ Links ]
5. MIKHIN NA, AZINOV SA. Spirochaetal jaundice of cattle in North Caucasus. Sovyet Vet 1935; 10: 23–27 (artículo original inexistente, un resumen en: Vet Bull 1937; 7:419). [ Links ]
6. JOHNSON D. Epidemiology of Weil's disease. Br Med J 1943; 2: 659. [ Links ]
7. JUNGHERR E. Bovine leptospirosis. J Am Vet Med Assn 1944; 105: 276–281. [ Links ]
8. BRYAN NS, BOLEY LE. Sudies on leptospirosis in domestic animals. IV. Survival of Leptospira Pomona in bovine semen extender. Mich State Univ Vet 1955; 16: 27–29. [ Links ]
9. ELLIS WA, NEILL SD, O'BRIEN JJ, HANNA J, BRYSON DG. The isolation of a strain of Leptospira serogroup Icterohaemorrhagiae from an aborted bovine foetus. Br Vet J 1977; 133: 108–109. [ Links ]
10. THIERMANN A. Experimental leptospiral infections in pregnant cattle with organisms of the Hebdomadis serogroup. Am J Vet Res 1982; 43: 780–784. [ Links ]
11. MICHNA SW, CAMPBELL RSF. The isolation of Leptospira Sejroe from the kidneys of aborting cattle. Vet Rec 1969; 84: 83–86. [ Links ]
12. ROTH EE, GALTON MM. Isolation and identification of Leptospira Hardjo from cattle in Louisiana. Am J Vet Res 1960; 21: 422–427. [ Links ]
13. AYANEGUI–ALCERRECA MA, WILSON P, MACKINTOSH CG, COLLINS–EMERSON JM, HEUER C, MIDWINTER AC et al. Leptospirosis in farmed deer in New Zealand: A review. N Z Vet J 2007; 55: 102–108. [ Links ]
14. ZUERNER RL, ELLIS WA, BOLIN CA, MONTGOMERY JM. Restriction fragment length polymorphisms distinguish Leptospiraborgpetersenii serovar Hardjo type Hardjo–bovis isolates from different geographical locations. J Clin Microbiol 1993; 31: 578–583. [ Links ]
15. SALOMÓN SA, ZERMEÑO G, MOLES CLP. Aislamiento de leptospiras a partir de animales de granja. Informe de Servicio Social. México DF: Universidad Autónoma Metropolitana, 1988. [ Links ]
16. MYERS M. Manual para el diagnóstico de laboratorio de la leptospirosis OPS. Centro Panamericano de Zoonosis. Buenos Aires: Organización Panamericana de la Salud, 1985. Nota técnica No 30. [ Links ]
17. LUNA AMA, MOLES CLP, GAVALDÓN RD, VÁSQUEZ NC, SALAZAR GF. Estudio retrospectivo de seroprevalencia de leptospirosis bovina en México considerando las regiones ecológicas. Rev Cubana Med Trop 2005; 57: 28–31. [ Links ]
18. CHÁVEZ TR. Análisis de los resultados obtenidos en el diagnóstico serológico de leptospirosis en animales, de 1989 a 2004 en el departamento de Microbiología e Inmunología, Facultad de Medicina Veterinaria y Zootecnia. UNAM (tesis de licenciatura). México DF: Universidad Nacional Autónoma de México, 2006. [ Links ]
19. BOOM R, SOL CJ, SALIMANS MM, JANSEN CL, WERTHEIM–VAN DILLEN PM, VAN DER NOORDAA J. Rapid and simple method for purification of nucleic acids. J Clin Microbiol 1990; 28: 495–503. [ Links ]
20. GRAVEKAMP C, VAN DE KEMP H, FRANZEN M, CARRINGTON D, SCHOONE GJ, VAN EYS GJ et al. Detection of seven species of pathogenic leptospires by PCR using two sets of primers. J Gen Microbiol 1993; 139: 1691–1700. [ Links ]
21. MENA BR. Análisis in silico de los genes gspDL y gspEL, del sistema de secreción tipo II de Leptospira biflexa serovariedad Patoc (tesis de maestría). México DF: Universidad Nacional Autónoma de México, 2006. [ Links ]
22. ALTSCHUL SF, GISH W, MILLER W, MYERS EW, LIPMAN DJ. Basic local alignment search tool. J Mol Biol 1990; 215:403–410. [ Links ]
23. SATOU N, NEI M. The neighbor joining method: a new method for reconstructing phylogenetic trees. Mol Biol Evol 1987; 4:406–425. [ Links ]
24. TAMURA K, DUDLEY J, NEI M, KUMAR S. MEGA 4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0. Mol Biol Evol 2007; 24: 1596–1599. [ Links ]
25. AHMED N, DEVI SM, VALVERDE M DE L, VIJAYACHARI P, MACHANG'U RS, ELLIS WA et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Ann Clin Microbiol Antimicrob 2006; 23: 5–28. [ Links ]
26. XIA X, XIE Z. DAMBE: Data analysis in molecular biology and evolution. J Hered 2001; 92: 371–373. [ Links ]
27. HANSON LE. Bovine leptospirosis. J Dairy Sci. 1976; 59:1166–70 [ Links ]
28. PEROLAT P, MERIEN F, ELLIS WA, BARANTON G. Characterization of Leptospira isolates from serovar Hardjo by ribotyping, arbitrarily primed PCR, and mapped restriction site polymorphisms. J Clin Microbiol 1994; 32: 1949–1957. [ Links ]
29. ALONSO–ANDICOBERRY C, GARCÍA–PEÑA FJ, ORTEGA–MORA LM. Epidemiología, diagnóstico y control de la leptospirosis bovina (Revisión). Invest Agr: Prod Sanid Anim 2001; 16: 205–226. [ Links ]
30. RAMADASS P, MARSHALL RB, JARVIS BDW. Species differentiation of Leptospirainterrogans serovar Hardjo strain Hardjobovis from strain Hardjoprajitno by DNA slot blot hybridisation. J Res Vet Sci 1990; 49: 194–197. [ Links ]
31. ALT DP, ZUERNER RL, BOLIN CA. Evaluation of antibiotics for treatment of cattle infected with Leptospira borgpetersenii serovar Hardjo. J Am Vet Med Assoc 2001; 219: 636–639. [ Links ]
32. THIERMANN AB, HANDSAKER JW, FOLEY JW, WHITE FH, KINGSCOTE BF. Reclassification of North American leptospiral isolates belonging to serogroups Mini and Sejroe by restriction endonuclease analysis. Am J Vet Res 1986; 47: 61–66. [ Links ]
33. THORNLEY CN, BAKER PM, WEINSTEIN P, MAAS EW. Changing epidemiology of human leptospirosis in New Zealand. Epidemiol Infect 2002; 128:29–36. [ Links ]
34. DE LA PEÑA–MOCTEZUMA A, BULACH D, KALAMBAHETI T, ADLER B. Comparative analysis of the LPS biosynthetic loci of the genetic subtypes of serovar Hardjo: Leptospirainterrogans subtype Hardjoprajitno and Leptospiraborgpetersenii subtype Hardjobovis. FEMS Microbiol Lett 1999; 177: 319–326. [ Links ]
35. ZAMORA J, RIEDEMANN S, MONTECINOS MI, CABEZAS X. Aislamiento en Chile de Leptospirainterrogans serovares Hardjo y Kennewicki en bovinos aparentemente sanos. Arch Med Vet. 1991; 23: 131–135. [ Links ]
36. SMITH CR, CORNEY BG, MCGOWAN MR, MCCLINTOCK CS, WARD W, KETTERER PJ. Amoxycillin as an alternative to dihydrostreptomycin sulphate for treating cattle infected with Leptospiraborgpetersenii serovar Hardjo. Aust Vet J 1997; 75: 818– 821. [ Links ]
37. FERESU SB, STEIGERWALT AG, BRENNER DJ. DNA relatedness of Leptospira strains isolated from beef cattle in Zimbabwe. Int J Syst Bacteriol 1999; 49: 1111– 1117. [ Links ]
38. VASCONCELLOS SA, OLVEIRA JCF, MORAIS ZM, BARUSELLI PS, AMARAL R, PINHEIRO SR et al. Isolation of Leptospirasantarosai, serovar Guaricura from buffaloes (Bubalus bubalis) in Vale do Ribeira, Sao Paulo, Brazil. Braz J Microbiol 2001; 32: 298–300. [ Links ]
39. TORTEN M, BIRNBAUM S, KLINGBERG MA, SHENBERG E. Epidemiologic investigation of an outbreak of leptospirosis in the Upper Galilee, Israel. Am J Epidemiol 1970; 91:52–8 [ Links ]
40. VEDHAGIRI K, NATARAJASEENIVASAN K, PRABHAKARAN SG, SELVIN J, NARAYANAN R, SHOUCHE YS et al. Characterization of Leptospiraborgpetersenii isolates from field rats (Rattus norvegicus) by 16S rRNA and lipL32 gene sequencing. Braz J Microbiol 2010; 41: 150–157. [ Links ]
41. MOLES CLP, CISNEROS PMA, GAVALDÓN RD, ROJAS SN, TORRES BJI. Estudio serológico de leptospirosis bovina en México. Rev Cubana Med Trop 2002; 54: 24–27. [ Links ]
* Este manuscrito es parte del trabajo de tesis de Doctorado en Ciencias de la Producción y de la Salud Animal del primer autor.
** Carestream Health, Inc. Nueva York, Estados Unidos de América.
*** Gene Codes Corporation. Brighton, Inglaterra.
**** Roche Applied Science. Mannheim, Alemania.
***** Millipore Corporation. MA, Estados Unidos de América.
† Macrogen Inc., Seúl, Corea del Sur.














