Servicios Personalizados
Revista
Articulo
Indicadores
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Veterinaria México
versión impresa ISSN 0301-5092
Vet. Méx vol.42 no.3 Ciudad de México jul./sep. 2011
Artículos científicos
Resistencia a penicilina G y oxacilina, de cepas de Staphylococcus aureus aisladas de mastitis bovina subclínica
Penicillin G and oxacillin resistance in Staphylococcus aureus strains isolated from bovine subclinical mastitis
Michael Zschöck* Amr El–Sayed** Nawara Eissa* Christoph Lämmler*** Hugo Castañeda–Vazquez†
* Landesbetrieb Hessisches Landeslabor, Abteilung II (Veterinärmedizin), Schubertstr. 60, 35392 Giessen, Alemania. Contacto: Dr. Michael Zschöck, Correo electrónico: michael.zschoeck@lhl.hessen.de
** Faculty of Veterinary Medicine, Departament of Internal Medicine and Infectious Diseases, Giza Square, Cairo University, Egipto.
*** Institut für Pharmakologie und Toxikologie, Justus–Liebig–University, Frankfurter Str. 107, 35392 Giessen, Alemania.
† Laboratorio de Mastitis y Diagnóstico Molecular, Departamento de Medicina Veterinaria, Centro Universitario de Ciecias Biológicas y Agropecuarias, Universidad de Guadalajara, km 15.5 de la Carretera Guadalajara–Nogales, Zapopan, Jalisco, México.
Recibido el 6 de mayo de 2010.
Aceptado el 14 de marzo de 2011.
Abstract
Sixty–eigth out of 530 different S. aureus field strains isolated from subclinical cases of bovine mastitis from Germany (n = 26), Indonesia (n = 16), Mexico (n = 16) and Brazil (n = 10), respectively, were selected to be studied in the present work. The strains were tested phenotypically as well as genotypically for the presence of penicillin G– and oxacillin–resistance. For the primary phenotypical species identification of the 530 S. aureus strains, plasmacoagulase–test and Api 32 Staph system was applied. This was confirmed by molecular detection of the S. aureus specific genes encoding 23 S rRNA, thermostable nuclease (nuc), clumping factor (clfA), coagulase (coa) and protein A region Xr (spa). The selection of the 68 strains was carried out by the random selection of one strain per herd; additionally, only strains with different macrorestriction profiles were included here. Genotypic resistance to semisynthetic penicillins (methicillin/oxacillin) and penicillin G was studied through the identification of mecA– and blaZ–genes, respectively. The mecA gene was detected in only one S. aureus isolate from Brazil, which was not phenotypically resistant against methcillin, as shown by the use of standard disc diffusion method, BBL–Chromoagar and MIC–determination by Vitek II. In contrast penicillin–resistance of strains based on the presence of the blaZ–gene could be observed in 50 (73.5%) of the investigated strains. However, only 40 (58.8%) of these 50 blaZ–positive strains were phenotypically penicillin–resistant. According to the presented data, resistance to semisynthetic penicillins in S. aureus field strains seems to be not a major problem in dairy herds of the investigated countries despite the long–term use of these antibiotics in the field.
Key words: Staphylococcus aureus, bovine mastitis, Germany, Indonesia, Mexico, Brazil, oxacillin, penicillin G, resistance.
Resumen
De 530 diferentes cepas de S. aureus aisladas de casos de mastitis subclínica bovina, se seleccionaron 68 cepas de S. aureus procedentes de Alemania (n = 26), Indonesia (n = 16), México (n = 16) y Brasil (n = 10), para estudiarlas en la presente investigación. Las cepas fueron analizadas fenotípica y genotípicamente para observar su resistencia a penicilina G y oxacilina. Para una identificación inicial se utilizó el sistema Api 32 Staph y la prueba de coagulasa. El resultado se confirmó por la detección molecular de los genes específicos de S. aureus 23S rRNA, nucleasa termoestable (nuc), factor aglutinante (clfA), coagulasa (coa) y la proteína A (spa) región Xr. La selección primaria de las cepas sospechosas se hizo al azar, seleccionando una cepa por hato; además sólo se incluyeron en el estudio cepas con diferentes perfiles de macrorrestricción. La resistencia genotípica a meticilina y penicilina se estudió mediante la identificación de los genes mecA y blaZ, respectivamente. El gen mecA fue detectado sólo en un aislamiento de S. aureus de Brasil y no fue resistente fenotípicamente a la meticilina, lo cual se demostró mediante los métodos de difusión estándar en discos, el uso del Chromoagar–BBL y la determinación de la concentración mínima inhibitoria (MIC, por sus siglas en inglés) por Vitek II. La detección genotípica de la resistencia de las cepas a la penicilina se basó en la detección del gen blaZ; y se observó en 50 cepas investigadas (73.5%). Sin embargo, sólo 40 cepas (58.8%) fueron fenotípicamente resistentes a la penicilina. Los resultados obtenidos muestran que la resistencia de las cepas aisladas de campo S. aureus a las penicilinas semisintéticas, actualmente no es un problema importante en las vacas lecheras, a pesar del uso extensivo de esas sustancias antibióticas en el campo en los países investigados.
Palabras clave: Staphylococcus aureus, mastitis bovina, Alemania, Indonesia, México, Brasil, oxacilina, penicilina G, resistencia.
Introducción
El Staphylococcus aureus es uno de los patógenos principales causantes de una gran variedad de serias enfermedades en humanos y animales. Entre los principales problemas causados por S. aureus está la mastitis, que ocasiona graves pérdidas económicas a la industria lechera.1
Actualmente, la emergencia y la diseminación de cepas de Staphylococcus aureus resistentes a meticilina (MRSA) han llegado a ser un fuerte problema para la salud pública internacional. La diseminación del MRSA ha causado inefcacia en el tratamiento alternativo antes efectivo con drogas terapéuticas en los hospitales.2 Aparte de los casos en humanos, el MRSA fue aislado de varias especies animales tales como yeguas con metritis,3,4 infecciones de heridas,5–7 mastitis bovina,8 perros, gatos y conejos enfermos.5,9,10 Recientemente, el MRSA fue aislado de las líneas clonales ST398 y ST9 en cerdos.11 La trasmisión de MRSA se ha registrado entre humanos, animales y en el medio ambiente, en los lugares en los que existe un contacto estrecho entre animales y humanos portadores del MRSA. Respecto al papel del MRSA en el origen de la mastitis, se encontró que fue el causante esporádico en casos de mastitis en humanos12 y de mastitis subclínica bovina.8,13 Sin embargo, las cepas aisladas de las vacas no pudieron ser diferenciadas de aquellas obtenidas de los trabajadores en contacto estrecho con animales.8
La posible transmisión del MRSA de bovinos con mastitis bovina subclínica a humanos al consumir éstos la leche contaminada, es un problema real, ya que ha habido uso extensivo de la cloxacilina durante más de 30 años. Este tipo de penicilina semisintética está en uso en los países involucrados en esta investigación.
Para un diagnóstico rápido y seguro del MRSA se han desarrollado diferentes métodos moleculares y de cultivo.14–17
En el presente estudio, de 530 cepas de S. aureus se seleccionaron 68, procedentes de cuatro países ubicados en tres continentes, para ser investigadas fenotípica y genotípicamente, y observar su resistencia a oxacilina y penicilina G. Esos aislamientos se obtuvieron de vacas afectadas con mastitis subclínica en Alemania, Indonesia, México y Brasil. La selección de los aislamientos tuvo como base el tamaño del polimorfismo del gen coa y del gen spa en su región Xr y la heterogeneidad del perfil, identificados mediante el análisis de macrorrestricción con el uso de electroforesis en gel de campos pulsados. Únicamente fueron investigados los aislamientos con perfiles genéticos diferentes.
El objetivo del presente trabajo fue investigar la diversidad genética de cepas del Staphylococcus aureus aisladas de mastitis subclínica bovina, y su impacto en la salud pública. Además, los resultados de este estudio actualizarán la prevalencia de S. aureus resistente a penicilina G y oxacilina, asilado de mastitis subclínica, con base en una selección genotípica de cepas.
Material y métodos
Aislamientos bacterianos
Inicialmente se recolectaron 530 cepas de S. aureus de cuatro países correspondientes a tres continentes, tomadas de cultivos18 de muestras de leche de un cuarto de la ubre de vacas afectadas con mastitis subclínica (conteo de células somáticas* > 100,000 células/ml, sin signos macroscópicos de mastitis) en agar sangre de borrego Columbia al 5% con 1% de esculina.** Se incluyeron aislamientos de Alemania (n = 367), Indonesia (n = 35), México (n = 41) y Brasil (n = 87).
Después del aislamiento primario, se llevó a cabo un proceso de selección de las cepas de acuerdo con sus relaciones genéticas. El proceso se basó en los resultados del análisis de macrorrestricción del ADN,19 considerando publicaciones anteriores.20 Sólo se utilizaron en este estudio los aislamientos que no estaban relacionados genéticamente, identificados mediante el tamaño del polimorfismo del gen coa y de la región Xr del gen spa, y por la electroforesis en gel de campos pulsados (PFGE). La clasifcación genética para observar que no hubiese relación entre los aislamientos, se hizo por análisis de macrorrestricción; se definía que no había relación genética cuando mostraban una diferencia en seis o más bandas y un coeficiente de variación de correlación de 60% o menor. Los patrones de bandeo fueron comparados visualmente por dos observadores independientes.
La identificación de los cultivos se hizo mediante las características bioquímicas del cultivo y aplicando métodos moleculares. Para la caracterización primaria se efectuó la prueba de coagulasa en tubo, utilizando plasma de conejo con EDTA,** mediante la prueba directa en tubo.21 La prueba fue evaluada a las 4 h y 24 h después de haber sido incubada a 37°C. Enseguida se realizó la caracterización bioquímica usando el sistema de identificación comercial API32Staph.*** Se realizó una confirmación molecular mediante la amplificación por PCR de los segmentos específicos de especie de los genes 23S RNAr, nuc, coa, clfA y spa.7,22–27 Los iniciadores u oligonucleotidos usados y los programas de ciclos se enlistan en el Cuadro 1.
Reacción en cadena de la polimerasa, PCR
La PCR se utilizó para confirmar los aislamientos de S. aureus por medio de detección de las secuencias específicas de S. aureus,1,28 y para investigar en las cepas la presencia de los genes blaZ y mecA resistentes a los antibióticos.30,31 Las cepas utilizadas como testigo fueron S. aureus ATCC 27626 y S. aureus ATCC 25923, respectivamente.
Para la extracción del ADN genómico se tomaron cinco colonias recién subcultivadas de S. aureus y fueron suspendidas en 50 μl de solución amortiguadora TE (10 mM de Tris HCl/l, 1 mM de EDTA/l, pH 8.0). Enseguida se agregó 1 μl de lysostafna (1.8 U/l). La solución fue incubada a 37°C durante 60 min. Después de 60 min se le agregó 1 μl de proteinasa K (15.1 mg/ml) y la suspensión fue incubada durante 2 h a 56°C. Para inactivar la proteinasa K se hirvió la suspensión durante 10 min y luego se centrifugó a 10 000 g durante 5 min. Finalmente, el sobrenadante fue enfriado a 4°C en hielo, para después utilizarlo en la PCR. La mezcla de reacción para la PCR (20 μl) contenía 0.7 μl de ambos iniciadores (10 pm/μl), 0.4 μl de trifosfato deoxinucleósido (10 mM/l; MBI), 2.0 μl de 10 x amortiguador termofílico, 1.2 μl de MgCl2 (25 mM/l), 0.1 μl de Taq ADN polimerasa (5 U/μl), y 12.9 μl de agua destilada. Finalmente, se agregaron 2.0 μl de la muestra de ADN. Para la visualización de los productos de la PCR se hizo una electroforesis de 10 μl de cada producto de la reacción en un gel con 2% agarosa en solución amortiguadora 1X TAE (40 mM Tris–HCl, 1 mM EDTA/l, 1.14 μl/l ácido acético glacial, pH 7.8) con un voltaje de 70–100, enseguida se tiñó el gel durante 5 min en 5 μl/ml de solución de bromuro de etidio.**** Los amplicones fueron visualizados en un transiluminador UV.*****
Detección de cepas no relacionadas genéticamente
Junto con la medición del polimorfismo del gen coa y la región Xr del gen spa, se efectuó el análisis de macrorrestricción por PFGE para seleccionar las cepas no relacionadas genéticamente. El ADN cromosomal de S. aureus se digirió con la enzima de restricción SmaI y los fragmentos se separaron utilizando la electroforesis en gel de campos pulsados (PFGE),19 con el uso del sistema de electroforesis de campos pulsados Chef–Dr II.***** Solamente se consideraron genéticamente no relacionados los aislamientos que tenían diferencias en el tamaño del polimorfismo del gen coa y en la región Xr del gen spa y que tenían patrones de restricción con al menos seis fragmentos diferentes, así como un coeficiente de variación de correlación de 60% o menor.20 Estos aislamientos se incluyeron en la presente investigación.
Control de la resistencia a antibióticos
La detección de la resistencia fenotípica y genotípica a la penicilina G y la oxacilina se realizó aplicando el método de la difusión estándar en disco30 y la prueba molecular con la PCR,29,30 respectivamente. La determinación de la sensibilidad de los aislamientos a los antibióticos antes mencionados se realizó haciendo cultivo de las cepas de S. aureus en caldo de Mueller–Hinton****** durante 4 h a 37°C. Subsecuentemente, 10 μl de la suspensión fue inoculada en agar Mueller–Hinton.****** Enseguida se colocó un disco con antibiótico, que contenía 10 UI de penicilina G o 1 mg de oxacilina en el centro del medio de cultivo. Los medios de agar se incubaron durante 24 h a 37°C. Se midió la zona de inhibición del crecimiento bacteriano alrededor del disco y se comparó con los parámetros establecidos para estas pruebas. Los resultados de las pruebas se interpretaron de acuerdo con las normas del National Committee for Clinical Laboratory Standards.18 Las cepas de S. aureus resistentes a oxacilina también fuero inoculadas en BBL Cromoagar MRSA.******* Los medios se incubaron de forma aerobia a 37°C y se leyeron después de 24 horas. Los MRSA tuvieron una coloración violeta y las otras colonias un color blanco, azul o azul–verde. Para verificar los resultados fenotípicos obtenidos, adicionalmente se determinó la concentración mínima inhibitoria MIC utilizando el sistema Vitek II.********
Resultados
Las 530 cepas de S. aureus de muestras de leche con mastitis fueron identificadas con el uso de la prueba de plasmacoagulasa y el sistema comercial de identificación API 32 Staph. Ello se confirmó fenotípicamente por la amplificación, mediante la PCR, de los segmentos específicos de S. aureus, que codifican para los genes 23S rRNA, nucleasa termoestable (nuc), clumping factor o factor aglutinante (clfA), coagulasa (coa) y el segmento del gen que codifica para la región respectiva Xr y la región enlazante de IgG de la proteína A (spa). Dependiendo del tamaño del polimorfismo del gen coa y la región Xr del gen spa, y de acuerdo con los perfiles de macrorrestricción mediante PFGE (Figura 1), como resultado de este estricto proceso se seleccionaron 68 aislamientos de S. aureus de los 530 aislamientos de campo iniciales, para continuar con las investigaciones. Finalmente se seleccionaron 26 aislamientos de Alemania, 16 de Indonesia, 16 de México y 10 de Brasil.

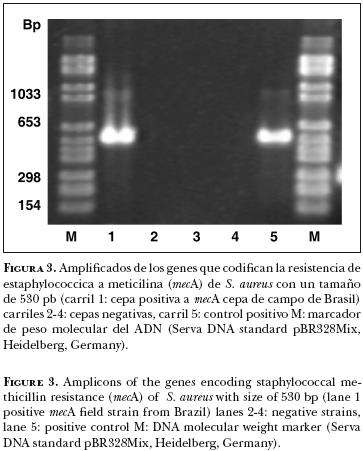
De acuerdo con los resultados de la resistencia a penicilina G y a oxacilina (Cuadro 2), 40 cepas mostraron mediante métodos fenotípicos resistencia a la penicilina G y 50 cepas fueron positivas genotípicamente al gen blaZ (Figura 2). La investigación genotípica para detectar el gen mecA en las 68 cepas de S. aureus analizadas reveló la presencia del gen sólo en un aislamiento originario de Brasil (Figura 3), si bien este resultado no concuerda con resultado negativo obtenido mediante el método estándar de difusión en disco de BBL–Cromoagar y la determinación de MIC sensible a oxacilina, por Vitek II.

En contraste a la resistencia a oxacilina y el gen mecA, el examen genotípico y fenotípico de la resistencia a penicilina G y al gen blaZ, la mayoría de las cepas de S. aureus mostraron la diseminación del gen blaZ, al hacer la prueba genotípica por PCR. En la mayoría de las cepas de S. aureus investigadas se localizó el gen blaZ 50 (73.5%). Detalladamente, 18 cepas de Alemania (65.2%), 11 de México (68.75%), 8 de Indonesia (80.0%) y 13 de Brasil (81.3%) fueron positivas al gen blaZ. En contraste, en el análisis fenotípico, únicamente 40 cepas (58.8%) fueron resistentes a la penicilina G. La distribución detallada fue: 10 cepas de campo en Alemania (38.5%), 5 en México (13.3%), 5 en Indonesia (50%) y 10 en Brasil (62.4%).
Discusión
Los antibióticos β–lactámicos son los agentes antimicrobianos más ampliamente utilizados en el tratamiento de la mastitis bovina. El objetivo del estudio fue investigar la presencia de diferentes linajes genéticos del Staphylococcus aureus, su papel en el origen de la mastitis bovina, y comparar la eficiencia de las PCR específicas de MRSA con la de los medios selectivos para MRSA, que requieren más tiempo para la detección de los aislamientos positivos a MRSA, así como también a los antibióticos β–lactámicos, por métodos fenotípicos y genotipicos . Para lograr ese propósito se incluyeron en el presente estudio 68 cepas aisladas de S. aureus sin relación genética alguna, que representaban 68 hatos de 530 aislamientos de S. aureus provenientes de cuatro países. Las 68 cepas mostraron perfiles genéticos heterogéneos, como se observó mediante el uso del tamaño del polimorfismo del gen coa, de la región Xr del gen spa, y del análisis de macrorrestricción por PFGE. El aislamiento previo de MRSA en casos de humanos se realizó en Indonesia,15,32 México,16,33 Brasil34–37 y Alemania.38 A excepción de Alemania,39 el MRSA no ha podido ser aislado de vacas con mastitis. En la presente investigación se encontro únicamente una cepa positiva al gen mecA, aislada de un caso de mastitis bovina, pero fenotípicamente fue negativa. El origen de este aislamiento de MRSA no fue claro. En informes previos sobre aislamientos de MRSA de muestras veterinarias8,40 y de muestras de alimentos,41 se concluyó que esos aislamientos no eran de origen animal, la mayoría procedía de humanos que estaban en contacto cercano con animales. Además hay otros estudios que informan sobre la posibilidad de una trasmisión de S. aureus de humanos a bovinos.42–44 El origen de la cepa positiva de mecA, descrita en la presente investigación, no pudo ser identificado. Investigaciones anteriores compararon el uso de los métodos de cultivo con los moleculares in vitro para la determinación de la resistencia a antibióticos.14,17,45 Las recomendaciones dadas por esos autores no son claras. Mientras que Van Hal et al.17 informan que los métodos moleculares fueron más confiables para detectar el MRSA que los métodos convencionales basados en agares, Flayhart et al.14 indican que el uso del C–MRSA es confiable y más seguro que otros métodos, ya que alcanza un promedio de especificidad de 99.7% en esa investigación. En los presentes resultados el análisis fenotípico de resistencia a oxacilina y la detección molecular del gen mecA fueron comparables. Pero pudo observarse una gran variación entre los métodos fenotípicos y genotípicos para la detección de la resistencia a penicilina G. Los presentes resultados no concuerdan con los resultados obtenidos por Flayhart et al.,14 quienes registraron 97.6% de coincidencia entre los diferentes métodos moleculares y de cultivo. Por otra parte, si bien el uso de los métodos moleculares es limitado debido a los altos costos de la PCR, ahorra tiempo, ya que los resultados pueden obtenerse en 2 o 3 horas, que son muy pocas, comparadas con las 18 a 24 horas necesarias para los métodos de cultivo.17 Alternativamente, los datos aquí presentados indican la necesidad de combinar el uso de diferentes métodos para lograr un diagnóstico seguro. Además, se recomienda debido a la confusión y a la gran variación encontradas entre la detección fenotípica y genotípica de la resistencia a antibióticos –como se confirma en el presente trabajo–, y también por la detección de falsos positivos y de resultados de cultivo negativos, como se ha informado previamente.46
La presente investigación apoya a otras publicaciones que mencionan la ausencia de MRSA o su incidencia muy baja en muestras de leche. Ello indica que el MRSA actualmente no es un patógeno principal en el ganado bovino lechero, a pesar de que durante mucho tiempo se han utilizado las penicilinas semisintéticas en la terapia de mastitis. La baja incidencia de aislamientos de MRSA en muestras de leche sugiere una posible contaminación de las muestras de leche por cepas de humanos o medioambientales.
Referencias
1. EL–SAYED A, ALBER J, LAEMMLER Ch, JAEGER , S, WOLTER W, CASTAÑEDA VH. Comparative study on genotypic properties of Staphylococcus aureus isolated from clinical and subclinical mastitis in Mexico. Vet Mex 2006; 37, 165–179. [ Links ]
2. WALDVOGEL FA. Staphylococcus aureus (including toxic shock syndrome). In: MANDELL GL, BENETT JE, DOLIN R, MANDELL DS, editors. Douglas and Benett's Principles and Practice of Infectious Disease. New York: Churchill Livingstone: 1754–1777. [ Links ]
3. ANZAI T, KAMADA M, KANEMARU T, SHUGUTA S, SHIMIZU A, HIGUCHI T. Isolation of methicillin–resistant Staphylococcus aureus from mares with metritis and its zooepidemiology. J Equine Vet Sci 1996; 7: 7–11. [ Links ]
4. SHIMIZU A, JAWANO J, YAMAMOTO CH, KAKUTANI O, ANZAI T, KAMADA M. Genetic analysis of equine methicillin–resistant Staphylococcus aureus by pulse–feld–gel– electrophoresis. J Vet Med Sci 1997; 59: 935–937. [ Links ]
5. RICH M, ROBERTS L. Methicillin–resistant Staphylococcus aureus isolates from companion animals. Vet Rec 2004; 154: 310. [ Links ]
6. SEGUIN JC, WALKER RD, CARON JP, KLOOS WE, GEORGE CG, HOLLIS RJ et al. Methicillin–resistant Staphylococcus aureus outbreak in a veterinary teaching hospital. Potential human to animal transmission. J Clin Microbiol 1999; 37: 1459–1463. [ Links ]
7. STEPHAN R, ANNEMÜLLER C, HASSAN AA, LÄMMLER CH. Characterization of eneterotoxigenic Staphylococcus aureus strains isolated from bovine mastitis in North–east Switzerland. Vet Microbiol 2001; 78: 373–382. [ Links ]
8. JUHAZ–KASZANVITZKY E, JANOSI S, SOMOGVI P, DAN A, VAN DER GRAAF–VAN BLOOIS L, VAN DUIJKEREN E et al. MRSA transmission between cows and humans. Emer Infect Dis 2007; 13: 630–632. [ Links ]
9. GORTEL K, CAMPBELL KL, KAKOMA I, WHITTEM T, SCHAEFFER DJ, WEISIGER RM. Methicillin–resistance among Staphylococci isolated from dogs. Am J Vet Res 1996; 60: 1562–1530. [ Links ]
10. TOMLIN JM, PEAD J, LLOYD DH, HOWELL S, HARTMAN F, JACKSON HA et al. Methicillin–resistant Staphylococcus aureus infections in 11 dogs. Vet Rec 1999; 144: 60–64. [ Links ]
11. KEHRENBERG C, CUNY C, STROMMENGER B, SCHWARZ S, WITTE W. Methicillin–resistant and susceptible Staphylococcus aureus strains of clonal lineages ST398 and ST9 from swine carry the multidrug resistance gene cfr. Antimicrob Agents Chemother 2009; 53: 779–781. [ Links ]
12. SCHOENFELD EM, MCKAY MP. Mastitis and methicillin–resistant Staphylococcus aureus (MRSA): the calm before the storm? J Emerg Med 2010; 38: 31–34. [ Links ]
13. DEVRIESE LA, HOMMEZ F. Epidemiology of methicillin resistant Staphylococcus aureus in dairy herds. Res Vet Sci 1975; 19: 23–27. [ Links ]
14 . FLAYHART D, HINDLER J, BRUCKNER D, HALL G, SHRESTHA R, VOGEL SH et al. Multicenter evaluation of BBL CHROMagar MRSA medium for direct detection of methicillin–resistant Staphylococcus aureus from surveillance cultures of the anterior nares. J Clin Microbiol 2005; 43: 5536–5540. [ Links ]
15. CHONGTRAKOOL P, ITO T, MA X, KONDO Y, TRAKULSOMBOON S, TIENSASITORN CH et al. Staphylococcal cassette chromosome mec (SCCmec). Typing of methicillin–resistant Staphylococcus aureus strains isolated in 11 Asian Countries: a proposal for a new nomenclature for SCCmec elements. Antimicrob Agents Chemother 2006; 50: 1001–1012. [ Links ]
16. ECHANIZ–AVILES G, VELAZQUEZ–MEZA M E, AIRES–DE–SOUSA M, MORFIN–OTERO R, RODRIGUEZ–NORIEGA E, CARNALLA–BARAJAS N et al. Molecular characterization of a dominant methicillin–resistant Staphylococcus aureus (MRSA) clone in a Mexican hospital (1999–2003). Clin Microbiol Infect 2006; 12: 22–28. [ Links ]
17. VAN HAL S J, STARK D, LOCKWOOD B, MARRIOTT D, HARKNESS J. Methicillin–resistant Staphylococcus aureu s (MR SA) detection: Comparison of two molecular methods (IDI–MRSA PCR assay and GenoType MRSA direct PCR assay) with three selective MRSA agars (MRSA ID, MRSASelect, and CHROMagar MRSA) for use with infection–control swabs. J Clin Microbiol 2007; 45: 2486 –2490. [ Links ]
18. NATIONAL COMMITTEE FOR CLINICAL LABORATORY STANDARDS. Methods for disk diffusion: approved standard M2–A8: performance standards for antimicrobial disk susceptibility tests. Wayne (PA): The Committee, 2003. [ Links ]
19. TOSHKOVA K, SAVOY A, SOEDARMANTO L, LÄMMLER CH, CHANKOVA D, VAN BELKUM A et al. Typing of Staphylococcus aureus isolated from nasal carriers. Zentralbl Bakteriol 1997; 286: 547–559. [ Links ]
20. TENOVER FC, ARBEIT R, ARCHER G, BIDDLE J, BYRNE S, GOERDING R et al. Comparison of traditional and molecular methods of typing isolates of Staphylococcus aureus. J Clin Microbiol 1995; 32: 407–415. [ Links ]
21. HARMON RJ, EBERHARDT RJ, JASPER DE, LANGLOIS BE, WILSON RA. Microbiological procedures for the diagnosis of bovine udder infections. 3rd ed. Arlington, VA: National Mastitis Council, 1990. [ Links ]
22. STRAUB JA, HERTEL C, HAMMES WP. A 23S rRNA–targeted polymerase chain reaction– based system for detection of Staphylococcus aureus in meat starter cultures and dairy product. J Food Prot 1999; 62: 1150–1156. [ Links ]
23. BRAKSTAD OG, AASBAKK K, MAELAND JA. Detection of Staphylococcus aureus by Polymerase chain reaction, amplification of the nuc gene. J Clin Microbiol 1992; 30: 1654 –1660. [ Links ]
24. FRENAY H ME, BUNSCHOTEN LM, SCHOULS WJ V, LEEUWEN CMJ, GRAULS EV, VERHOEF J et al. Molecular typing of methicillin–resistant Staphylococcus aureus on the basis of protein A gene polymorphism. Eur J Clin Microbiol Infect Dis 1996; 15: 60–64. [ Links ]
25. HOOKEY J, RICHARDSON VJF, COOKSON BD. Molecular typing of Staphylococcus aureus. Basis on PCR restriction fragment length pholymorphism and DNA sequence analysis of the coagulase gene. J Clin Microbiol 1998; 36: 1083–1089. [ Links ]
26. AKINEDEN Ö, ANNEMÜLLER C, HASSAN A A, LÄMMLER CH, WOLTER W, ZSCHÖCK M. Toxin genes and other characteristics of Staphylococcus aureus isolates from milk of cows with mastitis. Clin Diagn Lab Immunol 2001; 8: 959–964. [ Links ]
27. EL–SAYED A, ALBER J, LAMMLER CH, BONNER B, HUHN A, KALETA EF et al. PCR–based detection of genes encoding vir ulence determinants in Staphylococcus aureus from birds. J Vet Med 2005; 52: 38–44. [ Links ]
28. REINOSO EB, EL–SAYED A, LÄMMLER CH, BOGNI C, ZSCHOCK M. Genotyping of Staphylococcus aureus isolated from humans, bovine subclinical mastitis and food samples in Argentina. Microbiol Res 2006; 163: 314 – 3 22. [ Links ]
29. MARTINEAU F, PICARD FJ, GRENIER L, ROY PH, OUELLETTE M, TRIAL E et al. Multiplex PCR assays for the detection of clinically relevant antibiotic resistance genes in staphylococci isolated from patients infected after cardiac surgery. J Antimicrob Chemother 2000; 46: 527–533. [ Links ]
30. LEE JH. Methicillin (Oxacillin)–resistant Staphylococcus aureus strains isolated from major food animals and their potential transmission to humans. Appl Environ Microbiol 2003; 69: 6489– 6494. [ Links ]
31. BARRY AL, FUCHS PC, ALLEN SD, THORNSBERRY C, GERLACH EH, JONES N. Quality control limits for the standard anaerobic reference agar dilution susceptibility test procedure of the National Committee for Clinical Laboratory Standards. J Clin Microbiol 1989; 27: 192–195. [ Links ]
32. SEVERIN JA, LESTARI E S, KUNTAMAN K, MELLES D C, PASTINK M, PEETERS JK et al. Antimicrobial resistance in Indonesia, prevalence and prevention study group. Unusually high prevalence of Panton–Valentine leukocidin genes among methicillin–sensitive Staphylococcus aureus strains carried in the Indonesian population. J Clin Microbiol 2008; 46: 1989–1995. [ Links ]
33. VELAZQUEZ–MEZA ME, DE SOUSA M, ECHANIZ–AVILES G, SOLORZANO–SANTOS F, MIRANDA–NOVALES G, SILVA–SANCHEZ et al. Surveillance of methicillin–resistant Staphylococcus aureus in a pediatric Hospital in Mexico City during a 7–year period (1997 to 2003): Clonal evolution and impact of infection control. J Clin Microbiol 2004; 42: 3877–3880. [ Links ]
34. WEY SB, CARDO DM, HALKER E, CARRATU FP, SAES AC. Distribution and analysis of 8.268 nosocomial infections at the Hospital São Paulo: 1985–1989. Rev Hosp São Paulo 1990; 1: 169–174. [ Links ]
35. SADER HS, PIGNATARI AC, HOLLIS RJ, LEME I, JONES RN. Oxacillin– and quinolone– resistant Staphylococcus aureus in Sao Paulo, Brazil: a multicenter molecular epidemiology study. Infection control and hospital epidemiology. Infect Control Hosp Epidemiol 1993; 14: 260–264. [ Links ]
36. CALA FFA FILHO MP, LIMA SI, SINTO A, ANDRIOLO A, MENDES CMF. Avaliação da sensibilidade à teicoplanina e vancomicina, em Staphylococcus aureus estaflococos coagulase negativos. Rev Assoc Med Bras 1994; 40: 77–80. [ Links ]
37. TRINDADE PA, MCCULLOCH JA, OLIVEIRA GA, MAMIZUKA EM. Molecular techniques for MRSA typing: Current issues and perspectives. Braz J Infect Dis 2003; 7: 32–43. [ Links ]
38. SCHMITZ FJ, STEIERT M, TICHY HV, HOFMANN B, VERHOEF J, HEINZ HP et al. Typing of methicillin–resistant Staphylococcus aureus isolates from Düsseldorf by six genotypic methods. J Med Microbiol 1998; 47: 341–351. [ Links ]
39. MONECKE S, KUHNERT P, HOTZEL H, SLICKERS P, EHRICHT R. Microarray based study on virulence–associated genes and resistance determinants of Staphylococcus aureus isolates from cattle. Vet Microbiol 2007; 125: 128–140. [ Links ]
40. HARMON FA, TROSTLE SS, KLONEN AA. Isolation of Methicillin–resistant Staphylococcus aureus from a postoperative wound infection in a horse. J Am Vet Med Assoc 1997; 211: 590–592. [ Links ]
41. KITAI S, SHIMIZU A, KAWANO J, SATO E, NAKANO C, UJI T et al. Characterization of methicillin–resistant Staphylococcus aureus isolated from retail raw chicken meat in Japan. J Vet Med Sci 2005; 67: 107–110. [ Links ]
42. FOX LK, GERSHMANN M, HANCOOK DD, HUTTON CT. Fomites and reservoirs of Staphylococcus aureus causing intramammary infections as determined by phage typing: the effect of milking time hygiene practices. Cornell Vet 1991; 81: 183–193. [ Links ]
43. ROBERSON L, FOX K, HANCOOK DD, GAY JM, BESSER TE. Ecology of Staphylococcus aureus isolated from various sites on dairy farms. J Dairy Sci 1994; 77: 3354–3364. [ Links ]
44. ZADOKS R, VAN LEEUWEN W, BARKEMA H, SAMPIMON O, VERBRUGH H, SCHUKKEN Y H et al. Application of Pulse–fledgel electrophoresis and binary typing as tools in veterinary clinical microbiology and molecular epidemiologic analysis of bovine and human Staphylococcus aureus isolates. J Clin Microbiol 2000; 38: 1931–1939. [ Links ]
45. FARLEY J, STAMPER PD, ROSS T, SPESER S, CAI M, CARROLL KC. Comparison of the IDI–MRSATM assay to culture using BBLTM CHROMagarTM MRSA for detection of MRSA from nasal surveillance cultures in an at–risk population. 107th General Meeting of the American Society for Microbiology; 24 March 2007; Toronto, Canada. Toronto, Canada: American Society for Microbiology, 2007:1–4. [ Links ]
46. PAPE J, WADLIN J, NACHAMKIN I. Use of BBL CHROMagar MRSA medium for identification of methicillin–resistant Staphylococcus aureus directly from blood cultures. J Clin Microbiol 2006; 44: 2575–2576. [ Links ]
* Fossomatic, Aarhous, Dinamarca.
** Merck, Darmstadt, Alemania.
*** BioMerieux, Nürtingen, Alemania.
**** Sigma, Taufkirchen, Alemania.
***** Bio–Rad, Munich, Alemania.
****** Merck, Darmstadt, Dinamarca.
******* BBL; Becton Dickinson, Sparks, MD.
******** BioMerieux, Nürtigen, Alemania.














