Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Veterinaria México
Print version ISSN 0301-5092
Vet. Méx vol.40 n.4 Ciudad de México Oct./Dec. 2009
Artículos científicos
Covarianzas genéticas y fenotípicas para días abiertos y características de la curva de lactancia en vacas Holstein en el norte de México
Genetic and phenotypic covariances for days open and lactation curve characteristics in Holstein cows from northern Mexico
Reyes López–Ordaz* Héctor Castillo–Juárez** Hugo H. Montaldo**
* Departamento de Genética y Bioestadística, Facultad de Medicina Veterinaria y Zootecnia, Universidad Nacional Autónoma de México, 04510, México, D. F.
** Universidad Autónoma Metropolitana–Xochimilco, Calzada del Hueso 1100, Col Villa Quietud, 04960, México, D. F.
Responsable de la correspondencia:
Tel.: (55) 5622–5894, Fax: (55) 5622–5956.
Correo electrónico: montaldo@servidor.unam.mx.
Recibido el 7 de agosto de 2008
Aceptado el 11 de mayo de 2009
Abstract
The aim of this study was to estimate genetic and phenotypic (co)variances for lactation curve traits and for days open in Holstein cows. Data included 1 579 lactations from 766 cows, daughters of 126 sires in a dairy herd in northern Mexico. The studied traits within lactation were days open (DO), peak milk production (PMAX), days to peak milk production (DPMAX), 305–day milk production (MP305), lactation persistency (based on Wood equation) (PERSW), lactation persistency expressed as the natural logarithm of the Wood equation persistency (LNPERSW), and lactation persistency measured as (production at day 305/PMP) x 100 (PERS). Covariance components were obtained by single trait and bivariate mixed linear models using restricted maximum likelihood. In general, heritabilities estimated using the repeatability model yielded lower values than those obtained based on within lactation analysis. Average heritabilities estimated with single trait models within lactation were 0.13 ± 0.09, 0.28 ± 0.09, 0.28 ± 0.09, 0.17 ± 0.10 and 0.22 ± 0.10, for DO, MP305, PMAX, DPMAX, and LNPERSW, respectively. Genetic correlations between MP305 and DO (0.66±0.57) and between PMAX and DO (0.55 ± 0.71) were unfavorable for first lactation cows, but with large standard errors. Results confirmed a low heritability for DO, but with estimates possibly larger in younger cows. Genetic correlation between MP305 and PMAX was 0.89 ± 0.09, and LNPERSW and DPMAX was 0.98 ± 0.21 for the third lactation, indicating that DPMAX is a good measure of persistency. No evidence for genetic correlation between MP305 and LNPERSW was found.
Key words: Dairy Cattle, Genetic Parameters, Peak Milk Production, Milk Production, Lactation Persistency, Animal Model, REML.
Resumen
El objetivo de este estudio fue estimar covarianzas genéticas y fenotípicas para características de la curva de lactancia y días abiertos en vacas Holstein. Los datos incluyeron 1 579 lactancias de 766 vacas, hijas de 126 sementales, en un hato en el norte de México. Las características estudiadas por lactancia fueron días abiertos (DO), producción máxima de leche (PMÁX), días a la producción máxima (DPMÁX), producción de leche de 305 días (MP305), persistencia de la lactancia por Wood (PERSW), persistencia de Wood expresada como logaritmo natural (LNPERSW) y persistencia medida como ((producción al día 305/PMÁX) x 100) (PERS). Los componentes de covarianza fueron obtenidos mediante el uso de modelos univariados y bivariados con máxima verosimilitud restringida. Las heredabilidades estimadas para las características con el modelo de repetibilidad tuvieron valores generalmente inferiores a los estimados con los análisis dentro de lactancia. Las heredabilidades promedio estimadas con modelos univariados dentro de lactancia para DO, MP305, PMÁX, DPMÁX y LNPERSW fueron 0.13 ± 0.09, 0.28 ± 0.09, 0.28 ± 0.09, 0.17 ± 0.10 y 0.22 ± 0.10, respectivamente. Las correlaciones genéticas entre MP305 y DO (0.66±0.57) y entre PMÁX y DO (0.55 ± 0.71) fueron desfavorables para las primeras lactancias, pero con errores estándar elevados. Los resultados confirman una heredabilidad baja para DO, pero con valores posiblemente mayores en vacas jóvenes. La correlación genética entre MP305 y PMÁX fue de 0.89 ± 0.09 y entre LNPERSW y DPMÁX fue 0.98 ± 0.21 para la tercera lactancia, lo que indica que DPMÁX es buen estimador de la persistencia. No se encontró evidencia de correlación genética entre MP305 y LNPERSW.
Palabras clave: Ganado lechero, Parámetros genéticos, Producción máxima de leche, Producción de leche, Persistencia de la lactancia, Modelo animal, REML.
Introducción
El mejoramiento genético de las poblaciones de vacas lecheras requiere el conocimiento sobre las covarianzas genéticas y fenotípicas de las características económicamente importantes.1,2 Tradicionalmente, la producción de leche ha sido el principal criterio de selección en las vacas lecheras, porque se relaciona de manera directa con la ganancia económica para los productores.3,4 Recientemente se ha incrementado el énfasis dado al estudio de varias características reproductivas y de salud con el objetivo de obtener una reducción en los costos de producción y mejorar el bienestar animal.5–7 Algunos autores han evaluado las características de la curva de lactancia, como persistencia, la producción máxima y la producción de leche para determinar su posible inclusión como criterios de selección.8–11 Varios estudios han demostrado que, genética y fenotípicamente, la eficiencia reproductiva y la producción de leche tienen relaciones genéticas y fenotípicas desfavorables.12–16 Estos estudios también demostraron valores de heredabilidades por debajo de 0.10 para los días abiertos. Las heredabilidades para días abiertos y el intervalo entre partos en México han sido estimados en un rango de cero hasta 0.038,17,18 mientras que no hay estimaciones disponibles para las covarianzas genéticas que implican las características de la curva de lactancia y de reproducción para el ganado lechero mexicano. El objetivo de este estudio fue estimar los componentes de covarianza genética y fenotípica (heredabilidades, repetibilidades y correlaciones genéticas y fenotípicas) para los días abiertos (DO), la producción máxima de leche (PMÁX), los días a la producción máxima (DPMÁX), la persistencia de la lactancia estimada con la metodología de Wood (PERSW), la persistencia de la lactancia estimada con la metodología de Wood en forma logarítmica (LNPERSW), la persistencia medida como 100x(producción del día 305/producción máxima de leche) (PERS) y la producción de leche a 305 días (MP305), con datos de un hato comercial de vacas Holstein en la región árida del norte de México.
Material y métodos
Población y manejo
La información se obtuvo de un hato de ganado lechero en Bermejillo, Durango, México, localizado a 25° 49' latitud N, con 103° 29' longitud O y 1 325 msnm. El clima se clasifica como muy árido, semicálido, con lluvias en verano y temperatura extrema. La precipitación anual (r) es de 261 milímetros y la temperatura media anual (t) es de 18.6°C, el índice medio de la humedad (r/t) es de 14% y la oscilación anual del promedio de la temperatura mensual es de 10.5°C.19
Los animales fueron criados en un sistema intensivo con sombra disponible. La alimentación se basó en concentrado, alfalfa, ryegrass, ensilaje de maíz y complementos minerales. El concentrado fue ofrecido durante el ordeño dos veces al día. La sala de ordeño es un tándem doble con capacidad para 48 vacas.
Los registros analizados inicialmente se obtuvieron entre 1981 y 1993. La información incluyó los registros mensuales de la producción de leche por día y los eventos reproductivos de 2 112 partos de 891 vacas Holstein. La identificación del padre y de la madre también estuvo disponible.
La reproducción en el hato se basó en la inseminación artificial usando sementales de Canadá y de Estados Unidos de América, seleccionados con base en sus evaluaciones genéticas para la producción de leche. Las becerras de reemplazo fueron obtenidas del mismo hato.
Variables analizadas
Se usaron datos mensuales del día de prueba para modelar curvas de lactancia para lactancias individuales, usando la ecuación de Wood:20

Donde:
Yn = Producción de leche al n–ésimo día;
a, b y c = parámetros de la ecuación;
a = factor de posicionamiento que indica la producción al principio de la lactancia;
b = tasa del incremento hasta alcanzar el punto de la producción máxima;
c = índice de la disminución gradual a partir de la producción máxima;
e = base de los logaritmos naturales ln.20
Estos parámetros fueron obtenidos de la forma lineal de la ecuación 1, mediante regresión múltiple, usando mínimos cuadrados ordinarios, como sigue:
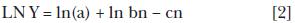
La producción de leche de 305 días (MP305) se obtuvo por medio de lactancia, sumando la producción de leche diaria predicha con el modelo a partir de los días 1 al 305 con la ecuación:
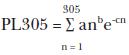
La producción de leche máxima (PMÁX) fue:
PMÁX = a (b/c) exp (–b). El día de la producción máxima (DPMÁX) fue: b/c días después del parto. La persistencia de Wood (PERSW) fue: PERSW = (b + 1) ln (c) + ln (b + 1).
Adicionalmente se calculó el logaritmo natural de PERSW (LNPERSW) para analizar una variable con una distribución más próxima a la normal.21
Se calculó también la persistencia de la lactancia (PERS), como el porcentaje que representa la producción de leche acumulada para el día 305 (Y305) sobre la producción de leche máxima como PERS = (Y305/ PMÁX) x 100.
Sólo se utilizaron lactancias con curvas típicas con respecto a la descripción de Wood para el análisis final;20 es decir, curvas que presentaron incremento inicial hasta alcanzar la producción del pico y después una disminución de la producción de leche hasta el final de la lactancia. Aproximadamente 20% de las lactancias analizadas fueron eliminadas debido a comportamiento atípico. Se presume que esas curvas corresponden a vacas con problemas de falta de información, datos equivocados o problemas de manejo.
El coeficiente de determinación (R2) del modelo de Wood para los datos analizados, varió de 0.42 a 0.99 para las lactancias individuales, con promedio de 0.60. Después de editar todos los datos, los análisis finales fueron realizados con un archivo de pedigrí con 1 063 animales con información de padre y madre, con 126 padres y un archivo de datos de producción y eventos reproductivos de 1 579 partos a partir de 766 vacas.
Estimación de covarianzas
Se utilizaron modelos univariados de repetibilidad como el siguiente, para la estimación de las heredabilidades:

donde:
yijk = observación para las variables dependientes: DO, MP305, PMÁX, DPMÁX, PERSW, LNPERSW y PERS.
µ = promedio general;
fi = efecto fijo del i–ésimo año–estación de parto;
nj = efecto fijo del j–ésimo número de parto;
ak = efecto genético aleatorio del k–ésimo animal;
pk = efecto ambiental permanente aleatorio para el k–ésimo animal;
eijk = error aleatorio (NIID).
Para el análisis se definieron dos estaciones de parto: a) otoño–invierno, de agosto a enero y b) primavera–verano, de febrero a julio. Las vacas con cinco o más partos fueron agrupadas en una categoría.
Todos los análisis se realizaron con el programa ASREML.22 En todos los análisis se utilizó la matriz de parentescos aditivos A.
Los componentes de varianza inicialmente estimados para cada característica, se usaron como valores iniciales para realizar análisis con modelos multica–rácter de repetibilidad, con los mismos efectos usados en los modelos univariados, sin alcanzar la convergencia, debido a la presencia de heterogeneidad de varianzas y covarianzas entre lactancias. Por ello se realizaron análisis multicarácter, en las tres primeras lactancias con modelos animales con el efecto fijo de año–estación y el efecto aleatorio de animal.
Se seleccionaron las variables DO, MP305, PMÁX, DPMÁX, LNPERSW para los análisis multicarácter, pues se consideró que representaban mejor los aspectos estudiados, porque fueron las que tuvieron mayor variación genética, mejores propiedades de distribución y para evitar redundancias. Se usaron modelos multicarácter con distinto número de variables, debido a que aquellas con heredabilidades iguales a cero se excluyeron de estos análisis para permitir la convergencia.
Resultados
El Cuadro 1 muestra los estadísticos descriptivos de las características estudiadas por lactancia y para todas las lactancias; el Cuadro 2, las heredabilidades, repetibilidades y las varianzas fenotípicas obtenidas con los modelos univariados de repetibilidad.

Las heredabilidades para los modelos dentro de lactancia fueron mayores que para los modelos de repetibilidad, aunque la heredabilidad de ciertas características fue cero para algunas lactancias (Cuadros 2, 3 y 4). Las heredabilidades obtenidas con los modelos univariados dentro de lactancia (Cuadro 3) fueron muy similares a las obtenidas con los modelos multicarácter (Cuadro 4).
Las correlaciones genéticas por lactancia se muestran en el Cuadro 5 y las correlaciones fenotípicas por lactancia en el Cuadro 6.
Discusión
Producción de leche
La heredabilidad estimada para MP305 con el modelo de repetibilidad (0.16) fue inferior al promedio de los valores obtenidos por lactancia (0.28). El rango de estimados con los distintos modelos (0.16–0.31) (Cuadros 2, 3 y 4), está aproximadamente dentro del rango de las estimaciones obtenidas en estudios anteriores, de 0.14 a 0.40.23–25
Estas diferencias reflejan la presencia de heterogeneidad de covarianzas a través de lactancias, que pueden hacer los modelos de repetibilidad menos adecuados para este análisis, como se demuestra por la falta de convergencia hallada en los análisis multicarácter de repetibilidad. La heredabilidad para MP305 encontrada aquí en la primera lactancia de 0.16 (Cuadro 2), es cercana a la obtenida por Ravagnolo y Misztal,23 de 0.14 bajo condiciones climáticas similares. Para las lactancias posteriores, los valores de heredabilidad obtenidos (0.29 y 0.31) se encuentran dentro del rango de los valores para el ganado lechero, publicados previamente para Holstein en México, de 0.18 a 0.48.26,27
Las heredabilidades promedio fueron 0.15 para DO, 0.25 para la MP305, 0.27 para PMÁX, 0.14 para DPMÁX y 0.13 para LNPERSW (Cuadro 4). Muir et al.,28 usando datos de primera lactancia en Holstein, encontraron heredabilidades de 0.07 para intervalo entre partos, 0.45 para la producción de leche, 0.09 para DPMÁX y 0.18 para la persistencia definida como la pendiente de la curva de lactancia después del rendimiento de leche al pico. Rekaya et al.,21 con datos de primera lactancia, encontraron heredabilidades de 0.26, 0.14, 0.26, y 0.05 para la producción de leche de 305 días, para la persistencia medida de acuerdo con la ecuación de Wood, para producción al pico, y para los días a la producción máxima en leche, respectivamente.
La repetibilidad para MP305 fue 0.33 (Cuadro 2). Este valor es inferior al observado por Palacios Espinosa et al.,26 (0.45) y también al obtenido por Valencia et al.27 y Montaldo y Torres,29 (cercano a 0.50). Sin embargo, está dentro del rango de 0.25 a 0.48 observado por López y Lara,30 en una revisión de parámetros genéticos de ganado lechero para las regiones áridas de México.
Persistencia de la lactancia
Las heredabilidades para las distintas características de persistencia de la lactancia (PERS, PERSW, LNPERSW), estimadas con los modelos univariados de repetibilidad fueron todas cercanas a cero (Cuadro 2). Este resultado podría sugerir que tales características dependen principalmente de la variación ambiental. Esto, sin embargo, podría estar asociado con la estructura de covarianzas porque cuando el análisis se realizó dentro de lactancia, los valores de heredabi–lidad se estimaron en un rango que va de 0.14 a 0.27. Otros autores han encontrado valores que van desde 0.05 hasta 0.14.21,31–33 Las heredabilidades para PERS, PERSW, LNPERSW obtenidas aquí fueron similares a las halladas por otros autores con un modelo univariado por lactancia;31–33 sin embargo, en la tercera lactancia son mayores a los observados por Sölker y Fuchs,11 de 0.21. Los estimados para la tercera lactancia (0.14–0.27), son aproximadamente similares a los observados por Sölker y Fuchs,11 de 0.21, Van der Linde et al.,34 de 0.15 y de Jakobsen et al.,35 de 0.21, pero menores al encontrado por Swalve y Gengler,36 de 0.30.
Van der Linde et al.,34 de 0.15 y de Jakobsen et al.,35 de 0.21, pero similares para LNPERSW en el análisis multicarácter para la primera lactancia y un valor menor al encontrado por Swalve y Gengler,36 de 0.30. La repetibilidad para PERS fue de 0.15 (Cuadro 2); ésta fue similar al valor de 0.18 obtenido por Tekerli et al.37
Producción máxima de leche
La heredabilidad estimada para PMÁX en este estudio con el modelo univariado de repetibilidad fue 0.16 (Cuadro 2), valor ligeramente inferior al estimado por Rekaya et al.38 de 0.26. Sin embargo, este valor es menor al obtenido en este estudio (0.41 a 0.45) para la tercera lactancia con un modelo multicarácter (Cuadros 3 y 4).
La repetibilidad para PMÁX fue 0.24 (Cuadro 2), valor similar al estimado por Rekaya et al.21 y Tekerli et al.,37 de 0.26.
Días abiertos
La heredabilidad obtenida para DO en el modelo unicarácter de repetibilidad (0.008) (Cuadro 2), es similar a los valores hallados por Dematawewa y Berger,39 de 0.04; Kadarmideen et al.,40 de 0.012 a 0.028; de Haile–Mariam et al.32 de 0.03; Abdallah y Mc Daniel,24 de 0.03; Demeke et al.,41 de 0.04; y de Olori et al.,42 de 0.04. Los análisis dentro de lactancia arrojaron valores de heredabilidad entre 0.10 y 0.18 (Cuadros 3 y 4). La repetibilidad de DO fue de 0.12 (Cuadro 2), similar a los valores observados por Damatawewa y Berger,39 de 0.12; y Demeke et al.,41 de 0.14. En México, Montaldo y Torres,17 estimaron un valor de 0.09.
Correlaciones genéticas
MP305 y la PMÁX son prácticamente la misma variable genéticamente hablando, con una rg cercana a 1 (Cuadro 6). Rekaya et al.21 obtuvieron un valor similar de 0.90; sin embargo, Jakobsen et al.43 encontraron un valor de 0.47 y Van der Linde et al.34 encontraron un valor de 0.52. LNPERSW tuvo una correlación genética de 0.20 con MP305 para los datos de primera lactancia (Cuadro 6), que es similar a las estimaciones de Rekaya et al.,21 de 0.33 y Muir et al.,28 de 0.21 también para datos de primera lactancia. MP305 tuvo una correlación genética favorable con LNPERSW en la primera lactancia, pero no en la tercera lactancia. La correlación genética entre LNPERSW y PMÁX fue de 0.18 para datos de primera lactancia y –0.44 aunque no significativa (P > 0.05), para datos de tercera lactancia (Cuadro 6). Rekaya et al.,21 estimaron dicha correlación genética como –0.14. Hay evidencias de heterogeneidad de las asociaciones entre estas variables entre lactancias (Cuadro 6), lo que sugiere que este tipo de estudios debería hacerse por lactancia, empero, esto podría indicar características particulares de esta base de datos.
Los errores estándar para las correlaciones genéticas de LNPERSW con otras características fueron grandes, ello se explica, en parte, por la pequeña variación genética aditiva de LNPERSW y por el número de las observaciones.24,44 Cabe destacar que Rekaya et al.21 estimaron dicha correlación genética como –0.14. La correlación genética entre LNPERSW y DPMÁX fue cercana a 1 (Cuadro 6). La correlación genética entre MP305 y DPMÁX fue 0.41 para datos de primera lactancia y –0.30 para datos de tercera lactancia pero con un error estándar mayor al estimador del parámetro. Muir et al.28 encontraron un valor de 0.63 y Rekaya et al.21 de 0.59, para esta correlación usando datos de primera lactancia. La correlación genética entre DO y MP305 fue 0.66, aunque no significativa (P > 0.05). Muir et al.28 encontraron una correlación genética de 0.51 entre MP305 y DO para datos de primera lactancia.
Los estudios realizados por Veerkamp et al.44 y Abdallah y McDaniel,24 también sugieren un antagonismo genético entre las características entre MP305 con DO con valores positivos de las correlaciones genéticas. La correlación genética para datos de segunda lactancia fue de –0.27; Otros estudios,45–47 sin embargo, han demostrado una correlación genética cercana a cero (0.01 a 0.04) entre estas características.
Hay evidencias de asociaciones genéticas desfavorables de DO con PMÁX, LNPERSW y DPMÁX en la primera lactancia, pero no en las lactancias posteriores (Cuadro 5) La correlación genética de DO con LNPERSW fue de 0.51 para la primera lactancia. Muir et al.28 estimaron la correlación genética entre intervalo entre partos y LNPERSW como 0.17. La correlación genética entre DPMÁX y DO fue 0.65 para datos de primera lactancia, mientras que Muir et al.28 encontraron un valor de 0.31 entre intervalo entre partos y DPMÁX.
Correlaciones fenotípicas
La correlación fenotípica entre MP305 y PMÁX y entre LNPERSW y DPMÁX fue cercana a 0.9 (Cuadro 5). Estos resultados son similares a los obtenidos por Rekaya et al.21
La correlación fenotípica entre MP305 y DPMÁX para la primera lactancia fue 0.18 y 0.17 para la tercera; Muir et al.28 estimaron un valor de 0.10. La correlación fenotípica entre LNPERSW y MP305 fue 0.20. Jakobsen et al.43 encontraron un valor de 0.54, mientras que Rekaya et al.21 lo estimaron como 0.40. La correlación fenotípica entre PMÁX y LNPERSW se estimó entre 0.01 y 0.08 (Cuadro 5), en tanto que Rekaya et al.21 la estimaron como –0.03. Las correlaciones fenotípicas fueron positivas para DO con MP305 (0.10), LNPERSW (0.23), PMÁX (0.01 a 0.08) y DPMÁX (0.17 a 0.18). Muir et al.28 estimaron los valores para intervalo entre partos con MP305 como 0.17, con LNPERSW como 0.23 y con DPMÁX como 0.09.
Implicaciones
Los valores de heredabilidad estimados para todas las características con el modelo de repetibilidad fueron menores a los estimados con el modelo dentro de lactancias. La falta de convergencia obtenida usando modelos de repetibilidad multicarácter indica que la estructura de (co)varianzas difiere entre lactancias, que representa una forma de interacción genotipo–medio ambiente. Esto puede indicar la conveniencia de realizar evaluaciones genéticas multicarácter considerando las lactancias como diferentes características, en lugar de usar un modelo de repetibilidad.
Las heredabilidades obtenidas aquí confirman que MP305, PMÁX y LNPERSW son características con suficiente variación genética aditiva para obtener cambios por selección. En este contexto, MP305 y PMÁX son características con una correlación genética alta y positiva, lo cual indica que la selección para una mayor producción de leche a 305 días incrementará el pico de producción, como consecuencia ello puede conducir a problemas metabólicos posparto. La correlación genética entre la producción de leche y LNPERSW fue positiva. Los resultados indican que la selección directa para las características reproductivas y de persistencia en esta población en el norte de México, tendría respuestas pequeñas a la selección. A corto plazo, estas características pueden ser mejoradas manipulando las condiciones ambientales.
Las correlaciones genéticas de DO con MP305, PMÁX, LNPERSW y DPMÁX fueron positivas para datos de primeras lactancias, indicando un antagonismo entre la producción de leche y la reproducción. Los resultados para la segunda lactancia indican, sin embargo, relaciones genéticas favorables entre DO con MP305 y PMÁX, aunque no fueron estadísticamente significativas. A este respecto, Ruiz et al.48 no encontraron para México evidencias de antagonismos entre fertilidad medida como intervalo entre partos y la producción de leche, usando datos de primeras lactancias de vacas Holstein.
Debido a la importancia económica relativamente alta de las características reproductivas en las vacas,49,50 así como los valores muy bajos de heredabilidad observados en este estudio, podría ser de utilidad buscar criterios de selección indirectos, como consumo de alimento o condición corporal, que afectan tanto la reproducción como la curva de lactancia14 o perfiles hormonales.16 Estas características pueden tener valores más altos de heredabilidad; por tanto, las respuestas indirectas de la selección para las características reproductivas podrían ser mayores a las obtenidas por selección directa, si fuese posible medirlas en toda la población y si las correlaciones genéticas con fertilidad fuesen suficientemente altas.
Agradecimientos
Los autores agradecen a Marcia Castillo Mendoza por revisar la versión en inglés de este artículo.
Referencias
1. HOFER A. Variance component estimation in animal breeding: a review. J Anim Breed Genet 1998; 115:247–265. [ Links ]
2. HANSEN LB. Consequences of Selection for Milk yield from a geneticist's viewpoint. J Dairy Sci 2000; 83:1145–1150. [ Links ]
3. VEERKAMP RE Selection for economic efficiency of dairy cattle using information on live weight and feed intake: a review. J Dairy Sci 1998; 81:1109–1119. [ Links ]
4. MOURITS MCM, HUIRNE RBM, DIJKHUIZEN AA, KRISTENSES AR, GALLIGAN DT. Economic optimization of dairy heifer management decisions. Agric Sys 1999; 61:17–31. [ Links ]
5. HOLMANN FJ, SHUMWAY CR, BLAKE RW, SCHWART RB, SUDWEEKS EM. Economic value of days open for Holstein cows of alternative milk yields with varying calving intervals. J Dairy Sci 1984; 67:636–643. [ Links ]
6. STRANDBERG E, OLTENACU PA. Economic consequences of different calving intervals. Acta Agric Scand 1989; 39: 407–420. [ Links ]
7. OLESEN I, GROEN AF, GJERDE B. Definition of animal breeding goals for sustainable production systems. J Anim Sci 2000; 78, 570–582. [ Links ]
8. GROSSMAN M, HARTZ SM, KOOPS WJ. Persistency of lactation yield: A novel approach. J Dairy Sci 1999; 82:2192–2197. [ Links ]
9. DEKKERS JCM, TEN HAG JH, WEERSINK A. Economic aspects of persistency of lactation in dairy cattle. Livest Prod Sci 1998; 53:237–252. [ Links ]
10. GENGLER N. Persistency of lactation yields: A review. Workshop on genetic improvement of functional traits in cattle. Interbull Bull 1996; 12:87–95. [ Links ]
11. SÖLKNER J, FUCHS W. A comparison of different measures of persistency with special respect to variation of test–day milk yields. Livest Prod Sci 1987; 16:305–319. [ Links ]
12. CASTILLO–JUAREZ H, OLTENACU PA, BLAKE RW, MCCULLOCH CE, CIENFUEGOS–RIVAS EG. Effect of herd environment on the genetic and phenotypic relationships among milk yield, conception rate and somatic cell score in Holstein cattle. J Dairy Sci 2000; 83: 807–814. [ Links ]
13. DECHOW CD, ROGERS GW, CLAY JS. Heritabilities and correlations among body condition scores, production traits, and reproductive performance. J Dairy Sci 2001; 84: 266–275. [ Links ]
14. VEERKAMP RF, KOENEN EPC, DE JONG G. Genetic correlations among body condition score, yield, and fertility in first–parity cows estimated by random regression models. J Dairy Sci 2001; 84:2327–2335. [ Links ]
15. DECHOW CD, ROGERS GW, CLAY JS. Heritability and correlations among body condition score loss, body condition score, production and reproductive performance. J Dairy Sci 2002; 85: 3062–3070. [ Links ]
16. PRYCE JE, ROYAL MD, GARNSWORTHY PC, MAO IL. Fertility in the high–producing dairy cow. Livest Prod Sci 2004; 86:125–135. [ Links ]
17. MONTALDO HH, TORRES S. Parámetros genéticos y fenotípicos de la producción de leche e intervalo entre partos de una población de vacas Holstein en México. Acta Universitaria 1992; 2:37–43. [ Links ]
18. LEMUS–FLORES C, BECERRIL–PEREZ CM, ORTIZ–SOLORIO CA, ESPINOZA–VELAZQUEZ J. Effect of percentage of white coat color on milk production and reproduction of first parturition Holsteins cows in some climates of Mexico. Agrociencia 2002; 36: 23–30. [ Links ]
19. GARCIA E. Modificaciones al sistema de clasificación climática de Köppen. México DF: Instituto de Geografía, UNAM. 1988:220. [ Links ]
20. WOOD PDP. Algebraic model of the lactation curve in cattle. Nature 1967; 216:164–165. [ Links ]
21. REKAYA R, CARABAÑO M.J, TORO M.A. Bayesian analysis of lactation curves of Holstein–Friesian cattle using a nonlinear model. J Dairy Sci 2000; 83:2691–2701. [ Links ]
22. GILMOUR AR, CULLIS B R, WELLIAM S J, THOMPSON R. ASREML. Reference manual. Harpenden, U. K: IACR–Rothamsted Exp. Station, 2002. [ Links ]
23. RAVAGNOLO O, MISZTAL I. Effect of heat stress on nonreturn rate in Holstein cow: genetic analyses. J Dairy Sci 2002; 85:3092–3100. [ Links ]
24. ABDALLAH JM, MC DANIEL BT. Genetic parameters and trends of milk, fat, days open, and body weight after calving in North Carolina Experimental herds. J Dairy Sci 2000; 83:1364–1370. [ Links ]
25. DRUET T, JAFFRÉZIC F, BOICHARD D, DUCROCQ V. Modeling lactation curves and estimation of genetic parameters for first lactation test–day records of French Holstein cows. J Dairy Sci 2003; 86:2480–2490. [ Links ]
26. PALACIOS–ESPINOSA A, RODRÍGUEZ–ALMEIDA F, JIMÉNEZ–CASTRO J, ESPINOZA–VILLAVICENCIO JL, NÚÑEZ–DOMÍNGUEZ R. Evaluación genética de un hato Holstein en Baja California Sur, utilizando un modelo animal con mediciones repetidas. Agrociencia 2001; 35:347–352. [ Links ]
27. VALENCIA PM, RUIZ FJ, MONTALDO HH. Genetic and environmental variance components for milk yield across regions, time periods and herd levels for Holstein cattle in Mexico. Revista Científica–FCV–LUZ. 2004; 14:404–411. [ Links ]
28. MUIR BL, FATEHI J, SCHAEFFER LR. Genetic relationships between persistency and reproductive performance in first–lactation Canadian Holsteins. J Dairy Sci 2004; 87:3029–3037. [ Links ]
29. MONTALDO HH, TORRES S. Repetibilidad de la producción de leche e intervalo entre partos en una población de vacas Holstein en México. Arch Zoot 1993; 42: 361–366. [ Links ]
30. LÓPEZ OR, LARA FV. Caracterización del mejoramiento genético de bovinos lecheros en México (tesis de licenciatura). Chapingo, México: Universidad Autónoma Chapingo, 1996. [ Links ]
31. GENGLER N, TIJANI A, WIGGANS GR, PHILPOT JC. Indirect estimation of (co)variance functions for test–day yields during first and second lactations in the United States. J Dairy Sci 2001; 84:542–570. [ Links ]
32. HAILE–MARIAM M, BOWMAN PJ, GODDARD ME. Genetic and environmental relationship among calving interval, survival, persistency of milk yield and somatic cell count in dairy cattle. Livest Prod Sci 2003; 80:189–200. [ Links ]
33. ROOS APW, HARBERS AGF, DE JONG, G. Random herd curves in a test–day model for milk, fat, and protein production of dairy cattle in the Netherlands. J Dairy Sci 2004; 87:2693–2701. [ Links ]
34. VAN DER LINDE R, GROEN A, DE JONG G. Estimation of genetic parameters for persistency of milk production in dairy cattle. Interbull Bull 2000; 25:1–4. [ Links ]
35. JAKOBSEN JH, REKAYA R, JENSEN J, SORENSEN DA, MADSEN P, GIANOLA D et al. Bayesian estimate of covariance components between lactation curve parameters and disease liability in Danish Holstein cows. J Dairy Sci 2003;86:3000–3007. [ Links ]
36. SWALVE HH, GENGLER N. Genetic of lactation persistency. J Anim Sci 1999; 24:75–82. [ Links ]
37. TEKERLI M, AKINCI Z, DOGAN I, AKCAN A. Factors affecting the shape of lactation curves of Holstein cows from the Balikesir Province of Turkey. J Dairy Sci 2000; 83:1381–1386. [ Links ]
38. REKAYA R, CARABAÑO MJ, TORO MA. Use of test day yields for the genetic evaluation of production traits in Holstein Friesian cattle. Livest Prod Sci 1999;57:203–217. [ Links ]
39. DEMATAWEWA CMB, BERGER PJ. Genetic and phenotypic parameters for 305–day yield, fertility, and survival in Holstein. J Dairy Sci 1998; 81:2700–2709. [ Links ]
40. KADARMIDEEN HN, THOMPSON R, COFFEY MP, KOSSAIBATI MA. Genetic parameters and evaluations from single– and multiple– trait analysis of dairy cow fertility and milk production. Livest Prod Sci 2003; 81:183–195. [ Links ]
41. DEMEKE S, NESER FWC, SCHOEMAN SJ. Estimates of genetic parameters for Boran, Friesian and crosses of Friesian and Jersey with the Boran cattle in the tropical highlands of Ethiopia: reproduction traits. J Anim Breed Genet 2004; 121:57–65. [ Links ]
42. OLORI VE, MEUWISSEN THE, VEERKAMP RF. Calving interval and survival breeding values as measure system with seasonal calving. J Dairy Sci 2002; 85:689–696. [ Links ]
43. JAKOBSEN JH, MADSEN P, JENSEN J, PEDERSEN J, CHRISTENSEN LG, SORENSEN DA. Genetic parameters for milk production and persistency for Danish Holstein estimated in random regression model using REML. J Dairy Sci 2002;85:1607–1616. [ Links ]
44. VEERKAMP RF, OLDENBROEK JK, VAN DER GAAST HJ, VAN DER WERF JHJ. Genetic correlation between days until start of luteal activity and milk yield, energy balance, and live weights. J Dairy Sci 2000; 83:577–583. [ Links ]
45. RAHEJA KL, BURNSIDE EB, SCHAEFFER LR. Relationships between fertility and production in Holstein dairy cattle in different parity. J Dairy Sci 1989; 72:2670–2678. [ Links ]
46. OLTENACU PA, FRICK A, LINDHÉ B. Relationship of fertility to milk yield in Swedish cattle. J Dairy Sci 1991; 74. 264–268. [ Links ]
47. ROMAN MR, WILCOX CJ. Bivariate animal model estimates of genetic, phenotypic, and environmental correlations for production, reproduction, and somatic cells in Jerseys. J Dairy Sci 2000; 83:829–835. [ Links ]
48. RUIZ LF, VALENCIA PM, MONTALDO HH, CASTILLO–JUAREZ H. Genetic parameters for somatic cell counts, production and reproduction trails in primiparous Mexican Holstein cows. 8th World Congress on Genetics Applied to Livestock Production; 2006 August 13–18: Belo Horizonte, MG, Brazil. Belo Horizonte, MG, Brazil: Universidad de Minas Gerais y EMBRAPA – Cenargen FAO. CD – ROM– 636.0820063 page 21. [ Links ]
49. DU PLESSIS M, ROUX CZ. A breeding goal for South African Holstein Friesians in terms of economic weights in percentage units.S Afr J Anim Sci 1999; 29:237–244. [ Links ]
50. WOLFOVA M, PRIBYL J, WOLF J. Economic weights for production and functional traits of Czech dairy cattle breeds. Czech J Anim Sci 2001; 46:421–432. [ Links ]
Este trabajo es resultado de la tesis de Maestría en Ciencias del primer autor.














