INTRODUCCIÓN
El advenimiento de bases de datos genómicos muy grandes ha hecho que el análisis filogenético de las secuencias de aminoácidos en proteínas sea un tema atractivo y desafiante, ya que las inferencias con respecto a la selección natural pueden proporcionar información funcional importante [1]. Hay disponibles muchos programas de computadora para el análisis de datos filogenéticos. Actualmente el programa más popular cubre una amplia gama de opciones, mediante un código adaptable y fácil de usar [2], ofreciendo así a los usuarios máximos resultados con un mínimo esfuerzo. El objetivo final de establecer el poder de la selección darwiniana para mejorar la función de la proteína aún no se ha alcanzado [3-5]. Sin embargo, los esfuerzos para cuantificar los relojes moleculares que se iniciaron en la década de 1960 por Pauling y otros ya ha dado resultados positivos [6]. En términos más generales, hay muchas dificultades en filogenómica, y se ha dicho que "más secuencias no son suficientes" [7].
La filogenia cuenta aminoácidos idénticos o similares en sitios específicos utilizando el código BLAST, y se puede refinar de muchas maneras [2], pero todas estas están limitadas por la restricción a sitios únicos. Existe una alternativa a los métodos de sitio único, teniendo la selectividad darwiniana como una característica implícita, y que ha sido corroborada por la identificación de la criticalidad auto-organizada en las áreas superficiales accesibles a solventes (SASA, del inglés “solvent-accessible surface areas”) para más de 5000 segmentos de aminoácidos de proteínas pertenecientes a la moderna Protein Data Base [8,9].
Esta criticalidad autoorganizada puede entenderse en el contexto más amplio de los sistemas complejos. En estos sistemas, la auto-organización se refiere a la capacidad de un sistema para generar un comportamiento colectivo emergente partiendo de interacciones entre sus constituyentes. Un ejemplo típico en física lo son las fases de la materia, donde las moléculas se auto-organizan al imponérseles condiciones externas como presión o temperatura, dando lugar a comportamientos colectivos, como lo puede ser por ejemplo la resistencia a fluir, lo cual determina si un material se comporta como líquido, sólido o gas. Al variar las condiciones externas aplicadas a un sistema, los cambios entre estos comportamientos no son suaves; de hecho implican discontinuidades en las propiedades termodinámicas, lo cual da lugar a las llamadas transiciones de fase. En 1869 el profesor de química Thomas Andrew encontró que existen condiciones de presión y temperatura donde la diferencia entre fases deja de existir. Así, arriba de cierta presión y temperatura, no es posible distinguir el agua de su vapor. A estos puntos se le llama críticos. Un poco después de su descubrimiento experimental, el físico holandés Van der Waals logró mostrar su existencia de manera teórica. Más aún, Van der Waals logró demostrar que si el fluido se describe escalando las variables termodinámicas en términos de los parámetros en el punto crítico, el fluido llevarse a una descripción universal que ya no depende del sistema en particular. Más allá de la termodinámica, la criticalidad es un comportamiento robusto al cual se llega sin importar los parámetros del modelo y sin necesidad de que el sistema esté en equilibrio. Hay dos características importantes de la criticalidad auto-organizada: una es la capacidad del sistema para mantenerse cerca de ese estado, y la otra es la existencia de la invariancia de escala (fractalidad) [9]. Esta invariancia indica que el sistema se ve igual cuando se observa a una escala diferente. Un ejemplo típico lo constituye la costa de un país, la cual se ve muy parecida al examinarse en mapas de diferente escala. Ello se debe a que existe un mecanismo de erosión y una auto-organización que mantiene a la costa es un estado crítico. Otro ejemplo son las nubes, las cuales también están en un estado crítico y por ello se ven blancas, ya que dispersan la luz de manera igual para todas las longitudes de onda. Como veremos, las proteínas también presentan invariancia de escala [8,9].
En vista de la discusión del párrafo precedente, es claro que el descubrimiento de la criticalidad auto-organizada de la SASA ya implica una selección darwiniana para acercar el proteoma a puntos críticos funcionales. Esto proporciona una plataforma para analizar la evolución de las proteínas individuales, tales como el lisozima c de la clara de huevo de gallina [10], la neuroglobina [11] y muchas otras [12].
El primer paso para desarrollar un nuevo método es probarlo en muchos casos específicos, examinando cada uno de los nuevos datos que proporciona. Debido a que cada familia de proteínas tiene una o más funciones diferentes, uno aprende algo nuevo en cada caso. Sin embargo, ciertos aspectos han ido surgido hasta ahora. Uno de ellos se refiere a la naturaleza del SASA de segmentos auto-organizados estudiados por Moret y Zebende [8]. Para entender este descubrimiento, en la Figura 1 se detallan los pasos seguidos por Moret y Zebende en el año 2007 [8].
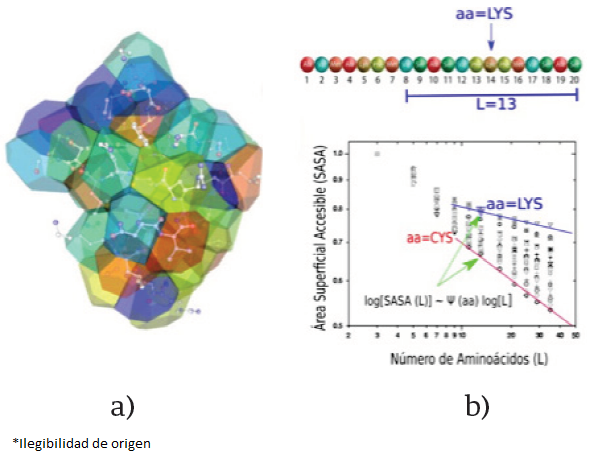
Figura 1. a) Fragmento de la estructura de una proteína, indicada por esferas unidas por barras. Sobrepuesta a ella se aprecian los poliedros de Voronoi, los cuales delimitan las regiones del espacio en la cuales todos los puntos contenidos dentro del poliedro están más cercanos al átomo situado en el centro del poliedro que a cualquier otro átomo. La superficie exterior neta de estos poliedros permite definir el área superficial accesible a las moléculas del agua (SASA). En la parte de arriba del panel b) se muestra la secuencia de aminoácidos (aa) de la proteína. Para estudiar su relación con la SASA, se escoge un aminoácido, en este caso cistina (aa=LYS), y se considera un intervalo de L aminácidos. Realizando una estadística sobre muchos fragmentos para diferentes L y proteínas, se obtiene la gráfica log-log de la parte b). Para L>9, los datos en la gráfica log-log pueden ajustarse con una recta, indicada en azul, y cuya pendiente da el parámetro Ψ(aa). Considerando otros aa se obtienen rectas similares, por ejemplo para aa=CYS se obtiene la línea roja. Estas pendientes definen la escala de hidropatía MZ del 2007, donde la CYS es el aa más hidrofóbico y LYS el aa más hidrofílico.
Para ello consideraron que la hidropatía está determinada de acuerdo al área accesible al agua, la cual a nivel molecular en la cercanía de la proteína tiene una estructura más parecida al hielo que al agua. El área accesible puede obtenerse usando la construcción de Voronoi, en la cual el espacio se subdivide en poliedros, cada uno centrado en un átomo de la estructura. El poliedro de Voronoi se define de modo que todos los puntos en su interior están más cercanos al átomo central del poliedro que de cualquier otro átomo. Dada la estructura de la proteína, esto permite obtener una imagen como la que se aprecia en la Figura 1a), y obtener así la SASA, que corresponde al área externa de los poliedros. Posteriormente, Moret y Zebende estudiaron como variaba la SASA para secuencias de aminoácidos (aa) de diferentes tamaños centrados en un aa dado, tal y como se muestra en la Figura 1b. Las longitudes de sus pequeños segmentos L =2N + 1 variaron de 3 a 45, pero el rango interesante resultó ser M <=9 ≤ L ≤ M>=35. A través de éste, encontraron un comportamiento lineal en un diagrama log-log (es decir, una ley de potencias y por lo tanto, auto-similar) para cada uno de los 20 aminoácidos centrados en un segmento dado. Es decir,
Aquí Ψ(aa) es un parámetro del índice de hidropatía para cada aa, y como puede verse en la Figura 1b), se obtiene de la pendiente de la recta que surge al realizar la gráfica log-log de L contra la SASA. Surge porque los segmentos más largos se repliegan sobre sí mismos, ocluyendo el SASA del aa central. El aspecto más sorprendente de esta oclusión plegada auto-similar es su casi universalidad en promedio a través del proteoma celular, y que es casi independiente del pliegue de la proteína individual. Esta es una demostración dramática del poder de la selectividad darwiniana involucrada en la formación acuosa de proteínas globulares, como se discute en detalle en la referencia [12]. Además, el carácter segmentario de la nueva escala [8] tiene un eco darwiniano: para cada familia de proteínas se puede identificar un ancho de ventana móvil optimizado W*, sobre el cual Ψ(aa) se promedia mejor; este promedio se denota por Ψ(aa,W*). Los perfiles de Ψ(aa,W*) muestran las características funcionales modulares optimizadas por la evolución [12].
El centro del rango de la SASA (una ley de potencias, y que describe estructuras auto-similares), 9 ≤ L ≤ 35, es 21. Las proteínas de membrana funcionan en el lado citoplásmico de la membrana celular [13], esta también descrita como un sustrato catalítico que soporta interacciones de tipo proteína-proteínas en el espacio fronterizo interfacial [14]. Las diferencias evolutivas entre la proteína gigante Hub Sr y la tirosina quinasa Syk se describen mejor mediante W* = 21 (+/- 5%) [15]. Es posible considerar a la actina como la proteína de membrana arquetípica, debido a su función esquelética en el soporte de membranas, al mismo tiempo que tiene suficiente flexibilidad para permitir cambios de forma funcionales termodinámicamente clasificados como de segundo orden. Además, la actina empuja las membranas hacia adelante durante el crecimiento celular [16,17], lo cual es termodinámicamente de primer orden.
La actina ha evolucionado muy poco. Se puede comparar con la ubiquitina, la cual tiene sólo 76 aminoácidos, y se encuentra sin cambios en mamíferos, aves, peces e incluso gusanos. La ubiquitina marca las proteínas enfermas para el reciclaje mediante una cascada de enzimas en tres etapas, iniciada por Uba (E1). Debido a su pequeño tamaño, además de su papel central en biología, la ubiquitina se ha convertido en uno de los más importantes sistemas modelo para estudiar la dinámica de proteínas y ha sido objeto de numerosos estudios, incluidos algunos que combinan enfoques experimentales y computacionales. Un estudio reciente con simulaciones de dinámica molecular a gran escala (> 5000 moléculas de agua) de la Ub globular identificó movimientos conformacionales lentos que implican correlaciones globulares estabilizadoras de cadenas β cerca de los terminales n y c en la escala de tiempo de microsegundos a milisegundos [18]. La función de etiquetado de la Ub es consistente con su perfil hidrofóbico y sugiere un modelo de red elástica agrietada para el blanco común compartido por muchas proteínas enfermas [19]. La Uba (E1) es 14 veces más grande que la Uba y también ha evolucionado muy poco, pero esa pequeña evolución rastrea la nivelación de los extremos hidrofóbicos del parámetro Ψ (aa,W*) [20]. Esta nivelación pivotante refleja la optimización de la dinámica de proteínas por su evolución [12,21].
RESULTADOS
La actina es una proteína de tamaño mediano con 377 aminoácidos, y es tan estable que para obtener cambios evolutivos sustanciales, se deben comparar humanos con algas (con 84% de identidades según BLAST, 92% positivas). Primero observamos la función Vr(W), es decir, la razón algas / humanos de las varianzas de Ψ(aa,W*), que se muestra en la Figura 2, la cual contiene la primera sorpresa de la actina (de hecho, las funciones de las proteínas son todas diferentes, por lo que generalmente hay sorpresas, especialmente con proteínas extremadamente estables, "casi perfectas" como la actina y la ubiqutina). La mayoría de las veces, W* es el valor de W que maximiza la razón de la varianza Vr, pero aquí W* corresponde a un máximo en la derivada dVr/ dW. ¿Qué ha ocurrido? La actina realiza dos funciones, la estabilización de las formas de las células después de pequeñas distorsiones de segundo orden durante el funcionamiento, y la polimerización para empujar las membranas hacia adelante durante el crecimiento celular [16,17], lo cual es es termodinámicamentedeprimerorden.Aparentemente, ambas funciones se optimizan al equilibrar los valores de W pequeños para W <21 (importante para la polimerización) [16,17], frente a los valores de W grandes de W (importante para estabilizar formas de células grandes). Este equilibrio elimina el máximo en Vr y genera, en cambio, un máximo en dVr/dW en W* = 21.
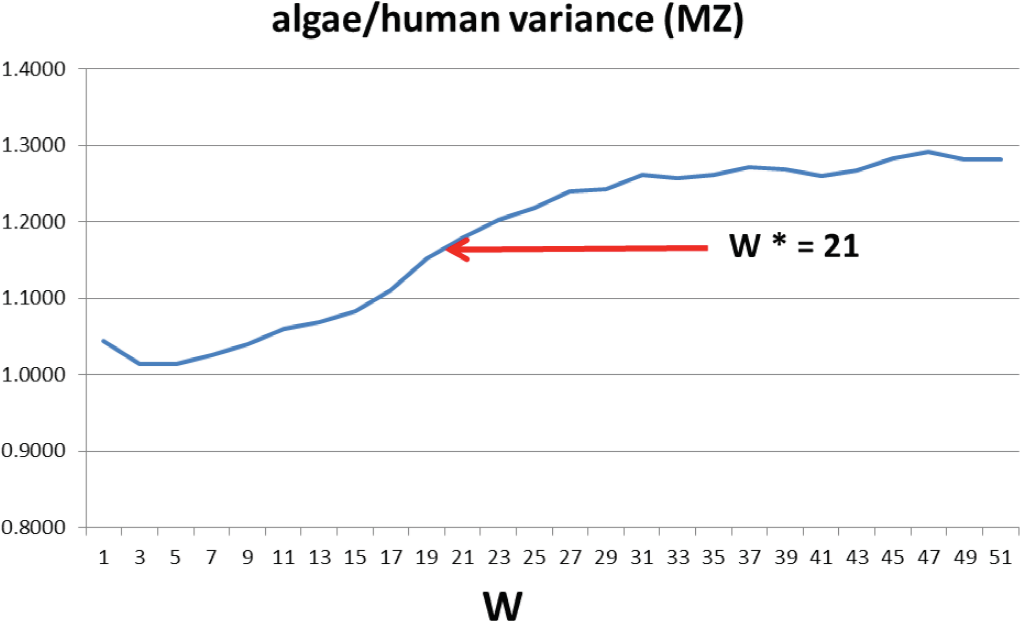
Figura 2. Razones de varianza de la actina en función del ancho W de la ventana móvil promediadora. El máximo en dVr/dW ocurre en W*=21. Secuencia humana P68133, secuencia de algas P53500.
Claramente esta imagen resulta atractiva, sin embargo, por sí misma parece poco convincente. Se puede probar al usarlo para perfilar con Ψ(aa,W*=21) los cambios evolutivos de la actina de las algas a los humanos (Figura 3). Por supuesto, con la conservación del sitio ~ 90%, estos cambios son pequeños, pero son sorprendentemente consistentes con estudios previos de nivelación de extremos hidrofóbicos (pivotes elásticos) [12,21]. Nótese la nivelación de los extremos hidropáticos en la región central 180-280 para humanos en comparación con la actina de algas. El pico correspondiente a un comportamiento hidrofílico cerca del sitio 110 es más profundo en humanos. Finalmente, los puntos extremos de los terminales n y c son ambos más hidrofílicos en la actina humana, lo que facilita la construcción de filamentos y bandas filamentosas más grandes [16,17].
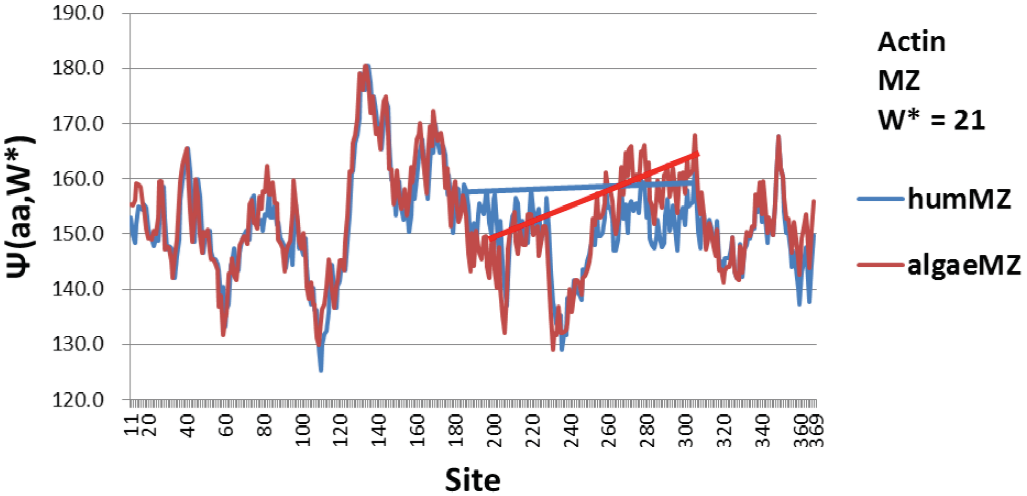
Figura 3. Debido a la fuerte conservación evolutiva, los perfiles de Ψ(aa,W*) para la actina en algas y humanos difieren poco, aunque la naturaleza de las diferencias muestra importantes mejoras darwinianas (evolución positiva) (ver texto). Aquí la hidrofobicidad aumenta con valores crecientes de Ψ(aa,W*), y es hidroneutral en 155.
Esta prueba evolutiva se puede llevar un paso más allá al comparar humanos Ψ(aa, 21) con Ψ(aa, 19) en la región central. Por supuesto, las diferencias son pequeñas, por lo que esta comparación es una prueba severa de la precisión de ambas ventanas móviles y la nivelación pivotal impulsada por la evolución Darwiniana.
Como vemos en la Figura 4 y en su leyenda correspondiente, la nivelación pivotante se mejora con W* = 21.
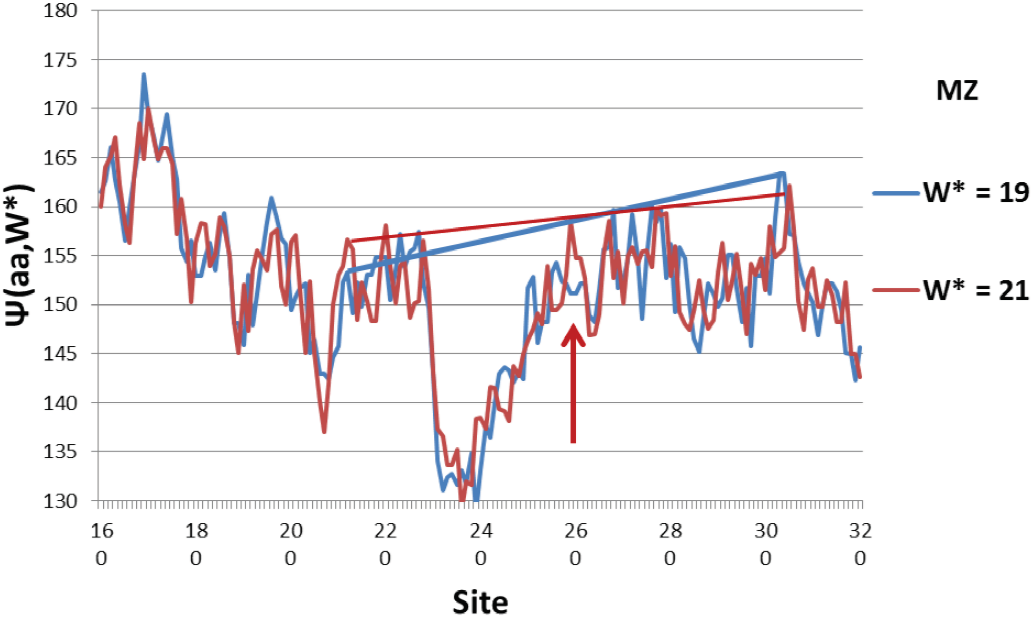
Figura 4. Los perfiles de Ψ(aa,W*) para W*=19 y W*=21 son necesariamente muy similares. En la región central que se muestra aquí (con líneas para guiar el ojo), los máximos hidrofóbicos (pivotes elásticos) están más nivelados con W*=21. Cerca del centro de la región de nivel, el perfil W*=21 tiene un pico de nivel extra cerca 260, que está ausente del perfil W*=19. Notar que el mínimo hidrófilo profundo cerca de 240, que está bien conservado de las algas a los humanos, también aquí se cambia poco. En caso de ser fotografías enviarlas en la calidad nativa de la cámara o del medio en donde fueron capturadas.
Tal nivelación pivotante precisa se puede utilizar para probar la significancia de las escalas alternativas de hidrofília-hidrofóbia. En el período clásico de la biofísica (antes del 2000), se propusieron no menos de 127 escalas de hidrofília-hidrofóbia. Cada escala tenía sus méritos y se basaba en, como mucho, solo unas pocas docenas de mediciones. Pocos intentos se hicieron para comparar sus exactitudes o aplicabilidad a propiedades distintas de las utilizadas en sus definiciones [12]. Las correlaciones entre escalas fueron típicamente ~ 70%. La escala estándar para las tasas de mutación (BLOSUM 62, utilizada en BLAST) exhibe un mínimo hidroneutral profundo en las tasas de mutación cerca de su centro [22]. Con la escala de MZ del 2007, este mínimo se asocia con alanina (A), glicina (G), el aminoácido más pequeño e histidina (H). La Tabla I de [8] muestra que ninguna de las escalas antiguas coloca los tres aminoácidos en su centro. En términos de la raíz de desviaciones cuadráticas medias respecto al valor promedio de cada escala, las diferencias fuera del centro son 7 veces o más mayores para las otras escalas que para la escala MZ. Por mucho, la escala más popular del período clásico es la escala de 1982 basada en la diferencia de entalpía del agua al aire de los péptidos cortos [23]. Esta escala KD ocupa el segundo lugar después de la escala MZ. En otras palabras, los esfuerzos anteriores al 2000 implicados en la construcción de 127 escalas exploraban en una buena dirección, pero las proteínas son tan complejas que el éxito solo fue posible bio-informáticamente después de que las estructuras PDB se hicieron numerosas y más precisas [8].
A la luz de esta perspectiva histórica, las grandes diferencias en los perfiles hidrofílicos de la actina con la escala de MZ de 2007 y la escala de KD de 1982 que se muestran en la Figura 5 no son sorprendentes. Una de las ventajas fundamentales de la escala MZ es que su carácter fractal está asociado con el enfoque evolutivo de las proteínas hacia una funcionalidad óptima (un punto crítico termodinámico [10]). La criticalidad es característica de la funcionalidad de redes neuronales [24-27] y las proteínas en general [28]. Las matemáticas fractales son bien conocidas por los matemáticos, pero su aplicación a las proteínas se ha desarrollado lentamente [29,30]. Tal característica de la evolución a veces ha sido objetivo en el diseño de redes informáticas [31].
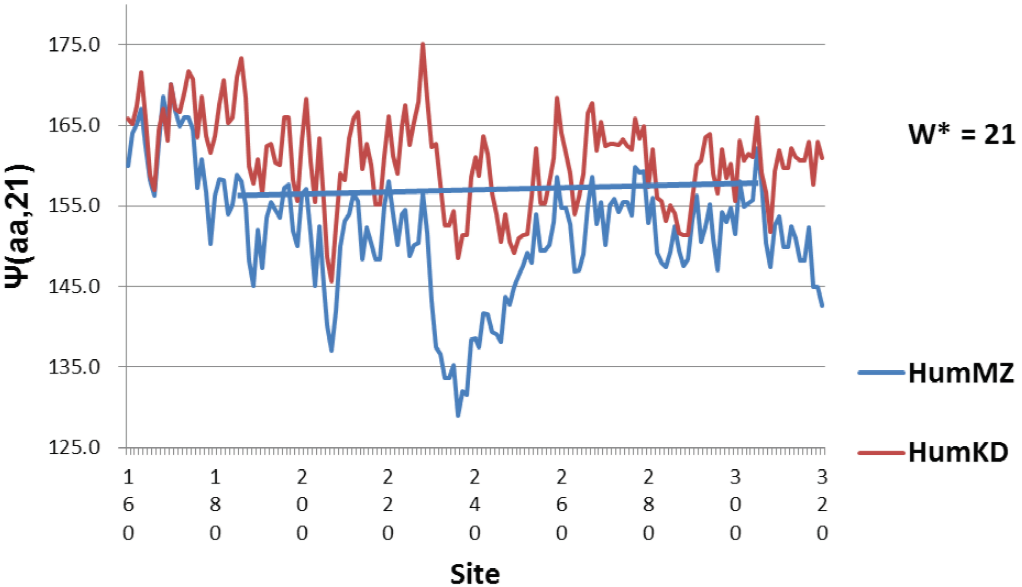
Figura 5. Los perfiles humanos se comparan utilizando la escala KD de 1982 (la mejor de las 127 escalas clásicas) y la moderna escala MZ del 2007. Los detalles más fino de la nivelación de pivote están ausentes del perfil KD. Más importante aún, el perfil de KD ha perdido por completo el mínimo hidrofílico profundo cerca de 240, que está bien conservado de algas a humanos (Figura 3). Esto significa que la evolución Darwiniana se puede reconocer en perfiles hidropáticos solo mediante el uso de la escala MZ.
La similitud de sitios ~ 90% se encuentra no solo para algas / humanos, sino también para levaduras / humanos e incluso levaduras / algas. Los perfiles hidropáticos para los dos últimos pares se muestran en las Figuras 6 y 7. Note la nivelación de dos mínimos hidrofílicos en levaduras. La comparación de levaduras y algas en la Figura 7 enfatiza el segmento hidrofóbico estabilizador de algas 265-281. La similitud de sitios 271-281 es <30%, por lo que estas grandes diferencias localizadas ocurren a pesar del 90% de la similitud general de sitios.
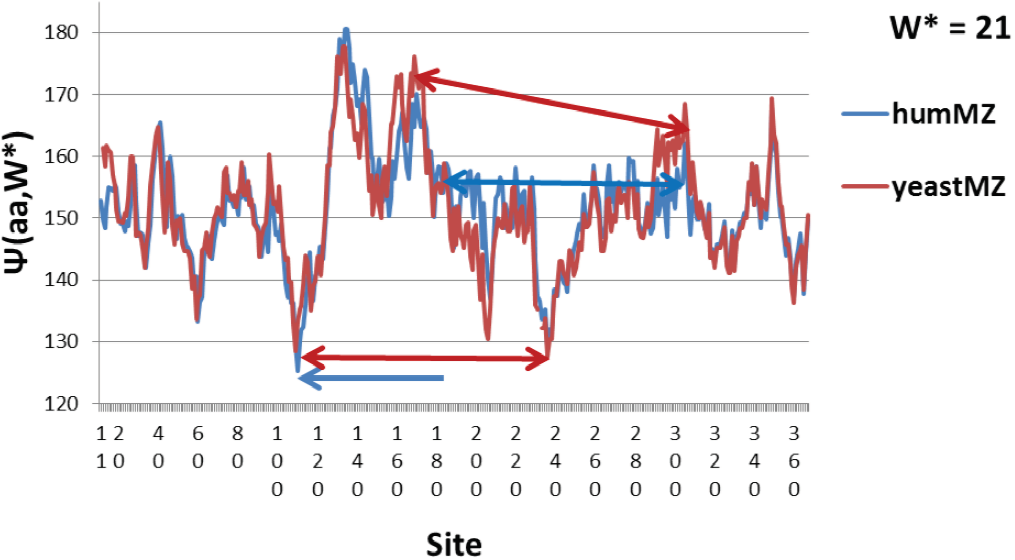
Figura 6. Los perfiles de humanos y levaduras se comparan utilizando la moderna escala MZ de 2007. Las diferencias más importantes están marcadas y discutidas en el texto. Secuencia de levadura P60010.
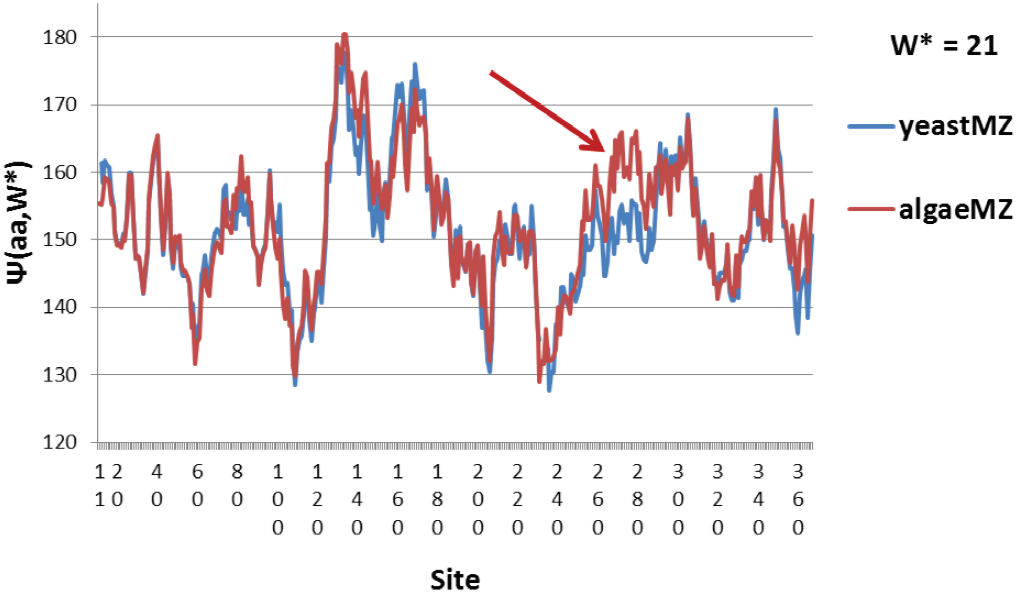
Figura 7. Las algas se estabilizan con relación a la levadura mediante un pico hidrofóbico adicional cerca del sitio 275.
¿Qué sucede cuando aumentamos la similitud a más del 98%? Este es el caso al comparar la actina humana con la del pez cebra. Las razones de varianza R de la Figura 8 poseen características interesantes. Se pudo haber anticipado el extremo en W* = 21, pero ¿qué pasa con los extremos de Vr en M <= 9 y de dR / dW en M> = 35? ¿Por qué los valores de corte en la escala MZ de hidropatía aparecen en Vr para la actina de pez cebra y humano? Una razón es que la actina ha evolucionado de pez cebra a humano al optimizar su hidrostructura para dos estados, el globular tal y como se preparó y los polimerizados funcionales [16,17]. Esto satisface dos condiciones a gran escala y topológicamente opuestas introduciendo correlaciones adicionales más allá de W* = 21, usando exactamente el rango de auto-similitud de MZ para M <≤ 9 ≤ M> = 35. Sin duda, es mucho más que una asombrosa coincidencia.
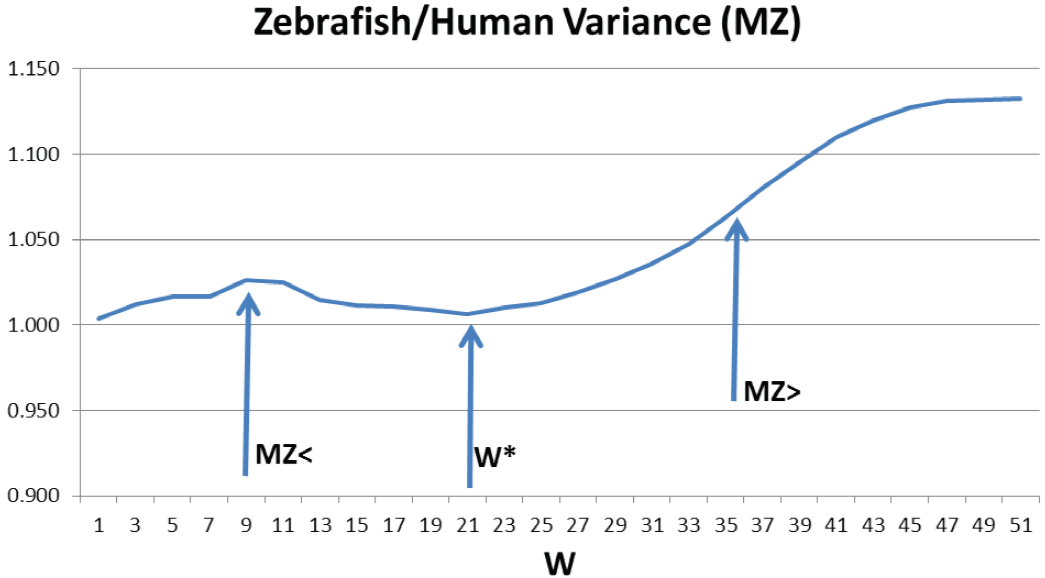
Figura 8. La comparación de las proporciones de varianza Vr del pez cebra y humano muestra un extremo en W*=21. Más aún, la función tiene características analíticas en los límites inferior y superior del rango fractal MZ: un extremo en MZ <, y un máximo en dVr/dW en MZ>. La secuencia de pez cebra es la AAH71401.
En los 377 sitios de aminoácidos de la actina de pez cebra/humano, solo hay 5 mutaciones diferentes. La comparación del perfil W* = 21 en la Figura 9 muestra que estos no son accidentales. Las 5 mutaciones diferentes profundizan tres extremos hidrofílicos cerca de 110, 160 y 290. Estos también son las casi expuestas vueltas en la estructura globular en 110, 168 y 283, PDB 1J6Z [32]. En el pez cebra, los mínimos en 110 y 236 están al mismo nivel, mientras que en la actina humana el mínimo en 110 es más profundo.
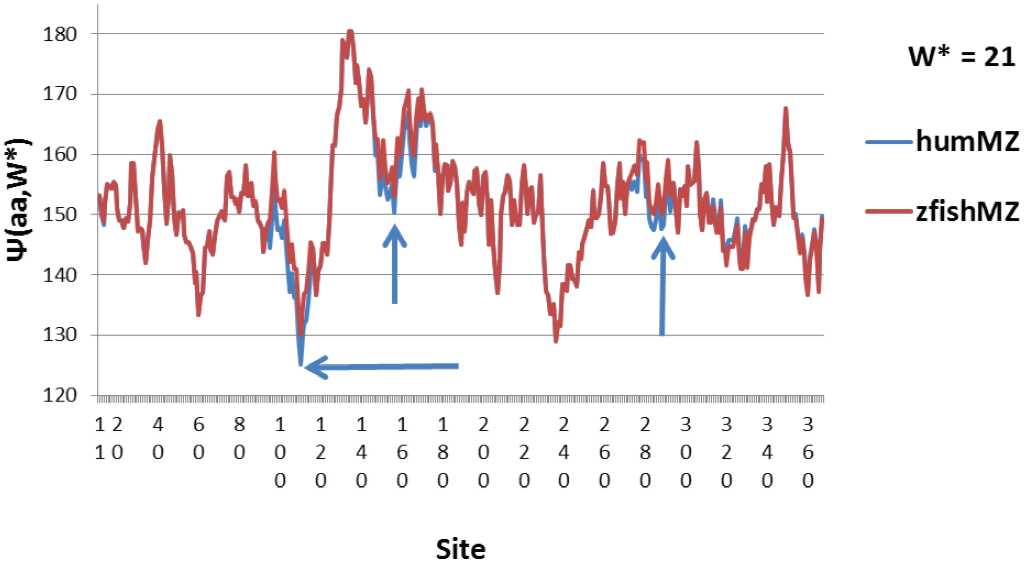
Figura 9. Las pequeñas diferencias entre la actina humana y del pez cebra se concentran cerca de tres extremos hidrofílicos.
La forma hidropática de la actina es inusual porque sus estados duales, a decir, globulares y polimerizados, deben ser estables y funcionar de manera reversible. En la Figura 8 vimos que esta dualidad se refleja en los extremos de Vr en W* = 21 y M <= 9 y un extremo de dVr/dW en W = 35 con la escala MZ. Tal vez tales características analíticas son razonables, dado que la escala MZ en sí misma es fractal y refleja la criticalidad autoorganizada, también rasgo característico del citoesqueleto en la escala celular [33]. ¿Se conserva alguna de esta estructura cuando utilizamos la escala KD [23]? Los resultados que se muestran en la Figura 10 son bastante inesperados. Se esperaría ver una Vr(W) cualitativamente diferente, pero esta función diferente todavía tiene puntos críticos en M <, W* = 21 y M>. Una explicación plausible es que la escala KD se basa en las diferencias de entalpía agua-aire, y estas energías de primer orden están involucradas en la polimerización mediante la unión de los terminales n y c [16,17].
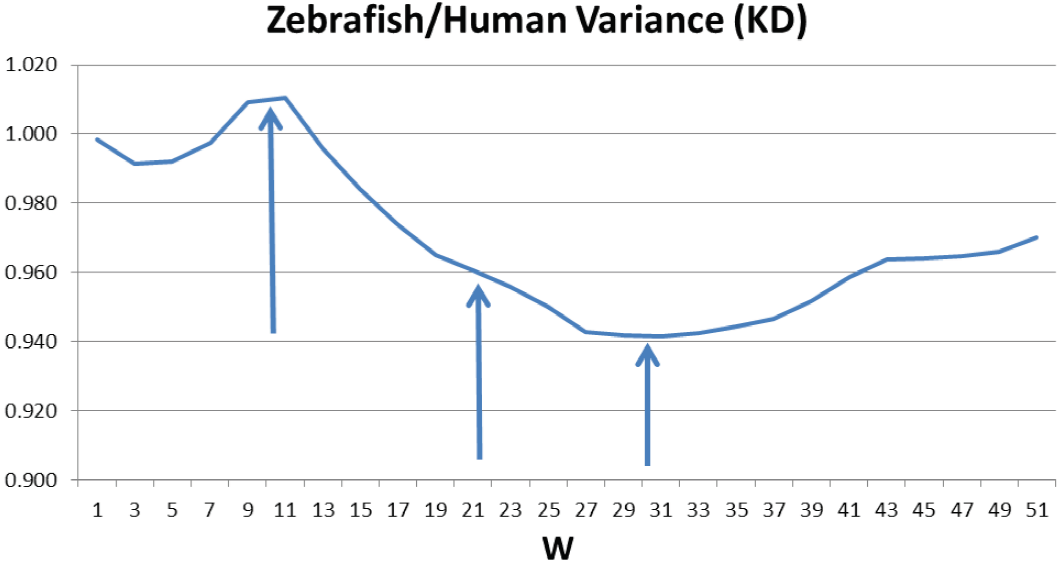
Figura 10. La forma de la relación de varianza Vr(W) se muestra aquí usando la escala KD; es cualitativamente diferente de los resultados mostrados en la Figura 8 usando la escala MZ, pero todavía muestra puntos críticos analíticos cerca de M<(máximo), W*=21 mínimo en dVr/dW y M> (mínimo).
DISCUSION
El descubrimiento de fractales termodinámicos en el SASA de> 5000 segmentos de proteínas [8], junto con promedios modulares utilizando ventanas móviles W, ya ha llevado a la observación de muchas conexiones cercanas entre la secuencia y la función de muchas proteínas, especialmente para proteínas de membrana [12]. La selección darwiniana positiva generalmente ha hecho que sea fácil optimizar W y encontrar W*, estableciendo así la selección Darwiniana de pasada. Aquí hemos encontrado tres valores de MZ >> 1 de W*, lo que sugiere que se han producido dos papeles para la evolución de la actina, una para la actina estabilizada en su forma globular y otra para la polimerización de la actina que estabiliza los citoesqueletos celulares. En general, ninguno de estos resultados evolutivos están presentes cuando W = 1 (por ejemplo, las Figuras 2 y 8), por lo que la filogenia ha sido incapaz de identificar la evolución Darwiniana a nivel molecular [2-7].
El uso de términos como pivotes y bisagras sugiere modelos elastométricos. Estos son intuitivamente atractivos [34,35]. La red creciente de actina se ha modelado como un sistema autoorganizado en criticalidad, en el que los esfuerzos mecánicos de largo alcance que surgen de la interacción con la membrana de plasma proporcionan la presión selectiva que lleva a la organización. La sincronización del citoesqueleto aparece naturalmente como resultado de la criticalidad autoorganizada [35], y se ve facilitada por la nivelación de los pivotes hidropáticos [12]. Este modelo celular se ha reformulado cuantitativamente a nivel molecular evolutivo aquí.
No hemos podido imaginar simulaciones que pudieran derivar estas características críticas de la actina, las cuales involucrarían al menos dos moléculas fusionadas: ~ 750 aminoácidos + agua. Las simulaciones más avanzadas de dinámica molecular en ubiquitina (77 aminoácidos) ahora revelan correlaciones de largo alcance de movimientos consistentes con escalamiento termodinámico [36]. También se puede suponer que la escala fractal universal de las interacciones agua-proteína podría conducir a las características críticas dobles de la actina. Incluso cuando se impone la condición relativamente suave de elasticidad de la columna rígida (por su nombre en inglés, "blackbone elasticity") para la percolación, las fracciones de escala irracionales asociadas con las caminatas aleatorias se reemplazan por fracciones simples [37].











 nueva página del texto (beta)
nueva página del texto (beta)


