INTRODUCCIÓN
En la actualidad, nuevas bases de datos genómicos son puestas al dominio público por distintos centros de investigación y laboratorios alrededor del mundo [1]. Lo anterior genera en la comunidad científica pertenecientes al campo de la biología y biomedicina la necesidad de extraer la mayor cantidad de información posible de estas bases de forma rápida y confiable [2,3,4,5].
El análisis de la información en una base de datos de secuencias de ADN permite la extracción de características a través de las correlaciones existentes entre las secuencias de ADN, sus periodicidades o en sus motif (patrones recurrentes en el ADN [6]), entre otros [7].
En el campo de la bioinformática se han desarrollado diversos algoritmos que resuelven algunas de estas tareas [3, 8]. Sin embargo, la bioinformática presenta ciertas limitaciones para realizar ciertos análisis como es la búsqueda de periodicidad. En este contexto una solución viable es la implementación de herramientas matemáticas propias del procesamiento de señales genómicas [9].
El procesamiento de señales genómicas es un área multidisciplinaria que utiliza herramientas propias del procesamiento digital de señales para extraer información con un significado biológico específico [10,11]
Dentro de este campo, uno de los hallazgos más estudiados e importantes en las regiones codificantes de proteínas es la existencia de un pico en la frecuencia f/3 y la ausencia de este pico en las no codificantes [12, 18].
Sin embargo, las regiones codificantes solo representan el 1% del genoma humano y por varios años el resto del genoma fue considerado ADN “basura” [19, 20]. El proyecto de la Enciclopedia de Elementos del ADN (ENCODE, por sus siglas en inglés) le asignó funciones reguladoras a alrededor del 80% del genoma humano [21,22,23].
Estas funciones controlan la lectura de la transcripción mediante la promoción, potenciación o silenciando genes, entre otras funciones de regulación [24].
En este trabajo, se presenta un análisis de los momentos estadísticos de los espectros en frecuencia de las regiones reguladoras propuestas por Ernst et al [23] que forman parte de la base de datos del ENCODE. La hipótesis de este trabajo es que existen diferencias estadísticas entre los espectros de frecuencia de las 15 regiones reguladoras y que probablemente existan picos de frecuencia característicos como es el caso de las regiones codificantes.
METODOLOGÍA
La metodología que se siguió para el desarrollo de ese trabajo se encuentra esquematizada en la Figura 1.
El primer paso que se realizó fue la elección de los datos. La base de datos que se seleccionó corresponde a los estados de cromatina propuestos por Ernst et al [23], la cual se puede consultar en https://genome.ucsc.edu/cgi-bin/hgFileUi?db=hg19&g=wgEncodeBroadHmm (Junio 2011).
Se descargaron los archivos correspondientes a las 15 regiones reguladoras (RRs) (ver Tabla 1) en los 22 autosomas y el cromosoma X en las nueve líneas celulares (CLs) disponibles en la base de datos (ver Tabla 2).
De acuerdo a la Figura 1, luego de seleccionar una base de datos, el siguiente paso consistió en “mapear” las secuencias. Las secuencias de las RRs fueron extraídas de la construcción del genoma de http://hgdownload.cse.ucsc.edu/goldenPath/hg19/encodeDCC/referenceSequences/ y posteriormente fueron mapeadas con la técnica de Neighbor Joining propuesta por Borrayo et al [25].
Se entiende por mapeo el proceso de trasladar la información contenida en las secuencias de ADN (secuencias ordenadas de los nucleótidos, representados por las letras A, T, C, G) a una representación numérica (señal genómica) con la menor pérdida de información [7]. El mapeo de Neighbor Joining [25] consiste en dos etapas, la primera es explicada por la Ecuación 1:
para i = 1,2,3,…, l donde l es la longitud de la secuen cia de ADN y Si ∈ {A,T,C,G} que a su vez representan los nucleótidos adenina (A), timina (T), citosina (C), gua nina (G); Ŝi ∈ {x1,x2,…,x16} y los valores de x1,2,…,16 son enteros equidistantes, el nuevo vector Ŝ tiene una longitud de l - 1.
La segunda etapa del mapeo consiste en realizar un ventaneo del vector Ŝ y es calculada por la Ecuación 2:
donde Ŝ es el vector resultado de la Ecuación 1,
Una vez mapeadas las secuencias a señales genómicas, se procedió a calcular la transformada de Fourier de cada una de ellas. La transformada de Fourier (ver Ecuación 3) es una herramienta matemática que permite calcular la periodicidad de una señal [9].
A continuación, agrupamos los espectros de las señales genómicas en 3105 conjuntos donde cada conjunto representa las señales genómicas que cumplen una de las RRs de las enlistadas en la Tabla 1, pertenecen a una LC de las presentadas en la Tabla 2 y a uno de los 23 cromosomas analizados.
La magnitud de cada coeficiente de Fourier (valor absoluto) fue calculada para obtener números reales y reducir la complejidad del análisis. Después se realizó una normalización de los datos con el objetivo de realizar una comparación de los mismos sin que la cantidad de puntos pudieran producir ruidos o valores atípicos. Los datos fueron normalizados entonces en función de 3 criterios: la longitud, la energía y la banda de frecuencia:
Normalización por longitud: consiste en interpolar lineal cada espectro de Fourier a un valor de 800 puntos que representa la mediana de tamaño del conjunto total de señales genómicas del estudio.
Normalización por energía: consiste en dividir el espectro normalizado por longitud entre el valor del tamaño original del espectro.
Normalización por banda de frecuencia: recordando que las señales genómicas fueron agrupadas en conjuntos que pertenecen a la misma RR, CL y cromosoma. Para cada conjunto las bandas de frecuencia (800) son divididas por el valor máximo de la banda evaluada.
Luego de realizar las normalizaciones pertinentes, se procedió de acuerdo al paso 3 de la Figura 1 al cálculo de momentos estadísticos. Sobre los espectros de frecuencia se calcularon los cuatro primeros momentos centrales (media, desviación estándar, asimetría y curtosis), y dos momentos estadísticos más (mediana y varianza).
La media es el primer momento estadístico central y es considerada el valor representativo de la distribución de coeficientes el cual puede ser interpretado como el valor medio de energía, donde un valor mayor representa coeficientes con una organización posiblemente definida y no aleatoria. La mediana se refiere al valor medio de energía de la distribución y puede ser semejante a la media si es una distribución normal.
La desviación estándar y varianza son medidas estadísticas de dispersión y que nos describen la tendencia a mantener el orden (consideramos orden a la cualidad de que existan picos frecuenciales que sobresalen sobre el resto). Una baja desviación estándar, es decir dispersión baja, corresponde a la posible presencia de picos frecuenciales distinguibles.
La asimetría es el tercer momento estadístico central y nos describe las colas en una distribución lo que está relacionado con la existencia de valores atípicos. La curtosis describe la persistencia a concentrarse los datos de la distribución en el valor medio de energía, lo cuál reforzaría la idea de la presencia de picos frecuenciales representativos en los espectros. Las ecuaciones de los momentos estadísticos descritos se encuentran en la Tabla 3.
RESULTADOS Y DISCUSIÓN
El objetivo de este trabajo es encontrar las diferencias estadísticas entre los espectros de frecuencia de las 15 regiones reguladoras. Para conseguir esto se calcularon seis momentos estadísticos: media (
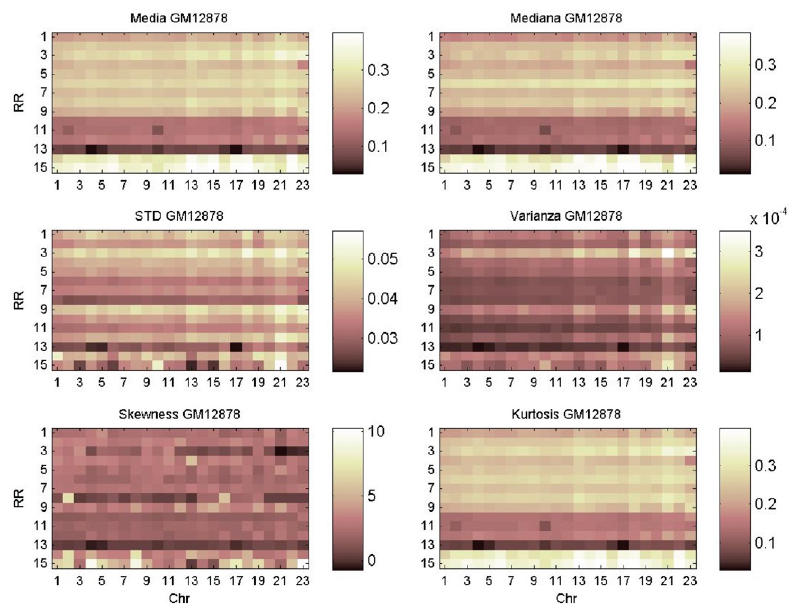
En la Figura 2 se presentan los momentos estadísticos de la LC GM12878 en todas las RRs y cromosomas. Se puede observar la existencia de 3 grupos de regiones reguladoras en la media, mediana y curtosis. Esto es interpretado como grupos de RRs con secuencias con un grado de orden (tendencia a estar ordenados y no dispuestos aleatorios) determinado.
El primer grupo está conformado por las RRs Repetitive I y Repetitive II interpretado como RRs altamente ordenadas. Por el contrario el grupo de RRs con bajo orden o alta aleatoriedad está conformado por las RRs Transcriptional Elongation, Weak Transcribed, Polycomb-repressed, Heterochromatin. Esta observación es congruente con el principio de mínimo esfuerzo donde secuencias largas, como lo son las pertenecientes a estas RRs, implican un gasto energético muy alto.
El resto de RRs están medianamente ordenadas (refiriéndonos a las secuencias de ADN).
Con respecto a la desviación estándar y la varianza, visualmente parecen no contener información relevante. Sin embargo, observando el rango en las que oscilan (0 - 0.05), podemos inferir que el orden, ya sea alto, medio o bajo, es preservado debido a que se puede considerar ambos valores de los momentos estadísticos como bajos. El asimetría nos muestra un comportamiento homogéneo en la presencia de valores atípicos en las primeras 13 RRs. Las Repetitive I y II tienen valores de asimetría mayores, es decir, que no todas las secuencias de ADN pertenecientes a estas RRs son altamente ordenadas. Este comportamiento se puede observar a través de las otras ocho LCs (ver figuras suplementarias S24-S31*).
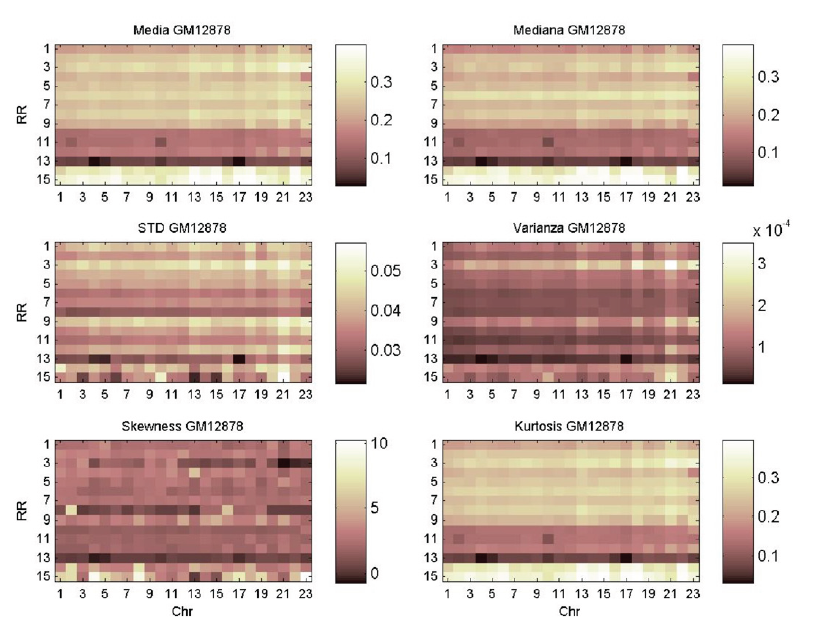
El comportamiento descrito anteriormente puede ser observado en la Figura 3, donde se analizan los datos pertenecientes a un cromosoma a través de todas las RRs y las LCs. El resto de los cromosomas se pueden ver en las figuras suplementarias S1-S23*. En todas ellas se refuerza la observación descrita.
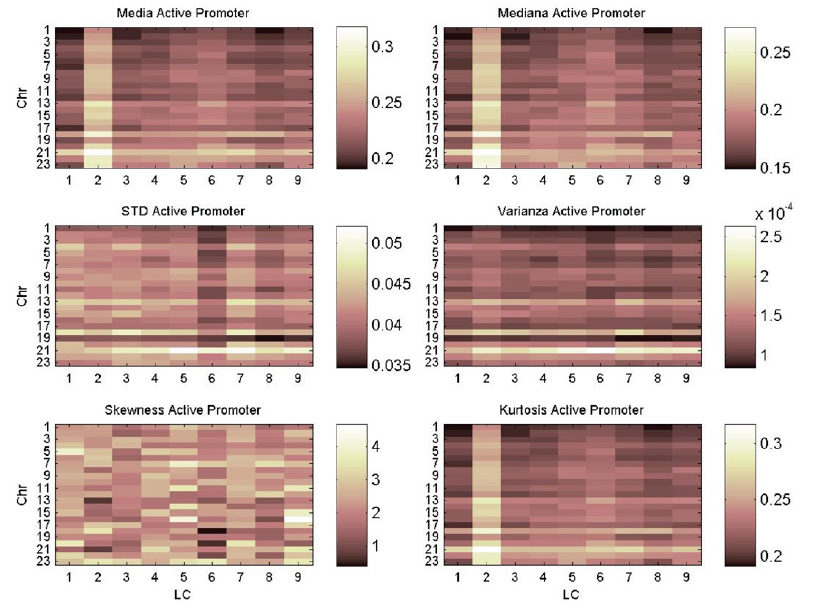
Analizando una única RR en todas las LCs y cromosomas, por ejemplo Active Promoter (ver Figura 4 y Figura 5) se puede observar que un comportamiento en la LC 2 (H1esc) es distinto al resto de las LCs. Las secuencias que son Active Promoter son las encargadas de promover la expresión de genes. Por el contrario, las secuencias con la función de Poised Promoter se encargan de reprimir o silenciar la expresión de los genes. Estas características son descritas en [26,27,28] y corresponden a las de las células madres.
Los valores altos en el caso de la RR Active Promoter en la media, en la mediana y en las curtosis son significativos, porque los promotores representan secuencias altamente ordenadas y, probablemente, accesadas continuamente.
En el caso contrario, de los bajos valores de los mismos momentos en la RR Poised Promoter se puede inferir que son altamente desordenadas. Esto es relevante porque son secuencias no necesarias en las células madres. Revisitando la idea de que una célula madre expresa la mayoría de los genes y silencia pocos genes, caso contrario de células diferenciadas como las otras 8 LCs, que expresa una cantidad pequeña de genes y silencia una gran cantidad [26,27,28].
El comportamiento de los momentos estadísticos de nuestro trabajo y la descripción dada por la literatura es relevante porque conecta el comportamiento frecuencial de las secuencias con características biológicas no determinadas con esta metodología lo cual da un argumento a favor para el uso del procesamiento de señales como una herramienta para análisis de datos genómicos en la disciplina del procesamiento de señales genómicas.
Como una línea de investigación futura a este trabajo puede implementarse una etapa de clasificación o clustering extrayendo una cantidad mayor de información que está contenida dentro de los espectros de frecuencia y no puede ser abordada solamente con índices estadísticos.
CONCLUSIONES
El análisis de datos genómicos es crucial dado que la comunidad científica deja al alcance del dominio público cada vez más bases de datos. Sin embargo poca información es extraída de estas. El procesamiento de señales genómicas es una disciplina poco explorada hasta el momento, aunque ha dado resultados interesantes como el descubrimiento de la frecuencia f/3 en la secuencias codificantes.
En este trabajo, hemos implementado el procesamiento de señales genómicas para analizar estadísticamente las frecuencias de secuencias no codificantes con 15 funciones biológicas específicas.
Los resultados obtenidos en este trabajo reflejan que los espectros de frecuencia de las señales son distintos entre funciones biológicas y que ciertas características biológicas, como en el caso de la línea celular H1esc, se pueden analizar con esta metodología.
Los resultados de este trabajo nos han permitido comprobar que el procesamiento de señales genómicas es una herramienta poderosa para la extracción de características. Además, y da pauta a que más herramientas nativas del procesamiento de señales puedan ser adoptadas a las señales genómicas para obtener información biológica relevante y que aún permanece oculta con los métodos tradicionales de bioinformática y genética.











 nueva página del texto (beta)
nueva página del texto (beta)