Introducción
La digoxina es un glucósido cardiotónico perteneciente a los digitálicos, compuestos amplia-mente utilizados como fármacos para el tratamiento de insuficiencia cardíaca congestiva y taquiarritmia auricular [1]. La digoxina actúa como un inhibidor de la actividad enzimática de la Na+/K+-ATFasa en cardiomiocitos [2]. El corazón genera una respuesta fisiológica como consecuencia de esta inhibición: se aumenta la fuerza máxima de contracción muscular y la velocidad de llenado ventricular, efecto conocido como inotrópico positivo; y se reduce la frecuencia cardíaca, efecto conocido como cronotrópico negativo.
En la actualidad, las enfermedades cardiovasculares son la principal causa de defunción en todo el mundo, de acuerdo con la Organización Mundial de la Salud. Para combatir estos padecimientos, una de las propuestas es el desarrollo de nuevos fármacos que deben ser probados en organismos modelos antes de ser utilizados en humanos. La elección del organismo modelo a utilizar dependerá del estudio que se hará, en este trabajo se utilizó el corazón de T. stultorum debido a que presenta un bajo costo de adquisición, es fácil de manipular y tiene una respuesta uniforme [3,4]. Al igual que en corazones humanos, la superficie endocárdica de corazones de moluscos es similar a los músculos suaves de mamíferos, además de que su frecuencia cardíaca puede llegar a valores comparables a los de humanos (60 latidos por minuto), este valor puede ser modificado por factores de estrés o compuestos químicos como fármacos [5,6].
Un modelo in silico que prediga la respuesta fisiológica que se obtiene al administrar una cantidad de fármaco podría complementar a los modelos in vivo; de esta manera se reduciría el sacrificio de animales, así como los experimentos en laboratorio. Por esta razón, la predicción de la cardioactividad específica que se obtiene al administrar una cantidad de digoxina puede ser de gran interés en el campo de la farmacología, así como en el de biosimulación.
Las redes neuronales artificiales (RNA) son un método computacional cuyo diseño y funcionamiento está basado en el comportamiento de las neuronas biológicas del cerebro humano. Éstas se han utilizado ampliamente en aplicaciones enfocadas a modelado de sistemas biológicos complejos, debido a su capacidad de reconocer patrones y relaciones de tipo no lineal, procesar variables complejas y modelar funciones de predicción [7,8]. Estas redes están formadas por varias unidades elementales llamadas neuronas, interconectadas entre sí, donde cada una tiene un peso. El perceptrón es un tipo de RNA, aunque también puede entenderse como la unidad básica del mismo; éste suma todas las señales de entrada y las multiplica por los pesos previamente inicializados de manera aleatoria. Mediante un proceso de entrenamiento, el perceptrón es capaz de distinguir patrones de entrada complejos [8].
Las RNA de topología perceptrón multicapa (PMC) son un método que ha demostrado un buen funcionamiento en el área del modelado farmacocinético y farmacodinámico debido a su capacidad de simular sistemas no lineales y procesar variables fisiológicas dependientes e independientes [7,9]. Éstas redes han sido ampliamente utilizadas en sistemas complejos que requieren predicción, sistema de soporte de decisiones, pacientes tratados con digoxina, entre otros [10-13].
En el presente trabajo se analizaron los datos de experimentos in vivo de la respuesta fisiológica de corazones de T. stultorum. Posteriormente se construyeron diversos modelos de RNA que fueron entrenados con los datos de los experimentos in vivo para predecir la actividad cardíaca de la almeja T. stultorum después de agregar una cantidad específica de digoxina. El objetivo de este trabajo es desarrollar un modelo predictivo que describa la relación farmacodinámica entre la concentración de digoxina y la respuesta fisiológica del ventrículo de T. stultorum.
Metodología
Conjunto de datos
Las lecturas de la actividad cardíaca de 25 ventrículos de T. stultorum fueron proporcionadas por el Laboratorio de Farmacología Marina, contando con un conjunto de datos de 4, 113 × 10, en donde se encontraba la fuerza de contracción del músculo cardíaco respecto con el tiempo, haciendo una distinción en el tiempo antes y después de agregar una cantidad de digoxina. Las diez variables utilizadas antes de la adición de la digoxina fueron: máximo y mínimo de contracción, tiempo de llenado ventricular, frecuencia cardíaca antes de la digoxina, peso, volumen, largo y ancho del corazón, concentración de digoxina y volumen utilizado para la dilución de digoxina; mientras que para las cuatro salidas se consideraron el máximo y mínimo de contracción, tiempo de llenado y frecuencia cardíaca, después de agregar la digoxina.
Preparación de la información
Las lecturas contenían más de un millón de datos en crudo correspondientes a los 25 corazones, obtenidos utilizando un transductor de fuerza Thornton tipo 420. La configuración del sistema se describe en [4]. Cada máximo local se tomó como un máximo de fuerza de contracción y cada mínimo local, como un mínimo de fuerza de contracción; el tiempo de llenado se tomó como el tiempo que tarda la cámara ventricular en llenarse de fluido sanguíneo y vaciarse, esto es, el tiempo que transcurre entre un latido y otro; y la frecuencia cardíaca se tomó como la cantidad de latidos en un minuto. El resto de las variables de entrada fueron proporcionadas de los experimentos in vivo. Las RNA fueron alimentadas con los 4, 113 datos de entrada extraídos de las 25 lecturas de la actividad cardíaca, estandarizados a valores de z (media = 0, desviación estándar = 1). Los datos de salida fueron normalizados de una forma centralizada en el rango [−1, 1].
Modelo predictivo basado en RNA
Se introdujeron diez variables como entradas de las RNA, y cuatro variables de salida. Se utilizó la tangente hiperbólica como función de activación, es decir, la función que genera la salida [8]. Esta es una función sigmoide que ayuda a que las RNA aprendan más rápido en relación con otras funciones [14], se utiliza cuando la normalización de los datos va de −1 a 1.
La topología de PMC que se utilizó se describe como 10 − N − 4, en donde 10 y 4 son el número de entradas y salidas, respectivamente, y N es el número de neuronas en la capa oculta del PMC. N es un número par desde 2 hasta 30.
Para validar las RNA se utilizó el método de submuestreo aleatorio, un tipo de validación cruzada que consiste en dividir de manera aleatoria los datos en subconjuntos para entrenar y probar las redes; diferentes para cada iteración [15]. A su vez se utilizó un método conocido como validación hold-out [15] para el cual se utilizó el criterio de parada early-stopping, que consiste en detener el entrenamiento de la red cuando el error del subconjunto de validación deja de disminuir y comienza a aumentar [15-17]. Para esta validación, el conjunto de datos se dividió en tres subconjuntos: 50 % para el entrenamiento, 25 % para validación, y el 25 % restante para probar el desempeño de la red. Cuando no se utilizó la validación hold-out, las redes se entrenaron hasta llegar a 100 épocas utilizando el 75 % de los datos, mientras que el 25 % restante se utilizó para probar la red. Para entrenar a las RNA-PMC se utilizaron los algoritmos de entrenamiento de Levenberg-Marquardt (LM), Regulación Bayesiana (RB), Propagación Resiliente (PR) y Gradiente Conjugado Escalado (GCE), versiones modificadas del algoritmo de retropropagación [8]. Cada arquitectura de red diferente se corrió 20 veces con y sin validación hold-out.
Para evaluar el desempeño y la precisión de la predicción del modelo se utilizaron los parámetros de error medio cuadrado (EMC), raíz del error medio cuadrado (REMC), y el coeficiente de regresión R 2; estos parámetros fueron un promedio de las 20 corridas para cada arquitectura, con el fin de reducir cualquier sesgo sobre la selección aleatoria de los datos [14]. El entrenamiento se optimizó en relación con el criterio donde se asume que mientras más bajo sea el EMC, el modelo simula mejor.
Evaluación de las contribuciones relativas
Este método consiste en calcular la magnitud de la contribución de variable de entrada sobre la salida [14]. Para obtener los valores de las contribuciones se usan los pesos de las neuronas y se asocian con las variables de entrada [10]. Las variables con una importancia relativa baja pueden ser omitidas del modelo sin tener una pérdida significativa del desempeño del mismo, resultando en redes con menor número de variables de entrada, reduciendo así el tiempo de procesamiento necesario para resolver la red [15].
Resultados y discusión
En la Tabla 1 se muestran los valores de EMC, REMC y los coeficientes R 2 para cada variable de salida de las mejores configuraciones de perceptrones. La configuración con el rendimiento más bajo fue la del algoritmo PR con 28 neuronas sin validación hold-out, obteniendo un EMC de 0.051440; por otro lado, la mejor configuración fue obtenida a través del algoritmo RB con 24 neuronas y validación hold-out, obteniendo un EMC de 0.002143 y coeficientes de R 2 por encima de 0,97 a excepción del tiempo de llenado del corazón, donde se obtuvo un valor de 0,5442, el cual muestra que la relación del tiempo de llenado con las entradas de las redes es baja en comparación con la frecuencia cardíaca y el máximo y mínimo de contracción.
Tabla 1: Algoritmos de entrenamiento y parámetros de desempeño para el conjunto de datos evaluados en las mejores redes neuronales artificiales.
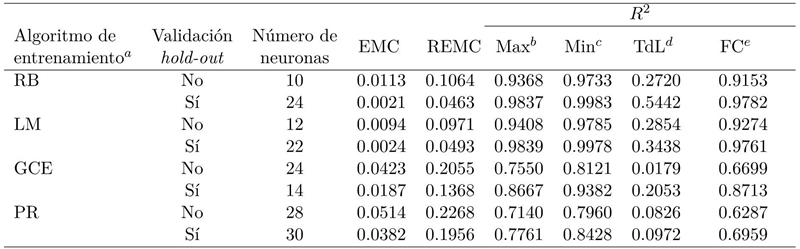
aRegulación Bayesiana (RB), Levenberg-Marquardt (LM), Gradiente Conjugado Escalado (GCE) y Propagación Resiliente (PR); bMáximo nivel de contracción ventricular; cMínimo nivel de contracción ventricular; dTiempo de llenado del corazón y eFrecuencia cardíaca.
El desempeño más alto de las redes sin validación hold-out se vio dividida entre los algoritmos RB y LM en relación con GCE y PR como se muestra en la Figura 1, este comportamiento muestra que los algoritmos con menor tiempo de procesamiento consiguen valores de EMC altos, mientras que los algoritmos de mayor tiempo obtienen EMC bajos. De igual forma se presenta un comportamiento similar a las configuraciones con validación hold-out (Figura 2), a excepción de las últimas configuraciones de LM y RB; ya que, a partir de las 20 neuronas en la capa oculta, se tiene un aumento del EMC, entendiéndose como un sobre entrenamiento de la red, provocando una pobre predicción de los valores de salida.

Figura 1: Valores promedios del error medio cuadrado de las 20 corridas de los 4 algoritmos de entrenamiento sin validación hold-out de acuerdo con el número de nodos en la capa oculta.

Figura 2: Valores promedios del error medio cuadrado de las 20 corridas de los 4 algoritmos de entrenamiento con validación hold-out de acuerdo con el número de nodos en la capa oculta.
En la Tabla 2 se muestran los resultados del análisis de las contribuciones relativas de las mejores seis redes determinadas a partir de la Tabla 1; dicho análisis determina la influencia de las variables de entrada respecto con las de salida. Los valores mostrados se obtuvieron del promedio de 20 iteraciones de cada configuración independientes a las usadas para determinar el desempeño de las redes. Estas contribuciones muestran una distribución equilibrada en su mayoría, lo que determina que las variables de entrada en el modelo no pueden ser omitidas del mismo. Al evaluar las contribuciones relativas se encontró que todas las variables de entrada tienen una contribución similar una con otra en relación con las variables de salida; esto es de esperarse, ya que la digoxina afecta directamente a la actividad cardíaca, y el perfil farmacocinético del corazón depende en gran medida de sus parámetros físicos.
Tabla 2: Importancia relativa ( %) de las variables de entrada*.

*Los valores mostrados son los promedios de 20 corridas independientes a las usadas para determinar el desempeño de las redes.
aRegulación Bayesiana (RB), Levenberg-Marquardt (LM), Propagación Resiliente (PR) y Gradiente Conjugado Escalado (GCE).
bMáximo de fuerza de contracción (MaFC), mínimo de fuerza de contracción (MiFC), tiempo de llenado (TL), frecuencia cardíaca (FC), volumen del corazón (VolC), largo del corazón (Lar), ancho del corazón (Anc), volumen de la dilución de digoxina (VolD) y concentración de digoxina (ConcD).
El parámetro del EMC de las 20 corridas de las configuraciones que se obtuvieron se acercaron al cero. Para el algoritmo RB se observa un máximo de EMC cuando el número de neuronas en la capa oculta aumenta, este fenómeno para ese algoritmo concuerda con otros trabajos [14-17], y puede deberse a que una alta cantidad de neuronas aumenta la complejidad del modelo innecesariamente y reduce su capacidad de generalización. Para la variable de tiempo de llenado los valores obtenidos fueron menores a 0.54, lo que sugiere que para esta variable, las entradas no están altamente relacionadas; esto puede deberse a que en los experimentos in vivo, la digoxina no afectó significativamente al tiempo de llenado ventricular, por lo que no existe una diferencia significativa entre éste parámetro antes y después de la adición del fármaco. La capacidad tan alta de generalización obtenida con las RNA se debe en gran medida al preprocesado de los datos, la estandarización de las entradas y la normalización de las salidas.
El algoritmo de entrenamiento PR con y sin validación hold-out, muestra un rendimiento considerablemente menor que los otros tres, tomando en cuenta los parámetros EMC y R 2, lo cual también es consistente con otros trabajos [14-15]. Sin embargo, el tiempo computacional requerido para el algoritmo PR fue el menor de los cuatro utilizados.
Para el modelo con la capacidad de predicción más alta (RB con 24 neuronas y validación hold-out), los coeficientes R 2 cercanos a la unidad indican una correlación muy fuerte entre la cantidad de fármaco añadido y la respuesta farmacodinámica del corazón reflejada en las variables fisiológicas de tiempo de llenado ventricular, frecuencia cardíaca, y máximo y mínimo de contracción ventricular.
Conclusiones
Debido a la relación farmacológica entre las variables de entrada estudiadas y los parámetros de la actividad cardíaca de salida, las entradas influyeron significativamente en éstas, a excepción del tiempo de llenado del corazón, que tuvo una correlación con las entradas considerablemente más baja que las otras salidas. Esta relación consiste en que la digoxina aumenta la fuerza máxima de contracción muscular, mientras que reduce la frecuencia cardíaca y el tiempo de llenado. Una de las variables de entrada que mostró una importancia de interés fue la concentración de digoxina como se muestra en la Tabla 2.
Las RNA-PMC son un método computacional eficiente para predicciones en el área de la farmacología cuando se entrenan con el algoritmo adecuado. Para este trabajo, el mejor algoritmo fue RB, seguido de LM.
La predicción de la actividad cardíaca de corazones de T. stultorum es difícil debido a la cantidad de factores que influyen en el fenómeno; sin embargo, esta predicción fue posible utilizando un modelo de RNA-PMC con la configuración adecuada. Se lograron obtener varios modelos con una alta capacidad de predicción de los parámetros biofísicos de corazones de T. stultorum (Tabla 1). Los modelos obtenidos presentaron una pobre eficiencia en el tiempo de llenado del corazón, como se describió anteriormente. Dichos modelos obtenidos pueden ser de ayuda en el estudio de los efectos de fármacos haciendo pruebas antes de suministrarse en humanos. Como trabajo futuro, se pueden utilizar métodos numéricos para reconstruir la señal de la actividad cardíaca a partir de los parámetros predichos.











 nova página do texto(beta)
nova página do texto(beta)


