Introducción
Tumores cerebrales
Los tumores cerebrales se originan a partir de las células gliales y pueden ser clasificados de acuerdo a su origen o grado de agresividad. Los tumores que inician en el cerebro se conocen como tumores cerebrales primarios, mientras que los tumores que se originan en otras partes del cuerpo se denominan metástasis. Los tumores cerebrales pueden dañar otras células de tejido cerebral sano debido a la inflamación que producen y por la presión que ejercen en otras partes del cerebro. Un tumor cerebral benigno se encuentra conformado por células anormales de crecimiento lento, que generalmente tienen bordes definidos y rara vez se propagan. Cuando se observa este tipo de tumor en el microscopio, las células tienen un aspecto casi normal. La cirugía por sí sola puede ser un tratamiento eficaz, sin embargo, si el tumor se encuentra en un área vital se clasifica como maligno a pesar de contar con las características anteriores. Por otra parte, un tumor maligno generalmente es de crecimiento rápido, invasivo y potencialmente mortal. No obstante, rara vez se propaga fuera del cerebro y la médula espinal [1].
Los gliomas son los tumores cerebrales primarios más frecuentes y la Organización Mundial de la Salud (World Health Organization) los clasifica en cuatro grados: I, II, III y IV [2]. Los tumores de grado I y II, es decir, aquellos de bajo grado (BG) y que corresponden a astrocitomas y oligodendrogliomas, pueden ser considerados tumores semi-malignos por presentar un mejor pronóstico en la salud del paciente. Por otro lado, los tumores de grado III y IV son gliomas de alto grado (AG), y corresponden a astrocitomas anaplásicos y a glioblastomas multiformes (GBM), los cuales son tumores malignos que tienen altas probabilidades de ocasionar la muerte del paciente [3]. Los gliomas grado IV (GBM) son de los más agresivos y permanentes. La forma en la que invaden es muy diversa y la mejor manera de atenderlos clínicamente es mediante la extracción completa a través de cirugía, sin embargo esto suele provocar una recurrencia en tumores y puede conducir a la muerte del pacientes entre 15 y 21 meses después de la cirugía [4]. A pesar de los recientes avances en Neuro-Oncología, diagnosticar pacientes con gliomas de AG sigue siendo complicado. Por lo tanto, existe la motivación de utilizar técnicas de procesamiento de imágenes médicas y reconocimiento de patrones para mejorar la definición en la segmentación de la región afectada, y así poder obtener un diagnóstico y plan de tratamiento más adecuado para el paciente [5].
Imágenes de resonancia magnética
La imagenología médica ha sido utilizada como herramienta de diagnóstico por más de 30 años en un amplio rango de aplicaciones médicas. Las imágenes de resonancia magnética (IRM) son una técnica no invasiva que permite la visualización de estructuras internas del cuerpo [6].
En particular, las IRM son actualmente una técnica estándar en el diagnóstico de tumores cerebrales, siendo necesaria la adquisición de diferentes secuencias de IRM para poder segmentar en su totalidad el tumor cerebral, incluyendo todas las subregiones del mismo. Dentro de estas secuencias se incluyen: a) IRM T1-ponderada (T1), la cual nos permite realizar un análisis estructural y verificar con cierta facilidad tejido sano; b) IRM T1-ponderada con realce de contraste (T1c), en este tipo de secuencia los bordes del tumor se muestran en la imagen hiperintensos, por lo que pueden diferenciarse fácilmente la región de necrosis y la parte activa de un tumor; c) IRM T2-ponderada (T2), en esta secuencia se puede observar como una región intensa la extensión del edema que rodea al tumor; así como d) IRM T2 con recuperación de la inversión atenuada de fluidos (T2FLAIR), la cual permite separar la región del edema del fluido cerebro-espinal debido a que suprime la señal producida por presencia de agua libre [2].
Ahora bien, el caracterizar de manera correcta un glioma es crucial para poder dar un buen diagnóstico y para que el experto clínico pueda decidir sobre el tratamiento más adecuado, lo cual podría ampliar la expectativa de vida de los pacientes. Las IRM nos permiten observar la presencia de gliomas, y sus subregiones, a partir de las cuales podemos obtener información morfológica y volumétrica de cualquier glioma, para posteriormente poder correlacionar esta información con el grado de agresividad o malignidad de los mismos. Así mismo, a partir de las IRM se puede detectar y analizar la extensión de regiones de necrosis, lo cual es muy característico en gliomas de AG [7].
Antecedentes
El análisis sobre la relación que puede existir entre la morfología de un tumor, segmentado a partir de una imagen digital, y su posible grado de malignidad ha sido un área de gran interés en la investigación científica. Este interés de estudiar y analizar la forma de los tumores está ligado a la naturaleza de los mismos. Los tumores benignos, o bien, de BG de malignidad usualmente tienen una forma compacta, son poco amorfos y su superficie está en general bien definida. Por otra parte, los tumores de mayor grado de malignidad presentan una forma más irregular, con superficies no tan lisas y son menos compactos. De ahí el interés de poder contar con descriptores de forma que permitan caracterizar un tumor cerebral.
Se encuentran reportados diversos trabajos de investigación donde se proponen distintos descriptores morfométricos, cada uno de ellos enfocado a alguna característica morfológica específica del tumor, tanto para el caso particular de tumores cerebrales como para otro tipo de tumores en general, por ejemplo en el caso de tumores de mama, pulmón e hígado, sólo por nombrar algunos [8-10]. Cabe mencionar que varios de estos descriptores trabajan con mediciones clásicas y no adecuadas para análisis discretos como sería el caso requerido en objetos discretizados obtenidos a partir de imágenes digitales [11]. En la Tabla I se muestra una lista de algunos descriptores morfométricos que se han reportado en general para el análisis y estudio sobre la forma en tumores, así como una breve descripción de los mismos.
En referencia al estudio en tumores cerebrales, se han propuestos algunos descriptores morfométricos con el interés de lograr establecer una relación entre el grado de malignidad de un glioma y algún índice morfométrico. Yang et.al [12] presentan un estudio de morfometría 3D para poder diferenciar entre glioblastomas multiformes y metástasis a partir de imágenes por tensores de difusión. Nuestro grupo de investigación ha realizado un análisis morfométrico 3D en cuanto a la compacidad discreta (Cd) como descriptor de forma entre gliomas de diversos grados de malignidad, teniendo como resultados una primera discriminación entre ellos significativa [13].
Corvatura discreta
La tortuosidad es una propiedad que poseen todas las curvas y que nos permite ver la variación de curvaturas en las mismas, es decir, los cambios que se puedan tener presentes entre curvas cóncavas y convexas. Una propuesta para medir la tortuosidad de una curva discretizada bidimensional es por medio de la tortuosidad discreta (Td) descrita por Bribiesca [14]. La medición de la Td de una curva 2D se lleva a cabo a través de un código cadena de cambios de pendientes (SCC por sus siglas en inglés, Slope Chain Code). Una cadena a está definida por una secuencia ordenada de n elementos y se representa como:

Figura 1 a) Curva continua y curvatura discreta representada por segmentos de recta, donde ɷ es el ángulo de contingencia entre dos segmentos continuos. b) Normalización del ángulo de contingencia ɷ en rango de [0,-1) y [0,1) [14].
Tortuosidad discreta 2D
La tortuosidad discreta (Td) se calcula a través de la suma de los valores absolutos de todos los ángulos de contingencia normalizados de la curva discretizada, esto es, del código cadena, Ecuación (1)[14].
Por ejemplo, se puede observar en la Figura 2 un círculo continuo que ha sido discretizado por seis segmentos. Para cada vértice entre pares de segmentos se muestra el valor del ángulo de contingencia normalizado (ɷ), donde el cambio de pendientes se ha considerado en un sentido horario por lo que los valores son negativos. El código cadena correspondiente que caracteriza a la curva discretizada a partir del vértice A es: -0.33, -0.33, -0.33, -0.33, -0.33, -0.33, y el valor de la Td es igual a 2.

Figura 2 Ejemplo de una curva continua discretizada con seis segmentos. En cada vértice se muestran los valores correspondientes a los ángulos de contingencia normalizados entre segmentos y el valor de Td es 2.
Ahora bien, dado que estamos trabajando con imágenes discretas, se ha considerado que las regiones de interés ya se encuentran a su vez discretizadas. Por lo tanto, la longitud de los segmentos de discretización está basada de acuerdo a la resolución del tamaño de los voxeles que conforman la imagen, como se puede observar en la Figura 3.
Tortuosidad discreta aplicada a volúmenes
La aportación en este trabajo es proponer una adecuación de la medición de la Td para dos dimensiones presentada en la sección anterior y poderla aplicar a volúmenes para obtener una tortuosidad para 3 dimensiones (Td-3D), dado que en nuestro caso el interés se centra en el análisis de forma de los volúmenes que se obtienen de los diferentes tumores cerebrales.
Los volúmenes de interés se obtienen a partir de imágenes tridimensionales, las cuales constan de un cierto número de cortes o rebanadas. Para este estudio se propone obtener los contornos por cada corte axial en el que se tenga presente el tumor cerebral. Posteriormente se calculan los valores de cada cambio de pendiente entre los voxeles contenidos en el contorno, como se muestra en la Figura 3.
Para obtener el valor de la Td-3D se propone primero concatenar los códigos cadena de todos los contornos pertenecientes al objeto discretizado, los contenidos en cada corte y a su vez de todos los cortes, de tal forma que el código cadena resultante contiene información de curvatura de todo el objeto. Posteriormente se realiza la sumatoria definida en la ecuación (1), para tener una aproximación de la Td-3D.
Materiales y metodología
Base de datos
Se consideró una base de datos de uso libre de IRM de 20 pacientes que fueron diagnosticados con presencia de gliomas, de los cuales 10 pacientes presenta gliomas de bajo grado (BG, cuyo diagnóstico clínico fueron astrocitomas u oligoastrocitomas) y los otros 10 pacientes fueron diagnosticados con presencia de gliomas de alto grado (AG, cuyo diagnóstico clínico menciona como astrocitomas anaplásico y glioblastoma multiforme).
Las imágenes de RM T1, T1c, T2 y T2FLAIR fueron adquiridas en cuatro centros clínicos diferentes: Bern University, Debrencen University, Heidelberg University y Massachusetts General Hospital de acuerdo a Menze et. al [15], donde para homogeneizar los datos todas las imágenes adquiridas las co-registraron a la T1c y se ajustaron para que tuvieran una resolución isotrópica de 1 mm por voxel en todas las secuencias de RM que se adquirieron.
Segmentación de las regiones de interés
En este trabajo de investigación se busca estudiar y analizar la morfología de las diferentes regiones que componen a un glioma, así como las regiones cerebrales circundantes que puedan ser afectadas. Se consideraron cuatro regiones prioritarias de estudio: el edema, el tumor entero, la región activa y la región de necrosis. La base de datos con la que se realiza el estudio incluye el protocolo de la segmentación de la región del edema, la cual fue extraída a partir de la secuencia T2; así como la segmentación del tumor entero a partir de las secuencias T1 y T1c [15].
Dado que se tiene interés en realizar el análisis morfométrico también en la región activa y en la región de necrosis para el caso de gliomas de AG de malignidad, como lo sería en un glioblastoma multiforme, llevamos a cabo la segmentación de dichas regiones mediante métodos de umbralización utilizando el software Brainvisa - Anatomist.
En la Figura 4, se muestran las segmentaciones de las regiones de interés para un glioma de BG en la parte superior de la figura y para un tumor con AG (glioblastoma multiforme) de malignidad en la parte inferior.

Figura 4 En la parte superior se muestra el caso de un paciente con un glioma de BG, mientras que en la parte inferior se muestra el caso para un paciente con glioma de AG. Se observan diferentes secuencias de RM así como regiones de interés, de acuerdo a las columnas: (a) IRM T1c resalta las regiones activas y de necrosis, sobre todo en el caso de un glioma de AG; (b) IRM T2 resalta la región del edema para ambos casos; y (c) muestra las segmentaciones de las distintas regiones de interés (edema en color morado, región activa en color amarillo, región de necrosis en color azul y la región del tumor entero está conformada por la región activa y la región de necrosis).
Detección de contornos y obtención de curvaturas
Se realizó un proceso de detección de bordes en base al algoritmo de Moore (Moore-Neighborhood) [16] a partir de las máscaras binarias obtenidas de las segmentaciones de cada región de interés mencionadas en la sección anterior, con el objetivo de tener los contornos discretizados. Dado que cada objeto discretizado en realidad representa el volumen del tumor, se obtuvieron contornos por cada corte axial contenido en el volumen, como se muestra en la Figura 5. Se puede apreciar en la Figura 5 que en ocasiones hay presencia de contornos externos y contornos internos en las regiones de interés, sobre todo es el caso de la región activa y la región de necrosis.

Figura 5 En la parte superior se muestran las máscaras de las segmentaciones de las regiones de interés. En la parte inferior se muestran los contornos obtenidos: (a) región del edema, (b) región del tumor entero, (c) región activa y (d) región de necrosis, respectivamente.
Una vez que se tienen los contornos que componen el volumen del tumor, se procede a obtener los ángulos de contingencia normalizados (ɷ) para cada par de voxeles. Debido a que se pueden tener presentes más de un contorno en cada corte, el cálculo de la Td de hace a partir de la concatenación de estos múltiples contornos obteniendo una tortuosidad discreta 2D general, por corte.
Código cadena de cambio de pendiente y tortuosidad discreta aplicado a volúmenes
Para determinar la Td-3D, primero se concatenan todos los códigos cadena de los ángulos de contingencia obtenidos en los diferentes contornos, presentes en todos y cada uno de los cortes que representan el volumen del tumor. Para ellos se define un punto inicial y punto final para cada corte del objeto 3D. Posteriormente se calcula la Td-3D mediante la sumatoria de los valores absolutos del código cadena global, de acuerdo a la ecuación (1), para cada uno de los pacientes. De esta forma, la Td-3D caracteriza la tortuosidad discreta global de cada objeto volumétrico. Con el fin de poder realizar comparaciones de tortuosidad entre distintos objetos es necesario normalizar los valores de tortuosidad que se han obtenido. En el método de Td (SSC) de Bribiesca [14], la normalización propuesta considera que todos los objetos (2D) se discretizan con el mismo número de segmentos n, por lo que al final se divide la tortuosidad entre n para normalizar. En este trabajo, sin embargo, se propone una variante para normalizar que consiste en dividir la tortuosidad entre el área envolvente del objeto, medida como el número de voxeles contenidos en la superficie del volumen a analizar.
Resultados y discusión
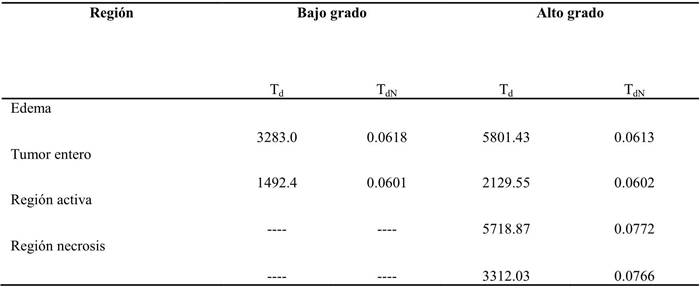
Se calculó la Td-3D para cada uno de los volúmenes de los gliomas en los 20 pacientes. Para los 10 pacientes con gliomas de AG se obtuvieron las cuatro regiones de interés mencionadas (edema, tumor entero, región activa y región de necrosis), mientras que para los 10 pacientes con gliomas de BG solo se tienen las regiones de edema y tumor entero. En la Tabla II se presentan los valores de Td-3D promedio, antes y después de normalizar, obtenidos para cada uno de los volúmenes de acuerdo al grado de malignidad correspondiente.
Las tortuosidades que permiten comparar los resultados son las que están normalizadas (TdN). Estos valores normalizados muestran que la tortuosidad de las regiones de edema son muy similares entre los caso de BG y AG. Lo mismo puede decirse de las tortuosidades de las regiones del tumor entero, que son muy parecidas. Este resultado llama la atención ya que los tumores de AG, al estar más vascularizados, se esperaría que fueran más tortuosos. Sin embargo, no se aprecian diferencias significativas en este caso. Una posible causa de esto se debe a la etapa de segmentación de las regiones de los distintos volúmenes, las cuales se realizaron de manera manual y es posible que se haya suavizado la curvatura real del objeto. Así mismo, está el hecho de que las imágenes utilizadas fueron pre-procesadas para estandarizar la referencia espacial y homogeneizar los datos [15], por lo que en algunos casos los ajustes pueden provocar cambios en los detalles de los contornos de los objetos. A partir de esto, se debe reconocer que este método, así como el cálculo de la tortuosidad discreta en general, es sensible a la resolución y a la definición con que se detectan los contornos. Estos factores podrían conducir a errores en la medición de la tortuosidad, una forma de disminuirlos sería a través de técnicas de segmentación más robustas para detectar los contornos con mayor precisión.
En la Tabla II también se muestran las tortuosidades de las regiones activas del tumor y de las regiones de necrosis para el caso de los gliomas de AG, siendo el valor de tortuosidad de la región activa en promedio más tortuosa que el resto de las regiones. De manera global, la región del tumor es la menos tortuosa, tanto para el caso de gliomas de BG como de AG, sin embargo cabe remarcar la alta sensibilidad que presenta el valor de la tortuosidad normalizada.
En la Figura 6 se muestra la gráfica de los valores de TdN para el caso de los gliomas de BG correspondiente a las regiones del edema y del tumor entero. Para efectos de presentación de los resultados, los datos se han ordenado de manera ascendente de acuerdo a los valores de TdN que se obtuvieron en la región del edema. En esta gráfica se puede apreciar que los valores que se obtuvieron de tortuosidad (TdN) para la región del edema son mayores a los valores de tortuosidad obtenidos en la región del tumor entero en la mayoría de los casos. Se puede observar además que siguen en términos generales un comportamiento parecido ambos valores de edema y tumor entero.

Figura 6 Valores de tortuosidad discreta normalizada obtenidos para cada uno de los pacientes que presentaron gliomas de bajo grado.
En la Figura 7 se presentan los valores de TdN para las cuatro regiones presentes en el caso de pacientes con gliomas de AG (edema, región del tumor entero, la región activa y la región de necrosis).

Figura 7 Valores de tortuosidad discreta normalizada obtenidos para cada uno de los pacientes que presentaron gliomas de alto grado.
Como puede observarse en la Figura 7, los valores normalizados de tortuosidad en la región del edema siguen siendo por lo general mayores que los valores normalizados de tortuosidad en la región del tumor, siguiendo un comportamiento creciente similar en ambas regiones. Sin embargo, se puede observar que los valores correspondientes a la región activa y a la región de necrosis presentan valores de tortuosidad más altos que los valores de tortuosidad de las regiones del edema y tumor, existe a su vez mayor variedad entre los mismos valores, tanto de la región activa como de necrosis.
Es importante mencionar que los valores de TdN que se muestran en las gráficas son relativamente pequeños, por lo que se puede pensar que no hay diferencias significativas entre ellos. Si bien la TdN es un valor considerado entre un rango de [0,1), el incremento en los valores de tortuosidad no llevan un comportamiento lineal. Es por ello que el valor promedio obtenido de TdN para gliomas de BG aparentemente es muy parecido al valor de TdN obtenido para gliomas de AG, esto es debido a la manera en cómo se está llevando a cabo la normalización de los valores de tortuosidad; se consideró TdN = T d /n [14], siendo n la superficie envolvente, calculada a partir del número de segmentos con los que ha sido discretizado el contorno del objeto. En nuestro caso, el tamaño del voxel es isotrópico de 1mm, por lo que el número de voxeles contenido en el perímetro de los contornos del volumen es relativamente mucho mayor que el valor de Td para cada uno de los casos y se tiene como resultado valores muy pequeños de TdN. La TdN de las regiones activa y de necrosis muestran una pequeña diferencia que en promedio no se considera significativa, como se muestra en la Tabla II; sin embargo, en algunos casos puntuales hay diferencias importantes que sí son significativas, Figura 7.
Conclusión
Se propone una variante de tortuosidad discreta aplicada a superficies volumétricas. Se obtuvieron y analizaron los valores de tortuosidad en las regiones de edema, tumor entero, región activa y región de necrosis para el caso de gliomas con bajo y alto grado de malignidad. Los valores de tortuosidad en las regiones del edema y el tumor entero presentan un comportamiento de tortuosidad similar, tanto en bajo como en alto grado. Para el caso de tumores de AG se observó que los valores de tortuosidad en la región activa son de mayor valor, seguidos por la región de necrosis, si bien los valores presentan mayor variabilidad entre ellos. Se observa que para encontrar una relación entre los valores de la tortuosidad discreta normalizada y la malignidad de un tumor cerebral primario, tomando en cuenta sus características morfológicas, se requiere de una segmentación robusta de las regiones del tumor donde los contornos no lleguen a ser alterados o suavizados de manera significativa.











 nueva página del texto (beta)
nueva página del texto (beta)