Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ingeniería biomédica
versión On-line ISSN 2395-9126versión impresa ISSN 0188-9532
Rev. mex. ing. bioméd vol.33 no.2 México dic. 2012
Artículo de investigación original
Estudio de la Actividad Biológica de Células de Melanoma en un Medio de Cultivo Mediante Técnicas de Biospeckle
Study of the Biological Activity of Melanoma-cells on a Culture Medium by Means of the biospeckle technique
R. A. Martínez-Celorio*, R.J. González-Peña**, R.M. Cibrián**, R. Salvador**, R. Gil-Benso***, T. San Miguel***, O.L. Sánchez-Muñoz****
*Grupo de Bioingeniería, DICIS, Universidad de Guanajuato, México.
**Grupo de Biofísica y Física Medica. Dep. Fisiología. Facultad de Medicina y Odontología. Universidad de Valencia.
***Dep. Patología. Facultad de Medicina y Odontología. Universidad de Valencia.
****Instituto de Ciencia Molecular, Universitat de València.
Correspondencia
R.A. Martínez-Celorio
Grupo de Bioingeniería, DICIS,
Universidad de Guanajuato, México.
Correo electrónico: rcelorio@salamanca.ugto.mx
Fecha de recepción: 04/Julio/2012.
Fecha de aceptación: 14/Noviembre/2012.
Resumen
Se presenta un estudio de la actividad biológica de células de un melanoma de piel ante la presencia de diferentes concentraciones del medicamento Colcemid mediante la técnica de Biospeckle. El estudio fue realizado sobre cuatro muestras colocadas en pocillos plásticos preparados para tales fines. Las muestras fueron: el medio de cultivo RPMI-1640 Gel; el medio de cultivo con 105 células de la línea MEL − RC08 y lo anterior con 1 µg y 2 µg de medicamento Colcemid, respetivamente. Estas muestras fueron estudiadas a la vez, mediante la técnica de Biospeckle que consiste en analizar la evolución temporal de los patrones de speckle de las mismas. Para esto, se diseño un experimento que consiste en iluminar las muestras con un haz de luz láser de He-Ne y adquirir un conjunto de 96 imágenes durante 24 horas usando una cámara de alta resolución Firewire. Las imágenes adquiridas son procesadas usando el método de las diferencias temporales de patrones de speckle para analizar su actividad biológica. Los resultados obtenidos muestran cambios en el comportamiento de la actividad biológica de las muestras en dependencia de la concentración de medicamento aplicada. Se discuten también las ventajas y desventajas del método empleado.
Palabras clave: Biospeckle, actividad biológica, método de diferencias temporales.
Abstract
We are reporting a study of the response of a biological tissue with a malignant melanoma induced of the cell line MEL-RC08 by means of Biospeckle’s technique. The study was performed on four prepared samples, which were placed in plastic wells prepared for such purposes. The used samples were: RPMI-1640 Gel; RPMI1640 plus 105 cells of the cell line MEL − RC08, and the other two plastic wells was added a concentration of 1 mg and 2 mg of Colcemid drug, respectively. The samples were tested at the same time by analyzing the temporal evolution of speckle patterns in each of them. For that, we designed an experiment, which consisted of to illuminate the samples with an He-Ne laser and acquire a set of 96 images for 24 hours by using a high resolution camera. Next, a temporal difference method for processing dynamic speckle patterns was implemented with the aim of analyzing the biological activity. The obtained results show that exist a change in the behavior of the biological activity of the samples depending on concentration of the applied drug. The advantages and disadvantages of the method are discussed.
Keywords: Biospeckle, biological activity, temporal difference method.
INTRODUCCIÓN
Cuando un haz de luz coherente se refleja o atraviesa un medio de índice de refracción no uniforme se obtiene una distribución estadística de la irradiancia luminosa conocida como patrón de speckle [1]. Estos patrones han sido ampliamente implementados en mediciones de desplazamientos, deformaciones, etc. [2, 3]. Una de las aplicaciones más novedosas de los patrones de speckle está en el estudio de la evolución de los sistemas biológicos, donde la técnica recibe el nombre Biospeckle o speckle dinámico [4]. Algunos ejemplos de aplicaciones del Biospeckle en sistemas biológicos las encontramos en el análisis de especies botánicas [5], en la evaluación de la motilidad del esperma congelado de bovino [6], en el conteo de células en la sangre [7], en el estudio de las células del endotelio en la córnea [8]. También estas técnicas han sido aplicadas para medir la presencia de hongos en especies de frijoles, tal como reportaron Braga y col. [9]. En estudios patológicos esta técnica ha sido también recurrida, ya que permite estudiar las actividades biológicas en especies de lento crecimiento, tal como el Bacilo de Calmette-Guérin, [10]. Otro trabajo publicado en dichos estudios muestra una relación entre el cambio del índice de refracción y la concentración bacteriana dado por Machado y col. [11], quienes lograron medir la constante de tiempo del crecimiento bacterial.
Existen diferentes métodos para el procesamiento de los patrones de speckle dinámicos. Algunos construyen imágenes auxiliares que representan la historia temporal de los patrones de speckle (HTPS), [12-14]. Estos métodos de procesamiento recurren en muchos casos al estudio de la textura de la imagen auxiliar reconstruida. En esta dirección, por ejemplo, Haralick y col. [15] propone el uso de la matriz de coocurrencia para analizar la textura de la imagen. Otras proposiciones usan el histograma suma o diferencia de la imagen [16], la transformada de wavelet [17], etc. Recientemente, Martí y col., [18] propusieron un procedimiento basado en el método de las diferencias temporales entre patrones de speckle dinámicos. La proposición se basa en detectar el cambio de la estructura del patrón de speckle mediante la diferencia entre patrones de speckle separados en el tiempo.
En este trabajo se muestra un estudio de la actividad biológica (AB) de un cultivo de tejido biológico mediante técnicas de Biospeckle. Al cultivo le fue inducido la línea celular MEL-RC08 [19] de un melanoma metastático analizando su respuesta ante la presencia de diferentes concentraciones del medicamento Colcemid. El procedimiento utilizado es el análisis de la AB mediante el método de las diferencias temporales. La importancia de este trabajo está en las aplicaciones de la técnica propuesta (Biospeckle) a los estudios patológicos por ser no-invasiva y que unida al procesamiento de señales, permite utilizar diferentes formas de análisis. A lo anterior se añade que el no-contacto evita la contaminación de las muestra y de los operarios.
Para lograr nuestro objetivo, este artículo está dividido en seis secciones. En la segunda sección se establecen los principios básicos del trabajo. En las secciones 3 y 4 se explican el procedimiento para obtener las muestras y el arreglo experimental usado. La sección 5 muestra los resultados obtenidos y los mismos se discuten. Finalmente, en la sección 6 se brindan las conclusiones del trabajo.
TÉCNICA DE BIOSPECKLE
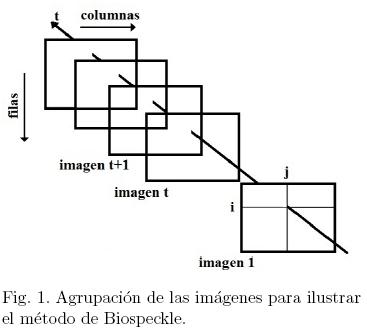
Para analizar el comportamiento de la AB en el tiempo mediante las técnicas de Biospeckle, primero es necesario realizar la captura de un conjunto lo suficientemente grande de imágenes a intervalos de tiempo regulares y durante todo el estudio. Seguidamente, estas imágenes se agrupan de manera consecutiva, una a continuación de la otra, tal como se representa en la Fig. 1. Así, calculando el módulo de la diferencia entre dos imágenes consecutivas, por ejemplo: en un tiempo t y t + 1 cualesquiera se reflejará entonces el cambio de la AB en la imagen. Este cálculo se expresa por [18]:
I (i, j, t)= |I (i, j, t + 1) − I (i, j, t)| (1)
donde: I(i, j, t +1) e I(i, j, t) representan los valores de irradiancia de los píxeles de coordenadas (i, j) de las imágenes correspondientes a los instantes t +1 y t, respectivamente.
Un conjunto de magnitudes pueden ser definidas para medir la AB en un área determinada de la muestra de tamaño M × N. Estas magnitudes son: 1.) actividad media en el área, 2.) actividad acumulativa y 3.) correlación espacial de la actividad. Estas magnitudes pueden ser calculadas como:
• actividad biológica media:

• actividad acumulativa:
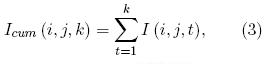
• correlación espacial de la actividad:

Estas magnitudes pueden servir en aplicaciones específicas. Por ejemplo, si se desea determinar el comportamiento de la AB media en una zona de la muestra se elige la zona en cuestión de área de M x N y en la misma podemos aplicar la Ec. (2). Una historia acumulativa de la evolución temporal de la AB en la muestra puede realizarse mediante la Ec. (3) . Si deseamos correlacionar dos áreas de la muestra se puede implementar mediante la Ec. (4) . Nosotros en este trabajo usamos la Ec. (1) para correlacionar dos patrones de speckle y detectar cambios de la AB. También usamos el valor máximo de la AB en una región; así como el cálculo de la actividad media mediante la Ec. (2).
PREPARACIÓN DE MUESTRAS
Se estudió una muestra de la línea celular MEL-RC08, cultivadas "in-vitro", de un melanoma metastático para observar su AB ante diferentes concentraciones medicamentosas de Colcemid [ref. 15212. Gibco, Invitrogen, Life Technologies LTD, Paisley, UK]. Las células se reprodujeron en un medio químico de cultivo RPMI-1640 [(Rosell Park Memorial Institute) ref 42401 Gibco, Invitrogen, Life Technologies LTD, Paisley, UK] con un pH entre 7-7.2 y bajo condiciones ambientales de temperatura (37 °C) y humedad controladas. Este medio de cultivo está suplementado (MCS) con un 10% de suero bovino fetal [ref. 10270. Gibco, Invitrogen, Life Technologies LTD, Paisley, UK], 1% de L-glutamina [ref 25030. Gibco, Invitrogen, Life Technologies LTD, Paisley, UK] y 1% de antibióticos para cultivo: penicilina y estreptomicina [PenStrep ref 15070. Gibco, Invitrogen, Life Technologies LTD, Paisley, UK].
Para el estudio, las muestras fueron colocadas en cuatro placas (pocillos) estériles de plástico tratado para cultivo, donde en cada pocillo se depositaron 1.5 ml de MCS. Además, en tres de los pocillos les fueron sembradas, en su centro, 105 células de melanoma metastático de la línea MEL-RC08, agregándoles a dos de ellos, en su centro, un volumen de Colcemid de 0.1mL (de 10µg/mL=1µg) y 0.2mL (de 10µg/mL=2µg), respectivamente. Una vez preparadas las muestras, las mismas fueron estudiadas a las 48 horas momento en que las células estaban en período de semiconfluencia. En la Fig. 2 se muestra un área de la muestra al inicio del ensayo y observada en un microscopio de contraste de fase.
ARREGLO EXPERIMENTAL
En la Fig. 3 se muestra un diagrama en bloques del arreglo óptico experimental para estudiar la respuesta de la AB de las células de melanoma al medicamento Colcemid. Como fuente de iluminación fue usado un láser de He-Ne, de la firma Spectra physics, con una longitud de onda de 633 nm y 25 mW de potencia. La luz del láser es expandida usando un filtro espacial y luego difundida por un vidrio esmerilado para iluminar uniformemente la muestra. Seguidamente, la iluminación atraviesa las muestras de estudio y pasa por un objetivo fotográfico SIGMA de aumento 0.76, distancia focal 50 mm y con un F# 16, que fue elegido para tener un tamaño del grano de speckle de aproximadamente 28.5 µm, el cual permite satisfacer el teorema del muestreo; o sea, que el tamaño del speckle sea por lo menos dos veces el período del píxel. Finalmente, esta iluminación es introducida en una cámara digital BASLER de alta resolución (1600 × 1200 píxeles) la cual funciona a una velocidad de 14 fps y con un tamaño de píxel de 4.6 µm. Para el estudio fueron capturadas un total de 96 imágenes en un tiempo de 24 horas.
Las imágenes capturadas (usando la herramienta adquisición de imágenes del programa Matlab) fueron procesadas primeramente llevando la resolución a 1024 × 1024 píxeles. A continuación, la imagen se divide en 4 imágenes con igual resolución de 512 × 512 píxeles para cada una de las muestras. Seguidamente, se seleccionó un área en el centro de cada imagen con resolución de 256 × 256 píxeles con el objetivo de observar en esta la AB mediante variación del patrón de correlación de speckle dado por la Ec. (1). Una vez detectada las variaciones se obtiene dentro de esta zona el valor de la actividad media mediante la Ec. (2) y el pico máximo de la variación en dicha zona. Finalmente, filtramos las imágenes para representar la zona de actividad biológica. Este procedimiento fue realizado a un total de 10 muestras de cada solución utilizando para el procesamiento el programa Matlab versión 10.0 para Windows.
RESULTADOS Y DISCUSIÓN
La Fig. 4 muestra el proceso de correlación implementado para obtener la información de la AB mediante la técnica de Biospeckle. En la Fig. 4a, b se observan los patrones de speckle de las 4 muestras estudiadas, que fueron capturados en los instantes consecutivos de tiempo correspondientes a las 2.00 h y 5.30 h de comenzado el estudio, respectivamente. Se pueden distinguir de manera delimitada las zonas circulares de los 4 pocillos donde fueron colocadas las muestras bajo estudio. En la Fig. 4c se presenta la imagen resultante correspondiente a la correlación entre las imágenes anteriores obtenida usando la Ec. (1) después de filtrar las imágenes con un filtro de la mediana [20]. Observe que en el pocillo de la parte superior izquierda de la imagen no aparece un patrón de correlación, lo cual indica que no existe AB en el mismo; o sea, la muestra en el mismo se encuentra en estado estable, no sufre variación alguna. Este resultado se corresponde con el medio de cultivo RPMI-1640 Gel que sirvió como referencia en nuestro estudio [21]. Por otro lado, en los demás pocillos es visible un patrón de speckle de correlación, el cual se debe a cambios en el patrón de speckle debido a la AB detectada en los mismos. La operación de correlación fue realizada con las 96 imágenes capturadas de manera consecutiva.
Después de realizar el algoritmo de correlación, se aplicó un filtro de máscara sobre cada una de las imágenes de las diferencias para eliminar el ruido de speckle fuera de los pocillos. Seguidamente, dentro de cada pocillo fue seleccionada un área cuadrada en su centro donde se realiza el estudio. Esta área tiene una resolución de 256 × 256 píxeles y donde se pudo apreciar que la actividad tenía picos cada ciertos intervalos de tiempo. Por esta razón, se escogió en cada muestra el primer incremento significativo de la AB y en éste fueron analizados tres tiempos t1, t2 y t3, que se corresponden a los valores de irradiancia media donde comienza el incremento, en el pico máximo y cuando disminuye a la estabilidad de la irradiancia en dicha zona, respectivamente. Lo anteriormente explicado se resume en las gráficas de la Fig. 5, donde en la primera fila se muestran las gráficas de la irradiancia media en el área seleccionada para el pocillo que contiene en su interior solamente el medio de cultivo RPMI-1640. En la segunda fila, se muestran los resultados obtenidos para el pocillo que contiene al medio de cultivo y la línea MEL-RC08 del melanoma metastático.
Las filas 3 y 4 se corresponden a los pocillos que les fueron añadidos cantidades de 1 µg y2 µg del medicamento Colcemid, respectivamente. De las gráficas de esta figura podemos sacar conclusiones importantes:
1. El medio de cultivo es estable, ya que las imágenes obtenidas no se diferencian apreciablemente entre sí en ningún instante de tiempo (t1 = 2.5 h, t2 = 4.5 h y t3 = 8.5 h). Las pequeñas variaciones de irradiancia luminosa que aparecen son debidas a inestabilidades de la fuente láser y movimientos inducidos.
2. Existen variaciones apreciables en el valor de la irradiancia luminosa de las imágenes correspondientes a los pocillos restantes. Por ejemplo, para el pocillo que contiene al medio de cultivo y la línea MEL-RC08 del melanoma metastático, el valor de la irradiancia media fue superior comparado con los restantes pocillos, teniendo este un pico de irradiancia de 1.2 u.a. (unidades arbitrarias). Para el pocillo con 1 µg de Colcemid, fila 3 de la gráfica, el pico de irradiancia es de 1.0 u.a; mientras que cuando se añadió 2 µg de Colcemid, el pico fue de 0.35 u.a. Estos resultados indican que a mayor cantidad de medicamento la AB disminuye, lo cual es lógico debido a la función de este medicamento de inhibir la actividad biológica [22]. Debemos señalar que al aumentar la concentración del medicamento se incrementa el esparcimiento de la luz, lo cual hace disminuir la irradiancia. Sin embargo, si se analiza la amplitud en la variación del pico de irradiancia, ésta es de menor amplitud en la muestra de mayor concentración cuando se compara con los picos de las restantes muestras.
3. El primer pico de la AB en las muestras comienza a ocurrir en diferentes instantes de tiempo. Por ejemplo, para la muestra con medio de cultivo y la línea MEL-RC08 del melanoma metastático, comienza en el instante t1 =2.5 h, para la muestra con 1 µg de Colcemid en t1 =3.75 h; mientras que para la muestra con 2 µg de Colcemid empieza en t1 =4.75 h. De este resultado podemos deducir que el incremento en la concentración de medicamento retarda el comienzo de la actividad biológica.
4. El radio de la región de AB en las imágenes disminuye a medida que se incrementa la concentración de medicamento. Si observamos todas las filas para un mismo instante de tiempo, por ejemplo t1, podemos detectar que el ancho de la zona de AB es mayor en la fila 2, la cual se corresponde al pocillo sin medicamento. Esto significa que la velocidad de reproducción es mayor en dicho pocillo que en los restantes, o sea, que el medicamento en los demás pocillos inhibe la reproducción celular.
Por todo lo anterior podemos plantear que las técnicas de Biospeckle permiten analizar el comportamiento de la actividad biológica en muestras de tejidos biológicos con un melanoma inducido de la línea MEL-RC08. Los resultados obtenidos indican que la AB se ve disminuida bajo la presencia del medicamento Colcemid.
Un análisis de la desviación estándar, σ, para las 10 mediciones de la irradiancia pico por muestra fue realizado mediante el método de teoría de error [23]. El valor obtenido fue de σ ≈ 0.137 u.a. Este valor de corrobora la veracidad del método implementado. Los errores cometidos al implementar el método propuesto se deben a varios factores. En primer lugar, a las condiciones ambientales que pueden acelerar o retardar la AB de las muestras. Para resolver esto, tanto los procesos de crecimiento como el estudio de las muestras se desarrollaron en un ambiente con control de temperatura y humedad relativa. Un segundo factor es el tiempo de realización del estudio; sin embargo, con los tiempos manejados experimentalmente se garantizó que este estudio fuese desarrollado en su totalidad en el período de semiconfluencia. Otras fuentes de error presentes menos trascendentes fueron: estabilidad del láser, vibraciones mecánicas, procesos de aproximación en los cálculos.
CONCLUSIONES
Se demostró que el método de Biospeckle permite caracterizar a la AB celular midiendo el comportamiento temporal de los cuadros de speckle en muestras biológicas. Los resultados obtenidos permitieron corroborar que la AB en células de un melanoma metastático de la línea MEL-RC08 puede dilatarse cuando se le añade a la muestra el medicamento Colcemid. El método es viable para el estudio de análisis clínicos, ya que al ser una técnica no-invasiva y de no-contacto presenta una alta seguridad y no contamina a la muestra. Por otro lado, el uso de técnicas de procesamiento de señales permite implementar diferentes formas de procesamiento para el análisis de los resultados.
Reconocimientos
Los autores quieren agradecer a los Departamentos de Patología y Biofísica de la Facultad de Medicina de la Universidad de Valencia (UV) por las facilidades en el uso de sus instalaciones. RAMC, desea agradecer la beca de investigación bajo proyecto otorgada por la UV. Agradecemos a los árbitros anónimos por sus comentarios y sugerencias.
REFERENCIAS
1. Goodman J.M. "Speckle and Metrology"; Chap. 8 in Speckle phenomena in optics: theory and application, Roberts and Company Publishers, Greenwood Village CO (2007). [ Links ]
2. Sirohi RS. "Speckle Metrology". Marcel Dekker, New York (1993). [ Links ]
3. Martínez-Celorio R.A., Dávila A., Kaufmann G. H. and Mendiola G. "Extension of the displacement measurement range for the electronic speckle-shearing pattern interferometry using carrier fringes and a temporal phase unwrapping method". Optical Engineering 2000; 39(3): 751-757. [ Links ]
4. Rabal H.J. y col., "Transient phenomena analysis using dynamic speckle patterns". Optical Engineering 1995; 35(1): 57-62. [ Links ]
5. Xu Z., Joenathan C., Khorana B.M. "Temporal and spatial properties of the time-varying speckles of botanical specimens". Optical Engineering 1995; 34: 1487-1502. [ Links ]
6. Carvalho P.H.A. y col., "Motility parameters assessment of bovine frozen semen by biospeckle laser (BSL) system". Biosystems Engineering 2009; 102(1): 31-35. [ Links ]
7. Serra J. "Aplicación de la Morfología Matemática a la telemedicina y la Biotecnología: Caracterización Morfológica de Células de sangre y Análisis de cDNA Microarrays" en Reconocimiento de Patrones Avances y Perspectivas, editado por J. L. Díaz de León y S. C. Yánez Márquez del Centro de Investigación en Computación Instituto Politécnico de Computación México., 1, 39-50.(2002). [ Links ]
8. Ayala G., Díaz M E., Martínez-Costa M. "Granulometric moments and corneal endothelium status". Pattern Recognition 2001; 34(6): 1219-1227. [ Links ]
9. Braga Jr R A, Rabelo GF, Granato LR, Santos EF, Machado JC, Arizaga R, y col. "Detection of fungi beans by the laser biospeckle technique". Biosystems Engineering 2005; 91(4):465-9. [ Links ]
10. Jardim D. F y col., "Observing bacterial activity interferometrically". European Biophysics Journal 2003; 32(2): 159-162. [ Links ]
11. Machado R.R. y col., "Metabolic activity interferometer: description and calibration of an interferometric method to measure growth of mycobacteria". European Biophysics Journal 2008; 38(1): 111-119. [ Links ]
12. Oulamara A., Tribillon G. and Duvernoy J. "Biological activity measurements on botanical specimen surfaces using a temporal decorrelation effect of laser speckle". Journal of Modern Optics 1989; 36: 165-179. [ Links ]
13. Braga Jr R A, Olivera B, Rabelo G, Marques R, Machado A, Cap N, y col. "Reliability of biospeckle image analysis". Optics and Lasers in Engineering 2007; 45: 390-395. [ Links ]
14. Sánchez-Arévalo F. M., Aldama-Reyna W., Lara-Rodriguez A.G., García-Fernández T., Pulos G., Trivi M., Villagran-Muniz M. "Use of time history speckle pattern and pulsed photoacoustic techniques to detectthe selfaccommodating transformation in a Cu-Al-Ni shape memory alloy". Materials Characterization 2010; 61: 518-524. [ Links ]
15. Haralick R., Shanmugam M.K. and Dinstein L. "Textural features for images classification". IEEE Transactions on System, Man and Cybernetics. SMC-3 1973. [ Links ]
16. Unser M. "Sum and Difference Histogram". IEEE Transactions on Pattern Analysis and Machine Intelligence 1986; 8(1): 118-125. [ Links ]
17. Fernández M., Mavilio A., "Wavelet transform analysis of dynamic speckle patterns texture". Applied Optics 2002; 41(32): 6745-6750. [ Links ]
18. Martí-López L., Cabrera H., Martínez-Celorio R.A., González-Peña R. "Temporal difference method for processing dynamic speckle patterns". Optics Communications 2010; 283(24): 4972-4977. [ Links ]
19. Gil-Benso R, Monteagudo C, Cerdá-Nicolás M, Callaghan RC, Pinto S, Martínez-Romero A, Pellín-Carcelén A, San-Miguel T, Cigudosa JC, López-Ginés C., "Characterization of a new human melanoma cell line with CD133 expression". Human Cell 2012; 25(2): 61-7. [ Links ]
20. González R. and Woods R., "Digital Image Processing", 3rd. Edition. Prentice-Hall, 2008. [ Links ]
21. Wang Y.J., Liu H.L., Guo H.T., Wen H.W., Liu J. "Primary hepatocyte culture in collagen gel mixture and collagen sandwich". World Journal of Gastroenterology 2004; 10(5): 699-702. [ Links ]
22. Yang H., Ganguly A., Cabral F. "Inhibition of Cell Migration and Cell Division Correlates with Distinct Effects of Microtubule Inhibiting Drugs". The Journal of Biological Chemistry 2010; 285(42): 32242-32250. [ Links ]
23. Berne J.L., Chueca-Pazos M., Herraez J. "Teoría y errores de instrumentación", Ed. 1, Editorial: Thomson Paraninfo, S.A., ISBN: 8428323089, 1996. [ Links ]