Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ingeniería biomédica
versión On-line ISSN 2395-9126versión impresa ISSN 0188-9532
Rev. mex. ing. bioméd vol.33 no.2 México dic. 2012
Artículo de investigación original
Rastreo de Imágenes y Reconstrucción de Volúmenes de Ultrasonido Médico
Image tracking and volume reconstruction of medical ultrasond
F. Torres1, Z. Fanti1, E. Lira1, C. García-Segundo1, B. Reyes-Ramírez1, E.J. Hazan2, R. Gerson3, F. Arámbula-Cosío1
1Centro de Ciencias Aplicadas y Desarrollo Tecnológico, Universidad Nacional Autónoma de México.
2Instituto Nacional de Rehabilitación.
3Hospital ABC, Ciudad de México.
Correspondencia:
Fernando Arámbula-Cosío
Centro de Ciencias Aplicadas y
Desarrollo Tecnológico. UNAM.
Apdo. postal 70-186.
Correo electrónico: fernando.arambula@ccadet.unam.mx
Fecha de recepción: 26/Octubre/2012.
Fecha de aceptación: 20/Diciembre/2012.
Resumen
En este trabajo presentamos los procedimientos necesarios para rastrear, en tiempo real, una sonda de ultrasonido clínico, así como dos métodos para reconstruir un volumen tridimensional a partir de un conjunto de imágenes bidimensionales modo B, rastreadas: un método basado en voxeles y otro basado en pixeles. Se presenta el problema de calibración de la sonda y los detalles de implementación del método de calibración de dos hilos cruzados, con el que obtuvimos un error de exactitud de 0.556 mm y una precisión de 0.249 mm, en la calibración. Como caso de estudio se presenta la reconstrucción de volúmenes de ultrasonido de maniquíes de mama con un tumor. Los dos métodos de reconstrucción implementados produjeron resultados similares.
Palabras clave: ultrasonido transoperatorio, ultrasonido 3D, calibración, reconstrucción, biopsias.
Abstract
In this work, the techniques available for real time tracking of a clinical ultrasound probe are reviewed. Two methods of volume reconstruction, from tracked images, have been implemented, and their performance has been compared: a pixel based method (PBM) and a voxel based method (VBM). It is also presented the ultrasound probe calibration problem and the details of implementation of the crossed wire method of calibration, which produced accuracy and precision errors of calibration of 0.556 and 0.249 mm respectively. As a case study for 3D reconstruction, the reconstruction of a phantom of a breast with a tumour, is presented. Both methods (PBM and VBM) produced similar reconstructions with comparable computational performance.
Keywords: transoperative ultrasound, 3D ultrasound, calibration, reconstruction, biopsies.
INTRODUCCIÓN
El ultrasonido es usado ampliamente para terapias y diagnósticos médicos y biológicos. Esta modalidad de imágenes es responsable de una de cada 5 imágenes médicas para el diagnóstico [1]. El uso de ultrasonido médico como técnica de imágenes tiene varias ventajas: no se expone al paciente o al personal médico a radiaciones ionizantes, como en el caso de la fluoroscopía intervencional; permite el acceso a buena parte de la anatomía del paciente; los equipos de ultrasonido son los más compactos, portátiles y económicos de entre las diferentes modalidades de imágenes médicas; la calidad de las imágenes ha mejorado mucho en los equipos más recientes [2].
El ultrasonido 3D permite la visualización y medición del volumen de estructuras tridimensionales de interés. Actualmente existen sondas de ultrasonido 3D, pero tienen la desventaja de ser equipos muy costosos en comparación a las sondas de ultrasonido 2D y el volumen visualizado se limita al tamaño de la sonda [3, 4]. Por otro lado existen técnicas para la reconstrucción de volúmenes de ultrasonido a partir de imágenes 2D de ultrasonido. Estas técnicas, conocidas como técnicas freehand, consisten en el rastreo de la sonda de ultrasonido para conocer la posición y orientación de planos de imagen adyacentes; mediante el procesamiento de los datos de las imágenes y los datos de orientación y posición de éstas, es posible la reconstrucción de un volumen de ultrasonido [3]. Actualmente se utilizan dos tipos principales de rastreo: óptico y electromagnético.
Los equipos de rastreo óptico calculan la posición 3D de un marcador, haciendo uso de un par estéreo de imágenes. La posición y orientación 3D de un instrumento (p.ej. Sonda de ultrasonido) se mide utilizando accesorios de rastreo que llevan al menos tres marcadores fijos en alguna configuración conocida, ya que es el mínimo requerido para definir un plano.
Los equipos de rastreo optico áctivos hacen uso de LEDS infrarrojos como marcadores, lo que requiere cables de conexión, mientras que los equipos pasivos hacen uso de esferas reflectoras de infrarrojo. Los rastreadores ópticos reportan errores de exactitud, dentro del volumen calibrado de operación, de fracciones de milímetro, mientras que su limitación principal es la necesidad de mantener una línea de vista entre cada accesorio de rastreo y las dos cámaras del rastreador. En la Figura 1, se muestra un rastreador óptico (A) así como los accesorios de rastreo colocados en una sonda de ultrasonido y una aguja de biopsia (B).
Los equipos de rastreo electromagnético miden continuamente la posición y orientación de un sensor, que consiste de una o varias bobinas, que detecta pulsos de radiofrecuencia generados por un transmisor, por lo que no dependen de una línea de vista, aunque es siempre necesario el uso de cables de conexión. El transmisor genera un campo magnético pulsado mediante varias bobinas y establece así el marco de referencia espacial para localizar cada sensor. La unidad de control del equipo calcula la posición de cada sensor dentro del volumen de medición de forma multiplexada, permitiendo así el rastreo de varios instrumentos. Los sensores se fabrican con dimensiones adecuadas para permitir el rastreo transoperatorio de la punta de un catéter o de una aguja de biopsia. Aunque los primeros rastreadores electromagnéticos eran afectados por la presencia de metales en el volumen de trabajo, los equipos modernos publican errores de localización de rastreo de fracciones de milímetro, dentro de un volumen calibrado que puede contener metales no ferromagnéticos.
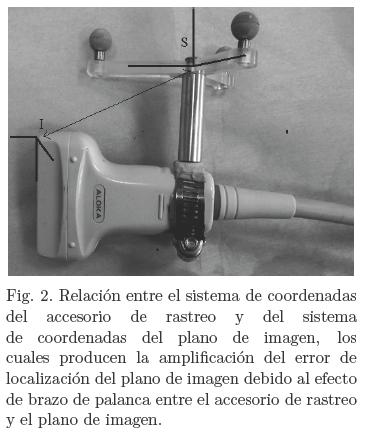
Independientemente de la técnica de rastreo de imágenes de ultrasonido utilizada, es importante tener en cuenta el efecto de palanca entre la posición del sensor o accesorio de rastreo, montado sobre la sonda, y el plano de la imagen. El efecto de palanca multiplica, en proporción a la longitud del brazo de palanca, el error de localización del plano de imagen, como se ilustra en la Figura 2, donde se muestra con I el origen del plano de imagen y con S el sistema del accesorio de rastreo.
Otra fuente de error en el rastreo de imágenes, es la desincronización entre la captura de la posición de la sonda y la captura de la imagen correspondiente. Esto es debido a que la obtención de posición por el rastreador es usualmente muy rápida, comparada con el tiempo de adquisición de la imagen. Lo cual podría introducir errores significativos para procedimientos que requieran de una exactitud de medición alta, cuando la sonda se mueve rápidamente.
La finalidad de este artículo es dar a conocer una serie de pasos esenciales para realizar reconstrucción de volúmenes de ultrasonido a partir de imágenes 2D, así como la comparación de dos métodos de reconstrucción de volúmenes. En las secciones siguientes se detalla la implementación del rastreo de una sonda de ultrasonido, modo B, con un sistema de rastreo óptico. Se presenta el problema de calibración de la sonda para obtener la posición 3D de un pixel en el plano de imagen, así como la reconstrucción de una sección de volumen por dos métodos: uno basado en pixeles y otro basado en voxeles. Como caso de estudio y prueba de los métodos, presentamos la reconstrucción de maniquíes de mama con insertos que simulan tumoraciones.
CALIBRACIÓN DE LA SONDA DE ULTRASONIDO
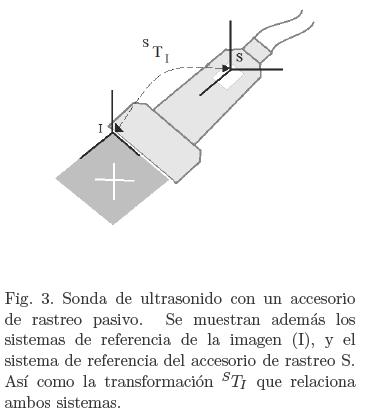
Como se describió en la sección anterior, un equipo de rastreo proporciona la posición y orientación de un accesorio o herramienta de rastreo, el cual, una vez montado rígidamente en una sonda de ultrasonido, determina un sistema de coordenadas local propio de la sonda; éste se muestra como el sistema S en la Figura 3.
Sin embargo, el objetivo del rastreo de imágenes es medir la posición y orientación 3D del plano de imagen (sistema local de coordenadas I en la Figura 3) con respecto al sistema de coordenadas de la sonda de ultrasonido. Para lo que es necesario encontrar la matriz de transformación rígida (STI) que mapea el plano de imagen al sistema de coordenadas local del accesorio de rastreo, así como los factores de escala horizontal y vertical del plano de imagen (ex, ey). Dicha transformación se podría calcular directamente de los dibujos de construcción de la sonda, con las dimensiones y posición del arreglo de ultrasonido dentro de la montura de ésta; sin embargo, usualmente esta información no está disponible y es necesario encontrar la transformación STI mediante un proceso de calibración experimental.
Existen varios métodos de calibración; en [5] se presenta la comparación de cuatro métodos de calibración basados en cuatro montajes experimentales diferentes: dos y tres hilos cruzados, pared única y accesorio Cambridge. De estos cuatro métodos el de dos hilos cruzados es el que resultó tener un menor error de reconstrucción. Se ha demostrado que los métodos de calibración de un solo punto, como lo es el de hilos cruzados, son los óptimos, debido a su fácil construcción y error de reconstrucción pequeño [6].
Para realizar la calibración de una sonda de ultrasonido por el método de dos hilos cruzados se utiliza una bandeja con agua en la que se sumergen dos hilos cruzados como se muestra en la Figura 4. El objetivo del procedimiento de calibración es determinar la transformación STI y los factores de escala horizontal (ex) y vertical (ey) en la Ecuación 1. En la Figura 5, se ilustra la adquisición de una imagen de ultrasonido rastreada del cruce de dos hilos sumergidos en agua.
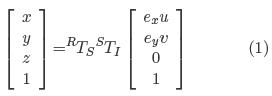
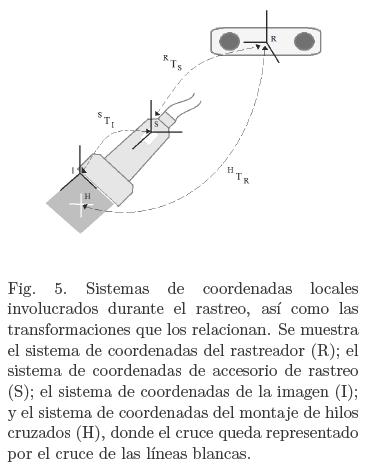
Por otro lado la transformación HTR mapea el origen del sistema de coordenadas del rastreador (R) al punto de cruce de los hilos (H). Esta transformación se puede omitir, pero al incluirla se evitan problemas de orientación y posición del volumen [5]. Al incluir latransformación HTR las nuevas coordenadas del punto de cruce de los hilos en una imagen de ultrasonido corresponderán al origen de un sistema de coordenadas (H) como lo describe la Ecuación 2.
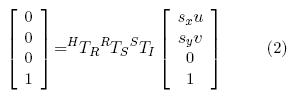
Cada una de las transformaciones corresponde a una matriz de transformación ortogonal con tres ángulos y tres desplazamientos (Ecuación 3):
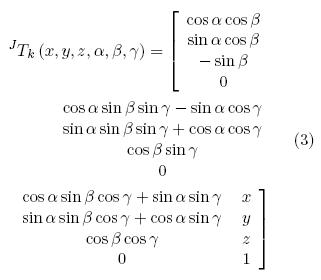
En la Ecuación 2, la transformación RTS la proporciona el rastreador; de esta manera, el problema de calibración consiste en determinar 11 parámetros: dos factores de escala exy ey, tres ángulos, tres desplazamientos correspondientes a STI; y tres desplazamientos de HTR, ya que las rotaciones se pueden omitir debido a que el origen de este sistema se tomó como el punto que corresponde al cruce de los hilos. Al establecer (u, v) en la Ecuación 2 como la posición del punto de cruce de los hilos cruzados en una imagen de ultrasonido se obtienen 3 ecuaciones no lineales, las cuales contienen los 11 parámetros que se desean estimar. Mediante la localización del punto de cruce de los hilos en diferentes planos de imagen, es posible la construcción de un sistema de ecuaciones no lineales sobredimensionado; este sistema de ecuaciones se resuelve con un método iterativo para ecuaciones no lineales como el de Levenberg-Marquardt [7]. Una vez que se determina la matriz STI podemos mapear cada plano de imagen de la sonda rastreada a un volumen, utilizando la Ecuación 1.
RECONSTRUCCIÓN DEL VOLUMEN A PARTIR DE UN CONJUNTO DE IMÁGENES RASTREADAS
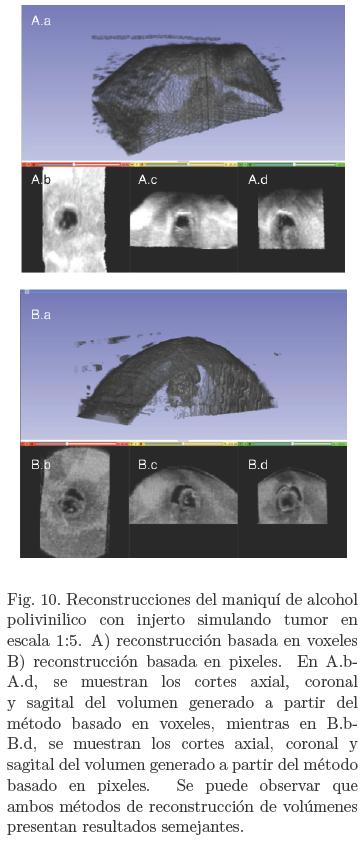
Para realizar la reconstrucción de un volumen a partir de un conjunto de imágenes de ultrasonido modo B, rastreadas, es necesario conocer las transformaciones correspondientes para mapear los pixeles en cada imagen a su correspondiente posición dentro de un arreglo de voxeles. Para esto se debe determinar, dependiendo de la aplicación, el origen y tamaño del volumen, así como el tamaño de los voxeles. Posteriormente el volumen se debe llenar haciendo uso de los valores de los pixeles de las imágenes adquiridas. Las dos clases principales de métodos para llenar un volumen a partir de planos de imágenes son: los métodos basados en voxeles (MBV), en los que se visita cada voxel del volumen y se le asigna un valor de nivel de gris calculado a partir de los pixeles de los planos vecinos al voxel; y los métodos basados en pixeles (MBP), en los que se visita cada pixel de cada plano de imagen y se asigna un valor al voxel más próximo. En este trabajo se presentan las reconstrucciones obtenidas, con ambas técnicas, de un maniquí de mama con un inserto que simula un tumor.
Los MBV llenan un volumen en un solo recorrido por todos los voxeles del mismo, haciendo una estimación del valor correspondiente a cada voxel. A diferencia de los MBP que requieren de dos etapas para el llenado de voxeles, ya que insertan la información de los pixeles en los voxeles correspondientes y posteriormente se debe de realizar un llenado de los voxeles que quedaron vacíos en el primer recorrido, [8].
Para calcular el tamaño del volumen se usan las coordenadas (x,y,z) menores y mayores de todo el conjunto de imágenes 2D localizadas en el espacio; con estos datos se genera un paralelepípedo que encierre a todas las imágenes, las coordenadas menores del paralelepípedo se utilizan como el origen del volumen. Una vez obtenido el tamaño del volumen se define el tamaño del voxel con respecto al tamaño del pixel de las imágenes haciendo uso de un factor de escala; a esta relación se le conoce como resolución [8].
Al realizar la reconstrucción de volúmenes a partir de imágenes rastreadas de ultrasonido, hay que tomar en cuenta varios aspectos, como son: el tamaño y posición del volumen así como el tamaño del voxel. La elección de estos parámetros dependerá del tipo de aplicación que se tenga, así como de las características de adquisición de las imágenes rastreadas. Si se requiere tener un volumen de alta resolución, la distancia entre las imágenes debe ser muy cercana, de tal manera que no queden espacios vacíos de más de un par de voxeles entre ellas; esto con el fin de que la interpolación no introduzca artefactos en el volumen; así mismo, el tamaño del voxel deberá de ser muy cercano al tamaño del pixel en las imágenes. Por otro lado, si se requiere tener volúmenes más manejables, el tamaño de éste puede solo contener una región de interés de las imágenes de ultrasonido y el tamaño del voxel ser mucho más grande que el tamaño del pixel.
Reconstrucción basada en voxeles
Trobaugh, et. al [9] proponen un MBV que consiste en calcular una normal del voxel hacia los dos planos más cercanos a éste y hacer una interpolación bilineal de los cuatro pixeles que rodean el punto de cruce entre la normal y el plano de la imagen. El valor final del voxel se calcula como el promedio ponderado por distancia del valor obtenido en la interpolación bilineal de los dos planos más cercanos. A continuación se detalla el algoritmo del MBV.
Algoritmo de reconstrucción basado en voxeles
Para cada voxel v en el volumen V:
• Encontrar la distancia normal de v a todos los planos de las imágenes, obteniendo un vector de distancias D.
• Obtener en D la mayor distancia y las dos menores distancias (dmax, d1 y d2 respectivamente), así como el plano al que corresponden las dos menores distancias I1 y I2.
• Para cada plano In, donde n =1, 2
— Se calcula el punto de cruce entre la normal que pasa por v y In
— Se realiza una interpolación bilineal (pn) de los 4 pixeles más cercanos al punto de cruce
— Se obtiene el peso correspondiente a cada plano In
wn =1 − dn/dmax
— El valor final de v se obtiene como el promedio de los resultados de la ponderación por distancia.
v =(w1 · p1 + w2 · p2)/2
Reconstrucción basada en pixeles
Generalmente la reconstrucción de volúmenes con el método basado en pixeles se divide en dos etapas; la primera recorre cada uno de los pixeles del conjunto de imágenes rastreadas y lo asigna a su ubicación correspondiente dentro del volumen. Cuando se da el caso, en que varios pixeles corresponden al mismo voxel, usualmente éstos se promedian, aunque existen varias propuestas de resolver el problema: dejar el pixel más reciente; dejar el máximo o mínimo [10]. A esta etapa se le conoce como "llenado de espacios" (bin filling). Una vez que los pixeles de las imágenes se han colocado en su posición correspondiente dentro del volumen, hará falta llenar los voxeles vacíos que quedaron entre las imágenes; en esta segunda etapa se realiza una interpolación del valor de los voxeles que no quedaron vacíos. Si la captura de imágenes se hace lo suficientemente cerca una de otra, es probable que el llenado de voxeles vacíos sea innecesario [11]. Por otro lado hay que tomar en cuenta que si quedaron regiones vacías, de proporciones grandes con respecto al tamaño del volumen, éstas se deben de considerar de una forma especial, o bien se deben de redefinir las propiedades del volumen, ya que al momento en que éstas son llenadas se pueden añadir artefactos que hagan al médico realizar un mal diagnóstico. Para esta etapa del método, diversos autores han propuesto, distintas metodologías a seguir.
El método de llenado de voxeles basado en interpolación de los vecinos más cercanos estima el valor del voxel vacío con el resultado de la interpolación de los voxeles no vacíos que se encuentren dentro de una ventana cúbica de tamaño fijo; en caso de que no existan voxeles no vacíos dentro de la ventana, ésta se puede crecer hasta encontrar voxeles no vacíos, o hasta llegar a un límite de tamaño; si este fuera el caso el voxel a interpolar se deja vacío. Otros métodos de interpolación son: el promedio de los voxeles dentro de la ventana [10], los dos voxeles más cercanos [12] y la media de los voxeles no vacíos dentro de la ventana cúbica [13]; todos ellos basados en el mismo principio.
Se debe de tomar en cuenta que en los métodos basados en pixeles se ejecuta de manera lineal en la primera etapa y de manera exponencial en la segunda, de tal manera que hacer la interpolación de gran cantidad de voxeles vacíos resultará en una reconstrucción lenta. A continuación se detalla el algoritmo de reconstrucción basada en pixeles haciendo el uso de interpolación del vecino más cercano dentro de una ventana cúbica en la segunda etapa, ya que es un método sencillo de implementar y tiene buenos resultados [8].
Algoritmo de reconstrucción basado en pixeles
Llenado de espacios con promedio (Primer etapa).
• Para cada imagen Ik, donde k — 1,2,..., número de imágenes rastreadas.
• Para cada pixel p en Ik
— Encontrar la posición del voxel v en el volumen V al que pertenece p.
— Si v no tiene un valor asignado
v = p; c = 1;
— Si v tiene un valor asignado
v = (v · c + p)/(c + 1); c = c + 1;
• Llenado de voxeles vacíos con interpolación (Segunda Etapa).
— Para cada v en V
— Si v no tiene un valor asignado
c = 0;
— Para cada voxel v' en una vecindad 3x3x3 o mayor alrededor de v
* Si v' tiene un valor asignado
c = c + 1; v = (v + v')/c;
En la siguiente sección mostramos los resultados de la calibración de una sonda de ultrasonido clínico 2D, así como los resultados de la reconstrucción 3D de un maniquí de mama, a partir de la adquisición de un conjunto de imágenes 2D rastreadas.
RESULTADO DE LA CALIBRACIÓN Y LA RECONSTRUCCIÓN DE VOLÚMENES
Nuestro sistema de adquisición y rastreo de imágenes de ultrasonido consiste en un equipo de ultrasonido clínico ALOKA 1000 (http://www.hitachi-aloka.co.jp), con una sonda modo B de propósito general de 7.5 MHz, a la que adaptamos un accesorio de rastreo pasivo, compatible con un equipo de rastreo óptico POLARIS (www.ndigital.com). Las imágenes de ultrasonido se capturan, con una tarjeta de adquisición de datos PXR 800, fabricada por CyberOptics Semiconductor, instalada en una PC Pentium de 3.0 GHz y 3GB de RAM. La reconstrucción de los volúmenes se realizó en una computadora MACPro, 2.8GHz Quad-Core Intel Xeon con 3GB de RAM.
Los métodos de calibración y de reconstrucción de volúmenes previamente mencionados fueron implementados en C++, utilizando las librerías de uso libre ITK (Insight Segmentation and Registration ToolKit http://www.itk.org) y VTK (Visualization ToolKit http://www.vtk.org). La adquisición de los datos del rastreador óptico y de las imágenes se realizó usando funciones de IGSTK (Image Guided Surgery ToolKit http://www.igstk.org) que es una librería de código abierto que provee los componentes básicos requeridos para desarrollar aplicaciones para procedimientos médicos guiados por imágenes.
Resultados de la calibración
Calibramos nuestra sonda de ultrasonido con el método de dos hilos cruzados como se describió en la Sección 2.
Exactitud
La exactitud de calibración se refiere al correcto posicionamiento de un punto en el sistema de coordenadas I, transformado al sistema de coordenadas R mediante la Ecuación 1.
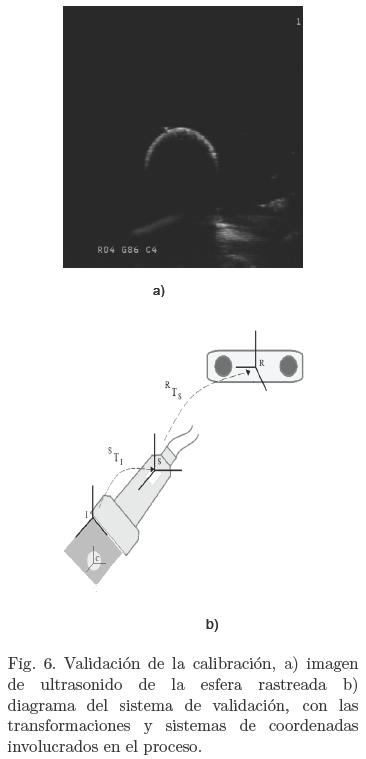
Para evaluar el error de calibración tomamos imágenes de ultrasonido de una esfera de dimensiones conocidas, cuyo centro es rastreado como se muestra en la Figura 6. De estas imágenes de ultrasonido se obtuvieron manualmente la posición de algunos puntos de la superficie de la esfera y se estimó una esfera haciendo uso de estos datos. Esto se realizó resolviendo un sistema de ecuaciones no lineales, en donde se obtiene una ecuación por cada punto sobre la superficie de la esfera, haciendo uso de la ecuación general de la esfera (Ecuación 4), donde x, y, z corresponden a los puntos marcados sobre la superficie; a, b, c corresponden al centro estimado de la esfera; r corresponde al radio estimado de la esfera. Una vez estimados el centro y el radio de la esfera se calculó la distancia entre el centro estimado y el centro rastreado; este dato es el que se reporta como la exactitud en la calibración E.
0 = (x — a)2 + (y—b)2 + (z — c)2— r2 (4)
Se adquirieron 10 imágenes de ultrasonido de una esfera rastreada y se utilizaron 2400 puntos adquiridos sobre la superficie de la esfera para estimar el centro y el radio de ésta. El error de exactitud obtenido fue: E = 0.556mm.
Precisión
La precisión de la calibración se refiere al error cometido por las transformadas obtenidas en el proceso de calibración al transformar un mismo punto visto desde diferentes planos. Para obtener este dato es necesario realizar la transformación, del sistema de coordenadas I al sistema de coordenadas S, de un punto visto desde diferentes planos y obtener el promedio de la desviación absoluta de la nube de puntos generada. Este dato se reporta como la precisión de la calibración (Pr). Para evaluar la precisión de la calibración no es necesario conocer la posición del punto en el espacio [9].
Para calcular la precisión de la calibración se obtuvieron 25 imágenes desde diferentes vistas del cruce de hilos, se seleccionó manualmente el punto de cruce de los hilos en cada imagen, para posteriormente transformar este punto y obtener el promedio de la desviación absoluta de la nube de puntos generada con las transformaciones. Con estos datos se obtuvo una precisión Pr = 0.249mm.
Repetibilidad
Otro dato importante en la validación del método de calibración es la repetibilidad del procedimiento [9]. Para medir la repetibilidad de nuestra calibración, dos usuarios realizaron 3 calibraciones cada uno, usando los mismos datos; se calculó la desviación estándar (σ) de los parámetros estimados mediante la calibración para conocer la variabilidad de los resultados. Estos resultados se reportan en la Tabla 1 donde (x, y, z) y (α, β, γ) corresponden a las traslaciones y rotaciones de la transformada 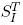 y (sx, sy) corresponden a los factores de escala en la Ecuación 1.
y (sx, sy) corresponden a los factores de escala en la Ecuación 1.
Reconstrucción 3D de volúmenes
Para evaluar la calidad de la visualización de nuestro sistema de rastreo y reconstrucción 3D utilizamos un maniquí de mama hecho de alcohol polivinílico [14]. La manufactura de dicho maniquí es tal que permite replicar la respuesta al esfuerzo mecánico del tejido real, así como su respuesta al ultrasonido, además de que posibilita la inserción de elementos que permiten simular de manera fidedigna diferentes formas y tamaños de alguna lesión tumoral. Se adquirieron 700 imágenes de ultrasonido modo B de 387x400 pixeles, tomadas con el maniquí sumergido en agua, rastreadas con un equipo POLARIS Aurora. Para fines de comparación, implementamos dos métodos de reconstrucción del volumen a partir de imágenes rastreadas: un método basado en voxeles; y otro basado en pixeles (descritos en la sección 3.1 y 3.2). En las Figuras 10 a) y 10 b) se pueden observar las reconstrucciones del maniquí de alcohol polivinílico con el MBV y el MBP respectivamente.
Para validar la reconstrucción de los volúmenes se realizaron reconstrucciones de un fantasma de ultrasonido (Ultrasound Resolution Phantom Model 044 CIRS), el cual incluye tres conjuntos de cilindros con diferentes características. El primer grupo de cilindros consiste en 6 cilindros de 1.5mm de diámetro y una separación entre ellos de 12.5mm; se obtuvieron 57 imágenes rastreadas de ultrasonido de este grupo. El segundo grupo de cilindros consiste de 9 cilindros de 3mm de diámetro y una separación de 12.5mm entre ellos; de este grupo se obtuvieron 63 imágenes rastreadas de ultrasonido. El tercer grupo consiste en 2 cilindros de 12mm de diámetro y 20mm de separación entre ellos, se obtuvieron 87 imágenes rastreadas de ultrasonido del grupo. Con las imágenes de ultrasonido se reconstruyeron 5 volúmenes con diferentes resoluciones (relación del tamaño del voxel, del volumen a reconstruir, con el tamaño del pixel de las imágenes de ultrasonido) de cada grupo. Se realizaron mediciones manuales de los cilindros en cada volumen reconstruido mediante el software de uso libre 3D Slicer (http://www.slicer.org/); se tomaron dos medidas de diámetro a cada cilindro (una medida en vertical y una medida en horizontal) y una medida de distancia entre cada par de cilindros del grupo. Las medidas adquiridas se realizaron en el plano de imagen que se debe de usar para que éstas sean correctas; dicho plano queda definido por dos líneas paralelas configuradas por el fabricante.
Reconstrucción basada en voxeles
Utilizamos el método basado en voxeles, propuesto por Trobaugh, et. al [9], para la reconstrucción de maniquí de mama con un injerto que simula un tumor (para lo que se utilizó una uva con semillas). El tamaño del volumen y el tiempo total de procesamiento para la reconstrucción con el MBV de arreglos de voxeles con diferentes resoluciones (Relación tamaño Pixel:Voxel) se muestra en la Tabla 2.
En la Figura 7 se muestra una rebanada de un volumen de un conjunto de cilindros del fantasma de ultrasonido en el cual se midió manualmente la distancia entre dos cilindros y el diámetro de un cilindro; en la imagen se pueden apreciar las dos líneas paralelas indicando que las medidas fueron obtenidas en el plano de imagen correcto.
En la Tabla 3 se muestran los promedios y las desviaciones estándar de las mediciones de diámetro y separación de los cilindros de cada grupo, en volúmenes con diferentes resoluciones reconstruidos con el método basado en voxeles. El promedio de error de todas las mediciones horizontales y verticales de los cilindros se muestra en la Tabla 4.
Reconstrucción basada en pixeles
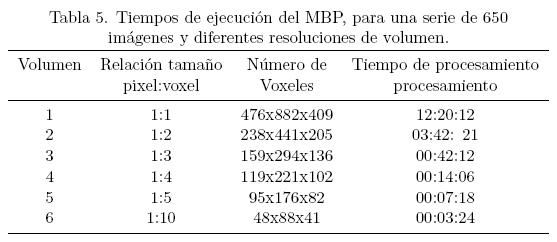
Se realizó una implementación del método basado en pixeles con un llenado de voxeles, con el promedio de éstos dentro de la ventana de Nelson & Pretorius [10]. Con este método se realizó la reconstrucción de volúmenes del maniquí de alcohol polivinílico con diferentes resoluciones. Los tiempos de procesamiento se muestran en la Tabla 5. Para la reconstrucción de volúmenes sin voxeles vacíos se utilizó una ventana de 23x23x23.
Al igual que con el MBV, se realizaron reconstrucciones de los fantasmas de ultrasonido con el método basado en pixeles, utilizando una ventana cúbica máxima de 23x23x23 para el llenado de voxeles. Se muestran los resultados de las mediciones en la Figura 8. En la Tabla 6 se muestran los promedios y las desviaciones estándar de las mediciones de diámetro y separación de los cilindros de cada grupo, en volúmenes con diferentes resoluciones reconstruidos con el método basado en pixeles. El promedio de error de todas las mediciones horizontales y verticales de los cilindros se muestra en la Tabla 7.
Se puede observar en la Figura 8 que con este método en las zonas donde se realizó llenado de huecos el volumen presenta baja nitidez. A su vez, en los bordes del volumen se presentan ciertos artefactos que no pertenecen al volumen.
El uso de una ventana pequeña o pocas imágenes causa, en altas resoluciones, huecos (voxeles vacíos) en los volúmenes, lo que produce artefactos negros como se muestra en la Figura 9. Para evitar los huecos en los volúmenes, la mayor distancia entre imágenes debe de ser menor al tamaño de la ventana. El aumento de tamaño en la ventana cúbica y en el número de imágenes elimina estos artefactos, pero aumenta considerablemente el tiempo de procesamiento de los volúmenes.
DISCUSIÓN Y CONCLUSIONES
Los experimentos realizados para la validación de la calibración de la sonda produjeron valores pequeños en los errores de exactitud (E = 0.556 mm) y precisión (Pr =0.249 mm). Es importante tener en cuenta que el error de exactitud en este caso fue calculado mediante la estimación de una sola esfera rastreada. Para que esta medición sea más fidedigna es necesario realizar la estimación de un mayor número de esferas en diferentes posiciones.
La repetibilidad del procedimiento de calibración mostró resultados satisfactorios, ya que se puede observar en la Tabla 1 que la variación de los datos obtenidos en diferentes calibraciones realizadas por diferentes usuarios no es significativa. Esto indica que la selección manual del punto de cruce de hilos en las imágenes de ultrasonido no afecta el resultado obtenido, haciendo que éste sea un método que no depende del usuario.
El fantasma de hilos cruzados presenta buenos resultados para la calibración, tanto en precisión como en exactitud. Sin embargo, hay factores que deben de ser tomados en cuenta para realizar una buena calibración, como son: tomar al menos 50 imágenes del cruce de hilos para la calibración desde diferentes posiciones y orientaciones de la sonda de US; colocar el cruce de hilos en diferentes posiciones dentro de la imagen; asegurar el hacer coincidir el plano de imagen del ultrasonido con el cruce de hilos; ser cuidadoso al segmentar el cruce de hilos de las imágenes adquiridas.
Con respecto a la reconstrucción de volúmenes, mediante el análisis de las Tablas 3 y 6 se puede ver que, en general, el error en las mediciones es de fracciones de milímetros, con excepción de las mediciones de los cilindros de 1.5mm de diámetro con la más baja resolución (1:10), ya que no fue posible identificar la forma del cilindro debido a la deformación que sufren los objetos en la reconstrucción. Se puede observar en estas Tablas 3 y 6 que el MBV tiende a aumentar el tamaño de los objetos, mientras que el MBP tiende a reducir el tamaño de éstos. El MBP reportó en promedio un mayor error en las mediciones horizontales; esto implica que los objetos tendrán una deformación mayor en este eje. Aunque el MBV también reporta un mayor error en las mediciones horizontales, la diferencia entre el promedio de las mediciones horizontales y verticales es mucho menor que en el MBP, implicando que la deformación que sufren los objetos sea menor al ser reconstruidos con el MBV. Se observa en las Tablas 4 y 7 que el error de las mediciones en el MBV es menor al error en las mediciones en el MBP.
El tiempo de procesamiento de los volúmenes haciendo uso del MBV es menor al tiempo de procesamiento con el MBP. Es importante mencionar que el tiempo de procesamiento del MBV depende únicamente del número de voxeles y el número de imágenes, mientras que el tiempo de procesamiento del MBP depende del número de voxeles, el número de imágenes, el número de pixeles en cada imagen y el tamaño de la ventana cúbica utilizada para la segunda etapa del método.
El MBP requiere un número mayor de imágenes para reconstruir volúmenes de calidad aceptable en comparación al MBV, el cual produjo reconstrucciones aceptables haciendo uso de solamente 131 imágenes y reduciendo considerablemente el tiempo de procesamiento. Es difícil concluir cuál de los dos métodos de reconstrucción implementados es mejor, ya que existen muchos factores que influyen en la calidad de los volúmenes y en el tiempo de procesamiento. Es importante destacar que el método y la resolución más adecuados dependerán directamente de la aplicación.
AGRADECIMIENTOS
Los autores agradecen el apoyo del Instituto de Ciencia y Tecnología del Distrito Federal, así como el apoyo del Consejo Nacional de Ciencia y Tecnología, de México, para la realización de este trabajo.
REFERENCIAS
1. Hallowell M. "A tutorial on ultrasonic physics and imaging techniques.", Proc Inst Mech Eng H., 2010; 224(2):127-42. [ Links ]
2. King A.P., Blackwall J.M., Penney G.P., Hawkes D.J., "Tracking liver motion using 3-D ultrasound and a surface based statistical shape model", Proceedings of the IEEE Workshop on Mathematical Methods in Biomedical Image Analysis, 2001; 145-152. [ Links ]
3. Rohling R, Gee A, Berman L. "Three-dimensional spatial compounding of ultrasound images", Med Image Anal. 1997; 1(3):177-93. [ Links ]
4. Krücker JF, Meyer CR, LeCarpentier GL, Fowlkes JB, Carson PL. "3D Spatial Compounding of Ultrasound Images Using Image-Based Nonrigid Registration". Ultrasound Med Biol. 2000; 26(9): 1475-1488. [ Links ]
5. Prager, R. W., Rohling, R. N. R., Gee, A. H. A., and Berman, L. L. "Rapid calibration for 3D freehand ultrasound". Ultrasound in medicine and biology, 1998; 24(6), 855-869. [ Links ]
6. Hsu, P. W., Prager, R. W., Gee, A. H., and Treece, G. M. "Freehand 3D Ultrasound Calibration: A Review", Advanced Imaging in Biology and Medicine, Heidelberg: Springer Berlin Heidelberg. (Berlin), 2009: 47-84. [ Links ]
7. More, J. "The Levenberg Marquardt algorithm: implementation and theory", Lecture Notes in Mathematics Volume, 1978; 630: 105-116. [ Links ]
8. Solberg, O., Lindseth, F., Torp, H., Blake, R., and Nagelhushernes, T. "Freehand 3D Ultrasound Reconstruction Algorithms– A Review", Ultrasound in Medicine & Biology, 2007; 33(7): 991-1009. [ Links ]
9. Trobaugh JW, Trobaugh DJ, Richard WD. "Three-dimensional imaging with stereotactic ultrasonography", Comput Med Imaging Graph, 1994; 18: 315-323. [ Links ]
10. Nelson, T. R. and Pretorius, D. H. "Interactive acquisition, analysis, and visualization of sonographic volume data", International Journal of Imaging Systems and Technology, 1997; 8(1): 26-37. [ Links ]
11. Gobbi, D. G. and Peters, T. M. "Interactive Intra-operative 3D Ultrasound Reconstruction and Visualization", Lecture Notes in Computer Science, 2002; 2489: 156-163. [ Links ]
12. McCann, H. A., Sharp, J. C., Kinter, T. M., McEwan, C. N., Barillot, C., y Greenleaf, J. F. "Multidimensional ultrasonic imaging for cardiology", Proceedings of the IEEE, 1998; 76(7): 1063-1073. [ Links ]
13. San José-Estépar, R., Martín-Fernández, M., Caballero-Martínez, P. P., Alberola-López, C., and Ruiz-Alzola, J. "A theoretical framework to three-dimensional ultrasound reconstruction from irregularly sampled data". Ultrasound in Medicine & Biology, 2003; 29(2): 255-269. [ Links ]
14. Quispe RM, Reyes B, Hevia N, García-Segundo C, Arámbula F, Sato R. "Implementation of the Polyvynil-alcohol mixed with nano-particles as a near representation of biological tissue: ultrasonic and photo-thermal study", J. Appl. Research and Technol, 2012; 10(1): 63-72. [ Links ]