Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de ingeniería biomédica
versión On-line ISSN 2395-9126versión impresa ISSN 0188-9532
Rev. mex. ing. bioméd vol.31 no.2 México dic. 2010
Artículo de investigación original
Segmentación y detección de glóbulos blancos en imágenes usando Sistemas Inmunes Artificiales
Erik Cuevas,* Valentín Osuna-Enciso, Diego Oliva, Fernando Wario, Daniel Zaldivar
* Universidad de Guadalajara, CUCEI, Departamento de Ciencias Computacionales, Av. Revolución 1500, Guadalajara, Jalisco, México.
Correspondencia:
Erik Cuevas.
E-mail: erik.cuevas@cucei.udg.mx
Artículo recibido: 27/mayo/2010.
Artículo aceptado: 30/noviembre/2010.
RESUMEN
Los glóbulos blancos (GB) o leucocitos juegan un papel importante en el diagnóstico de distintas enfermedades. Aunque técnicas de procesamiento digital de imágenes han ayudado parcialmente al análisis de tales células, prevalecen distintas complicaciones en la detección de GB, debido a variaciones en forma, tamaño, así como iluminaciones no adecuadas inherentes a la preparación del frotis sanguíneo. Por otra parte, los sistemas inmunes artificiales (SIA), basados en la manera en que el sistema inmunológico natural optimiza sus funciones para la detección de antígenos, han sido aplicados con éxito en la solución de distintos problemas de optimización, produciendo resultados superiores comparados con las técnicas clásicas. El enfoque más usado de SIA es el algoritmo de selección clonal (ASC), el cual permite proliferar a aquellas soluciones (anticuerpos) que mejor resuelven a una determinada función de desempeño (antígeno). Este artículo presenta un algoritmo para la segmentación, detección y medición de leucocitos en imágenes de frotis sanguíneo. El algoritmo usa dos sistemas ASC, uno para segmentación y el otro para detección de leucocitos. El problema de detección es considerado en este trabajo como un problema de optimización entre el leucocito y la forma circular que mejor lo aproxime. El algoritmo utiliza la codificación de tres puntos de la imagen de bordes para modelar los leucocitos candidatos. Una función de costo evalúa si tales leucocitos candidatos están realmente en la imagen. Siguiendo los valores de tal función de costo, el grupo de leucocitos candidatos es modificado usando ASC hasta aproximarse a los leucocitos reales presentes en la imagen de bordes. Los resultados obtenidos en comparación a otros algoritmos usados para la misma tarea validan la eficiencia de esta propuesta en exactitud, velocidad y robustez.
Palabras clave: Algoritmo de selección clonal (ASC), sistemas inmunes artificiales (SIA), procesamiento de imagen, detección de glóbulos blancos.
ABSTRACT
White blood cells also known as leukocytes play a significant role in the diagnosis of different diseases. Digital image processing techniques have successfully contributed to analyze the cells, leading to more accurate and reliable systems for disease diagnosis. However, a high variability on cell shape, size, edge and localization complicates the data extraction process. Moreover, the contrast between cell boundaries and the image's background varies according to lighting conditions during the capturing process. On the other hand, Artificial Immune Systems (AIS) have been successfully applied to tackle numerous challenging optimization problems with remarkable performance in comparison to classical techniques. One of the most widely employed AIS approaches is the Clonal selection algorithm (CSA) which allows to proliferate candidate solutions (antibody) that better resolve a determinate objective function (antigen). This paper is thus focused on the segmentation, localization and measurement of leukocytes from other different components in blood smear images. The algorithm uses two different CSA systems, one for the segmentation phase and other for the leukocyte detection task. In the approach, the detection problem is considered to be similar to an optimization problem between the leukocyte and its best matched circle shape. On the other hand, Clonal selection algorithm (CSA) is one of the most widely employed AIS approaches. The algorithm uses the encoding of three points as candidate leukocytes over the edge image. An objective function evaluates if such candidate leukocytes are actually present in the edge image following the guidance of the objective function's values. A set of encoded candidate leukocytes are evolved using the CSA so that they can fit into the actual leucocytes shown by the edge map of the image. The obtained results in comparison with the state-of-the-art algorithms validate the efficiency of the proposed technique with regard to accuracy, speed, and robustness.
Key words: Artificial immune systems (AIS), clonal selection algorithm (CSA), white blood cells detection, image processing.
INTRODUCCIÓN
El número, tamaño y deformación de los GB proveen información útil a los especialistas para el diagnóstico de enfermedades infecciosas, inflamatorias, cáncer, leucemias, y otros procesos1,2. El método tradicional para que un experto humano realice el conteo y medición de las células es una tarea monótona que consume demasiado tiempo. Por otro lado, el uso de técnicas de procesamiento de imágenes se ha desarrollado rápidamente en los últimos años, a tal punto que los hematólogos pueden utilizar imágenes de frotis sanguíneos para automáticamente obtener una primera aproximación en el diagnóstico de enfermedades. Tales técnicas han ayudado en el conteo de células sanguíneas en forma automática, sin embargo son incapaces de proveer información de características importantes de los leucocitos. Por ello, en este trabajo se presenta una propuesta que consiste de algoritmos SIA para segmentación y medición aproximada de leucocitos en frotis sanguíneos.
Muchos trabajos se han realizado en el área de segmentación y localización de GB. Entre los más comunes están la detección de bordes, crecimiento de regiones, filtrado, morfología matemática y clustering, o agrupamiento. En Ritter et al3 presentaron un método completamente automático para segmentación e identificación de bordes de todos los GB que no presentan traslapes en una imagen obtenida a partir de un frotis sanguíneo. Ongun et al.4 realizaron localización de GB por medio de un pre-procesamiento morfológico seguido por la aplicación del algoritmo «snake-baloon». Mientras que Jiang et al.5 propusieron un esquema de segmentación de GB en imágenes de color basado en técnicas de agrupamiento de características, considerando primeramente filtrado escalo-espacial para extraer los núcleos y agrupamiento para extracción de citoplasma. Leyza et al.6 usaron operadores morfológicos y examinaron las propiedades escalo-espaciales de un operador tensor para mejorar la exactitud de la segmentación. Scotti7 presentó un método morfológico automático que está basado en el análisis morfológico de GB, definiendo algunos índices morfológicos. Kumar et al.8 usaron el operador de energía «Teager» para la segmentación de núcleos basados en bordes, los cuales son efectivamente detectados por el operador de energía Teager, sin embargo el método está restringido a que exista al menos un espacio entre el glóbulo blanco y el fondo de la imagen. Por otra parte, Cseke introdujo un esquema de segmentación multi-escala9, en el que implementa el método de umbralización automático propuesto por Otsu10. Recientemente Wang et al.11 propusieron un algoritmo pesado computacionalmente para detección de GB basado en una red neuronal celular difusa. Este método aunque permite detectar con mucha precisión la forma de un solo glóbulo blanco, no ha sido probado para la detección de múltiples GB, además de degradar su desempeño cuando el algoritmo no es detenido en el número de iteraciones adecuadas. Número que dicho sea de paso no es claro cómo obtener.
La detección de formas circulares suele llevarse a cabo por medio de la transformada circular de Hough12. Sin embargo, la exactitud de los parámetros de los círculos detectados es pobre en presencia de ruido13. Además el tiempo de procesamiento requerido por la transformada circular de Hough hace prohibitivo su uso por algunas aplicaciones, en particular para imágenes digitales grandes y áreas densamente pobladas de pixeles borde. Para tratar de superar tales problemas, se han propuesto varios algoritmos basados en la transformada de Hough (TH), tales como la TH probabilística14,15, la TH aleatoria (THA)16 y la TH difusa (THD)17. En [18], Lu & Tan propusieron una aplicación basada en la THA llamada THA iterativa (THAI), que logra mejores resultados en imágenes complejas y ambientes ruidosos. El algoritmo aplica iterativamente la THA a regiones de interés en la imagen las cuales son determinadas a partir de la última estimación de los parámetros del círculo/elipse detectado. La detección de formas puede también ser realizada usando métodos de búsqueda estocástica, tal como los algoritmos genéticos (AG), los cuales han sido recientemente aplicados a importantes tareas de detección de formas. Por ejemplo, Roth y Levine propusieron el uso de AG para extraer primitivas geométricas en imágenes19. Lutton et al. realizaron mejoras al método anterior20, mientras que Yeo et al. usaron un AG multipoblación para detectar elipses21. En [18] fueron usados AG para buscar similitudes cuando el patrón a detectar ha estado sujeto a una transformación de afinidad desconocida. En Ayala Ramírez et al. se presentó un detector de círculos basado en AG22 que es capaz de detectar múltiples círculos en imágenes reales, sin embargo falla frecuentemente en detectar círculos imperfectos o en condiciones difíciles. Recientemente, Dasgupta et al. propusieron otro excelente trabajo23 de detección automática de círculos, usando un algoritmo de alimentación de bacterias como método de optimización. Considerando el caso de detección elipsoidal, Rosin propuso en [24] [25] un algoritmo de ajuste de elipses que usa cinco puntos.
Por otro lado, los métodos biológicamente inspirados han sido exitosamente transferidos a paradigmas computacionales, muestra de ello son los conocidos desarrollos en redes neuronales artificiales, algoritmos evolutivos, algoritmos de enjambres y otros. El sistema inmune humano (SIH) es un sistema adaptativo altamente evolucionado, paralelo y distribuido26, que exhibe importantes habilidades, las cuales pueden ser usadas en el campo de la computación. Este nuevo campo de estudio es conocido como sistemas inmunes artificiales (SIA)27, el cual es un sistema computacional inspirado en la teoría del sistema inmune y sus funciones, incluyendo principios y modelos. Los SIA han alcanzado recientemente considerable interés por parte de la comunidad científica28, enfocados en distintos aspectos, tales como optimización, reconocimiento de patrones, detección de anomalías, análisis de datos y aprendizaje de máquina. La optimización inmune artificial (OIA) ha sido exitosamente aplicada en la solución de numerosos problemas de optimización con un marcado rendimiento en comparación con técnicas clásicas29.
El algoritmo de selección clonal (ASC)30 es uno de los enfoques de OIA más empleados. El ASC es un algoritmo de optimización evolutivo relativamente nuevo que ha sido construido basado en el principio de selección clonal (PSC)31 del SIH. El PSC explica la respuesta inmune cuando un patrón antígeno es reconocido por un anticuerpo. En el ASC, el antígeno representa el problema a ser optimizado, así como sus restricciones, mientras que los anticuerpos son las soluciones candidatas del problema. La afinidad anticuerpo-antígeno indica la cercanía entre la solución candidata y el problema. El algoritmo realiza la selección de anticuerpos basados en afinidad, ya sea por comparación contra el patrón antígeno o evaluando el patrón por medio de una función objetivo. El ASC tiene la habilidad de salir de mínimos locales mientras opera simultáneamente sobre el espacio de búsqueda. No usa derivadas o información relacionada a ellas, sino reglas de transición probabilística. A pesar de su implementación simple y directa, ha sido extensamente utilizado para resolver distintos tipos de problemas complejos de ingeniería32-34.
Este artículo presenta un algoritmo para la segmentación y detección automática de GB en imágenes complicadas y con ruido de frotis sanguíneos. El algoritmo usa dos ASC diferentes, uno para segmentación y otro para detección de leucocitos. La técnica propuesta hace la segmentación de cada frotis en tres regiones: leucocitos, glóbulos rojos y fondo. Esto se logra modelando al histograma de los pixeles que pertenecen a las regiones de la imagen como una mezcla de Gaussianas, tal que cada una de las funciones Gaussianas que participan en la combinación representa una de las clases o región35,36. Los parámetros de las funciones Gaussianas son calculados automáticamente por medio del ASC con sólo el número de clases deseadas como información previa. Por otro lado, la detección de GB es considerado como un problema de optimización entre el leucocito y su mejor aproximación circular. El algoritmo utiliza la codificación de tres puntos no colineales de la imagen de bordes para modelar los leucocitos candidatos. Una función de costo evalúa si tales leucocitos candidatos están realmente en la imagen. Siguiendo los valores de tal función de costo, los leucocitos candidatos son modificados usando ASC hasta aproximarse a los leucocitos reales en la imagen de bordes. De este enfoque propuesto resulta un detector de precisión sub-píxel capaz de identificar leucocitos en imágenes, a pesar de que éstos presenten significativas porciones ocluidas o traslapadas. La evidencia experimental muestra la efectividad del método para detectar leucocitos bajo distintas condiciones. Comparaciones realizadas con algoritmos que conforman el estado del arte en la detección de GB11 demuestran un mejor desempeño del método propuesto.
Este trabajo está organizado de la siguiente manera: en la sección 2 se muestran los fundamentos del ASC, la sección 3 presenta el enfoque de segmentación usado. La sección 4 presenta el algoritmo de detección de GB propuesto. En la sección 5 se muestran los resultados obtenidos en la segmentación y detección de GB. En la sección 6 se prueba el rendimiento del enfoque propuesto mediante la comparación con otro enfoque considerado parte del estado del arte en la detección de GB. Finalmente, en la sección 7 son mostradas las conclusiones.
ALGORITMO DE SELECCIÓN CLONAL
En sistemas inmunes naturales sólo los anticuerpos que son capaces de reconocer al cuerpo extraño o antígeno (individuos no-propios), son seleccionados para proliferar por medio de clonación26. Por tanto, la base del método de optimización clonal establece que sólo los anticuerpos más capaces son clonados. Particularmente, los principios del ASC tomados del PSC son los siguientes:
• Mantenimiento de una memoria de individuos, los cuales están desconectados de la población.
• Selección y clonación de los anticuerpos más estimulados.
• Eliminación de los individuos no estimulados.
• Maduración de afinidades y re-selección de los clones que muestren las mayores afinidades.
• Valores de mutación proporcional a las afinidades de los anticuerpos.
De los conceptos de inmunología se tiene que un antígeno es cualquier sustancia que forza al sistema inmune a producir anticuerpos contra ella. Considerando los sistemas de ASC, el concepto de antígeno, se refiere al problema de optimización, en este caso se refiere a las soluciones candidatas de los parámetros, ya sea de las funciones Gaussianas o de los círculos que aproximan a los GB. En el ASC los anticuerpos son llamados simplemente individuos. En este trabajo, un anticuerpo es una representación de una solución candidata para un antígeno, es decir, el leucocito-círculo. Un mecanismo de selección garantiza que tales individuos (soluciones) que reconocieron mejor al antígeno y por lo tanto dieron mejores respuestas, sean seleccionados para tener una vida más prolongada. Por lo tanto, tales individuos son llamados individuos de memoria (M).
Diferencias de ASC y algoritmos genéticos
Las principales características de los algoritmos genéticos son37:
• Codifica las soluciones.
• La búsqueda se realiza a través de toda la población.
• Usa una función objetivo como criterio de desempeño.
• Usa operadores estocásticos tales como crossover y mutación.
El ASC comparte características con los AG, como lo son: la codificación de parámetros, el uso de una población y el operador estocástico de mutación, el cual en general alcanza porcentajes mayores en el ASC que en los AG. Sin embargo, la principal diferencia radica en la forma en la cual evolucionan los individuos para encontrar una nueva aproximación al óptimo. Mientras el operador de crossover en AG provoca que la solución muestre tendencias altamente marcadas por el mejor individuo de la población y por lo tanto, a arrastrar a toda la población de cromosomas hacia una sola solución38,39, en el ASC existe la desconexión del repertorio de individuos, por lo cual es posible encontrar más de una solución en el espacio de búsqueda debido a una gran diversidad en la población de anticuerpos: de esta manera, el ASC evita quedar atrapado en mínimos locales40, lo que no siempre resulta en los AG. En este sentido es que se considera que el ASC tiene la capacidad de converger hacia múltiples óptimos del problema, situación que obligaría a los AG a realizar varias corridas para encontrar los mismos resultados. Es por esto que el ASC es capaz de manejar efectivamente complejas tareas de optimización multimodal41-44. Por todo lo anterior es que el ASC es adoptado en este trabajo para encontrar: a) los valores de segmentación de una imagen usando una mezcla de funciones Gaussianas y b) los parámetros de los círculos (punto central y radio) que mejor aproximan a los leucocitos en la imagen.
Definiciones
Para describir el ASC, la notación incluye letras mayúsculas en negritas indicando matrices y letras minúsculas en negritas indican vectores. Algunos conceptos también relevantes son mencionados a continuación:
(i) Antígeno: El problema a ser optimizado y sus restricciones (leucocito real a detectar).
(ii) Anticuerpo: Las soluciones candidatas del problema (posibles leucocitos).
(iii) Afinidad: El valor de la función objetivo para un anticuerpo (correspondencia entre el leucocito real y el candidato).
La cadena d es la variable de codificación del vector x, es decir, d = encode(x); y x es llamada la decodificación del anticuerpo d o x = decode(d).
El conjunto l es llamado el espacio anticuerpo, es decir, d∈l, el cual es entonces definido como:

donde el entero positivo m es el tamaño de la población de anticuerpos D = {d1,d2,...,dm}, el cual es un grupo m-dimensional de anticuerpos d, estando en un punto dentro del espacio anticuerpo l.
Operadores de ASC
Considerando como base[45], el ASC implementa tres diferentes operadores: el operador de proliferación clonal (TPC), el operador de maduración de afinidad (TMA) y el operador de selección clonal (TSC). A (k) es la población de anticuerpos en el instante k que representa el conjunto de anticuerpos a, tal que A(k) = {a1(k),a2(k),...,an(k)}. El proceso de evolución del ASC puede describirse como:

Operadores de proliferación clonal (TMA)
Suponiendo que:

donde Y(k) = TPC(A(k)) = ei •ai(k), i = 1,2,...,n, y ei es un qi-dimensional vector columna. Existen varios métodos para calcular qi. En este trabajo se calcula de la siguiente manera:

La función round(x) obtiene el entero más próximo a x, donde NC es llamado el tamaño clonal. El valor de qi (k) es proporcional al valor de F(a1(k)). Después de la proliferación clonal, la población se convierte en

donde

Operador de maduración de afinidad (TMA)
El operador de maduración de afinidad es implementado por mutación, esto es, cambios aleatorios son introducidos a los anticuerpos, tal como sucede en el sistema inmune natural. Dichos cambios pueden llevar a incrementar la afinidad. Por tanto, la mutación es realizada por el operador (TMA) el cual se aplica a la población Y(k) que se obtuvo después de la proliferación clonal z(k) = TMC(Y(k)).
El factor de mutación es calculado en base a la siguiente ecuación, propuesta en [28]:

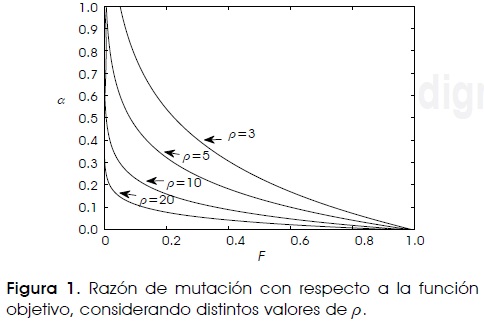
siendo a la razón de mutación, F el valor de la función objetivo correspondiente a cada anticuerpo (a1) y que está normalizada en el intervalo (0,1), mientras que ρ es un factor fijo. En [46] es demostrada la importancia de incluir el factor ρ en la ecuación (7) para mejorar la respuesta del algoritmo. La manera en que ρ modifica la razón de mutación es mostrada en la Figura 1.
El número de mutaciones realizadas a cada clon con respecto del valor de la función objetivo F, es igual a L•α, considerando a L como la longitud del anticuerpo. En otras palabras, mientras mayor sea la afinidad, menor será la mutación realizada, es decir, menos bits pertenecientes a ese anticuerpo en particular, serán modificados; el caso contrario será para individuos con baja afinidad.
Aplicando el operador de maduración de afinidad, la población se transforma en:

donde (TMA) es el operador tal como fue definido en la ecuación (7) y aplicado sobre el anticuerpo yij.
Operador de selección clonal (TSC)
Si se define bi(k) ∈ Zi(k) ∀i = 1,2,... n como el anticuerpo con la mayor afinidad en Zi(k), entonces a¡(k + 1) = TSC(Zi(k)∪ai(k)). Donde TSC es definido como:

De esta manera son seleccionados los n anticuerpos con mejor desempeño con respecto a la función a optimizar F(•).
Los pasos del ASC pueden ser definidos de la siguiente manera:
1. Se inicializa aleatoriamente una población (Pinit). Esto es, un conjunto de h = Pr+n soluciones candidatas donde n es el número de individuos de memoria (M), que se suma a la población remanente (Pr), resultando como población total PT=Pr+M.
2. Se seleccionan los n mejores individuos de la población total PT para construir A(k), de acuerdo a la medición de afinidad (función objetivo).
3. Empleando (TPC) se reproduce la población A(k), proporcionalmente a su afinidad con el antígeno (Ecuación 4), y se genera una población temporal de clones Y(k).
4. La población Y(k) de clones es mutada usando el parámetro TMA de acuerdo a la afinidad entre el anticuerpo y el antígeno (Ecuación 7). De lo anterior resulta una población madurada de anticuerpos Z(k).
5. Usando (TSC) se seleccionan los n mejores individuos de Z(k) para componer el nuevo conjunto de memoria M=A(k+1).
6. Se añaden Pr nuevos anticuerpos (introducción de diversidad) a los nuevos individuos de memoria M para construir la nueva población PT.
7. El algoritmo se detiene si cierto criterio de afinidad o un número de generaciones es alcanzado; de otra manera, se regresa al paso 2.
La Figura 2 muestra el flujo básico del algoritmo de selección clonal.
El número de anticuerpos en la memoria n, se selecciona haciendo un compromiso entre costo computacional y rapidez esperada del algoritmo. Dicho número en general depende del número de variables usadas en la optimización (número de elementos contenidos en el anticuerpo). Una buena aproximación como se propone en [47,48], resulta en considerar a n aproximadamente diez veces el número de variables contenidas en el anticuerpo.
La propiedad de similaridad49 dentro de los anticuerpos puede afectar la velocidad de convergencia del ASC. La idea de la incorporación de anticuerpos aleatorios (paso 6) se basa en la teoría de redes inmunes y es introducida para proveer diversidad a los individuos recién creados en M, los cuales podrían ser similares a los que se encuentran en la vieja memoria.
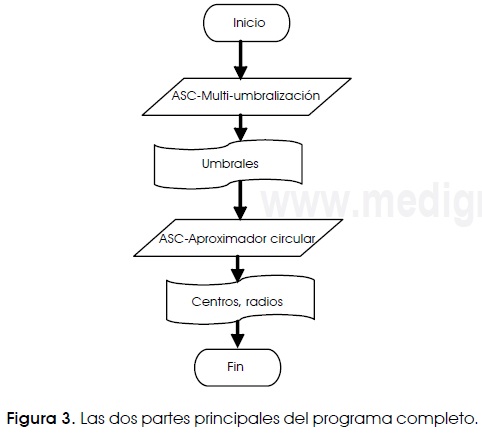
El algoritmo propuesto se compone de dos partes, una que corresponde a un ASC que es usado para encontrar los valores de la mezcla Gaussiana. En esta parte se obtienen los valores de umbral que segmentan a los glóbulos blancos. En la segunda parte, es usado otro ASC; durante esta fase, son encontrados los parámetros de los círculos que mejor aproximan a los leucocitos. En la Figura 3 se muestra el enfoque propuesto de segmentación y detección de GB.
ALGORITMO DE SEGMENTACIÓN
Aproximación gaussiana
Asumiendo una imagen con L niveles de intensidad (0,...,L-1) siguiendo una distribución que puede mostrarse por el histograma h(g). Para simplificar la descripción, el histograma está normalizado y es considerado como una función de densidad de probabilidad:
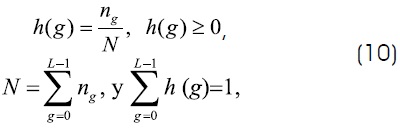
Además se asume que ng representa el número de pixeles con un nivel de gris g, mientras que N es el número total de pixeles en la imagen. La función del histograma puede ser modelada como una mezcla de funciones Gaussianas35,36, de tal manera que:
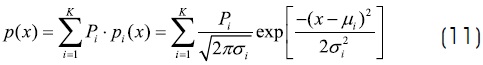
considerando que Pi es la probabilidad a priori de la clase i, pi(x) es la función de densidad de probabilidad de una variable aleatoria x en la clase i, mientras que µi y σi son la media y la desviación estándar de la i-th función Gaussiana, y Kes el número de clases dentro de la imagen. Además se deberá satisfacer la restricción 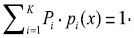 La función de costo usada en este artículo en la segmentación, para medir las afinidades antígeno-anticuerpo es
La función de costo usada en este artículo en la segmentación, para medir las afinidades antígeno-anticuerpo es

donde se asume un histograma de Np puntos como en [50] y es la penalidad asociada a la restricción 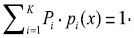
En general la estimación de los parámetros que minimicen el error producido por la mezcla Gaussiana no es un problema trivial. Un método simple es considerar las derivadas parciales de la función de error igualadas a cero, obteniendo un conjunto simultáneo de ecuaciones trascendentales. Sin embargo, es difícil de obtener una solución analítica debido a la naturaleza no-lineal de las ecuaciones. Los algoritmos, por lo tanto, hacen uso de aproximaciones iterativas basadas en la información del gradiente o la estimación de máxima probabilidad, como el algoritmo de expectación-maximización (EM). Desafortunadamente, tales métodos pueden fácilmente caer en mínimos locales. Por tales motivos, el ASC es usado en este trabajo para encontrar los parámetros de cada función Gaussiana, y los valores de umbralización correspondiente.
Cálculo de parámetros con el ASC
Para calcular los parámetros Pi, µi y σi que representan a cada función Gaussiana en la ecuación (11) se emplea el ASC. Aquí, i (∈1, 2, 3) define los objetos que serán segmentados: glóbulos blancos, glóbulos rojos y fondo. Cada anticuerpo será representado como una cadena de 22 bits de la forma:

donde Pi y µi son codificadas con 8 bits cada uno, mientras que o usa sólo 6 bits. Cada elemento del anticuerpo tiene los siguientes rangos:

donde I representa la imagen y L la escala de intensidad. La población inicial es creada considerando:

n representa el número de anticuerpos a ser creados, y 22 es el tamaño de cada cadena que representará al anticuerpo; de esta manera, están siendo creadas n cadenas de 22 números reales entre 1 y -1, que representan a toda la población inicial. Luego, a esta población inicial es aplicada la función signo con el objetivo de formar las cadenas binarias.
Para transformar una cadena binaria al valor real R es usada la siguiente expresión:

donde Rmaz es uno de los valores máximos definidos en las ecuaciones (14) para cada parámetro Pi, µi y σi.
Determinación de los valores de umbral
Una vez determinados los parámetros Pi, µi y σi de la función de probabilidad (11) que minimizan la función objetivo (12), el siguiente paso es determinar los valores óptimos de umbral. Considerando que las clases de pixeles están ordenadas tal que µ1 <µ2 <µ3, los valores de umbral pueden obtenerse calculando el error de probabilidad global para dos funciones Gaussianas adyacentes, siguiendo:

Considerando
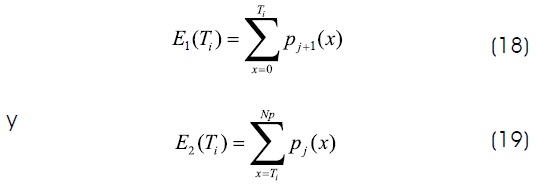
Donde E1(Ti) es la probabilidad de clasificar incorrectamente los pixeles de la clase j-th en la clase (j + 1 )-th, mientras E2(Ti) es la probabilidad de clasificar erróneamente los pixeles de la clase (j + 1 )-th en la clase j-th. Pj son las probabilidades a priori dentro de la mezcla de densidades de probabilidad, y Ti es el valor de umbral entre la clase j-th y la clase ( j + 1)-th. Los valores límite de cero y Np en (18) y (19) representan los límites representables en el histograma. El valor umbral T el cual minimiza el error E(Ti) es encontrado resolviendo:

considerando

Aunque la ecuación cuadrática anterior tiene dos soluciones posibles, sólo una de ellas es positiva y se encuentra dentro del intervalo. La Figura 4 ilustra el proceso de determinación del umbral entre dos clases.
ALGORITMO DE DETECCIÓN
Método
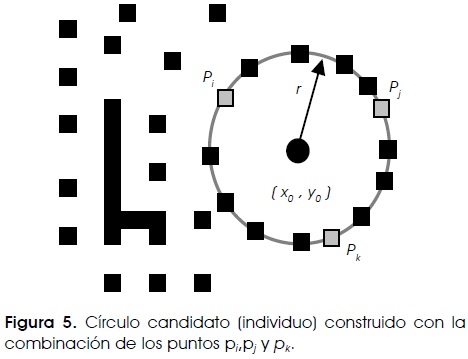
En este artículo cada leucocito es aproximado por el círculo que más se ajuste a su forma. Los círculos candidatos son modelados usando la ecuación de segundo grado (Ecuación 22), la cual define el centro y radio de cada circunferencia, a partir de las coordenadas de tres puntos no colineales51. Este enfoque además de ser computacionalmente económico, representa el número de puntos mínimo para definir una primitiva geométrica circular52.
Una vez segmentados los leucocitos usando el método descrito en la sección 3, se obtiene el mapa de bordes correspondiente, mediante la aplicación del gradiente morfológico50. Las coordenadas (xi, yi) de los puntos de borde obtenidos son luego almacenadas en un vector 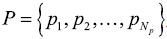 siendo Np el número total de pixeles.
siendo Np el número total de pixeles.
Para construir cada círculo-leucocito candidato (o anticuerpos, dentro del marco de SIA), los índices i1 i2 e i3 de tres puntos borde son combinados, asumiendo que la circunferencia del círculo pasa a través de los puntos  Un número de soluciones candidatos es generado aleatoriamente para la población inicial. Las soluciones evolucionarán a través de la aplicación del ASC y conforme el algoritmo itere sobre la población, un mínimo será alcanzado y el mejor individuo (que corresponde a ese mínimo) será considerado como la solución al problema de aproximación circular.
Un número de soluciones candidatos es generado aleatoriamente para la población inicial. Las soluciones evolucionarán a través de la aplicación del ASC y conforme el algoritmo itere sobre la población, un mínimo será alcanzado y el mejor individuo (que corresponde a ese mínimo) será considerado como la solución al problema de aproximación circular.
Aplicar métodos clásicos basados en la transformada de Hough o en contornos activos para detección de los GB requiere de una gran cantidad de memoria además de consumir demasiado tiempo de cómputo. Tales métodos también requieren un paso de recolección de primitivas geométricas potenciales, como el implementado en este artículo. Sin embargo, en la propuesta aquí presentada conforme el proceso evoluciona, la función objetivo mejora con cada generación, discriminando los círculos no-plausibles, evitando así visitar puntos innecesarios en la imagen. En la siguiente sección se describen los pasos requeridos para formular la tarea de detección de GB como un problema de optimización basado en SIA.
Representación de los individuos
En el ASC usado, cada anticuerpo C de la población usa tres puntos borde como elementos. En esta representación, los puntos de borde son almacenados de acuerdo a un índice relativo a su posición dentro del arreglo P. El procedimiento codifica un anticuerpo como el círculo que pasa a través de tres puntos Pi, Pj y Pk (C = {Pi, Pj, Pk}). Los parámetros del círculo candidato tales como el centro (x0 ,y0) y el radio r que corresponden al anticuerpo C se obtienen a partir de la siguiente ecuación:

Considerando

siendo det(.) el determinante y d ∈ {i, ¡, k}. Sin embargo, los resultados de las ecuaciones (24) y (25) deben de ser redondeados para que puedan ser usados en el contexto de la aplicación.
La Figura 5 ilustra los parámetros definidos por las Ecuaciones 22 a 25. En este caso, es posible representar los parámetros [x0, y0,r] como una transformación R del vector de índices i, ¡ y k.
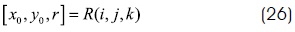
De esta forma se recorre el espacio de la imagen de bordes buscando los parámetros que mejor aproximen a las formas circulares contenidas, usando como mecanismo de optimización el ASC. Este enfoque reduce el espacio de búsqueda eliminando soluciones no factibles.
Función objetivo
La existencia de una posible circunferencia en una imagen puede ser verificada como la manera en que una forma virtual circular concuerda con los puntos borde de un círculo real en la imagen. Una función objetivo validará la relación existente entre el círculo candidato C (anticuerpo) y el contenido presente en la imagen (antígeno). Para realizar tal prueba se considera un vector de puntos S = {s1, s2,...,sNs}, siendo Ns el número de puntos de prueba sobre los cuales se verificará la existencia de un punto borde.
El conjunto de prueba S es generado por el algoritmo de círculo de punto central (Midpoint Circle Algorithm-MCA)53. El MCA calcula considerando un radio r y las coordenadas del centro los Ns puntos requeridos para representar el círculo, produciendo el vector de prueba S. El algoritmo emplea la ecuación del círculo x2 + y2 = r2 con sólo el primer octante. Dibuja una curva iniciando en el punto (r, 0) y continúa arriba y hacia la izquierda usando sumas y restas de enteros. Ver detalles completos en [54]. Aunque el algoritmo es considerado el más rápido y que provee una precisión de sub-pixel, es importante asegurar que los puntos que caigan fuera del plano de la imagen no sean considerados ni incluidos en el vector S.
La función de costo o función objetivo J(C) calcula la correspondencia (o error) resultante de los pixeles S del círculo candidato y los pixeles realmente existentes en la imagen de bordes, resultando:
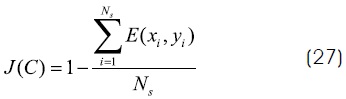
donde E(Xi, Yi) es una función que comprueba la existencia del pixel (Xi, Yi) esto es:

La función objetivo en (27) acumula el número de puntos que de acuerdo E(Xi, Yi) existen en la imagen de bordes. Los pixeles S bajo prueba, constituyen el perímetro del círculo que corresponden a C. Por tanto el algoritmo busca minimizar J(C) dado que un valor pequeño implica una mejor respuesta (o correspondencia) de la «circularidad». Debido a que la función objetivo (27) valida la relación existente entre el círculo candidato C, definido por la ecuación (22), y el círculo presente en la imagen, calculado a partir del algoritmo MCA; no es necesario añadir ninguna restricción adicional para la discriminación entre formas circulares y otros lugares geométricos como elipses, hipérbolas, etc.22,52.
Considerando este enfoque propuesto el algoritmo es capaz de hallar la figura circular que de acuerdo a J(C) presenta el valor más pequeño; además, es importante considerar que cualquier otra figura presente en la imagen que no correspondan a GB (ruido asociado u otros elementos), ya habrán sido descartadas en el paso de segmentación del algoritmo propuesto.
Finalmente, el proceso de optimización se detiene después de un número máximo de iteraciones o cuando la diferencia entre una figura circular y los anticuerpos sea menor que un umbral predefinido.
RESULTADOS EXPERIMENTALES
Segmentación
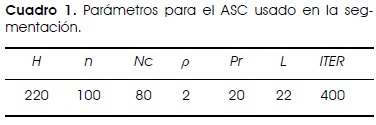
En esta sección son mostrados los resultados correspondientes a la segmentación de las imágenes del frotis sanguíneo, esta umbralización resulta en la segmentación de glóbulos blancos, glóbulos rojos y fondo. Una mejora previa de la imagen puede conducir a una respuesta del algoritmo más adecuada (vea por ejemplo [55]), aunque en los resultados presentados ningún algoritmo de pre-procesamiento fue aplicado. Las pruebas fueron realizadas usando los parámetros mostrados en el Cuadro 1, los cuales fueron determinados después de excesiva experimentación. Las Figuras 6 y 7 muestran los resultados obtenidos.
Cada anticuerpo es tridimensional, definido como  donde h representa el número de individuos, mientras que el subíndice indica el tamaño de cada anticuerpo en bits. Los individuos son inicializados aleatoriamente como establece el ASC y una vez realizadas las iteraciones son encontrados los valores de cada Gaussiana. Con esta información se obtienen los umbrales que permiten clasificar a los GB. Finalmente, la imagen con los bordes obtenidos por el algoritmo del gradiente morfológico50 es alimentada al siguiente ASC que realiza la detección.
donde h representa el número de individuos, mientras que el subíndice indica el tamaño de cada anticuerpo en bits. Los individuos son inicializados aleatoriamente como establece el ASC y una vez realizadas las iteraciones son encontrados los valores de cada Gaussiana. Con esta información se obtienen los umbrales que permiten clasificar a los GB. Finalmente, la imagen con los bordes obtenidos por el algoritmo del gradiente morfológico50 es alimentada al siguiente ASC que realiza la detección.
Detección
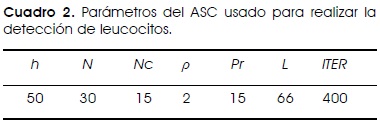
La imagen que contiene los bordes de los GB se convierte en la entrada del ASC que realiza la detección. Los mejores parámetros encontrados, usados para la detección de círculos por el ASC son mostrados en el Cuadro 2.
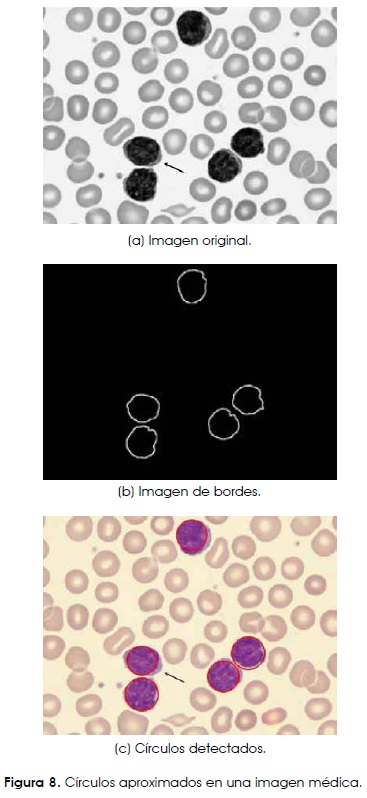
En imágenes reales es poco común encontrar círculos perfectos, incluso cuando los círculos físicos lo son, esto debido principalmente a que las características de la cámara, lentes, iluminación y otros factores tienen implicaciones al momento de tomar la imagen. Por otro lado, varios problemas suelen presentarse en la detección de GB tales como la presencia de altos niveles de ruido, el traslape de estructuras o bien su oclusión, lo cual incrementa notablemente la complejidad del reconocimiento. El ASC propuesto es capaz de encontrar el individuo candidato que presenta una mayor afinidad al GB contenido en la imagen considerando todos estos factores. El resultado del método de detección propuesto puede verse en la Figura 8.
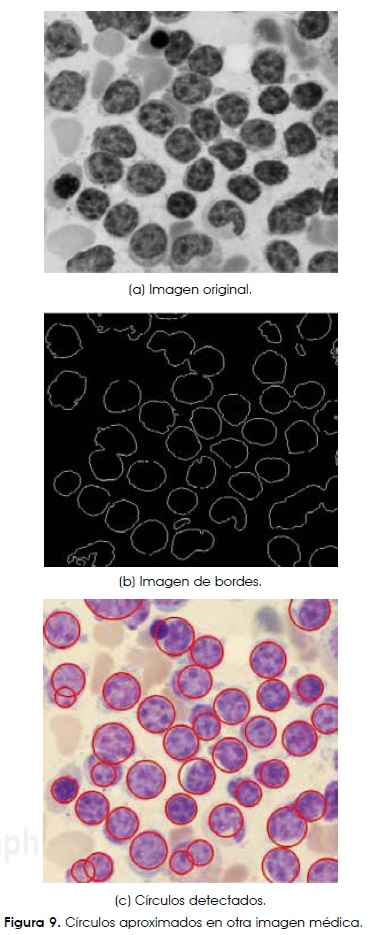
Un caso más complejo se muestra en la Figura 9, en ella pueden verse distintos contactos entre células, células parcialmente ocluidas, conglomerados de células entre otros; además, en las células que presentan una forma alargada, en general el algoritmo intenta aproximarlas con más de un círculo. Es importante notar que la información correspondiente a los parámetros de cada círculo (tal como sus centros y radios) pueden ser utilizados para el cálculo del área relacionada con cada una de las células encontradas o simplemente encontrar su posición relativa dentro del frotis.
COMPARACIÓN
Con el objetivo de medir el desempeño del método propuesto en este artículo, se compararon sus resultados con los obtenidos por el algoritmo propuesto por Wang et al. en [9], el cual es considerado como un algoritmo preciso y moderno para la detección de GB por parte de la comunidad biomédica.
Para el algoritmo propuesto en [11] la estructura de la red y el valor de parámetros se consideró siguiendo la configuración sugerida por los mismos autores en [11], mientras que para el método aquí propuesto se configuró a los parámetros de cada ASC con los valores reportados en los Cuadros 1 y 2.
La Figura 10 muestra el resultado obtenido por ambos métodos al considerar una imagen que contiene 2 GB. De la Figura 10b resulta evidente que el método Wang permite detectar sólo un GB. Lo anterior se debe a que este algoritmo en su evolución necesita información local y global de la imagen, lo que dificulta su generalización a varios contornos activos. Por otro lado, en la Figura 10c se muestra el resultado obtenido con el método propuesto en este artículo.
Una desventaja importante en el método propuesto por Wang es la falta de estabilidad del método. Esto es, si se deja iterar exageradamente el algoritmo, los contornos activos que cubren el GB degeneran en figuras delimitadas por otras estructuras vecinas a los GB. La Figura 11a muestra el resultado del algoritmo Wang si se permite iterar hasta 400 ciclos, en tanto la Figura 11b muestra el resultado del método propuesto permitiendo que el algoritmo itere hasta 1,000 generaciones.
Con el objetivo de comparar la precisión de ambos métodos, se consideró el área que ambos enfoques aproximan con respecto al área real del GB. Estas mediciones consideraron diferentes grados de evolución de los algoritmos, tomando en cuenta un solo GB (el único capaz de detectar el método Wang). Los resultados obtenidos son mostrados en el Cuadro 3. El Error reportado en el Cuadro 1 representa el promedio obtenido después de realizar 20 veces cada prueba.
CONCLUSIÓN
En este trabajo se presenta un método para detectar glóbulos blancos presentes en imágenes de frotis sanguíneos, mediante el uso del ASC. Tal enfoque se plantea en dos partes; la primera corresponde a segmentación y la segunda a la detección. En la segmentación la técnica propuesta divide a cada frotis en tres regiones: leucocitos, glóbulos rojos y fondo. Esto se logra modelando al histograma de los pixeles que pertenecen a las regiones de la imagen como una mezcla de Gaussianas, tal que cada una de las funciones Gaussianas que participan en la combinación representa una de las clases o región33,34. Los parámetros de las funciones Gaussianas son calculados automáticamente por medio del ASC con sólo el número de clases deseadas como información previa. Resultados experimentales muestran que la aproximación propuesta es capaz de producir resultados satisfactorios, dejando en claro la capacidad del algoritmo para realizar la selección de umbrales automáticamente, mientras preserva las principales características de la imagen.
La segunda parte corresponde a la detección, la cual opera con la información obtenida en el primer paso. La detección de GB es considerado como un problema de optimización entre el leucocito y su mejor aproximación circular, por lo que el algoritmo utiliza la codificación de tres puntos no colineales de la imagen de bordes para modelar los leucocitos candidatos. Una función de costo evalúa si tales leucocitos candidatos están realmente en la imagen. Siguiendo los valores de tal función de costo, los leucocitos candidatos son modificados usando el ASC hasta aproximarse a los leucocitos reales en la imagen de bordes. De este enfoque propuesto resulta un detector de precisión sub-pixel capaz de identificar leucocitos en imágenes, a pesar de que éstos presenten significativas porciones ocluidas o traslapadas.
Una característica importante de este trabajo es considerar la tarea de detección de GB como un problema de optimización. Tal enfoque permite al algoritmo explorar estructuras traslapadas u ocluidas, ya que el algoritmo encuentra la aproximación circular que mejor se relaciona de acuerdo a J(C) con la estructura.
Para probar el rendimiento del algoritmo propuesto se evaluó objetivamente la precisión obtenida comparado con el algoritmo propuesto por Wang et al.9, el cual es un enfoque considerado parte del estado del arte en la detección de GB. Resultados experimentales demostraron que aunque el algoritmo de Wang es ligeramente más preciso, el algoritmo propuesto es superior al permitir detectar múltiples GB y no presentar inestabilidad en los resultados.
RECONOCIMIENTO
El Primer autor agradece el financiamiento recibido por PROMEP-SEP para la realización de esta investigación.
REFERENCIAS
1. Mitchell S, Elkind M, Jianfeng C, Tanja R, Bernadette B, Ralph L. Leukocyte count predicts outcome after ischemic stroke: The Northern Manhattan Stroke Study. Journal of Stroke and Cerebrovascular Diseases 2004; 13(5): 220-227. [ Links ]
2. Pappua V, Bagchi P. 3D computational modeling and simulation of leukocyte rolling adhesion and deformation. Computers in Biology and Medicine 2008; 38(6): 738-753. [ Links ]
3. Ritter N, Cooper J. Segmentation and border identification of cells in images of peripheral blood smear slides. In Proceedings of the Thirtieth Australasian Conference on Computer Science 2007; 62: 161-169. [ Links ]
4. Ongun G, Halici U, Leblebicioglu K, Atalay V, Beksac M, Beksac S. Feature extraction and classification of blood cells for an automated differential blood count system. Neural Networks. Proceedings. IJCNN 2001; 4: 2461-2466. [ Links ]
5. Jiang K, Liao QM, Dai SY. A novel white blood cell segmentation scheme using scale-space filtering and watershed clustering. Machine Learning and Cybernetics, International Conference on, 5, 2003: 2820-2825. [ Links ]
6. Dorini LB, Minetto R, Leite NJ. White blood cell segmentation using morphological operators and scale-space analysis. In SIBGRAPI '07: Proceedings of the XX Brazilian Symposium on Computer Graphics and Image Processing. [ Links ]
7. Scotti F. Automatic morphological analysis for acute leukemia identification in peripheral blood microscope images. In 2005 IEEE International Conference on Computational Intelligence for Measurement Systems and Applications, CIMSA, 2005: 96-101. [ Links ]
8. Kumar B, Joseph D, Sreenivasc T. Teager energy based blood cell segmentation. Digital Signal Processing. DSP 14th International Conference on, 2, 2002: 619-622. [ Links ]
9. Cseke I. A fast segmentation scheme for white blood cell images. In Pattern Recognition. Conference Image, Speech and Signal Analysis, Proceedings. 11th IAPR International Conference 2002; 3: 530-533. [ Links ]
10. Otsu N. A threshold selection method from gray-level histograms. Automatica 1975; 11: 285-296. [ Links ]
11. Wang S, Korris FL, Fu D. Applying the improved fuzzy cellular neural network IFCNN to white blood cell detection, Neuro-computing 2007; 70: 1348-1359. [ Links ]
12. Muammar H, Nixon M. Approaches to extending the Hough transform. Proc. Int. Conf. on Acoustics, Speech and Signal Processing ICASSP-89, 1989; 3: 1556-1559. [ Links ]
13. Atherton TJ, Kerbyson DJ. Using phase to represent radius in the coherent circle Hough transform, proc. IEE Colloquium on the Hough Transform, IEE, London (1993). [ Links ]
14. Fischer M, Bolles R. Random sample consensus: A paradigm to model fitting with applications to image analysis and automated cartography. CACM 1981; 24(6): 381-395. [ Links ]
15. Shaked D, Yaron O, Kiryati N. Deriving stopping rules for the probabilistic hough transform by sequential analysis, comput. Vision Image Understanding, 1996; 63: 512-526. [ Links ]
16. Xu L, Oja E, Kultanen P. A new curve detection method: Randomized hough transform (RHT), pattern Recognition Lett. 1990; 11(5): 331-338. [ Links ]
17. Han JH, Koczy LT, Poston T. Fuzzy Hough transform. Proc. 2nd Int. Conf. on Fuzzy Systems, 1993; 2: 803-808. [ Links ]
18. Lu W, Tan JL. Detection of incomplete ellipse in images with strong noise by iterative randomized Hough transform (IRHT). Pattern Recognition 2008; 41(4): 1268-1279. [ Links ]
19. Roth G, Levine MD. Geometric primitive extraction using a genetic algorithm. IEEE Trans. Pattern Anal. Machine Intell. 1994; 16(9): 901-905. [ Links ]
20. Lutton E, Martinez P. A genetic algorithm for the detection 2-D geometric primitives on images. Proc. of the 12th Int. Conf. on Pattern Recognition, 1994; 1: 526-528. [ Links ]
21. Yao J, Kharma N, Grogono P. Fast robust GA-based ellipse detection, Proc. 17th Int. Conf. on Pattern Recognition ICPR-04, 2, Cambridge, UK, 2004: 859-862. [ Links ]
22. Ayala-Ramírez V, García-Capulin CH, Pérez-García A, Sánchez-Yáñez ER. Circle detection on images using genetic algorithms. Pattern Recognition Letters 2006; 27(6): 652-657. [ Links ]
23. Dasgupta S, Das S, Biswas A, Abraham A. Automatic circle detection on digital images whit an adaptive bacterial foraging algorithm. Soft Computing, 2009; DOI 10.1007/s00500-009-0508-z. [ Links ]
24. Rosin PL, Nyongesa HO. Combining evolutionary, connectionist, and fuzzy classification algorithms for shape analysis. Cagnoni, S. et al. (Eds.), Proc. EvoIASP, Real-World Applications of Evolutionary Computing, 2000: 87-96. [ Links ]
25. Rosin PL. Further five point fit ellipse fitting, proc. 8th British Machine Vision Conf., Cochester, UK, 1997: 290-299. [ Links ]
26. Goldsby GA, Kindt TJ, Kuby J, Osborne B.A. Immunology, fifth ed., Freeman, New York, NY, 2003. [ Links ]
27. de Castro LN, Timmis J. Artificial Immune Systems: A New Computational Intelligence Approach, springer, London, UK,2002. [ Links ]
28. Dasgupta D. Advances in artificial immune systems, IEEE Computational Intelligence Magazine 2006; 1(4): 40-49. [ Links ]
29. Wang X, Gao XZ, Ovaska SJ. Artificial immune optimization methods and applications - a survey, proceedings of the IEEE International Conference on Systems, Man, and Cybernetics, The Hague, The Netherlands, October 2004: 3415-3420. [ Links ]
30. de Castro LN, von Zuben FJ. Learning and optimization using the clonal selection principle, IEEE Transactions on Evolutionary Computation 2002; 6(3): 239-251. [ Links ]
31. Ada GL, Nossal G. The clonal selection theory. Sci Am 1987; 257: 50-57. [ Links ]
32. Coello-Coello CA, Cortes NC. Solving multiobjective optimization problems using an artificial immune system. Genet Program Evolvable Mach 2005; 6: 163-190. [ Links ]
33. Campelo F, Guimaraes FG, Igarashi H, Ramirez JA. A clonal selection algorithm for optimization in electromagnetics. IEEE Trans Magn 2005; 41: 1736-1739. [ Links ]
34. Weisheng D, Guangming S, Li Z. Immune memory clonal selection algorithms for designing stack filters. Neurocomputing 2007; 70: 777-784. [ Links ]
35. Cuevas E, Zaldivar D, Perez-Cisneros M. A novel multi-threshold segmentation approach based on differential evolution optimization. Expert Systems with Applications 2010; 37(7): 5265-5271. [ Links ]
36. Cuevas E, Zaldivar D, Perez-Cisneros M. Seeking multi-thresholds for image segmentation with Learning Automata. Machine Vision and Applications, 2010; DOI 10.1007/s00138010-0249-0. [ Links ]
37. Orhan E, Alper D. Artificial Immune Systems and applications in industrial problems. G.U. Journal of Science 2004; 17(1): 71-84. [ Links ]
38. Poli R, Langdon WB. Foundations of Genetic Programming. Springer, Berlin, Germany (2002). [ Links ]
39. Fabio F, Maurizio R. Comparison of artificial immune systems and genetic algorithms in electrical engineering optimization. COMPEL: The International Journal for Computation and Mathematics in Electrical and Electronic Engineering 2006; 25: 792-811. [ Links ]
40. Gao X, Wang X, Ovaska S. Fusion of clonal selection algorithm and differential evolution method in training cascade-correlation neural network. Expert Systems 2008; doi: 10.1016/j. neucom.2008.11.004. [ Links ]
41. Yoo J, Hajela P. Immune network simulations in multicriterion design. Structural Optimization 1999; 18(2): 85-94. [ Links ]
42. Wang X, Gao XZ, Ovaska SJ. A hybrid optimization algorithm in power filter design. Proceedings of the 31st Annual Conference of the IEEE Industrial Electronics Society, Raleigh, NC, 2005: 1335-1340. [ Links ]
43. Xu X, Zhang J. An improved immune evolutionary algorithm for multimodal function optimization. Proceedings of the Third International Conference on Natural Computation, Haikou, China, 2007: 641-646. [ Links ]
44. Tang T, Qiu J. An improved multimodal artificial immune algorithm and its convergence analysis. Proceedings of the Sixth World Congress on Intelligent Control and Automation, Dalian, China, 2006: 3335-3339. [ Links ]
45. Gong M, Jiao L, Zhang L, Du H. Immune secondary response and clonal selection inspired optimizers. Progress in Natural Science 2009; 19: 237-253. [ Links ]
46. Cutello V, Narzisi G, Nicosia G, Pavone M. Clonal selection algorithms: A comparative case study using effective mutation potentials. C. Jacob et al. (Eds.): ICARIS 2005, LNCS 2005; 3627: 13-28. [ Links ]
47. Dong W, Shi G, Zhang L. Immune memory clonal selection algorithms for designing stack filters. Neurocomputing 2007; 70(4): 777-784. [ Links ]
48. Gong M, Jiao L, Zhang L. Baldwinian learning in clonal selection algorithm for optimization. Information Sciences 2010; 180(8): 1218-1236. [ Links ]
49. Gong M, Jiao L, Zhang X. A population-based artificial immune system for numerical optimization. Neurocomputing 2008; 72: 149-161. [ Links ]
50. Gonzalez RC, Woods RE. Digital Image Processing. (3rd Edition), Prentice-Hall, Inc., Upper Saddle River, NJ, USA, 2006. [ Links ]
51. Fischer M, Bolles R. Random sample consensus: A paradigm to model fitting with applications to image analysis and automated cartography. CACM 1981; 24(6): 381-395. [ Links ]
52. Cuevas E, Zaldivar D, Pérez-Cisneros M, Ramírez-Ortegón M. Circle detection using discrete differential evolution optimization. Pattern Analysis & Applications, DOI: 10.1007/ s10044-010-0183-9, 2010. [ Links ]
53. Bresenham JE. A Linear Algorithm for Incremental Digital Display of Circular Arcs. Communications of the ACM 1987; 20: 100-106. [ Links ]
54. Van Aken JR. An efficient ellipse drawing algorithm. CG & A 1984; 4(9): 24-35. [ Links ]
55. Xu X, Zhang J. An improved immune evolutionary algorithm for multimodal function optimization. Proceedings of the Third International Conference on Natural Computation, Haikou, China, 2007: 641-646. [ Links ]
Nota
Este artículo también puede ser consultado en versión completa en: http://www.medigraphic.com/ingenieriabiomedica/