INTRODUCCIÓN
El cultivo de camarón es una industria productiva a nivel mundial con un crecimiento exponencial en la última década. El principal estado productor de camarón en nuestro país es Sinaloa con más de 96 mil toneladas, le sigue Sonora con 75 mil toneladas y Nayarit con 17 mil toneladas (García, 2020). En el país existen 1,447 granjas, con un área total de 45,000 hectáreas destinadas al cultivo de camarón; esta actividad es el sustento de miles de habitantes de las costas de Sonora, Sinaloa, Nayarit, Baja California Sur y Tamaulipas, además de otros 11 estados, lugares donde se concentra la producción de 237 mil 647 toneladas (CONAPESCA, 2018; García, 2020).
El mayor problema al que se había enfrentado la camaronicultura en el país eran las epizootias causadas por infecciones virales. Dentro de los patógenos que ocasionan las mayores pérdidas a la industria mexicana se encuentran los virus de la necrosis hipodérmica y hematopoyética infecciosa (IHHNV= Infectious hypodermal and hematopoietic necrosis virus), del síndrome de Taura (TSV= Taura syndrome virus) y el virus del síndrome de mancha blanca (WSSV=White spot syndrome virus) (Lightner & Redman, 1998; Flegel et al., 2008; Lightner, 2011). Sin embargo, desde el 2009 se presentó en Asia una enfermedad bacteriana emergente llamada síndrome de la muerte temprana (EMS= Early mortality syndrome) o enfermedad de la necrosis hepatopancreática aguda (AHPND= Acute hepatopancreatic necrosis disease), el agente causal es una cepa nueva de Vibrio parahaemolyticus (VP-AHPND+) (Flegel, 2012; Tran et al., 2013). La enfermedad EMS/AHPND se propagó en el 2010 hacia granjas del sur de China y la isla de Hainan; para principios de 2011 se había detectado en Vietnam y Malasia (Lightner et al., 2012), lo que ocasionó pérdidas billonarias en producción. En el noroeste de México (Nayarit, Sinaloa y Sonora) se presentaron epizootias severas en el cultivo de camarón desde el 2013 (SENASICA, 2014) las cuales generaron grandes pérdidas de hasta el 80% en la producción en el inicio del cultivo, debido a patologías similares a AHPND (Tran et al., 2013), en México ya existían reportes en Sinaloa (Soto-Rodriguez et al., 2015), donde actualmente continúan las epizootias (CESASIN, 2020). Por consiguiente, el comité Estatal de Sanidad Acuícola del Estado de Sonora A. C. (COSAES) reportó pérdidas millonarias, pues si en el 2009 la producción alcanzó 81,423 toneladas, en 2013 solo llegó a 13,192 toneladas, por lo que se recomendó cerrar las granjas en el siguiente ciclo 2014, aunque ya no es tan grave la enfermedad en la camaronicultura, la producción continúa con pérdidas (COSAES, 2017). Las estimaciones anteriores del impacto económico acumulativo de AHPND han oscilado entre $ 8 mil millones de dólares para Asia y $ 4 mil millones para Estados Unidos (Aranguren-Caro et al., 2020), país donde iniciaron las mortalidades en el 2017 (Dhar et al., 2019).
La enfermedad AHPND se caracteriza por el desprendimiento masivo de los túbulos de las células epiteliales del hepatopáncreas (HP), causada por las toxinas PirA y PirB de V-AHPND+. El diagnóstico preciso se basa en la detección de una región intergénica de PirA para validar la presencia específica del patógeno (Sirikharin et al., 2015). Es importante subrayar la resistencia a antibióticos que han desarrollado algunas cepas de VP-AHPND+ a la oxitetraciclina y tetraciclina relacionada con el gen tet, transportado por un plásmido; otros aislados presentan resistencia a las quinolonas, probablemente por la adquisición de plásmidos que median la resistencia a las quinolonas (PMQR) o debido a los cambios en expresión de las bombas de expulsión y porinas que controlan la cantidad de droga en la célula bacteriana (Han et al., 2015; Rortana et al., 2018).
Debido a la importancia que tiene AHPND, los objetivos del presente trabajo fueron (1) identificar los aislados bacterianos de las cepas causantes de epizootias mediante análisis por PCR con los primers AP3 específicos (2) realizar comparaciones genómicas de secuenciación de la región 16S ADNr de las cepas colectadas durante los ciclos de cultivo 2013 y 2014 para determinar si el agente causal de las mortalidades atípicas del camarón en las granjas del estado de Sonora son la misma o diferentes cepas de V. parahaemolyticus que causa la VP-AHPND+ en Asia; (3) Determinar el perfil genómico con elementos de PCR palindrómicos extragénicos repetitivos, conocido como rep-PCR, con los primers GTC5 y (4) evaluar la resistencia de VP-AHPND+ a antibióticos comerciales.
MATERIALES Y MÉTODOS
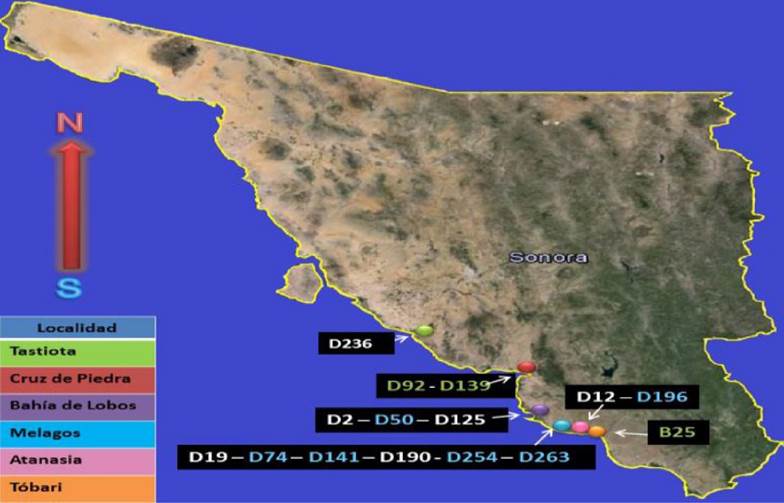
Área de estudio: En el ciclo de cultivo del 2013, los sitios de muestreo fueron nueve, seis de ellos ubicados en la Junta Local de Sanidad Acuícola (JLSA) de Cruz de Piedra en el centro de Sonora (27°57’3” Latitud Norte y 110°40’33” de Longitud Oeste), una en Tobari (27°03´00” LN y 109°53´00” LO) y dos en Atanasia (26° 47’ 2.59” LN y 109° 48’ 48.20” LO). Las muestras incluyeron camarón (silvestre y de cultivo), sedimento y agua.
Para las muestras del ciclo 2014 se monitorearon cuerpos de agua (esteros y drenes) circundantes a las granjas que presentaron mortalidad en el ciclo 2013, las localidades son en los esteros de Melagos (27° 09’ 46.90” LN, 110° 15’ 58.49” LO), Atanasia (27° 08’ 08.85” LN, 110° 11’ 27.67” LO) y El Riito (26° 47’ 2.59” LN, 109° 48’ 48.20” LO) (COSAES, 2017).
Colecta de muestras: Las muestras se colectaron asépticamente en bolsas estériles (Whirl-pack, Fort Atkinson, Wisconsin) y consistieron en 100 mL de agua y 50 g de sedimento de cada sitio, introduciéndolas en las columnas de agua o esteros adjuntos al área de suministro de las granjas. Éstas se almacenaron en hieleras para trasportarlas y analizarlas en el laboratorio como lo recomienda la Norma oficial Mexicana (NOM-230-SSA1-2002). Para la captura de los camarones silvestres o de cultivo, se utilizó el muestreo dirigido con una atarraya convencional, mediante, en el cual se colectaron organismos con características o signos anormales que presumiblemente estén asociados con alguna enfermedad (OIE, 2013). Para la transportación de los organismos vivos al laboratorio se utilizaron acuarios de plástico limpios con agua del mismo sitio (Joshi et al., 2014).
Análisis bacteriológicos de agua: se sembraron directamente 100 μL de la muestra en placa en el medio de cultivo TCBS (Thiosulfate Citrate Bile Salts Sucrose; DIFCO, BD, México), posteriormente se incubaron por 18 a 24 h a 28 - 30 °C (Gomez-Gil et al., 1998; Soto-Rodriguez et al., 2010). En el caso del sedimento este se mezcló 1:1 con agua marina estéril y se maceró con un mortero, posteriormente se centrifugó para separar las bacterias del sedimento para diluirlas en forma seriadas (10-1 - 10-6), sembrándose 100 mL en el medio de cultivo (Galaviz-Silva et al., 2018).
Análisis bacteriológico de postlarvas (PL´s): se pesó un gramo de muestra y se maceró en 10 mL de solución salina al 2% estéril, después se sembraron 100 μL en el medio de cultivo TCBS y se incubaron de 18 a 24 h a una temperatura de 28 a 30 °C (Gomez-Gil et al., 1998; Soto-Rodriguez et al., 2010). Para el análisis bacteriológico de HP en juveniles, se utilizaron pinzas de disección estériles y bajo condiciones asépticas se pesó 1 g de cada uno para macerarse en 10 mL de solución salina al 2% estéril en mortero; de este se cultivaron 100 μL en TCBS y se prosiguió como en el caso de las PL´s (Gomez-Gil et al., 1998; Soto-Rodriguez et al., 2010).
Aislamiento y purificación de cepas bacteriológicas: Para la pre-selección de colonias de VP se incluyó únicamente las colonias verdes del agar TCBS (crecimiento típico de V. parahaemolyticus) con o sin presencia de bioluminiscencia y se resembró en agar tripticaseina de soya (TSA; DIFCO, BD) + 2% de NaCl y en CHROMagar Vibrio que es un medio diferencial para VP, pero no para VP-AHPND+, donde crece de color malva (CHROMagar, Paris, Francia), el método que se utilizó fue por estría cruzada a una temperatura de 35°C por 24 h (Di-Pinto et al., 2011). Estas cepas de VP fueron agrupadas por área de colecta y tipo de muestra (sedimento, agua, PL´s o juveniles), seleccionándose solo una para la identificación definitiva por PCR con primers específicos para VP-AHPND+.
En el 2013 se revisaron 106 casos y en el 2014 fueron 496 (Tabla 1 y 2, respectivamente).
Tabla 1 Aislados bacterianos de Vibrio parahaemolyticus (no confirmados AHPND+) en agua, sedimento y camarón del ciclo de cultivo 2013 en tres Juntas Locales de Sanidad Acuícola (JLSA) en el sur de Sonora.
| JLSA | Agua (N/Pa) | Sedimento (N/P) | Larva (N/P) | HP b(N/P) | Total (N/P) |
| Atanasia | 8/6* | 7/5 | 5/3* | 3/3 | 23/17 |
| Mélagos | 8/5* | 5/4* | 7/7* | 3/3* | 23/19 |
| El Riito | 5/1 | 5/2 | 30/27 | 20/16 | 60/46 |
| Total | 21/12 | 17/11 | 42/37 | 26/22 | 106/82 |
a (N/P) = (Número de casos/Positivos); b HP= Hepatopáncreas. Se marcan con (*) el tipo de muestra donde se detectaron los casos AHPND+.
Tabla 2 Aislados bacterianos de V. parahaemolyticus (no confirmados AHPND+) en agua, sedimento y camarón durante el ciclo de cultivo 2014 en 11 Juntas Locales de Sanidad Acuícola (JLSA) en el estado de Sonora.
| JLSA | Agua (N/Pa) | Sedimento (N/P) | Larva (N/P) | HP b(N/P) | Total (N/P) |
| Aquiropo | - | - | - | 7/5 | 7/5 |
| Atanasia | 20/16 | 5/3 | 10/4 | 30/28 | 65/51 |
| Bahía de Kino | 4/2 | - | 1/1 | 7/6 | 12/9 |
| Bahía de Lobos | 40/30* | 20/15* | - | 25/22* | 85/67 |
| Cruz de Piedra | 10/7 | 10/5 | 10/1 | 10/5** | 40/18 |
| Mélagos | 40/39* | 15/10 | 15/11 | 70/61* | 140/121 |
| El Riito | 5/3 | 5/2 | - | 10/6 | 20/11 |
| Santa Bárbara | - | - | 5/1 | 5/1 | 10/2 |
| Siari | 2/2 | - | - | - | 2/2 |
| Tastiota | 10/2 | - | - | 10/9* | 20/11 |
| Tobari | 15/13* | 10/5 | 10/4* | 30/21 | 65/43 |
| *Sin definir | 10/8 | - | - | 20/14 | 30/22 |
| Total | 156/122 | 65/40 | 51/22 | 224/178 | 496/362 |
a(N/P) = Número de casos/Positivos; b HP = Hepatopáncreas; SM: Sin muestra. Se marcan con (*) el tipo de muestra donde se detectaron los casos AHPND+.
Patogenicidad de las cepas: Las colonias con crecimiento en CHROMagar Vibrio y positivas por PCR, fueron inoculadas en camarones de 1 g (Megalarvas de Sinaloa, S. de R.L.) para comprobar el segundo y tercer postulado de Koch (El microorganismo debe poder ser extraído y aislado de un organismo enfermo y cultivarse en un cultivo puro y el microorganismo que se ha cultivado en un cultivo debería poder causar la enfermedad una vez introducido en un organismo sano, respectivamente). Se inoculó un grupo de cinco camarones por cada cepa de VP-AHPND+ en acuarios previamente desinfectados y separados en agua marina sintética (35 ‰, Rangen, Buhl, ID, USA); los retos de patogenicidad se realizaron por inmersión por 15 min con cultivos frescos de las cepas (incubados 24 h a 30 °C, a una concentración de 0.5 de la escala de McFarland). Después de la inmersión, los camarones se regresaron al acuario correspondiente junto con el inóculo (Tran et al., 2013). El caldo soya tripticaseina (TSB) estéril + 2 % NaCl se usó como control negativo. El control positivo consistió en el inóculo con la cepa VP-AHPND+ MC32, proporcionada por Ricardo Sánchez Díaz. Los camarones retados en los bioensayos se mantuvieron en observación hasta la manifestación de los signos clínicos de la AHPND (anorexia, nado errático, HP pálido, intestino vacío y letargia (Soto-Rodríguez et al., 2015); de los camarones enfermos se obtuvieron muestras para histopatología y PCR.
Caracterización molecular: La extracción del ADN de las cepas bacterianas obtenidas de estómago, HP, agua y sedimento, se realizó con DNAzol (Invitrogen, Carlbad, CA., USA), según las instrucciones del fabricante. Se utilizaron 100 ng de ADN de las muestras cuantificadas en el espectrofotómetro Nanodrop 2000 (Thermo Fisher Scientific, Inc., Waltham, MA, USA) disueltas en 50 μL de agua estéril y se conservaron a -20°C hasta su uso (Galaviz-Silva et al., 2018).
Identificación por PCR: Se realizó la identificación de VP-AHPND+ por medio de PCR punto final. El coctel de reacción se preparó con una mezcla de amortiguador 5X, 1.5 mM MgCl2, 200 μM de cada dNTP, 0.2 μM de cada primer específico (AP3F) - 5’-ATGAGTAACAATATAAAACATGAAAC-3’ y (AP3R) - 5’-21 GTGGTAATAGATTGTACAGAA-3’, 3 μL de ADN de la muestra y 1 U de Taq polimerasa GoTaq (Promega, Madison, WI), para tener el volumen final de 25 μL con agua bidestilada (H2Od) (Sirikharin et al., 2015). Se utilizó un programa de precalentamiento de 94° por 5 min, seguido de 30 ciclos de desnaturalización de 94° por 30 s, anillamiento de 53°C por 30 s, extensión de 72°C por 40 s y una extensión final de 72 por 5 min en un termociclador (MultiGene thermal cycler, Labnet International, Inc., Edison, NJ). Se obtuvieron productos amplificados de 336 pb observados en un transiluminador de luz UV (Bio-Rad) en geles de agarosa al 1.5% teñido con bromuro de etidio (Sirikharin et al., 2015).
Análisis genómico de la región 16S rADN: La caracterización molecular del polimorfismo de las cepas se analizó amplificándose la región del gen 16S ADNr de las bacterias PCR positivas con los primers: U1F (5’-CTYAAAKRAATTGRCGGRRRSSC - 3’) y U1R (5’-CGGGCGGTGTGTRCAARRSSC-3’) (Rivas et al., 2004). Se preparó una mezcla de 25 μL con amortiguador 5X, 1.5 mM MgCl2, 200 μM de cada dNTP, 0.2 μM de cada primer, ADN de las bacterias (100 ng), 1 U de Taq polimerasa (GoTaq, Promega) y H2Od. El programa de amplificación inició con un ciclo de 95°C por 5 min, seguido de 35 ciclos de desnaturalización a 95°C durante 1 min, alineamiento a 55°C por 2 min y extensión a 72°C durante 1 min con una extensión final a 72°C por 7 min. Los productos se visualizaron en geles de agarosa al 1%, en un transiluminador UV (Bio-Rad). Posteriormente los amplicones fueron purificados con el estuche QIAquick PCR (Qiagen). La secuenciación se realizó en los Servicios Genómicos de CINVESTAV-LANGEBIO, Irapuato, Guanajuato. La secuencia de los productos amplificados se depositó en el GenBank (https://www.ncbi.nlm.nih.gov/genbank/) para compararlas con las registradas en el banco de datos y determinar la homología con Blast (https://blast.ncbi.nlm.nih.gov/). Se elaboró el dendrograma que determina la evolución filogenética y el grado de similitud con las descritas en otras regiones geográficas. Las ramas corresponden a particiones en al menos 500 réplicas por “bootstrap”. La distancia evolutiva se computó con el método “p-distance” y son el número de diferencia en las bases por sitio, involucrándose a 17 secuencias de nucleótidos. El análisis se realizó con Mega 7 (Saitou & Nei, 1987).
Variabilidad de las cepas por rep-PCR - (GTG)5: Para discriminar entre cepas PirA+ seleccionadas por PCR, se utilizó la técnica de rep-PCR (GTG)5, la cual se basa en la amplificación de secuencias palindrómicas repetidas intercaladas en el ADN. Se preparó una mezcla de 25 μL para PCR con amortiguador 5X Taq Green (Promega), 25 mM MgCl2, 25 mM desoxirribonucleótidos trifosfatados (dNTPs), 25 μM del primer (GTG)5 5’-GTGGTGGTGGTGGTG-3’, H2Od, ADN (100 ng/μL) y 1 U de ADN polimerasa (GoTaq, Promega). La amplificación comenzó con una pre desnaturalización a 95°C (7 min), seguido por 4 ciclos de 95°C (2 min), 36°C (2 min) y 72°C (2 min), más 30 ciclos de 95°C (1 min), 50°C (1 min) y 72°C (1 min). La elongación final fue a 72°C por 5 min. Las muestras se analizaron visualmente en electroforesis de geles de agarosa al 1.5% en 1X TBE (Tris Borato EDTA), a 100 V por 1 h y 30 min y de estos se seleccionaron las cepas con perfiles o patrones de bandas diferentes (Matsheka et al., 2005).
Resistencia a los antibióticos: Los antibióticos comerciales AVIMEX (Cd. De México, México) utilizados para los antibiogramas fueron: Oxi-blend (Oxitetraciclina: OXD), Flor-blend (Florfenicol: FLD), Enro-blend (Enrofloxacina: EBD), Magna-mix (Magnacina: MGX),Tri-mex (Sulfacloropiridacina y Trimetroprim: TX) y Aviflox (Enrofloxacina: AFX). El grado de sensibilidad se estableció de acuerdo al diámetro de inhibición: muy sensible (>17 mm), sensible (12-16 mm), intermedio (8-11 mm) y resistente (≤ 12-17 mm, según el antibiótico), de acuerdo al criterio establecido de CLSI (2005a; 2005b) ajustándose la turbidez de la suspensión bacteriana a 0.5 de la escala de McFarland en el medio de cultivo Mueller Hinton (DIFCO, DB) con 2% de NaCl por triplicado para cada antibiótico y bacteria evaluada (Yano et al., 2014).
Análisis estadístico: Para el análisis de datos de antibiogramas, se realizó un análisis de varianza (ANOVA) simple para comparar entre las variables analizadas, con pruebas de rangos múltiples y la prueba de (LSD) Fisher con el Paquete estadístico STATISTICA Versión 5.0.
RESULTADOS
Análisis bacteriano: De los muestreos en los esteros y en granja de camarón se aislaron colonias verdes característica en el crecimiento de VP en el agar TCBS (se ignoraron las colonias amarillas). Estas colonias se ordenaron según el tipo de muestra y localidad, agrupándose en 82 aislados en los ciclos de cultivo del 2013; la mayoría provino de El Riito con 46 cepas aisladas, 27 de larvas, 16 de HP, 2 de sedimento y una de agua (Tabla 1). Las muestras analizadas de granjas de cultivo en las JLSA durante el ciclo de cultivo 2014 ascendieron a 362 cepas de VP, la mayoría de Mélagos (121), Bahía de Lobos (67), Atanasia (51) y Tobari (43) sumando en ambos años 444 muestras procesadas (Tabla 2).
Todas estas cepas se resembraron en el medio de cultivo CHROMagar Vibrio y 186 presentaron características para VP con coloración malva, de los cuales se seleccionaron 83 cepas que representa el 18.6%; se descartaron aquellas que se repetían el lugar de procedencia y mismo tipo de muestra (PL´s, juveniles, sedimento o agua) para la identificación por PCR para VP-AHPND+ con el método AP3.
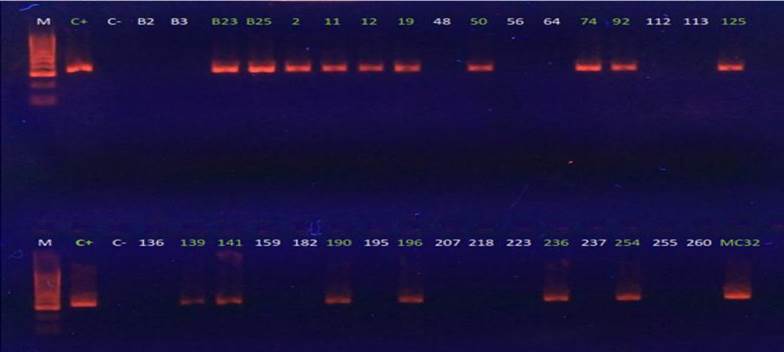
Identificación por PCR: Las cepas positivas por PCR fueron 16, mostrando los amplicones de 336 pb (Fig. 1), la procedencia de las muestras (agua, sedimento, PL´s o juveniles) y la localidad donde fueron obtenidas se describen en la Tabla 3.
Tabla 3 Cepas positivas para VP-AHPND+ por localidad y tipo de muestra.
| CLAVE | Localidad | Ciclo de cultivo | Muestra | PCR AP3 | Número de acceso al GenBank |
|---|---|---|---|---|---|
| Vp-B25 | Tóbari | Agua | + | MH091005.1 | |
| Vp-D2 | Bahía de Lobos | Sedimento | + | MH091006.1 | |
| Vp-D11F | Tóbari | Larva | + | MH091007.1 | |
| Vp-D12F | Atanasia | Larva | + | MH091008.1 | |
| D19F | Mélagos | Sedimento | + | MH091009.1 | |
| D50 | Bahía de Lobos | HP | + | MH091010.1 | |
| D74F | Mélagos | Larva | + | MH091011.1 | |
| D92 | Cruz de Piedra | HP | + | MH091012.1 | |
| D125 | Bahía de Lobos | Agua | + | MH091013.1 | |
| D139 | Cruz de Piedra | HP | + | MH091014.1 | |
| D141 | Mélagos | HP | + | MH091015.1 | |
| D190 | Mélagos | HP | + | MH091016.1 | |
| D196F | Atanasia | Agua | + | MH091017.1 | |
| D236pF | Tastiota | HP | + | MH091018.1 | |
| D254 | Mélagos | Agua | + | MH091019.1 | |
| D266 | Mélagos | Agua | + | MH091021.1 |
Se obtuvieron nueve cepas VP-AHPND+ de HP de camarones (HP y PL´s) de granjas que presentaban epizootias. De las muestras de agua se recuperaron otros cinco aislados y dos de sedimento en los estanques.
Caracterización bioquímica de las cepas: El perfil bioquímico de las cepas VP-AHPND+ muestra tres grupos diferentes. En la Tabla 3 se enlistan el grupo de cepas que mostraron el mismo perfil bioquímico y PCR positivo a AP3. El segundo grupo presenta diferencias por fermentar la ramnosa y una cepa fue positiva a ornitina. El tercer grupo muestra un perfil idéntico a la cepa de referencia de ATCC 17802 de V. parahaemolyticus AHPND -, el segundo y tercer grupo fueron descartados en el proceso de preselección de crecimiento en CHROMagar Vibrio.
Secuenciación de la región 16S ADNr: Las secuencias de la región 16S ADNr de las cepas aisladas de Sonora se depositaron en el GenBank (https://www.ncbi.nlm.nih.gov/) con los números de acceso citados en la Tabla 3. En el análisis evolutivo se definen tres grupos (Fig. 1): Las primeras que se originaron en Tailandia (3HP y 5HP) y China que surgieron en el 2009 (Fig. 2A) y se caracterizan por tener 15 secuencias (Fig. 2B) diferentes de nucleótidos en las regiones analizadas que enmarcan el polimorfismo de la región 16S ADNr y separan el origen evolutivo; en la segunda rama se separan las cepas de Sonora descritas en este trabajo y luego las Sinaloa.

Figura 2A Relación evolutiva de las taxas: (A) La historia evolutiva fue inferida con el método NJ (Neighbor-Joining) con 500 réplicas, indicándose la distancia de las cepas asiáticas, las de Sinaloa y Sonora.

Figura 2B El análisis evolutivo involucró 15 secuencias de nucleótidos que marcan el polimorfimo de las cepas. Todas las posiciones que contenían espacios vacíos (gaps) fueron eliminados. Había un total de 825 posiciones en la hoja de datos. El análisis evolutivo se realizó con MEGA7.
Variabilidad de cepas por PCR - (GTG)5: El análisis demuestra que existe polimorfismo entre las cepas de estudio representado primero por cinco grupos (A, B, C, D y E) que muestran el mismo perfil de amplicones (Fig. 3). La distribución de las cepas, el lugar de procedencia y la cantidad de cepas por JLSA muestra que la mayor variabilidad de las cepas VP-AHPND+ ocurrió en el sur de Sonora, donde se obtuvieron la mayoría de las cepas identificadas en el presente estudio, con una mayor variedad de biotipos, esto se debe a que se encuentran la mayor concentración de granjas de cultivo de camarón en el estado, de las 11 JLSA que se muestrearon, solo en 6 JLSA se obtuvieron las cepas de VP-AHPND+. En cuatro JLSA al sur de estado se identificaron 15 cepas positivas, dos cepas para el centro y una para el norte de Sonora (Fig. 4).

Figura 3 Perfil de amplicones para las cepas de VP-AHPND+ donde se utilizó la técnica de rep-PCR (GTG)5, la cual se basa en la amplificación de secuencias palindrómicas repetidas intercaladas en el ADN, la lectura del gel se realizó con el software GelAnalizer 2010.
Resistencia a los antibióticos: Los resultados de los antibiogramas con los antibióticos comerciales muestran que algunas cepas presentan un mayor grado de inhibición o resistencia a algún antibiótico (Fig. 5). En la Tabla 4 y Fig. 5 se registran los diámetros para todas las cepas evaluadas, incluso también con la cepa de referencia ATCC y MC32. Todas las cepas presentan diferencias significativas (P< 0.05) entre los halos de inhibición de los antibióticos evaluados, las cepas mostraron mayor sensibilidad a EVD, FLD, AFX y MGX. Doce cepas (75 %) presentaron resistencia a OXD, con halos de inhibición de 7 a 12 mm, (Fig. 5 y 6). En la Tabla 4 se muestran diferencias significativas entre las cepas en la inhibición con EBD, FLD, MGX y AFX. El 62.5 % (10 cepas) presentaron resistencia al antibiótico TX sin mostrar halos de inhibición.

Figura 5 Diámetro de inhibición (mm) de los antibióticos contra las cepas AHPND+. Letras distintas presentaron diferencias significativas (P<0.05).
Tabla 4 Antibiogramas de las cepas AHPND+ de Sonora.
* = Resistente; OXD= Oxitetraciclina; EBD= Enrofloxacina; FLD= Florfenicol; MGX= Magnacina; TX= Sulfacloropiridacina y Trimetroprim; AFX= Aviflox, Enrofloxacina.
 : Letras distintas presentaron diferencias significativas (P<0.05).
: Letras distintas presentaron diferencias significativas (P<0.05).
Patogenicidad de las cepas: Los retos con los camarones infectados demostraron la patogenicidad de las 16 cepas de VP-AHPND+ con variación en el periodo de incubación de la enfermedad y mortalidad, reconociéndose dos grupos principales; el grupo de los aislados Vp-B25, Vp-D2, Vp-D11, Vp-12F, D19F y D50 causaron signos clínicos a partir de las 8 horas de post infección con mortalidad que inició a las 18 h; en cambio, los retos con las cepas del segundo grupo (D74F, D92, D125, D139, D141, D190, D196F, D236pF, D254 y D266) mostraron signos clínicos a las 25 h con mortalidad inicial a las 72 h. Los signos de los ejemplares con AHPND incluyeron inicialmente nado errático, letargia, el intestino vacío y HP pálido (Fig. 7). Los resultados histopatológicos demuestran los túbulos hepatopancreáticos con infiltración hemocítica, necrosis epitelial, masas bacterianas dentro del lumen y descamación del epitelio de los túbulos HP (Fig. 8).
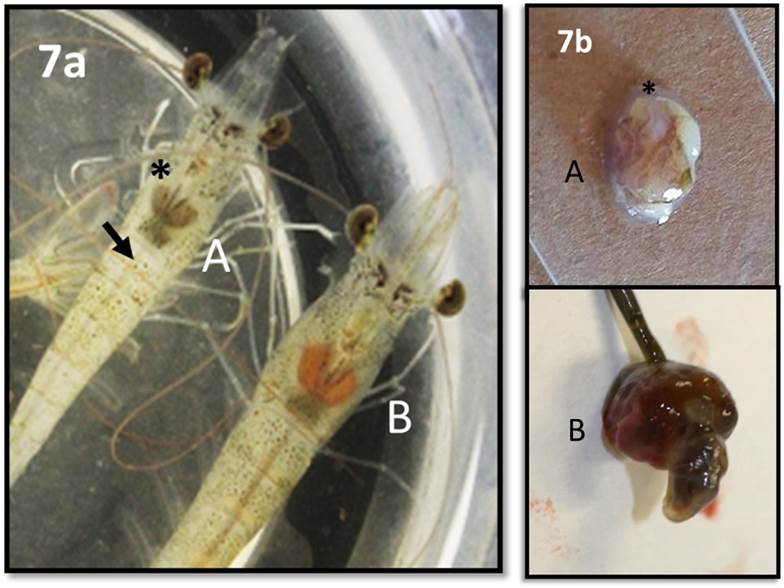
Figura 7 Signos macroscópicos característicos de la infección con las cepas VP-AHPND+. (7a) con Hepatopáncreas pálido (A, asterisco) e intestino vacío (flecha) infectado experimentalmente y (7b) HP pálido (A), disectado para histopatología. Los ejemplares sanos muestran HP rojo, del grupo control y controle negativo (B).

Figura 8 Histopatología del hepatopáncreas (HP) de camarones infectados con VP-AHPND+. Túbulos del HP con infiltración hemocítica (flecha corta), necrosis en epitelio tubular (flecha larga), lumen tubular con masas de bacterias patógenas (*) y lúmen con descamación del epitelio de los túbulos del HP (circulo). En el recuadro, corte histológico del control negativo con los túbuos sanos (vacuolas lipídicas y lumen de túbulos libres de infección.
DISCUSIÓN
De acuerdo al primer objetivo de este estudio, los análisis bacterianos en CHROMagar y por PCR con el método AP3 (Sirikharin et al., 2015) confirman la presencia de VP-AHPND+ en Sonora, lo cual se reafirma por los reportes de la presencia del patógeno en el vecino estado de Sinaloa (Soto-Rodriguez et al., 2015). El agente etiológico se distribuyó en muestras de agua, sedimento y camarón, tanto en las áreas de afuera, así como en el interior de las granjas de cultivo, debido a que VP es comúnmente encontrado en el medio ambiente marino aunado a que la bacteria es altamente patógena, porque contiene un plásmido fácilmente transmisible a otras cepas (Hastuti & Desrina, 2016) e incluso otras especies de bacterias como V. harveyi (Muthukrishnan et al., 2019) V. owensii (Liu et al., 2015), V. campbellii (Dong et al., 2017) y Vibrio punensis (Restrepo et al., 2018). Estos plásmidos expresan genes PirA y PirB que codifican las toxinas proteínicas (12.7 kDa and 50.1 kDa) que lisan el hepatopáncreas del camarón y le causan la muerte (Kondo et al., 2014). Los aislados de VP-AHPND+ de agua y sedimento explican la alta recurrencia de epizootias de AHPND en las granjas de camarón, al ser contaminadas con el medio ambiente local (Yang et al., 2019), debido a que las PL´s son certificadas como negativas por los laboratorios de diagnóstico recomendados por los Comités de Sanidad Acuícola locales. Estos resultados subrayan la importancia de mejorar los programas de buenas prácticas de manejo incluyendo uso de reservorios de agua tratada antes de la siembra (FAO, 2014).
En el transcurso de la identificación se descartaron las colonias amarillas que crecen en TCBS por ser de otras especies de Vibrio (V. cholerae, V. alginolyticus) aunque son muy comunes en el agua de mar por ser halofílicos, y además de ser cepas habituales y oportunistas en estómago e intestino de camarón, encontrándose frecuentemente en la fase tardía de la enfermedad junto con las cepas patógenas AHPND+ que crecen en colonias de color verde (Soto-Rodriguez et al., 2015).
Los signos clínicos macroscópicos e histopatológicos que causaron las cepas AHPND+ concuerdan con las que se describe en las cepas de Asia y América, así como la variabilidad en el tiempo en que se presentan los signos y el tiempo de sobrevivencia que oscila de 4 a 10 horas (cepas MO904 y M06-07) ó 2-4 días (BpShHep31 y BpShHep 24) (Tran et al., 2013; Soto-Rodríguez et al., 2015; Sirikharin et al., 2015; Muthukrishnan et al., 2019). Las comparaciones genómicas de secuenciación de la región 16S ADNr de las cepas colectadas durante los ciclos de cultivo 2013 y 2014 indican que fueron diferentes las cepas de V. parahaemolyticus que causaron la VP-AHPND+, respaldado así mismo por los análisis de “DNA fingerprint”, incluso se ha demostrado que la virulencia depende de la cepa, densidad, ruta de infección, tiempo de exposición, edad y condición del camarón (Soto-Rodriguez et al., 2015; Muthukrishnan et al., 2019) como en otras especies de Vibrio (Alday-Sanz et al., 2002). Así como las cepas de Sinaloa y Thailandia presentan elevada diversidad de polimorfismo en las cepas patógenas de V. parahaemolyticus, algunas con la máxima virulencia entre las 7-10 h con mortalidades del 50% y las menos virulentas con mortalidades a las 17 h en los retos experimentales (Joshi et al., 2014; Soto-Rodriguez et al., 2015). En nuestros resultados se diferencian de las cepas de Tailandia, China y el Noreste de México (Sinaloa y Sonora) con base en las secuencias de la región 16S ADNr, lo cual concuerda con reportes recientes que reconocen tres cladas filogenéticas basado en la alineación de secuencias nucleotídicas del plásmido pVA1 que hospedan (Latinoamericana, Asiática y Cosmopolita), diferenciándose en la ausencia del transposón Tn3 en la clada Asiática y además, algunas deleciones (pérdida de secuencias de ADN o nucleótidos) en la región pirAB encontrada en las cepas Latinoamericanas (González‑Gómez et al., 2020).
La evaluación de la resistencia a antibióticos del agente causal in vitro, mediante pruebas de inhibición de VP-AHPND+, demostró que se han desarrollado cepas con resistencia a OXD y TX. Las variaciones de inhibición obtenidas con el OXD pueden ser debido a que este antibiótico es el más utilizado en el cultivo de camarón en México, donde los antibióticos más comunes para contrarrestar las enfermedades producidas por bacterias del género Vibrio son OXD, FLD, trimetormetoprim-sulfametoxazol, saraoxacina (SARA) y EBD (Santiago et al., 2009). Existen escasos reportes de resistencia de VP-AHPND+ en México que reportan dos cepas resistentes a ampicilina, OXD y tetraciclina (Han et al., 2015). Los aislados de VP-AHPND+ de China han presentado perfiles de resistencia múltiple a antibibióticos como amikacina, gentamicina, tetraciclina, y ciprofloxacina (Yang et al., 2019). Este trabajo confirma los resultados de Han et al. (2015) de la resistencia a OXD, aumentando así la lista de cepas resistentes y reportamos por primera ocasión la resistencia a TX. Aunque también se presentaron cepas sensibles, se sugiere que la aplicación de fármacos requiere de un abordaje cuidadoso, con el fin de no incurrir en la generación de nuevas resistencias (Varela-Mejía & Alfaro-Mora, 2018). Por ende, lo más recomendable es minimizar el uso de antibióticos considerado como un último recurso en operaciones de cultivo de camarón. De acuerdo a la Norma Oficial Mexicana de Emergencia NOM-EM-05-PESC-2002 en la camaronicultura nacional, se prohíbe el uso de cloranfenicol y de furazolidona para la prevención y tratamiento de enfermedades y cualquier tratamiento con antibióticos treinta días antes de realizar la cosecha (Chávez-Sánchez & Higuera-Ciapara, 2003).











 nueva página del texto (beta)
nueva página del texto (beta)