Introducción
Las bacterias acido lácticas (BAL) son un amplio y heterogéneo grupo de microorganismos que se han empleado para la conservación de los alimentos (Stiles et al., 1997). El uso de estos microorganismos en los alimentos ha evolucionado, pasaron de ser utilizados en alimentos fermentados tradicionalmente a ser parte de procesos de fermentación controlada para la producción de metabolitos relevantes para la industria y su uso como probióticos que sirven como fábrica de metabolitos de interés biotecnológico (Rezac et al., 2018). Dentro de este grupo heterogéneo de bacterias se encuentra Lactococcus lactis, una bacteria Gram positiva, anaeróbica facultativa y no formadora de esporas. L. lactis es la especie mesófila predominante en la acidificación, dado que imparte sabor a ciertos productos lácteos fermentados (Cavanagh et al., 2015; Song et al., 2017). Se ha denominado cepa probiótica debido a que es una bacteria segura y a que se han realizado estudios que han mostrado su actividad inmunomoduladora, anticancerígena, antioxidante y antimicrobiana (Commane et al., 2005; Han et al., 2013; Kimoto et al., 2004; Zhang et al., 2011; Yerlikaya 2019).
Otra característica importante sobre L. lactis es que se ha empleado como cepa de expresión heteróloga de proteínas recombinantes (Jørgensen et al., 2014; Morello et al., 2008; Song et al., 2017). Uno de los sistemas empleados para la producción de estas proteínas es el sistema de expresión de genes controlado por nisina (NICE, por sus siglas en inglés), en donde utilizan los genes del sistema de censado de nisina y el promotor inducible con nisina (pNis) para lograr una sobreexpresión de las proteínas de interés (Kuipers et al., 1995). Además, este sistema incluye vectores que tienen como marcador de selección de grado alimenticio como lactosa y D-alanina para que tales cepas y sus productos puedan ser usadas en la industria alimentaria (Platteeuw et al., 1996). El empleo de estas cepas nos permite generar proteínas recombinantes con interés biotecnológico. Thurincina H es una bacteriocina producida por Bacillus thuringiensis, tiene muchas características favorables para ser considerada una proteína de interés; entre ellas destacan que tiene un amplio espectro de actividad contra diferentes bacterias tanto Gram positivas como Gram negativas, algunos de ellos contaminadores de alimentos; además, esta bacteriocina es activa en pH, que va de muy ácido hasta alcalino (pH 3.0-9.0), una ventaja con respecto a nisina, la cual actúa a pH ácido mayormente (Casados-Vázquez et al., 2017; Lee et al., 2009; Oros-Flores et al., 2018). También es resistente a altas temperaturas (hasta 80 °C) y a la liofilización (datos no publicados). Sin embargo, aunque tenga todas estas ventajas, no se puede emplear en alimentos debido a que la cepa productora no es reconocida como segura, a diferencia de L. lactis productora de nisina. Para la biosíntesis de thurincina H se necesitan al menos nueve genes que se encuentran integrados en un clúster denominado ThurH (thnP, thnE, thnD, thnR, thnA1, thnA2, thnA3, thnB, thnT, thnI), el cual hemos caracterizado previamente en nuestro laboratorio. Dentro de este clúster genético hay un pequeño operón constituido por los genes thnR, thnD y thnE, este es el responsable de brindar inmunidad a la cepa productora (Casados-Vázquez et al., 2018). Dado que nuestra proyección final es producir thurincina H en L. lactis -y esta cepa resultó ser sensible a ella-, es necesario buscar la manera de hacerla resistente. El objetivo de este trabajo fue generar una cepa de L. lactis resistente a thurincina H para que posteriormente podamos producir este péptido sin afectar a nuestra cepa heteróloga productora. Para conseguirlo, se generó una construcción donde se integraron los genes thnR, thnD y thnE en un vector con origen de replicación para L. lactis bajo el control del promotor pNis. La construcción generada brindó inmunidad a la cepa L. lactis incluso a concentraciones de 0.8 µg/ml; la cepa silvestre es sensible desde 0.2 µg/ml. Estos resultados son favorecedores porque se consiguió brindarle inmunidad a la cepa L. lactis contra thurincina H, y este hallazgo es importante dado que podría emplearse L. lactis resistente para la producción de thurincina H.
Materiales y métodos
Cepas y medios de cultivo
Lactococcus lactis NZ3900 es una cepa derivada de L. lactis subsp. cremoris. Esta cepa se creció a 30 °C sin agitación por 16 h en medio M17 suplementado con glucosa a una concentración final de 0.5% para fines de propagación y en medio Elliker (triptona 20 g/l, extracto de levadura 2.5 g/l, NaCl 4 g/l, acetato de sodio anhidro 0.5 g/l, ácido ascórbico 0.5 g/l) suplementado con lactosa a una concentración final de 0.5% para seleccionar las transformantes.
Bacillus cereus 183, B. thuringiensis subsp. morrisoni y B. thuringiensis subsp. kurstaki fueron crecidas en TSB (caldo soya tripticaseína) o TSA (agar soya tripticaseína) a 28 °C con agitación a 200 rpm durante 16 h.
Elaboración de construcciones
Para la clonación del clúster ThurH (integrado por los genes: thnP, thnE, thnD. thnR, thnA1, thnA2, thnA3, thnB, thnT y thnI), en el vector pNZ8149 se partió de la construcción pHT/ThurH, esta construcción contiene los 10 genes clonados en el vector pHT3101 (Oros-Flores et al., 2018). Se amplificaron dichos genes (que corresponden al clúster completo) con los oligonucleótidos:
ThnPFw/SpeI (5´GACTAGTTTATTGGGAAATCGCTTTATAGACATC 3´)
ThnIRv/SpeI (5´GACTAGTCTATATTTCTGAAGTATACAA 3´)
Se utilizó una polimerasa de alta fidelidad de acuerdo con las especificaciones del proveedor. El fragmento amplificado de 8141 bp y el vector pNZ8149 se digirieron con la enzima SpeI (NEB). El vector, además de ser digerido, también se desfosforiló con CIP (Calf Intestinal Phosphatase, NEB). Los fragmentos se ligaron con la enzima T4 DNA ligasa (NEB) en proporción 1:2 vector, inserto. La reacción de ligación se transformó en L. lactis NZ3900 electrocompetentes, y se seleccionaron colonias crecidas en Elliker con lactosa 0.5%. Las colonias seleccionadas se crecieron en medio líquido Elliker con lactosa 0.5%, y se extrajo DNA plasmídico (Birnboim & Doly, 1979). El DNA fue digerido con la enzima SpeI para identificar a las clonas positivas que además se confirmaron por PCR; la construcción completa se nombró pNZ/ThurH. A partir de esta última se elaboró la construcción pNZ/RDEP, la cual contiene al operón thnRDE (tres genes implicados en la inmunidad) y al gen thnP; los cuatro genes fueron clonados en el vector pNZ8149 siguiendo el mismo procedimiento que para pNZ/ThurH. Los oligonucleótidos utilizados para la amplificación fueron:
ThnRRv/PstI (5´CAAAACTGCAGTCATTTTTCATTCTCCTCCTC 3´)
ThnPFw/PstI (5´CAAAACTGCAGTTATTGGGAAATCGCTTTAT 3´)
El amplicón de 3502 bp y el vector fueron digeridos con la enzima PstI (NEB), el vector además fue desfosforilado. La ligación se realizó en una proporción de 1:5 (vector, inserto), y las colonias positivas se verificaron por digestión con la misma enzima y por PCR. Las construcciones transformadas en la cepa L. lactis NZ3900 fueron conservadas en glicerol al 10% a -80 °C.
Concentración mínima inhibitoria de thurincina H contra cepas del género Bacillus
Antes de determinar la concentración mínima inhibitoria (CMI) contra L. lactis, se determinó la CMI contra las cepas Bacillus cereus 183, Bacillus thuringiensis subsp. morrisoni y Bacillus thuringiensis subsp. kurstaki Cry-B, esta última transformada con pHT/ThurH. Se dejaron precultivos de cada cepa a partir de una colonia individual en medio TSB, se cultivaron durante 16 h a 28 °C con 200 rpm de agitación. Al día siguiente, los precultivos se diluyeron a una densidad óptica (DO600) de 0.1 en un volumen de 200 µl y se les adicionaron 50 µl de thurincina H en concentraciones 0 µg/ml, 0.2 µg/ml, 0.5 µg/ml, 0.9 µg/ml, 1.9 µg/ml, 3.8 µg/ml, 7.5 µg/ml y 15 µg/ml. Las cepas se incubaron a 28 °C con agitación y se tomaron lecturas de DO600 cada hora en un lector de placas (Synergy HTX, Biotek). El experimento se realizó por triplicado.
Concentración mínima inhibitoria de thurincina H contra L. lactis NZ3900
Una colonia de las cepas L. lactis NZ3900 silvestre, L. lactis transformada con pNZ8149 (vector vacío), pNZ/ThurH y pNZ/RDEP se sembraron en Elliker con lactosa 0.5%, y se incubaron sin agitación durante 16 h a 30 °C. Al día siguiente los precultivos se diluyeron a una densidad óptica (DO600) de 0.1 en un volumen de 200 µl y se les adicionaron 50 µl de thurincina H en concentraciones 0 µg/ml, 0.2 µg/ml, 0.5 µg/ml, 0.8 µg/ml. Las cepas se incubaron a 28 °C sin agitación en un lector de placas (Synergy HTX, Biotek) y se tomaron lecturas de DO600 cada dos horas durante 24 horas. El experimento se realizó por triplicado. Es importante mencionar que el ensayo se realizó sin nisina como inductor.
Resultados
Se logró la clonación del clúster ThurH dentro del vector pNZ8149 entre sitios SpeI. El clúster ThurH quedó integrado por los genes thnP, thnE, thnD. thnR, thnA1, thnA2, thnA3, thnB, thnT y thnI (Figura 1A). Es importante mencionar que este es un clúster que contiene una región promotora divergente entre los genes thnR y thnA1, lo que significa que los genes thnRDEP están codificados en la cadena menos del DNA, regulados por el promotor pThnR, y los genes thnA1A2A3BTI están codificados en la cadena más, regulados por el promotor pThur. Puesto que estos genes se clonaron en el mismo orden en que se encuentran en B. thuringiensis, el promotor pNis del vector pNZ8149 no será funcional para esta construcción (Figura 1B). La construcción pNZ/RDEP sí quedó regulada bajo el promotor pNis, y el orden de los genes quedó en sentido transcripcional correcto (Figura 1C).
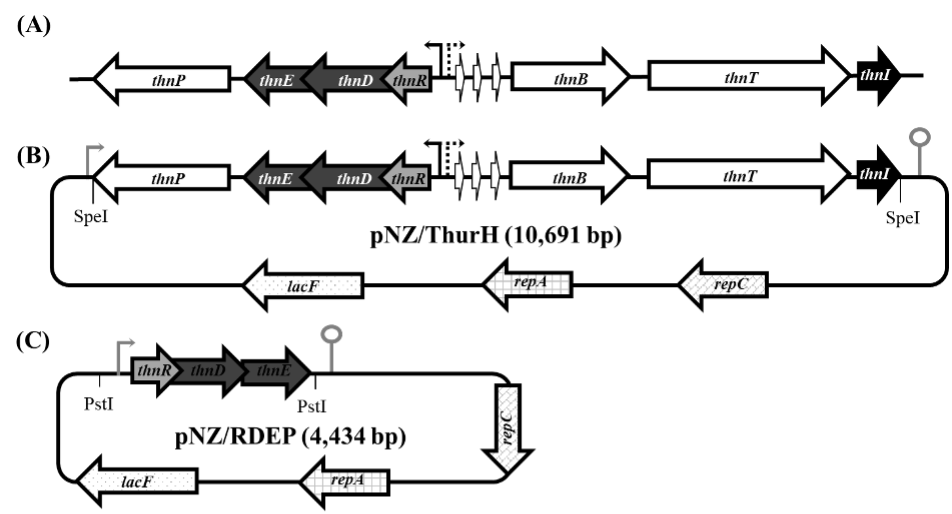
Fuente: Elaboración propia.
Figura 1 Esquema de las construcciones pNZ/ThurH y pNZ/RDEP. a) Orden de los genes del clúster ThurH en B. thuringiensis subsp. morrisoni; b) Acomodo de los genes del clúster ThurH clonados en el vector pNZ8149; c) Acomodo de los genes thnRDEP en el vector pNZ8149. Las flechas dobladas indican los promotores, el gris corresponde a pNis, el negro a pThnR y el punteado a pThur. La paleta ilustra el terminador. Se indican los sitios usados para la clonación. Las flechas lacF, rep A y repC corresponden al marcador de selección de grado alimenticio, al gen de replicación A y C, respectivamente.
Antes de realizar las pruebas de inmunidad de la cepa L. lactis transformada, se decidió determinar la CMI para cepas del género Bacillus, con el propósito de establecer la cantidad de thurincina H, que se usará en el ensayo contra L. lactis. Se utilizaron B. cereus 183 por ser la cepa indicadora modelo, una cepa sensible que se usa cotidianamente para determinar actividad antimicrobiana de thurincina H. Por su parte, B. thuringiensis subsp. morrisoni es la cepa nativa productora de thurincina H, esta cepa contiene el clúster ThurH completo y es inmune a su bacteriocina. Además, se incluyó B. thuringiensis subsp. kurstaki Cry-B (cepa curada de plásmidos naturales) transformada con pHT/ThurH, la cual también produce thurincina H y es inmune a esta porque la transformamos con la maquinaria completa; ya que, al estar en plásmido, contiene más copias que la cepa silvestre.
Para el ensayo se partió de una concentración de 15 µg/ml de thurincina H, y a partir de esta se hicieron diluciones seriadas hasta 0.2 µg/ml. Se observó que B. cereus 183 se inhibió completamente con 1.8 µg/ml (Figura 2a), ya que es una cepa muy sensible y se vio afectada incluso en concentraciones bajas como 0.23 µg/ml, 0.47µg/ml y 0.94 µg/ml. Aunque el efecto fue entre las horas 4 a 7, posterior a esto se recuperó el crecimiento y alcanzó la misma DO de la cepa silvestre. A excepción de la que tuvo el tratamiento a 0.94 µg/ml, esta alcanzó una DO de 0.48 ± 0.07, mientras que la silvestre llegó a una DO de 0.65 ± 0.02 (Tabla 1).
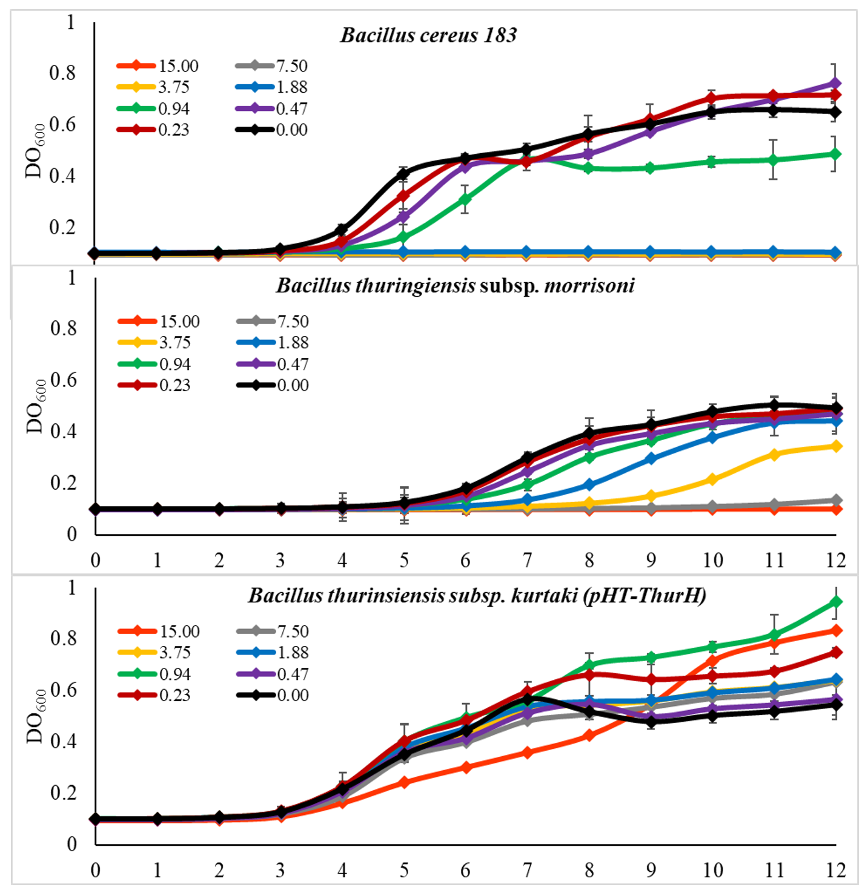
Fuente: Elaboración propia.
Figura 2 Gráficos de concentración mínima inhibitoria de thurincina H contra cepas del género Bacillus. B. cereus 183 es una cepa sensible a thurincina H; B. thuringiensis subsp. morrisoni es la cepa productora natural de thurincina H y B. Thuringiensis subsp. kurstaki está transformada con el clúster pThur. Las concentraciones de thurincina H se especifican en el inserto, están en µg/ml. Los ensayos se realizaron por triplicado.
Tabla 1 Efecto de las diferentes concentraciones de thurincina H sobre Bacillus thuringiensis y Lactococcus lactis.
| Cepa ⁄ Thur en μg/ml | 15 | 7.5 | 3.75 | 1.88 | 0.94 | 0.47 | 0.23 | 0.0 |
| Bacillus cereus 183 | 0.095 ± 0.001 | 0.098 ± 0.001 | 0.099 ± 0.001 | 0.1 ± 0.001 | 0.49 ± 0.07 | 0.76 ± 0.01 | 0.72 ± 0.01 | 0.7 ± 0.02 |
| Bacillus thuringiensis subsp. morrisoni |
0.1 ± 0.001 | 0.13 ± 0.002 | 0.34 ± 0.01 | 0.44 ± 0.006 | 0.47 ± 0.02 | 0.47 ± 0.01 | 0.49 ± 0.008 | 0.49 ± 0.03 |
| Bacillus thuringiensis subsp. kurstaki Cry-B/ pHT-ThurH |
0.83 ± 0.05 | 0.63 ± 0.03 | 0.64 ± 0.03 | 0.64 ± 0.03 | 0.94 ± 0.1 | 0.56 ± 0.01 | 0.74 ± 0.12 | 0.60 ± 0.02 |
| Cepa ⁄ Thur en μg/ml | 0.8 | 0.4 | 0.2 | 0.0 | ||||
| Lactococcus lactis/pNZ8149 | 0.002 ± 0.001 | 0.01 ± 0.002 | 0.15 ± 0.04 | 0.60 ± 0.02 | ||||
| Lactococcus lactis/pNZ-ThurH | 0.009 ± 0.002 | 0.14 ± 0.05 | 0.60 ± 0.01 | 0.64 ± 0.03 | ||||
| Lactococcus lactis/pNZ-RDEP | 0.61 ± 0.04 | 0.60 ± 0.01 | 0.63 ± 0.01 | 0.69 ± 0.08 | ||||
Nota. Los datos están reportados en densidad óptica a 600 nm. Para las cepas de Bacillus se midieron a las 12 h y para Lactococcus lactis a las 24 h. Thur: Thurincina H.
Fuente: Elaboración propia.
Para el caso de la cepa silvestre B. thuringiensis subsp. Morrisoni, la CMI fue 7.5 µg/ml (Figura 2b), y la cepa de B. thuringiensis subsp. kurstaki fue la más resistente, logrando crecer en todas las concentraciones, incluso con 15 µg/ml. Con esta concentración apenas disminuyó su crecimiento en un 50% con respecto al control sin thurincina H (Figura 2c). Con estos resultados se decidió usar tres concentraciones de thurincina H para calcular la CMI en L. lactis NZ3900. La cepa transformada con el vector vacío (pNZ8149) se inhibió completamente con la concentración de 0.4 µg/ml y resultó ser incluso más sensible a thurincina H que B. cereus 183 (Figura 3a). La cepa que contenía el operón de inmunidad (pNZ/RDEP) adquirió resistencia, soportó la concentración más alta probada (0.8 µg/ml) y su fase exponencial se alargó, comenzando a las 8 horas y a las 20 horas de crecimiento alcanzó casi la misma DO que la cepa sin thurincina H. A la cepa sin thurincina H le tomó 12 horas llegar a la fase estacionaria con una DO máxima de 0.7 ± 0.05; a la cepa tratada con 0.8 µg/ml le tomo 20 horas alcanzar la misma densidad óptica (Tabla 1). Esto indica que los genes de inmunidad están siendo reconocidos por la cepa y expresados sin inducción con nisina.
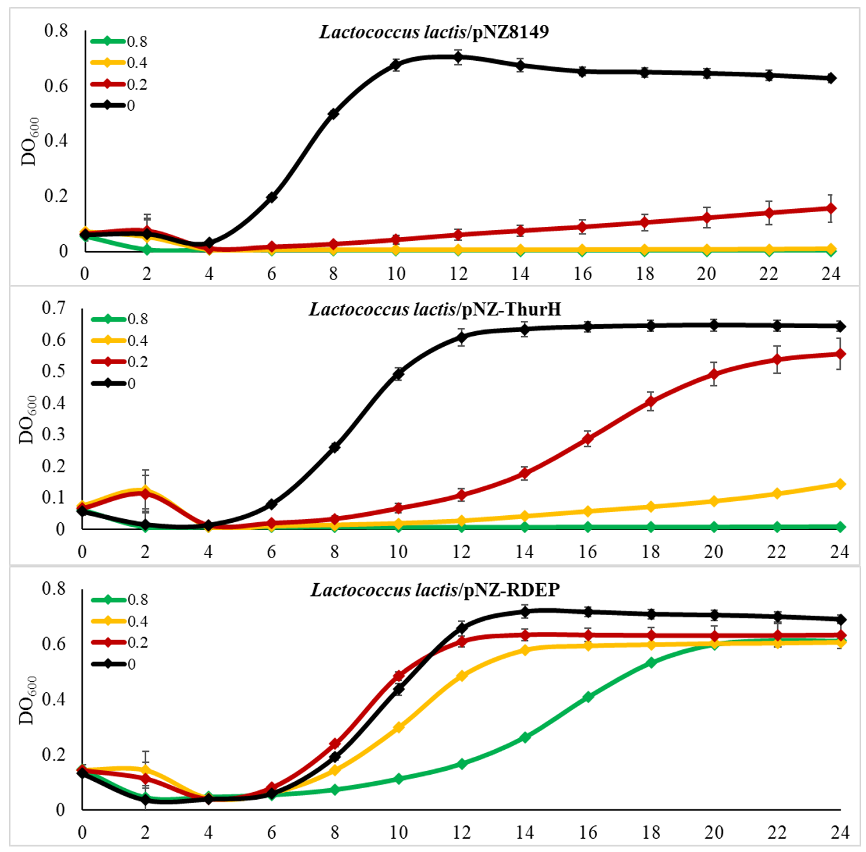
Fuente: Elaboración propia.
Figura 3 Gráficos de concentración mínima inhibitoria de thurincina H contra L. lactis. L. lactis/pNZ8149 es la cepa transformada con el vector vacío. L. lactis pNZ-ThurH está transformada con el clúster pThurH completo. L. lactis pNZ-RDEP esta transformada con los genes de inmunidad. Las concentraciones de thurincina H se especifican en el inserto, están en µg/ml. Los ensayos se realizaron por triplicado.
Además del operón de inmunidad, utilizamos el clúster completo para ver si L. lactis reconocía el promotor de B. thuringiensis y adquiría inmunidad. Cabe destacar que ambas construcciones (pNZ/RDEP y pNZ/ThurH) contienen los genes de inmunidad thnRDE entre otros, la diferencia es que en pNZ/RDEP están regulados por el promotor pNis y en pNZ/ThurH por el promotor nativo de B. thuringiensis. Se encontró que la cepa de L. lactis con pNZ-ThurH puede soportar un poco más que la cepa sin genes de inmunidad. Con la concentración de 0.2 µg/ml alcanzó una DO de 0.55 ± 0.01, mientras que la cepa sin thurincina H llegó a 0.65 ± 0.03 (Tabla 1). Al parecer L. lactis puede hacer uso del promotor de B.
Discusión
El clúster genético de thurincina H se encuentra dividido por un promotor divergente; en la cadena retardada del DNA tenemos a los genes thnRDEP, tres de ellos corresponden al operón de inmunidad (genes thnRDE), la regulación de estos genes la tiene el promotor pThnR (Figura 1a) (Casados-Vázquez et al., 2018), la otra mitad de genes que corresponden a thnA1A2A3BTI se encuentran en la cadena más y están regulados bajo el promotor pThur (Figura 1a). Estos genes se clonaron en este mismo orden en el vector pNZ8149, y por la orientación de estos se perdió la funcionalidad del promotor pNis (Figura 1b). Se trabajó con esta construcción en L. lactis para probar si los promotores nativos de B. thuringiensis (pThnR y pThur) podían ser reconocidos por L. lactis.
Como se puede ver, en la Figura 1b están incluidos todos los genes necesarios para la biosíntesis de thurincina H; sin embargo, no se logró obtener actividad antimicrobiana cuando se utilizó esta cepa (datos no mostrados). Una explicación a esto sería que el promotor de B. thuringiensis no es reconocido por L. lactis. Para probar esto, se decidió someter a la cepa de L. lactis transformada con pNZ/ThurH a tratamiento con thurincina H. Si esta muestra resistencia a la bacteriocina, indicaría que reconoce el promotor de B. thuringiensis. Interesantemente, se encontró que sí ganó un poco de resistencia, lo que sugiere que los promotores de B. thuringiensis están siendo reconocidos por la maquinaria de transcripción de L. lactis y se están expresando los genes de inmunidad; aunque cabe señalar que la cepa tardó mucho tiempo en recuperarse, y esto puede atribuirse a que el promotor es débilmente reconocido y, en consecuencia, la cantidad de proteínas de inmunidad es baja. Esto justificaría por qué no logramos obtener thurincina H con esta construcción. La bacteria puede estar sintetizando thurincina H a tan bajas concentraciones que es difícil detectar actividad con los métodos empleados.
Otro hallazgo interesante fue que el promotor pNis funciona incluso sin nisina. Esto se descubrió al hacer el ensayo de inmunidad usando la construcción pNZ/RDEP, donde la cepa transformada con estos genes se sometió a tratamiento con thurincina H y resultó resistente a thurincina H en todas las concentraciones usadas. Este dato es interesante dado que el uso de nisina como inductor incrementa el costo de producción de las proteínas recombinantes, además de que muchas veces se necesita hacer una purificación para la eliminación de la nisina (Mierau & Kleerebezem, 2005). La generación de una cepa de L. lactis resistente a thurincina H sin adición del inductor nos da una ventaja en el camino hacia la producción de thurincina H recombinante en una cepa segura. Aunque los resultados en el ensayo de inmunidad nos sugieren que la cepa de L. lactis puede expresar los genes thnRDE sin inductor, debemos dejar claro que esa cantidad no es suficiente para obtener proteínas de interés biotecnológico en cantidades adecuadas, la inducción con nisina sería necesaria para incrementar la cantidad de proteína recombinante.
Se puede destacar lo siguiente: la fuga residual de la expresión de los genes thnRDE es suficiente para brindar inmunidad; sin embargo, si quisiéramos purificar estas proteínas con interés biotecnológico, lo adecuado sería usar el inductor para incrementar su expresión. Ahora, hablando específicamente de thurincina H, el poder producirla en L. lactis y sobre todo de manera inducida nos daría dos ventajas: 1) obtener thurincina H en un sistema seguro y 2) obtenerla en cantidades significativas. Un estudio de este tipo se realizó con nisina, Hansen et al. (2009) insertaron los genes de inmunidad nisEFG y nisI en una cepa de B. subtilis para hacerla resistente e incrementar el rendimiento de nisina. Estos hallazgos confirman el propósito de nuestro trabajo, ya que se buscó obtener una cepa resistente para la producción de thurincina H. El paso por seguir en este contexto sería clonar los genes thnA1A2A3BT en el mismo vector bajo la regulación del promotor de nisina. El resultado obtenido sugiere que una construcción así nos permitiría obtener thurincina H, incluso sin adición del inductor. Otra ventaja de usar una construcción en plásmido es que el número de copias en la célula es mayor que si lo tuviéramos en cromosoma, y esto favorece a tener mayor cantidad de la proteína recombinante. En el caso de B. thuringiensis, cuando una cepa sensible a thurincina H se transforma con el plásmido pHT/ThurH, esta adquiere mayor resistencia que la cepa silvestre (Figura 2a y 2b), lo que genera una ventaja a la hora de producir thurincina H, puesto que nuestra cepa recombinante podría resistir mayor cantidad de thurincina H y, por lo tanto, obtener mejores rendimientos. Este trabajo nos arrojó muchos resultados interesantes y también nos dio la pauta para continuar trabajando en búsqueda del objetivo final, que es obtener thurincina H en la cepa heteróloga L. lactis.
Conclusiones
Se generó una cepa de Lactococcus lactis resistente a thurincina H haciendo uso del operón thnRDE sin necesidad de inducir con nisina. Ahora contamos con una cepa inmune a thurincina H que puede ser modificada para expresar los genes thnA1A2A3 con el propósito de obtener thurincina H recombinante en un sistema seguro.











 nueva página del texto (beta)
nueva página del texto (beta)


