INTRODUCCIÓN
Salmonella es un género bacteriano mayormente asociado con enfermedades en humanos. Se divide en dos especies: enterica y bongori. A su vez, Salmonella enterica se puede dividir en seis subespecies: 1) Salmonella enterica subsp. enterica, 2) Salmonella enterica subsp. salamae, 3) Salmonella enterica subsp. arizonae, 4) Salmonella enterica subsp. diarizonae, 5) Salmonella enterica subsp. houtenae y 6) Salmonella enterica subsp. indica. Alternativamente, las cepas de Salmonella enterica pueden clasificarse con base en sus antígenos de superficie O (lipopolisacárido) y H (flagelar). Con base en esta tipificación se han reportado más de 2500 serovariedades o serotipos (Fábrega y Vila 2013).
Dos de los principales síndromes clínicos causados por la infección con Salmonella en seres humanos son fiebre entérica o fiebre tifoidea, y gastroenteritis infecciosa. La fiebre entérica o tifoidea es una enfermedad invasiva sistémica causada por Salmonella enterica serotipos Typhi y Paratyphi A y B que afecta exclusivamente al ser humano. En contraste, hay muchas cepas de Salmonella no tifoidea (SNT) que causan enfermedad diarreica en el ser humano y puede, además, infectar una amplia gama de animales, siendo los serotipos frecuentemente relacionados con estos casos Salmonella Enteritidis y Salmonella Typhimurium (Fábrega y Vila 2013). Sin embargo, la prevalencia de los serotipos causantes de SNT podría ser dependiente de su localización geográfica y temporal (Pui et al. 2011).
De acuerdo con la Organización Mundial de la Salud, se estima que hay 153 097 991 casos de SNT y 56 969 muertes relacionadas al año, y se ha descrito que la mayoría de los casos de SNT se asocian con el consumo de alimentos o agua contaminados con la bacteria, lo que representa un tema importante de salud pública (Kirk et al. 2015). En México, la Dirección General de Epidemiología declara anualmente > 72 000 casos de gastroenteritis por infección de Salmonella (DGE 2015). Cabe señalar que los serotipos y la vía de infección vinculados con estos casos de SNT no están claramente definidos.
Los serotipos de SNT se caracterizan por una amplia especificidad de hospederos y condiciones de crecimiento diversas, lo cual favorece su introducción en el ambiente mediante contaminación fecal directa (heces de humanos o animales infectados) o indirecta (descarga agrícola o aguas residuales), pudiendo atravesar la cadena alimentaria para la contaminación del alimento (Chen et al. 2013, Li et al. 2014). Los ecosistemas acuáticos (ríos, lagos, costeras y estuarios) desempeñan un papel crucial en la difusión y retransmisión de bacterias, por lo que se consideran una fuente potencial de infecciones por Salmonella (Levantesi et al. 2012). Estudios epidemiológicos han descrito la presencia y capacidad de supervivencia de Salmonella en el medio acuático (Levantesi et al. 2012). En México, Salmonella es reconocida como un peligro acuático significativo de ríos (Jiménez et al. 2014) y como comensal de animales domésticos (Jiménez et al. 2011).
En consonancia con la contaminación del agua por Salmonella, el agua de riego se ha identificado como fuente de contaminación de cultivos hortícolas relacionados con brotes, y en México se ha vinculado con algunos de ellos (CDC 2008, 2013). En Estados Unidos, en 2008 y 2013, se notificaron brotes de SNT vinculados con el consumo de chiles serranos (Behravesh et al. 2011) y pepinos (CDC 2013), respectivamente. El serotipo Salmonella Saintpaul se identificó como agente causal de ambos brotes, y el agua de irrigación y el ambiente de las granjas de México se establecieron como la fuente común de contaminación (Behravesh et al. 2011, CDC 2013). Posteriormente, Jiménez et al. (2014) reportaron alta incidencia (85 %) de Salmonella y una gran diversidad de serotipos de SNT distribuidos en los ríos del noroeste de México.
Generalmente, la SNT es una enfermedad autolimitante, y se trata mediante reposición de líquidos y electrolitos. La recomendación del uso de antibióticos es para pacientes con enfermedad grave o alto riesgo de enfermedad invasiva (Pui et al. 2011, Chen et al. 2013). Sin embargo, la aparición de serotipos de SNT con resistencia a antibióticos (RA), especialmente a la primera línea de tratamiento con estos fármacos (ampicilina, amoxicilina, cloranfenicol y trimetoprim-sulfametoxazol) se ha convertido en un grave peligro para la salud (CENETEC 2012, Chen et al. 2013). Los mecanismos para la adquisición de RA en Salmonella involucran el uso inapropiado de antibióticos por parte del hombre (agricultura, medicina y veterinaria) (Su et al. 2004), así como el intercambio de elementos genéticos móviles que llevan genes de RA con el ecosistema colonizado por la bacteria (Paytubi et al. 2014). La detección de resistencia múltiple a antibióticos (RMA) en Salmonella aislada de fuentes de agua (Li et al. 2014) es de gran relevancia debido a su participación como vía importante en la transmisión de Salmonella RA a los seres humanos.
En México se ha informado sobre el aumento de RA en cepas de Salmonella obtenidas de muestras clínicas (Amábile-Cuevas 2010), animales domésticos (Jiménez et al. 2011) y alimentos (Miranda et al. 2009), lo cual se ha vinculado con el uso irracional de antibióticos. Sin embargo, la emergencia de cepas de Salmonella RMA obtenidas de recursos hídricos naturales requiere una mejor descripción, ya que éstos participan en la difusión de la bacteria. Por lo tanto, el objetivo de este estudio fue identificar el perfil de RA de diferentes serotipos de SNT aisladas de ríos del valle agrícola de Culiacán y establecer relaciones entre aislamientos, como base para determinar el papel de estos ecosistemas como reservorios y fuentes de propagación de Salmonella RA.
MATERIALES Y MÉTODOS
Colección de cepas de Salmonella
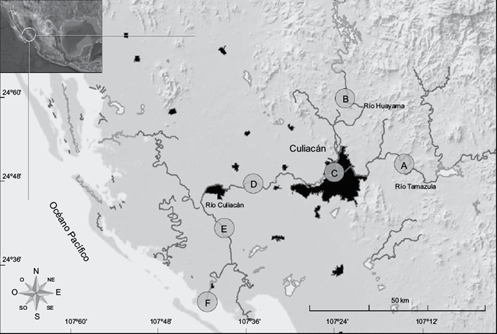
Se analizaron la prevalencia, la diseminación y el perfil de RA de 111 cepas de Salmonella recuperadas previamente de tres ríos (Humaya, Tamazula y Culiacán) situados en el valle de Culiacán, en la región noroeste de México. De estos ríos se seleccionaron seis sitios de muestreo (denominados A, B, C, D, E y F) con una separación de aproximadamente 25 km entre sí, para cubrir un área de estudio en la cual se llevan a cabo tanto prácticas agrícolas como actividades recreacionales. Además, estos sitios debían estar ubicados a menos de 1 km de una población rural donde se practicara la crianza de algún tipo de animal. Las muestras se recogieron con una frecuencia quincenal de julio de 2008 a junio de 2009. Las muestras de agua de río se tomaron aproximadamente 30 cm por debajo de la superficie en botellas de plástico estériles y se mantuvieron a 4 ºC por un periodo no mayor de 6 horas hasta su procesamiento. Las cepas de Salmonella aisladas de dichas muestras fueron previamente clasificadas por serología; se reportaron 23 serotipos completamente tipificados de Salmonella enterica sub. enterica (Agona, Anatum, Bovismorbificans, Braenderup, Give, Infantis, Javiana, Kiambu, Luciana, Minnesota, Montevideo, Münster, Newport, Oranienburg, Pomona, Poona, Saintpaul, San Diego, Senftenberg, Soahanina, Sundsval, Texas, Weltevreden) y tres serogrupos de la misma subespecie (E1, I, L). Además, se reportaron aislamientos de Salmonella enterica sub. houtenae (grupo IV) y cepas no tipificables (Jiménez et al. 2014).
Preparación del inóculo
Las cepas de Salmonella se almacenaron a -80 ºC hasta su cultivo en agar Hecktoen (BD Bioxon, México). Posteriormente, de una a tres colonias se transfirieron a caldo de soya triptona (Difco, México) y se incubaron a 37 ºC durante 24 h. Después de la incubación se preparó una suspensión bacteriana en agua peptona tamponada al 0.1 % (BD Bioxon, México), y se ajustó a la turbidez de 0.5 en escala de MacFarland.
Prueba de sensibilidad a antibióticos
La sensibilidad a los antibióticos de las cepas de Salmonella se determinó utilizando el método de difusión en disco de acuerdo con el Instituto de Estándares Clínicos y de Laboratorio (CLSI 2015). Se tomaron alícuotas de 0.1 mL de suspensión bacteriana y se extendieron por duplicado en agar Mueller Hinton (BD Bioxon, México). Posteriormente, los discos de antibióticos (Oxoid, Reino Unido) se colocaron sobre el agar inoculado y se incubaron a 37 ºC durante 24 h. El panel evaluado comprendió 16 antibióticos (Cuadro I). Para la interpretación de resultados, las cepas se clasificaron como susceptible, intermedia o resistente a antibióticos de acuerdo con el diámetro (mm) del halo de inhibición (Cuadro I) (CLSI 2015). Las cepas se consideraron con RMA si presentaban RA ≥ 2 antibióticos, independientemente de si éstos pertenecían a la misma familia. La cepa de E. coli ATCC 25922 se utilizó como referencia para verificar la calidad de la prueba.
CUADRO I DISTRIBUCIÓN DE LA RESISTENCIA DE LAS CEPAS DE Salmonella A LOS ANTIBIÓTICOS DE ACUERDO CON EL SITIO Y PERIODO DE AISLAMIENTO
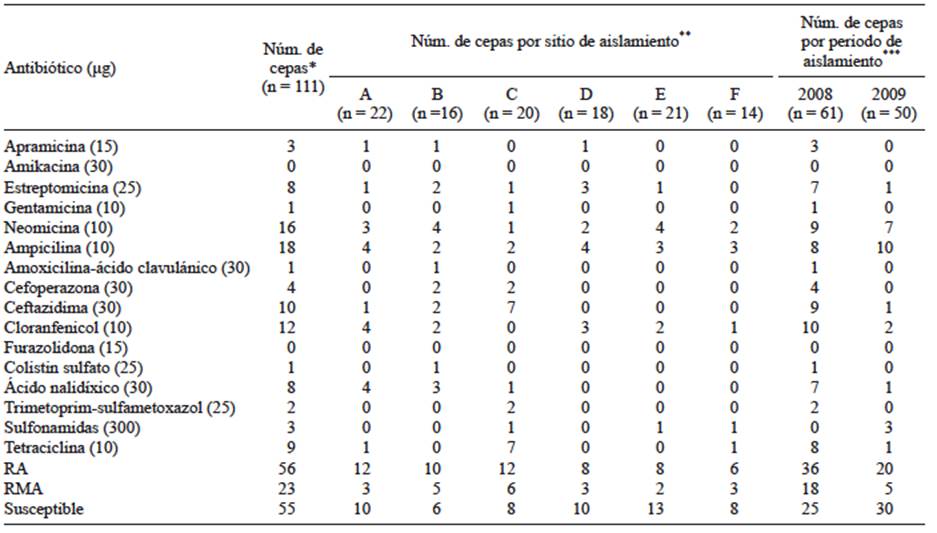
NA: no aplicable, RA: resistencia a un antibiótico, RMA: resistencia a múltiples antibióticos (≥ 2 antibióticos)
*Kruskal-Wallis: H = 99.11, p = 0.994; **Kruskal-Wallis: H = 3.80, p = 0.579; *** Kruskal-Wallis: H = 24.65, p = 0.314
Análisis estadístico
Los datos generados en este estudio se analizaron mediante el paquete estadístico para las ciencias sociales IBM SPSS Statistics v. 21. Las pruebas de chi cuadrada (Χ2) y Kruskal-Wallis se utilizaron para estimar las tasas de RA entre los serotipos de SNT, así como el origen y la fecha de aislamiento. Los valores de p ≤ 0.05 se consideraron estadísticamente significativos.
RESULTADOS
El 50.5 % (56/111) de las cepas de Salmonella presentaron RA ≥ 1 de los 16 antibióticos del panel evaluado; 41.1 % (23/56) de las cepas RA se identificaron con RMA (Cuadro I). Las cepas de Salmonella mostraron como principal fenotipo de RA a ampicilina (16.2 %), seguido de neomicina (14.4 %), cloranfenicol (10.8 %) y ceftazidima (9.0 %). Ninguna de las cepas de Salmonella mostró resistencia frente a amikacina y furazolidona. La prueba de Kruskal-Wallis no mostró diferencia significativa en la distribución de la RA entre las cepas, la fecha y el origen de aislamiento (Cuadro I). A pesar de la diferencia no significativa en la distribución de cepas RMA entre los periodos de estudio, se observó una reducción evidente de cepas de Salmonella con RMA aisladas en 2009 (5) comparadas con 2008 (18). Cabe señalar que las cepas de RA se asocian significativamente al periodo de aislamiento de 2008 (X2 = 32.34, p = 0.046).
Respecto a los serotipos de Salmonella evaluados en el estudio, el 82.1 % (23/28) y el 46.4 % (13/28) muestran cepas con RA y RMA, respectivamente (Cuadro II). No se observó diferencia significativa de RA entre los serotipos (H = 37.34, p = 0.138), lo que puede indicar que estos serotipos tienen el mismo potencial para desarrollar RA habitualmente prescritos. Los serotipos identificados como susceptibles al panel antimicrobiano pertenecían a Salmonella Bovismorbificans, Salmonella Braenderup, Salmonella Sundsvall, Salmonella Texas, y Salmonella I. El cuadro II describe la frecuencia de Salmonella RA por serotipo y una clasificación de los antibióticos evaluados según los mecanismos fundamentales de resistencia bacteriana (Mendoza 2011). Con base en esto, las cepas de Salmonella RA mostraron mayor resistencia a los antibióticos que inhiben la síntesis de la pared celular y la síntesis de proteínas. Curiosamente los serotipos predominantes, Salmonella Oranienburg y Salmonella Saintpaul, exhibieron RMA a 11 y nueve antibióticos, que están representados por cuatro y cinco clases de antimicrobianos diferentes, respectivamente (Cuadro II).
CUADRO II PREVALENCIA DE LA RESISTENCIA A ANTIBIÓTICOS ENTRE LOS SEROTIPOS de Salmonella NO TIFOIDEOS AISLADOS DE LOS RÍOS CONTRA CINCO CLASES DE ANTIBIÓTICOS
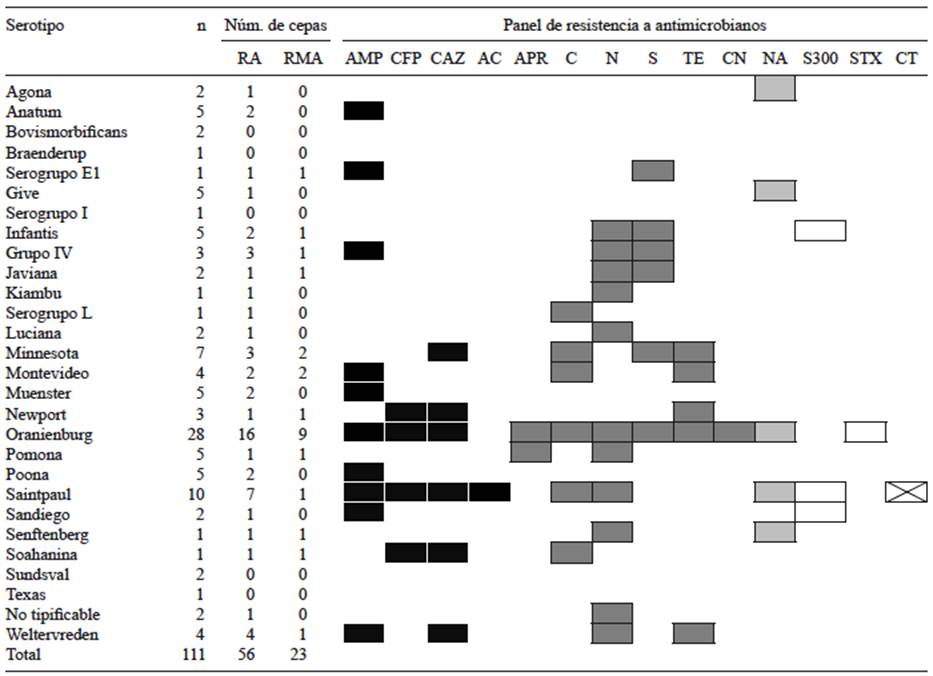
Panel de antibióticos: ampicilina (AMP), cefoperazona (CFP), ceftazidima (CAZ), amoxicilina-ácido clavulánico (AC), apramicina (APR), cloranfenicol (C), neomicina (N), estreptomicina (S), tetraciclina (TE), gentamicina (CN), ácido nalidíxico (NA), sulfonamidas (S300), trimetoprim-sulfametoxazol (STX) y colistín sulfato (CT). Los serotipos de Salmonella RA se clasificaron de acuerdo con el mecanismo de acción de resistencia: síntesis de inhibidores de la pared celular ( ), síntesis de inhibidores de proteínas (
), síntesis de inhibidores de proteínas ( ), inhibidores de síntesis de ácidos nucleicos (
), inhibidores de síntesis de ácidos nucleicos ( ), inhibidores metabólicos (
), inhibidores metabólicos ( ) y modificadores de la permeabilidad de la membrana celular (
) y modificadores de la permeabilidad de la membrana celular ( ).
).
RA: resistencia a un sólo antibiótico, RMA: resistencia ≥ 2 antibióticos
El antibiograma de 16 antibióticos evaluados reveló 26 patrones RA (R1-R26) entre los diferentes serotipos de SNT. El cuadro III resume la frecuencia de patrones de RA entre las cepas y serotipos, los cuales se clasifican principalmente en patrones RA constituidos por un antibiótico (R1-R4). En nuestro estudio, los principales patrones RA difundidos entre serotipos fueron R1 (28.6 %) y R2 (25.0 %) (Cuadro III). En particular, Salmonella Oranienburg se caracterizó por un mayor porcentaje de cepas de RMA (32 %) (Cuadro II) representado por la amplia variedad de patrones RA (13 patrones) (Cuadro III). La gran diversidad de patrones de resistencia encontrados en las cepas de estudio impidió establecer supuestos teóricos de relaciones entre serotipos o adquisición de resistencia en sitios de muestreo específicos, lo que podría significar el uso indiscriminado de antibióticos en la región noroeste de México.
CUADRO III PATRONES DE RESISTENCIA A ANTIBIÓTICOS DE LOS SEROTIPOS DE Salmonella AISLADOS DE LOS RÍOS EN EL NOROESTE DE MÉXICO

La codificación de antibióticos es igual a la que se presenta en el cuadro II. Los serotipos se codifican como: Agona (A), Anatum (B), E1 (C), Give (D), Infantis (E), IV (F), Javiana (G), Kiambu (H), L (I) , Luciana (J), Minnesota (K), Montevideo (L), Münster (M), Newport (N), Oranienburg (O), Pomona (P), Poona (Q), Saintpaul (R), Sandiego (S), Senftenberg (T), Soahanina (U), no tipificable (V), Weltevreden (W)
*Resistencia significativa a antibióticos
DISCUSIÓN
En México hay falta de conocimiento sobre los fenotipos de RA de diferentes serotipos de Salmonella recuperados a partir de ecosistemas acuáticos. Por lo tanto, los resultados de este estudio proporcionan una mejor comprensión de la frecuencia y diseminación del comportamiento antimicrobiano de los serotipos de SNT distribuidos a través de los principales ríos de la región noroeste de México. La alta incidencia de cepas Salmonella RA (50.5 %) denota una situación preocupante porque esta fuente de agua podría favorecer la aparición y propagación de estas cepas de Salmonella en el ambiente de la región (Fig. 1), lo que representa un aumento del riesgo de casos de SNT y la necesidad de tratamientos clínicos y veterinarios adecuados.
Varios estudios de vigilancia de la RA en Salmonella concuerdan con nuestros resultados, lo cual evidencia la marcada aparición de cepas de Salmonella RA recuperadas de medios acuáticos como agua de riego (Li et al. 2014), estuarios (Carvalho et al. 2013), canales (López et al. 2009), y aguas subterráneas (Phokela et al. 2011). Se ha documentado que los ríos del valle Culiacán se utilizan para prácticas agrícolas en el cultivo de hortalizas, granos, oleaginosas y frutas, entre otros plantíos (CODESIN 2015, López et al. 2009, Jiménez et al. 2014), lo cual expone los productos de exportación al riesgo de contaminación con cepas de Salmonella RA. Anteriormente, la CDC vinculó este hecho con un brote de SNT en Estados Unidos (CDC 2013).
La frecuencia de cepas de Salmonella resistentes a varios antibióticos, incluyendo ampicilina, tetraciclina, estreptomicina, trimetoprim-sulfametoxazol, cloranfenicol, y sulfonamidas, así como otros fármacos, es una realidad establecida ampliamente en la literatura (Levantesi et al. 2012, Abapka et al. 2014). Particularmente, se han reportado altos niveles de resistencia a estos antibióticos en SNT (Voss-Rech et al. 2016). En México, estudios previos dirigidos a la caracterización de la RA han descrito cepas de Salmonella de origen clínico (Amábile-Cuevas 2010) o alimentario (Miranda et al. 2009), o bien originadas en aguas de canal (López et al. 2009), resistentes a tetraciclina, ampicilina, cloranfenicol y ceftriaxona. Estos fenotipos de RA se observaron en las cepas de Salmonella incluidas en este estudio. Por lo tanto, se sugiere que estos fenotipos se mantienen entre las cepas de Salmonella como resultado de su dispersión o clonalidad, previamente descritas por Jiménez et al. (2014).
La vigilancia de la RA en serotipos aislados a partir del ambiente acuático refleja el entorno en el que crece el organismo (Li et al. 2014), y es un indicador útil de la mala gestión de los antimicrobianos en los entornos de producción de los animales, la actividad agrícola y la farmacoterapia en humanos (Singh et al. 2012, Cabello y Godfrey 2016). En este contexto, nuestros resultados mostraron que la prevalencia de los serotipos con RA (Cuadro III) se relaciona con la actividad humana como la principal fuente de antibióticos en los ríos, dado que los patrones de resistencia estuvieron mayormente asociados con ampicilina, cloranfenicol, ceftazidima y ácido nalidíxico. Cabe señalar que estos antibióticos se han señalado como los de mayor consumo para el tratamiento médico en México (Benavides-Plascencia et al. 2005). Por ello, grandes cantidades de estos fármacos son liberados en aguas residuales municipales por excreción o eliminación (Lien et al. 2016), y se han detectado en diferentes ambientes acuáticos como ríos (Zhang et al. 2015), agua para acuacultura (Pham et al. 2015), influentes y efluentes de plantas de tratamiento de aguas residuales (Rodríguez-Mozaz et al. 2015) y principalmente en los efluentes de hospitales (Castro-Pastrana et al. 2015). Por otro lado, se encontró una alta incidencia de cepas de Salmonella resistentes a tetraciclina, lo que podría estar relacionado con el amplio uso de este antibiótico para tratamiento humano, así como en actividades agrícolas y pecuarias (Borghi y Palma 2014). Además, se ha reportado que más del 70 % de este antibiótico es excretado después de la medicación, y que su alta hidrofobicidad y baja volatilidad, le permiten prevalecer por periodos prolongados en ambientes acuáticos (Daghrir y Drogui 2013).
La amplia distribución de fenotipos RA de Salmonella Oranienburg y Salmonella Saintpaul en la región podría ser reflejo de la recirculación de estos serotipos entre el medio ambiente y animales domésticos (Jiménez et al. 2011, 2014), así como un indicador potencial de la interacción de genes de RA entre poblaciones microbianas que colonizan estos ecosistemas (Carvalho et al. 2013, Abapka et al. 2014). Dado que el agua es el hábitat de las bacterias y una de las rutas más importantes para su diseminación, se ha propuesto que también desempeña un papel crítico como reservorio de genes que confieren resistencia a antimicrobianos (GRA) (Vaz-Moreira et al. 2014). De acuerdo con esto, los resultados de nuestro trabajo demuestran la gran variedad de patrones de resistencia a antibióticos que puede presentar Salmonella (Cuadro III). Por ello, se sugiere continuar con la caracterización molecular de genes implicados en la RA de al menos un representante de cada familia de antibióticos, principalmente aquellos que mostraron mayor incidencia (ampicilina, tetraciclina, estreptomicina, ceftazidima, trimetoprim-sulfametoxazol, cloranfenicol, ácido nalidíxico y sulfonamidas).
Este estudio muestra que la mayoría (82.0 %) de los serotipos de SNT presentan RA o RMA (Cuadro II), y que manifiestan una gran diversidad de patrones RA (Cuadro III), lo que implica la facultad indiscriminada de estos serotipos para adquirir resistencia contra diversas clases de antibióticos y correlacionar con los diversos mecanismos que utiliza Salmonella para inhibir el efecto antibiótico, especialmente Salmonella Oranienburg y Salmonella Saintpaul (Cuadro II). Diversos mecanismos de RA han sido descritos para Salmonella (Su et al. 2004), lo cual ha reflejado la capacidad de serotipos de SNT de origen ambiental para exhibir patrones de RMA > 4 clases de antibióticos (Carvalho et al. 2013).
Salmonella Typhimurium DT29 fue la primera cepa identificada con RMA (ampicilina, estreptomicina, sulfonamidas, tetraciclina y furazolidona) que fue ampliamente diseminada y responsable de una epidemia grave en el hombre y los animales. Después se sumaron esfuerzos para estudiar y controlar la RA de este serotipo, así como otros serotipos emergentes (Frye y Jackson 2013). En este sentido, se debe prestar especial atención a los serotipos que muestran RMA de origen acuático, incluyendo Salmonella Oranienburg, Salmonella Saintpaul, Salmonella Minnesota y Salmonella Muenchen, ya que son los principales serotipos dispersos en la región noroeste de México (López et al. 2009, Jiménez et al. 2011, 2014) y se relacionan con brotes mundiales de SNT (Behravesh et al. 2011, Pui et al. 2011, CDC 2013). En México, Salmonella Oranienburg fue el agente causal de un brote de origen alimentario que se caracterizó por presentar RMA, lo cual dificultó el tratamiento de la población afectada (Vázquez-Garcidueñas et al. 2014). Además, se ha reportado que este serotipo es altamente resistente al menos a nueve agentes antimicrobianos (Kaneene et al. 2010).
Por último, la aparición de cepas con RMA -exhibida por la mayoría de los serotipos de Salmonella- tiene un impacto negativo en la eficacia del tratamiento antimicrobiano; en consecuencia, puede conducir a aumentos en las tasas de morbimortalidad de la salmonelosis (Frye y Jackson 2013). Además, un aumento de la resistencia a la ampicilina entre los serotipos de Salmonella incluidos en este estudio se considera un problema de salud en México, ya que este antibiótico es uno de los más recetados. La RA observada en Salmonella exhibe la continuidad de un panorama alarmante, ya que muchos de estos antibióticos (ampicilina, sulfonamidas, fluoroquinolonas o cefalosporinas) son la primera línea de terapia antimicrobiana frente a la salmonelosis (CENETEC 2012, Chen et al. 2013). Debido a la presencia de cepas de Salmonella susceptibles a la familia de los nitrofuranos (furazolidona), éste podría considerarse como un tratamiento alternativo para la salmonelosis en la región de estudio (Chadfield y Hinton 2003).
CONCLUSIONES
La información descrita contribuye a mejorar el conocimiento sobre la RA en diferentes serotipos de SNT recuperados de ríos del noroeste de México, y puede utilizarse en estudios epidemiológicos sobre este patógeno de transmisión alimentaria. Estos hallazgos sugieren que los ríos ubicados en la región podrían servir como albergue y fuente de transmisión de diferentes serotipos de Salmonella con RA entre hospederos y/o ambientes. La RA de Salmonella frente a cloranfenicol, ampicilina, amoxicilina y trimetoprim-sulfametoxazol es un problema de salud pública debido a que estos antibióticos son considerados como el tratamiento de elección contra la SNT. La educación y estrategias sobre el control de la propagación de este patógeno, así como el uso adecuado de los antibióticos, son cruciales para limitar la aparición de cepas de Salmonella RMA.











 nueva página del texto (beta)
nueva página del texto (beta)