Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista internacional de contaminación ambiental
versão impressa ISSN 0188-4999
Rev. Int. Contam. Ambient vol.30 no.4 Ciudad de México Nov. 2014
Bioaerosoles y evaluación de la calidad del aire en dos centros hospitalarios ubicados en León, Guanaguato, México
Bioaerosols and air quality assessment in two hospitals located in León, Guanajuato, Mexico
María MALDONADO-VEGA, Juan José PEÑA-CABRIALES, Sergio DE LOS SANTOS VILLALOBOS. Andrea P. CASTELLANOS-ARÉVALO, David CAMARENA-POZOS, Bertha ARÉVALO-RIVAS, Laura VALDÉS-SANTIAGO, Laura J. HERNÁNDEZ-VALADEZ y Dora Linda GUZMÁN DE PEÑA*
Departamento de Biotecnología y Bioquímica. Unidad Irapuato-CINVESTAV-IPN Km 9.6 Libramiento Norte Irapuato-León. Gto. C.P. 36821 *Autora responsable: dguzman@ira.cinvestav.mx
Recibido marzo 2014;
aceptado agosto 2014
RESUMEN
La contaminación biológica dentro de los hospitales es de gran preocupación debido a que las bacterias y hongos son las causas más importantes de infecciones nosocomiales. Un gran número de bacterias y propágulos fúngicos son capaces de dispersarse vía aérea por lo que la exposición a estos patógenos potenciales debe ser controlado y para ello es necesario evaluar la composición y concentración de microorganismos aéreos en clínicas y hospitales. En este trabajo se realizó un estudio piloto para determinar la calidad del aire en dos hospitales en León, Guanajuato, México. Los objetivos de este trabajo fueron identificar, determinar y caracterizar a los propágulos fúngicos y aerobacterias dentro de estos sitios públicos, así como aislar e identificar organismos que pudieran comportarse como patógenos potenciales en el ambiente hospitalario, en áreas donde permanecen pacientes vulnerables sometidos a cirugía, quimioterapia y terapia intensiva. El número de aerobacterias y propágulos fúngicos fueron cuantificados por medio de cultivos selectivos y fueron reportados en términos de unidades formadoras de colonias por metro cúbico de aire (UFC/m3). Las concentraciones obtenidas indicaron que los dos hospitales se consideran como contaminados en ciertas áreas, ya que los niveles de bacterias y propágulos fúngicos estuvieron muy por arriba de los aceptables de acuerdo con lo establecido por la Organización Mundial de la Salud (WHO 1990). El hospital 1 presentó concentraciones de bacterias de 40 a 280 UFC/m3 con lo que su calidad de aire fue calificada como pobre, además de que en este aire se encontraron 17 géneros de bacterias y 15 de hongos. El hospital 2 con más años de servicio y mayor incidencia de pacientes presentó una mayor concentración microbiana tanto de propágulos fúngicos (32 a 442 UFC/m3) como de bacterias (90 a 548 UFC/m3). En este segundo hospital se identificaron 17 géneros de bacterias y 22 de hongos. En cuanto al aislamiento e identificación de organismos, se encontraron más del tipo Gram-negativos que Gram-positivos en ambos hospitales. Las enterobacterias como Escherichia coli, Enterobacter cancerogenus y Acinetobacter sp. fueron predominantes y de importancia clínica para los usuarios del hospital, mientras que las bacterias del género Bacillus fueron las Gram-positivas predominantes. Entre los hongos, Fusarium y Penicillium eran los más comunes. Asimismo se identificaron hongos de alta importancia clínica como Microsporum audouinii, Cladosporium oxysporum, Mucor ramosissimus, Alternaria arborescens y Cryptococcus albidus. La identificación y densidad de los microorganismos en el aire de estos hospitales son el primer paso para tomar medidas preventivas y reducir los niveles de microorganismos.
Palabras clave: bioaerosoles, muestra de aire, bacterias, propágulos fúngicos, hospital, calidad del aire, biopartículas.
ABSTRACT
Microbiological contamination in hospitals is of main concern since bacteria and fungi constitute a threat on the spreading of nosocomial infections. Airborne microorganisms in hospitals can cause negative health effects in immune-compromised people. Hence, it is very important to determine the density and types of microorganisms which live in the hospital environment. This study was aimed to measure the fungal and bacteria loads from air samples, as well as to identify them at the level of genus or species in two different hospitals of León, Guanajuato, Mexico. Microbial identification was run by molecular and standard microbiological techniques. Concentration of bacteria and fungus present in the air is reported in terms of the number of colony forming units per cubic meter of air (CFU/m3). Both hospitals were considered contaminated, since microbial density was significantly higher than the acceptable level reported by the World Health Organization (WHO 1990). Hospital 1 presented bacterial density values ranging from 40 to 280 CFU/m3. While, fungal density values ranging from 56 to 408 CFU/m3. Hospital 2 showed fungal density values ranging from 32 to 442 CFU/m3, and bacteria density values ranging from 90 to 548 CFU/m3. Bacterial identification revealed 9 genera, and fungal identification showed 17 genera in hospital 1 and 17 bacterial genera, 22 fungal genera in hospital 2. The predominant bacteria were Escherichia coli, Enterobacter cancerogenus, and bacteria of Acinetobacter genera. Fusarium and Penicillium were the most common fungal isolates. Likewise, fungus such as Mi-rosporum audouinii, Cladosporium oxysporum, Mucor ramosissimus, Alternaria arborencens and Cryptococcus albidus were found as medically important fungi. The identification and quantification of bioaerosols of these hospitals could be used to take action toward the reduction of bioaerosol concentration in order to protect the people who generally use hospital.
Key words: bioaerosols, bacteria, hospital, air quality, bioparticles.
INTRODUCCIÓN
Los bioaerosoles o biopartículas pueden definirse como partículas o microfragmentos de animales, plantas o microorganismos en el aire (Rose 1994, Cox y Wathes 1995). El análisis de la calidad del aire se recomienda para obtener información sobre la mayoría de los microorganismos relacionados con enfermedades infecciosas o alergénicas. Adicionalmente, la estimación de la densidad y diversidad de estos microorganismos en hospitales es un indicador de la calidad del ambiente (Burge 1990, Hoseinzadeh et al. 2013). Aunque los patógenos oportunistas aéreos pueden ser relativamente inofensivos para personas sanas, pueden causar efectos adversos en personas inmunocomprometidas (Augustowska y Dutkiewicz 2006). Se ha visto que muchas enfermedades infecciosas y alérgicas están asociadas con la exposición a biopartículas en edificios con húmedad o aire acondicionado sin mantenimiento (Burge 1990). En México se ha reportado una correlación entre los síntomas manifestados por los trabajadores y el tipo de población bacteriana aislada en los bioaerosoles (Castañeda Roldán et al. 2003, 2006). Por lo que el control de la calidad del aire en el interior de los hospitales adquiere un papel importante en la prevención de infecciones hospitalarias y puede ser útil para el diseño de estrategias que protejan tanto a los empleados del hospital, como a los pacientes, especialmente aquellos inmunocomprometidos e inmunosuprimidos (Leung y Chan 2006). El objetivo de este trabajo fue determinar la concentración y el tipo de microorganismos presentes en la atmósfera intramuros de dos hospitales de la ciudad de León Guanajuato, México.
MATERIALES Y MÉTODOS
Se seleccionaron dos hospitales en la ciudad de León, Guanajuato, México para evaluar la calidad del aire. El hospital 1 corresponde a un hospital de especialidades de primer nivel, mientras que el hospital 2, corresponde a un hospital regional de consulta familiar de segundo nivel. El primero se localiza en un área de menor impacto urbano, mientras que el segundo se localiza en un área céntrica y de mayor urbanización.
Obtención de muestras de aire
El muestreo y análisis de aire para determinar la calidad microbiológica consideró las recomendaciones metodológicas descritas por el Instituto Nacional de Seguridad e Higiene de España (NTP 409, 1990, NTP 608, 2001), pues en México no se tiene referencia de normas al respecto. El protocolo utilizado para la toma de muestras se realizó por duplicado y fue el siguiente: una vez en el punto de muestreo, el responsable se colocó los guantes desinfectados con una solución de etanol al 70 %. Asimismo, las piezas del equipo de monitoreo microbiológico ambiental fueron desinfectadas con toallas sanitizantes Millipore. Se colocó el casete (caja con medio de cultivo específico, surtido por Millipore) en la rejilla del equipo. Una vez terminado el tiempo de muestreo para cada medio de cultivo, los casetes fueron rotulados y sellados con papel parafilm y almacenados a 4 °C. Se hicieron las siguientes consideraciones: el tiempo transcurrido entre la toma de la muestra y su procesamiento en el laboratorio no fue mayor a 12 h. Se tomaron casetes no abiertos e incubados bajo las mismas condiciones como testigo. El equipo utilizado fue un muestreador microbiológico ambiental de la marca Millipore (Sistema M. Air T.) comparable al método Slit-To-Agar basado en las directivas de la Farmacopea de EUA para salas limpias (Fig. 1A). El equipo de captura de aire contenía placas de medio de cultivos de 73 mm de diámetro x 6 mm de alto (equivalente a un área de 44 cm2). En estas placas con 20 g de medio, se impactaron las partículas contenidas en el volumen de aire muestreado y se propagaron los organismos viables. Este método es frecuentemente usado para el aislamiento de hongos y bacterias que se encuentran en el ambiente, se basa en la cuantificación de los microorganismos contenidos en 1000 L ó 1 m3 de aire (Costa Baquiao et al. 2012). El sistema M. air T. se basa en el principio de inercia de impacto de Anderson y usa una serie de coladores con cerca de 1000 microperforaciones, lo cual reduce el potencial de colonias sobrelapadas y desecación del medio (Luksamijarulkul et al. 2012). El flujo de aire captado ya está preprogramado en el equipo, el cual fue de 140 L/min y el volumen de cada muestra de 100 o de 250 L, el tiempo de muestreo de 42 s y 1.47 min respectivamente. La altura del muestreo fue de 1.5 m. Una vez que se tomaron las muestras de aire, cada uno de los casetes con medio de cultivo se almacenaron a 4 °C hasta su procesamiento en el laboratorio. El medio utilizado para cada tipo de microorganismo fue específico para hongos y levaduras (agar rosa de Bengala (ARB)), así como selectivo para bacterias (agar soya tripticaseína (AST); Fig. 1B y 1C). El volumen de aire analizado de los hospitales se comparó con un control de aire externo en ambos sitios de estudio para determinar datos de concentración diferencial y ambiental. Como el sistema de captura microbiana consiste en recoger aquellas biopartículas viables, éstas se hacen impactar en medios nutritivos. Por ello las placas expuestas fueron incubadas a la temperatura correspondiente al crecimiento óptimo de los componentes biológicos. El agar soya de tripticaseína (AST) se utilizó para el cultivo de bacterias aerobias ya que provee un excelente soporte de crecimiento para estos organismos. Estos cultivos de bacterias fueron incubados a 37 °C por un periodo de 24 h para iniciar el conteo de colonias, las que fueron monitoreadas durante tres días para observar detalles de sus características morfológicas. El medio de cultivo ARB se utilizó para los hongos, las cajas de Petri fueron incubadas a 25 °C por un periodo de 5 días. El ARB fue utilizado debido a sus propiedades bacteriostáticas, así como a su efecto ligeramente fungistático sobre el crecimiento del micelio (Ottow 1972). Una vez transcurrida la incubación, se realizaron los conteos de colonias en cada una de las cajas de Petri, mismos que fueron reportados como unidades formadoras de colonias por metro cúbico de aire (UFC/m3) y calculados con la siguiente fórmula:
Conteos totales (UFC/m3) = [Colonias totales x 1000]/250 o 100 L, dependiendo del volumen de muestra tomado.
Una vez contadas las colonias observadas con mayor frecuencia, se aislaron en cajas de agar papa dextrosa (APD) en el caso de los hongos y de AST en el caso de las bacterias para su posterior identificación macro y microscópica.
Observaciones morfológicas
Los organismos aislados fueron cultivados durante siete días en APD a 25 °C. Para el caso de hongos se consideró la textura o aspecto, color, velocidad de crecimiento radial, tamaño (mm), pigmentación del micelio y al anverso del medio, forma, borde y elevación de la colonia. Para la identificación microscópica se observaron las conidias, hifas, conidióforos, esporas y cuerpos fructíferos. Para el caso de las colonias de bacterias se consideró el color, textura, forma, tamaño y bordes. En cuanto a la morfología microscópica se realizó la prueba de tinción de Gram.
Extracción de ADN de los cultivos
Los diferentes aislados de bacterias se sembraron en 3 mL de caldo soya tripticasa (CST), más un duplicado del medio sin inocular como testigo negativo. Todos los cultivos se incubaron en baño maría (marca Lab-Line Instrument, Inc. modelo 4682), con agitación (250 rpm) por 12 h a 37 °C. Transcurrido el tiempo de incubación se tomaron los 3 mL del CST para la extracción del ADN a través del ZR Fungal/ Bacterial DNA Kit™ siguiendo las indicaciones del fabricante. Se utilizó el mismo kit para la extracción del ADN fúngico.
Condiciones de la técnica de reacción en cadena de la polimerasa (PCR, por sus siglas en inglés)
La reacción de PCR se llevó a cabo en un volumen total de 50 μL bajo las siguientes condiciones: 5 μL de 10 x amortiguador PCR, 1.5 μL de MgCl2 (50 mM), 1 μL de solución de dNTP (10 mM), 1 μL de cada iniciador directo y reverso (30 mM), 0.25 μL de polimerasa Platinum Taq DNA 5 U/μL (Invitrogen). El programa se llevó a cabo en un Mastercycler Gradient (Eppendorf AG, Hamburg, Modelo: 5331), el templado fue inicialmente desnaturalizado a 95 °C por 5 min, seguido por 30 ciclos de amplificación de PCR con una desnaturalización a 94 °C por 30 s, alineamiento a 57 °C por 40 s, primera extensión a 72 °C por 1 min y una extensión final de 5 min a 72 °C.
Identificación de bacterias
Se realizó la amplificación y secuenciación de regiones conservadas del gen 16S ribosomal (16S RNA). Los oligonucleótidos utilizados fueron FD1: 5' CCG AAT TCG TCG ACAACAGAG TTT GAT CCT GGC TCA G 3' y RD1: 5' CCC GGG ATC CAA GCT TAA GGA GGT GAT CCA GCC 3' (Weisburg et al. 1991). Para la identificación del género y especie de las bacterias aisladas, las secuencias fueron alineadas y comparadas con las secuencias ARNr 16S de las bases de datos National Center for Biotechnology Information mediante el programa BLAST (Gasch et al. 2000) y para dar más confianza a los resultados y verificar si se trataba del mismo microorganismo se utilizaron tres bases de datos adicionales: Ribosomal Database Project II (http://rdp.cme.msu.edu/seqmatch/seqmatch_intro.jsp; Cole et al. 2005, Cole, et al. 2007) y Greengenes Project (http://greengenes.lbl.gov/cgi-bin/nph-blast_interface.cgi; DeSantis et al. 2006), SILVA database project (http://www.megx.net/gms/geographic-blast/). Las cuatro bases de datos fungieron como herramientas para el alineamiento y clasificación de las secuencias.
Identificación de propágulos fúngicos
La identificación molecular de los propágulos fúngicos se llevó a cabo mediante la amplificación de la región 5.8 S de los genes ribosómicos. La región ITS1-5.8S-ITS2 fue amplificada a partir del ADN genómico usando los oligonucleótidos específicos ITS1: (5' TCC GTA GGT GAA CCT GCG G 3'), ITS4: (5' GCT GCG TTC TTC ATC GAT GC 3'; Luo y Mitchell 2002). El programa de amplificación fue predesnaturalizado a 95 °C por 5 min; 35 ciclos a 95 °C por 30 s, 50/55/60 °C por 30 s y 72 °C por 1 min; y una extensión final a 72 °C por 10 min. Los datos de secuenciación fueron optimizados usando el programa BioEdit v. 7.0 y los alineamientos fueron creados utilizando CLUSTAL-X (Thompson et al. 1997). Para la identificación del género y la especie de los hongos aislados, las secuencias fueron analizadas en las siguientes bases de datos: Fungal ITS Pipeline (http://www.emerencia.org/fungalitspipeline.html; Nilsson et al. 2009), la base de datos para identificación molecular de hongos UNITE database (http://unite.ut.ee/; Koljalg et al. 2005, Nilsson et al. 2008) y la ITS2 database III (http://its2.bioapps.biozentrum.uni-wuerzburg.de/; Koetschan et al. 2010).
Secuenciación
La purificación del ADN amplificado durante la PCR se realizó usando el kit UltraClean® PCR Clean-Up siguiendo las indicaciones del fabricante. Se cuantificó en un Nanodrop (Thermo Scientific, Modelo: ND2000) para determinar la cantidad de ADN en cada una de las muestras. Finalmente el producto purificado se envió al laboratorio de secuenciación del CINVESTAV-Irapuato-México para secuenciar en la plataforma Illumina MiSeq.
RESULTADOS
De los bioaerosoles analizados se encontró una concentración de propágulos fúngicos que osciló en un intervalo de 56 a 184 UFC/m3 para el hospital 1, mientras que el testigo de ambiente exterior fue de 408 UFC/m3 (Fig. 2) Para el hospital 2, el intervalo fue de 32 a 442 UFC/m3, con el testigo de ambiente exterior de 404 UFC/m3 (Fig. 2). La mayor concentración de propágulos fúngicos se presentó en la mayoría de las secciones del hospital 2 con excepción del área de terapia intensiva de niños (Fig. 2). Las observaciones macroscópicas y microscópicas de algunos de los hongos aislados permitieron identificar propágulos fúngicos (datos no mostrados). Respecto a las aerobacterias encontradas en los bioaerosoles analizados, el hospital 1 presentó un intervalo de 40 a 232 UFC/m3, mientras que el testigo de ambiente exterior fue de 280 UFC/m3 (Fig. 3). En el hospital 2, fue de 90 a 548 UFC/m3 y el testigo de ambiente exterior presentó 122 UFC/m3 (Fig. 3). En el hospital 1 la mayor concentración de bacterias fue encontrada en el área de terapia intensiva de niños (232 UFC/m3), mientras que la menor concentración fue en el área de trasplantes (40 UFC/m3; Fig. 3). En el hospital 2 la mayor concentración de bacterias fue en el área de terapia intensiva de adultos (448 UFC/m3) y la menor concentración en el área de pediatría intensiva (32 UFC/m3; Fig. 3).
Las aereobacterias identificadas en los hospitales se presentan en el cuadro I. En el hospital 1 se identificaron 11 bacterias distribuidas en nueve géneros (Fig. 4A): Bacillus, Corynebacterium, Escherichia, Kocuria, Enterobacter, Proteus, Rhanella, Pseudomonas, Kluyvera. Entre los organismos aislados, se encontraron 11 especies (Cuadro I).

En el hospital 2 las bacterias estuvieron distribuidas en 17 géneros (Cuadro I y Fig. 4B): Bacillus, Escherichia, Knoellia, Kocuria, Micrococcus, Mycobacterium, Rothia, Klebsiella, Alcaligenes, Agrobacterium, Kluyvera, Neisseria, Pseudomonas, Acinetobacter, Enterobacter, Stenotrophomonas, Pandoraea. Entre los organismos aislados se encontraron 21 especies (Cuadro I).
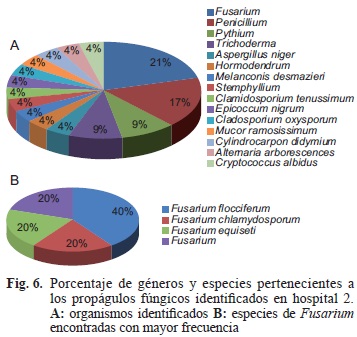
La abundancia de los propágulos fúngicos identificados para cada hospital se muestra en las figuras 5 y 6, respectivamente. Los aislados de hongos fueron obtenidos de los dos hospitales. Se observó el micelio de varios propágulos fúngicos en las muestras tomadas, algunas de las cuales fue difícil aislar debido a que hongos como los Zygomycetos cubren la superficie de las placas impidiendo el crecimiento de otros hongos. Sin embargo, fue posible aislar e identificar un total de 17 propágulos fúngicos en el hospital 1 y 22 en el hospital 2. Los resultados de secuenciación del ITS y algunas otras, permitieron la identificación de especies, principalmente con base en las las características micro y macromorfológicas tales como tipo de colonia, presencia o ausencia de micelio aéreo, color de la colonia y producción de pigmentos (Samsom et al. 2010). Algunos propágulos fúngicos pertenecieron a la misma especie. Sin embargo, al momento de aislarlos presentaban variaciones ligeras en las características de sus colonias. Los siguientes seis géneros fueron identificados en el hospital 1: Fusarium, Helminthosporium, Microsporum, Penicillum, Botryotichum, Achremonium (Fig. 5A). Mientras que en el hospital 2 los siguientes 15 géneros fueron identificados: Cladosporium, Penicillum, Pythium, Stemphyllium, Trichoderma, Fusarium, Melanconis, Hormodendrum, Epicoccum, Mucor, Clamidosporium, Cylindrocarpon, Alternaria, Cryptococcus, Aspergillus (Fig. 6A). En los hospitales 1 y 2, el género dominante entre las secuencias identificadas de hongos fue Fusarium con un 56 % y 21 % del total respectivamente. En el hospital 1, hubo una distribución homogénea entre los géneros Penicillum, Microsporum y Helminthosporium (11 % de cada uno; Fig. 5A). Algunos de los hongos reportados en la figura 5A no fueron identificados a nivel de especie debido a que no fue posible separarlos de otras colonias, lo cual se ha reportado como un problema recurrente en estudios de identificación de hongos (Nilsson et al. 2009).

DISCUSIÓN
Los microorganismos en el ambiente representan un peligro invisible ya que son un factor de riesgo para contraer y manifestar enfermedades. En años recientes, se han llevado a cabo diferentes estudios para evaluar la contaminación microbiana del aire en lugares de riesgo tales como hospitales, estas acciones son consideradas como aportación hacia la prevención de infecciones nosocomiales (Whyte, et al., 1992, Eickhoff, 1994). Dado que los niveles de microorganismos dependen de un gran número de variables, actualmente no existen niveles específicos de UFC/m3 aceptados por todas las instituciones (Gulliver y Briggs 2004, Pasquarella et al. 2000, Ho-seinzadeh et al. 2013). Sin embargo, se han propuesto estándares que son aceptados ampliamente. Hasta el momento, el medio más efectivo para cuantificar microorganismos en el aire está limitado al conteo de unidades formadoras de colonias (UFC); éste es el parámetro más importante ya que indica el número de microorganismos viables (capaces de multiplicarse) (Pasquarella, et al., 2000). La Organización Mundial de la Salud (OMS) ha sugerido que la concentración de microorganismos en lugares de uso común debe ser menor de 300 UFC/m3. Mientras que en lugares para individuos con sistemas inmunocomprometidos, la concentración debe ser menor de 100 UFC/m3 (WHO 1990). Adicionalmente, se ha propuesto una relación de los valores de concentración de microorganismos entre el ambiente interior (I) y el ambiente exterior (E) para determinar la procedencia de la contaminación. Se dice que una relación I/E mayor de 1 indica que la fuente de contaminación microbiana se encuentra en el ambiente interior (Brooks y Davis 1992). La relación I/E de los propágulos fúngicos para el hospital 1 (tomando el valor más alto reportado en la sala de espera 1) fue 184/408 = 0.45. Para el hospital 2 fue 442/404 = 1.09 (tomando el máximo valor de concentración microbiana encontrado en el 3er piso de cirugía). En el caso de aerobacterias para el hospital 1 la mayor concentración fue encontrada en terapia intensiva de niños, de tal modo que la relación fue de 232/280 = 0.82. Para el hospital 2 fue de 448/122 = 3.6 (tomando el valor más alto que fue encontrado en terapia intensiva de adultos). Con estas consideraciones es posible decir que la fuente de contaminación en la mayoría de las muestras de aire colectadas en el hospital 1 es mayor en el exterior (ya que la relación fue menor a 1) y para el hospital 2 la fuente de contaminación procedió del ambiente interior. Esta diferencia podría explicarse en función del tiempo de operación de cada uno de los hospitales.
Las concentraciones obtenidas indicaron que los dos hospitales se consideran como contaminados en ciertas áreas, ya que los niveles de bacterias y propágulos fúngicos estuvieron muy por arriba de los aceptables de acuerdo con lo establecido por la OMS (WHO 1990). Respecto a los niveles de microorganismos del hospital 1 se observa en la figura 2 que para propágulos fúngicos el área de quimioterapia, terapia intensiva (adultos), terapia intensiva (niños) y trasplante (presión 4to. piso) tuvieron concentraciones menores de 100 UFC/ m3, lo que de acuerdo con la OMS es aceptable para lugares donde se encuentran individuos con sistemas inmunosuprimidos. Para aerobacterias las mismas áreas tuvieron niveles menores de 100 UFC/m3 (Fig. 3) excepto terapia intensiva (niños) que presentó 232 ± 4 UFC/m3 y quimioterapia con 136 ± 16 UFC/m3. En cuanto al hospital 2, todas las áreas muestreadas estuvieron por arriba de 100 UFC/m3 excepto pediatría intensiva con 32 ± 4 para hongos y 90± 8 para bacterias (Figs. 2 y 3). De tal modo, el análisis microbiológico del ambiente de los dos hospitales indica que se superan los valores de los estándares para bacterias y hongos en el aire de estos centros hospitalarios de acuerdo con lo establecido por la OMS (Figs. 2 y 3).
La parte del ADN más utilizada para conocer la taxonomía e identificar bacterias es el gen 16S ARNr (Lansac et al. 2000, Weill et al. 2006). En microbiología clínica la identificación rápida y correcta de los agentes patógenos es un requisito esencial para el diagnóstico y la aplicación de un tratamiento adecuado. Los resultados muestran que en el hospital 1 más del 60 % de las bacterias aisladas pertenecen a la familia Enterobacteriaceae (Fig. 3A). Las especies que pertenecen a este grupo habitan ambientes naturales como agua limpia, aguas residuales, vegetales, suelo. Sin embargo, cuando incrementan su población pueden causar infecciones nosocomiales como las del tracto urinario y la bacteremia (Giammanco et al. 2011). Respecto a Enterobacter cancerogenus se ha reportado un incremento en infecciones nosocomiales causadas por esta bacteria (NNIS 2003), la cual además presenta una resistencia natural a aminopenicilinas (Rottman et al. 2002). En tanto que Enterobacter ludwigii es una de las seis especies asignadas a un complejo denominado Enterobacter cloacae que ha llamado la atención por el aumento en su incidencia como patógeno (Mezzatesta et al. 2012). Por otro lado el diagnóstico de Kluyvera asociado a infecciones es de muy baja frecuencia, produce enfermedades de manera aislada y es reconocido principalmente como un patógeno oportunista. Sin embargo, recientemente se han reportado casos en los que Kluyvera se comporta de forma más virulenta de lo que previamente se conocía (Farmer et al. 1981, Sarria et al. 2001, Carter y Evans 2005). Asimismo, se ha reportado que Kluyvera cryocrescens puede causar infecciones serias en pacientes con catéter venoso central, especialmente en inmunosuprimidos (Toprak et al. 2008). En el caso de Rahnella aquatilis se le han adjudicado varios tipos de infecciones como bacteremia (Hoppe et al. 1993, Oh y Tay 1995), sepsis (Goubau et al. 1988), infecciones respiratorias (Harrell et al. 1989) e infecciones del tracto urinario especialmente en pacientes inmunocomprometidos o que presentan enfermedades como endocarditis o enfermedad congénita el corazón (Matsukura et al. 1996). También se ha reportado un caso de sepsis causada por Rahnella aquatilis en un paciente inmunosuprimido (Chang et al. 1999). En hospedantes normales Proteus mirabilis no es un agente causal común en infecciones del tracto urinario (Tolkoff-Rubin y Rubin 1986), aunque sí puede infectar a una alta proporción de pacientes con tractos urinarios que presentan anormalidades anatómicas o con instrumentación crónica (Tolkoff-Rubin y Rubin 1986, Pearson et al. 2008).
Algunos géneros encontrados en el hospital 1 como Pseudomonas son bacterias ubicuas, específicamente las especies aisladas en este hospital no son reconocidas como patógenos de humanos. Sin embargo pueden presentarse como oportunistas (Palleroni 1993, Moore 2006).
Por su lado el hospital 2 mostró una amplia distribución de los organismos aislados comparada con el hospital 1 (Fig. 3B). Además de especies que pertenecen a la familia Enterobacteriaceae se encontraron: una especie del género Pandoraea y tres del género Neisseria, aunque ninguna de ellas tiene importancia como patógeno en humanos (Bennett et al. 2012), además de Alcaligenes, Kluyvera y Klebsiella.
Se ha reportado la presencia de Enterobacter aerogenes en agua limpia, aguas residuales, suelo, productos lácticos y en las heces de animales y humanos. Esta especie produce infecciones y bacteremia (Grimont 2006). Por otro lado, todas las especies del género Neisseria son primariamente comensales de membranas mucosas en mamíferos (Lansac et al. 2000). Sin embargo, sólo dos especies son consideradas de importancia clínica Neisseria meningitidis y Neisseria gonorrhoeae (Bennett et al. 2012). Las demás especies pueden estar presentes en la microbiota natural del tracto respiratorio superior, pero ocasionalmente causan infecciones oportunistas si llegan al torrente sanguíneo (Lansac et al. 2000). Existe un caso reportado en el que Alcaligenes faecalis causó absceso pancreático (Ashwath y Katner 2005). Kluyvera ascorbata ha sido el agente causal de infecciones en el tracto urinario (Isozaki et al. 2010), y septicemia (Moonah et al. 2010). Precisamente de este género es la especie aislada con mayor frecuencia en muestras clínicas (Sarria et al. 2001). Por su parte, Klebsiella pneumoniae es uno de los principales productores de infecciones intrahospitalarias cuya prevalencia va en aumento al volverse resistente, por lo que se le ha relacionado con estancias hospitalarias largas y con la mortalidad (Hoyos-Orrego et al. 2007, Echeverri Toro y Cartaño Correa 2010, Wollheim et al. 2011). Finalmente, Pandoraea pulmonicola se ha aislado en pacientes con fibrosis quística (Costello et al. 2011).
Respecto a los propágulos fúngicos identificados en el hospital 1 se observó que Fusarium equiseti, F. semitectum, F. solani y F. crookwellense fueron de las especies encontradas (Fig. 5B). Los hongos del género Fusarium se encuentran comúnmente en el suelo y han sido aislados de varios frutos y vegetales (Jain et al. 2008). Suelen ser fitopatógenos débiles de algunas especies de plantas, sin embargo, en los últimos años se han reportado infecciones invasivas en pacientes con tratamientos de quimioterapias, con trasplante de médula ósea o que sufren anemia aplásica (Anaissie et al. 1986, Vartivarian et al. 1993, Segal et al. 1998,). La mayoría de los casos de fusariosis invasiva han sido causados por F. solani, F. oxysporum, F. moniliforme y F. chlamydosporum (Guarro y Gene 1995, Segal et al. 1998). Ninguna de estas especies estuvo presente en las muestras aisladas del hospital 1, no obstante, en el hospital 2 se puede ver que F. chlamydosporum estuvo representada y que en general el género Fusarium cubrió el 21 % de todas las muestras (Fig. 6B). Por otro lado, la dermatofitosis causadas porMicrosporum audouinii son infrecuentes en países desarrollados (Fig. 5A). Sin embargo, el género Microsporum resulta de importancia como agente causal de tiña capitis, tiñea corpus y tiñea rosada entre otras dermatofitosis para los pacientes y el personal involucrado en las diversas actividades del hospital (Escutia et al. 2001). El género Penicillium está ampliamente distribuido en la naturaleza por lo que es muy común encontrarlo. Estudios recientes han indicado que las esporas de este hongo en el aire contribuyen a crear varios tipos de alergias respiratorias, específicamente asma y rinitis alérgica (Chow et al. 1999, Chow et al. 2000).
En lo que respecta al hospital 2 se encontró una mayor riqueza de hongos (14 en comparación con seis del hospital 1; Figs. 6A). Después de Fusarium (21 %), Penicillium (17 %) fue el género aislado con mayor frecuencia en el hospital 2 (Fig. 6A). Como ya se ha señalado previamente respecto a Penicillium, también las especies de Aspergillus (4 %) son consideradas prevalentes en el aire y pueden causar alergias en individuos inmunosuprimidos. Asimismo, las especies de este género son los hongos oportunistas más comunes que pueden causar infecciones pulmonares (Shen et al. 2007, Kousha et al. 2011). Otro de los géneros identificados en el aire del hospital 2, fue Cladosporium que también es considerado uno de los hongos más comunes en el ambiente y que es capaz de causar algunas respuestas alérgicas y queratomicosis (Chew et al. 2009). El siguiente de los propágulos fúngicos que podrían tener importancia clínica identificados en el hospital 2 son especímenes del género Pythium, que se asocian con la queratitis (Thanathanee et al. 2013). Entre los hongos que se han documentado como alergénicos están Epicoccum nigrum, Mucor, Alternaria y Cladosporium (Horner et al. 1995). Tomando en consideración estos resultados, vemos diferencias importantes entre los dos hospitales, las cuales pueden ser explicadas en términos del tipo y número de pacientes que reciben, así como de las instalaciones. La identificación de la diversidad de microorganismos que se encuentran en estos hospitales nos ayuda a identificar factores que pueden contribuir a mejorar la calidad del aire y con ello la prevención de infecciones nosocomiales. Ciertas acciones de higiene pueden incluso controlar la presencia de bacterias como Staphylococcus aureus (Rampling et al. 2001). Por ello, prácticas como la desinfección y limpieza de las diferentes áreas del hospital pueden reducir la transmisión de patógenos (Boyce 2007). Asimismo, el uso de sistemas de aire acondicionado con filtros efectivos puede ayudar a mantener los estándares de limpieza que eviten la propagación de microorganismos en el aire (Charkowska 2008, Tigli et al. 2013).
CONCLUSIONES
Esta investigación de la concentración y tipo de bioaerosoles en dos hospitales demuestra que la calidad del aire es pobre y que no se están llevando a cabo medidas esenciales para el control de los mismos. La identificación de propágulos fúngicos de géneros y especies como Cladosporium, Mucor ramosissimus, Alternaría arborencens y Cryptococcus albidus en el hospital 2 y Microsporum en el hospital 1, tienen alta importancia clínica por ser patógenos dermatofitos y agentes de enfermedades respiratorias. Kluyvera cryocrescens y Enterobacter cancerogenus encontradas en el hospital 1 y Alcaligenes faecalis, Kluyvera ascorbata, Klebsiella pneumoniae y Pandoraea pulmonicola presentes en el hospital 2, también se consideran de importancia clínica con posibilidad de incidencia para los usuarios de hospitales.
AGRADECIMIENTOS
Este trabajo fue financiado con fondos del proyecto Convenio: 11-01-A-70, Clave: 2011-04-164493 de CONCYTEG.
REFERENCIAS
Álvarez I. y Wendel J.F. (2003). Ribosomal ITS sequences and plant phylogenetic inference. Mol. Phylogenet. Evol. 29, 417-434. [ Links ]
Anaissie E., Kantarjian H., Jones P., Barlogie B., Luna M., López-Berestein G. y Bodey G.P. (1986). Fusarium. A newly recognized fungal pathogen in immunosuppressed patients. Cancer 57, 2141-2145. [ Links ]
Ashwath M.L. y Katner H.P. (2005). Pancreatic abscess secondary to Alcaligenes faecalis. Am. J. Med. Sci. 329, 54-55. [ Links ]
Augustowska M. y Dutkiewicz J. (2006). Variability of airborne microflora in a hospital ward within a period of one year. Ann. Agric. Environ. Med. 13, 99-106. [ Links ]
Bennett J.S., Jolley K.A., Earle S.G., Corton C., Bentley S.D., Parkhill J. y Maiden M.C. (2012). A genomic approach to bacterial taxonomy: an examination and proposed reclassification of species within the genus Neisseria. Microbiol. 158, 1570-1580. [ Links ]
Boyce J.M. (2007). Environmental contamination makes an important contribution to hospital infection. J. Hosp. Infect. 65, 50-54. [ Links ]
Burge H. (1990). Bioaerosols: prevalence and health effects in the indoor environment. J. Allergy. Clin. Immunol. 86, 687-701. [ Links ]
Brooks B.O. y Davis W. F. (1992). Indoor air sampling methods. En Undernstanding indoor air quality. ( B.O. Brooks y W. F. Davis Eds.) CRC Press, Londres, Reino Unido. Pp. 105-143. [ Links ]
Carter J.E. y Evans T.N. (2005). Clinically significant Kluyvera infections: a report of seven cases. Am. J. Clin. Pathol. 123, 334-338. [ Links ]
Castañeda Roldan E.I., Rivera Tapia J.A. y Lechuga Bautista K. (2003). Determinación de la calidad mi-crobiológica del aire en una industria textil. Revista Latino Americana de la Salud en el Trabajo 2, 21-24. [ Links ]
Castañeda Roldan E.I., Morales Polanco M., Avelino Flores F., Chávez Bravo E., Espinosa Texis A., y Morales Tepatl E. (2006). Cuantificación de bioserosoles en las áreas de proceso de una industria zapatera poblana y su relación con la salud de los trabajadores. Enfermedades Infecciosas y Microbiología 26, enero-marzo. [ Links ]
Cole J.R., Chai B., Farris R.J., Wang Q., Kulam S.A., McGarrell D.M., Garrity G.M. y Tiedje J.M. (2005). The Ribosomal Database Project (RDP-II): sequences and tools for high-throughput rRNA analysis. Nucleic Acids Res. 33, D294-D296. [ Links ]
Cole J.R., Chai B., Farris R.J., Wang Q., Kulam-Syed-Mo-hideen A.S., McGarrell D.M., Bandela A.M., Cardenas E., Garrity G.M. y Tiedje J.M. (2007). The ribosomal database project (RDP-II): introducing myRDP space and quality controlled public data. Nucleic Acids Res. 35, D169-D172. [ Links ]
Costa Baquiao A., Zorzete P., Alvez Reis T., Assuncao E., Vergueiro S. y Benedito C. (2012). Mycoflora and Mycotoxins in field samples of Brazil nuts. Food Control 28, 224-229. [ Links ]
Costello A., Herbert G., Fabunmi L., Schaffer K., Kava-nagh K.A., Caraher E.M., Callaghan M. y McClean S. (2011). Virulence of an emerging respiratory pathogen, genus Pandoraea, in vivo and its interactions with lung epithelial cells. J. Med. Microbiol. 60, 289-299. [ Links ]
Cox C. S. y Wather C. M. (1995). Bioaerosols Handbook. Lewis Publishers, Nueva York, EUA. 656 pp. [ Links ] Chang C.L., Jeong J., Shin J.H., Lee E.Y. y Son H.C. (1999). Rahnella aquatilis sepsis in an immunocompetent adult. J. Clin. Microbiol. 37, 4161-4162. [ Links ]
Charkowska A. (2008). Ensuring cleanliness in operating theatres. Int. J. Occup. Saf. Ergon. 14, 447-453. [ Links ]
Chew F.L., Subrayan V., Chong P.P., Goh M.C. y Ng K.P. (2009). Cladosporium cladosporioideskeratomycosis: a case report. Jpn. J. Ophthalmol. 53, 657-659. [ Links ]
Chow L.P., Su N.Y., Yu C.J., Chiang B.L. y Shen H.D. (1999). Identification and expression of Pen c2, a novel allergen from Penicillium citrinum. Biochem. J. 341, 51-59. [ Links ]
Chow L.P., Chiou S.H., Hsiao M.C., Yu C.J. y Chiang B.L. (2000). Characterization of Pen n13, a major allergen from the mold Penicillium notatum. Biochem. Biophys. Res. Commun. 269, 14-20. [ Links ]
DeSantis T.Z., Hugenholtz P., Larsen N., Rojas M., Brodie E.L., Keller K., Huber T., Dalevi D., Hu P. y Andersen G.L. (2006). Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB. Appl. Environ. Microbiol. 72, 5069-5072. [ Links ]
Echeverri Toro L.M. y Cartaño Correa J.C. (2010). Kleb-siella pneumoniae como patógeno intrahospitalario: epidemiología y resistencia. IATREIA 23, 240-249. [ Links ]
Eickhoff T.C. (1994). Airborne nosocomial infection: a contemporary perspective. Infect. Control. Hosp. Epidemiol. 15, 663-672. [ Links ]
Escutia B., Febrer I., Peman J., Oliver V. y Sanchez-Carazo J.L. (2001). [Tinea capitis by Microsporum audouinii]. Rev. Iberoam. Micol. 18, 88-90. [ Links ]
Farmer J.J., Fanning G.R., Huntley-Carter G.P., Holmes B., Hickman F.W., Richard C. y Brenner D.J. (1981). Kluyvera, a new (redefined) genus in the family En-terobacteriaceae: identification of Kluyvera ascorbata sp. nov. and Kluyvera cryocrescens sp. nov. in clinical specimens. J. Clin. Microbiol. 13, 919-933. [ Links ]
Gasch A.P., Spellman P.T., Kao C.M., Carmel-Harel O., Eisen M.B., Storz G., Botstein D. y Brown P.O. (2000). Genomic expression programs in the response of yeast cells to environmental changes. Mol. Biol. Cell. 11, 4241-4257. [ Links ]
Giammanco G.M., Grimont P.A., Grimont F., Lefevre M., Giammanco G. y Pignato S. (2011). Phylogenetic analysis of the genera Proteus, Morganella and Providencia by comparison of rpoB gene sequences of type and clinical strains suggests the reclassification of Proteus myxofaciens in a new genus, Cosenzaea gen. nov., as Cosenzaea myxofaciens comb. nov. Int. J. Syst. Evol. Microbiol. 61, 1638-1644. [ Links ]
Goubau P., Van Aelst F., Verhaegen J. y Boogaerts M. (1988). Septicaemia caused by Rahnella aquatilis in an immunocompromised patient. Eur. J. Clin. Microbiol. Infect. Dis. 7, 697-699. [ Links ]
Grimont F. y Grimont P. A. D. (2006). The genus Entero-bacter. Prokaryotes 6, 197-214. [ Links ]
Guarro J. y Gene J. (1995). Opportunistic fusarial infections in humans. Eur. J. Clin. Microbiol. Infect. Dis. 14, 741-754. [ Links ]
Gulliver J. y Briggs D.J. (2004) Personal exposure to par-ticulate air pollution in transport microenvironments. Atmos. Environ. 38, 1-8. [ Links ]
Harrell L.J., Cameron M.L. y O'Hara C.M. (1989). Rahn-ella aquatilis, an unusual gram-negative rod isolated from the bronchial washing of a patient with acquired immunodeficiency syndrome. J. Clin. Microbiol. 27, 1671-1672. [ Links ]
NTP 409 (1990). Ministerio de trabajo y asuntos sociales España. Contaminantes biológicos: criterios de valoración. [ Links ]
NTP 608 (2001). Ministerio de trabajo y Asuntos sociales España. Agentes biológicos: planificación de la medición. [ Links ]
Hoppe J.E., Herter M., Aleksic S., Klingebiel T. y Niethammer D. (1993). Catheter-related Rahnella aquatilis bacteremia in a pediatric bone marrow transplant recipient. J. Clin. Microbiol. 31, 1911-1912. [ Links ]
Horner W.E., Helbling A., Salvaggio J.E. y Lehrer S.B. (1995). Fungal allergens. Clin. Microbiol. Rev. 8, 161-179. [ Links ]
Hoseinzadeh E., Samarghandie M.R., Guiasian S.A., Alikhani Y. y Roshanaie G. (2013). Evaluation of bioaerosols in five educational hospitals wards air in Hamedan, During 2011-2012. Jundishapur J. of Microbiol. 6, e10704. [ Links ]
Hoyos-Orrego A., Rivera-Rivera O., Hoyos-Posada C., Mesa-Restrepo C. y Alfaro-Velásquez J. (2007). Características clínicas, epidemiológicas y de susceptibilidad a los antibióticos en casos de bacteriemia por Klebsiella pneumoniae en neonatos. Rev. CES 21, 31-39. [ Links ]
Isozaki A., Shirai K., Mimura S., Takahashi M., Furushima W. y Kawano Y. (2010). A case of urinary tract infection caused by Kluyvera ascorbata in an infant: case report and review of the literature. J. Infect. Chemother. 16, 436-438. [ Links ]
Jain A., Mohan J., Singh M. y Goswami B.K. (2008). Potentiality of different isolates of wilt fungus Fusarium oxysporum collected from rhizosphere of tomato against root-knot nematode Meloidogyne incognita. J. Environ. Sci. Health B. 43, 686-691. [ Links ]
Koetschan C., Forster F., Keller A., Schleicher T., Ruderisch B., Schwarz R., Muller T., Wolf M. y Schultz J. (2010). The ITS2 Database III--sequences and structures for phylogeny. Nucleic. Acids. Res. 38, D275-D279. [ Links ]
Koljalg U., Larsson K.H., Abarenkov K., Nilsson R.H., Alexander I.J., Eberhardt U., Erland S., Hoiland K., Kjoller R., Larsson E. y others (2005). UNITE: a database providing web-based methods for the molecular identification of ectomycorrhizal fungi. New. Phytol. 166, 1063-1068. [ Links ]
Kousha M., Tadi R. y Soubani A.O. (2011). Pulmonary aspergillosis: a clinical review. Eur. Respir. Rev. 20, 156-174. [ Links ]
Lansac N., Picard F. J., Menard C., Boissinot M., Ouellette M., Roy P.H. y Bergeron M.G. (2000). Novel genus-specific PCR-based assays for rapid identification of Neisseria species and Neisseria meningitidis. Eur. J. Clin. Microbiol. Infect. Dis. 19, 443-451. [ Links ]
Leung M. y Chan A.H. (2006). Control and management of hospital indoor air quality. Med. Sci. Monit. 12, SR17-23. [ Links ]
Luo G. y Mitchell T.G. (2002). Rapid identification of pathogenic fungi directly from cultures by using multiplex PCR. J. Clin. Microbiol. 40, 2860-2865. [ Links ]
Matsukura H., Katayama K., Kitano N., Kobayashi K., Kanegane C., Higuchi A. y Kyotani S. (1996). Infective endocarditis caused by an unusual gram-negative rod, Rahnella aquatilis. Pediatr. Cardiol. 17, 108-111. [ Links ]
Mezzatesta M.L., Gona F. y Stefani S. (2012). Enterobacter cloacae complex: clinical impact and emerging antibiotic resistance. Future Microbiol. 7, 887-902. [ Links ]
Moonah S., Deonarine K. y Freeman C. (2010). Multidrug resistant Kluyvera ascorbata septicemia in an adult patient: a case report. J. Med. Case Rep. 4, 197. [ Links ]
Moore E.R.B., Tindall B. J., Martins Dos Santos, V A. P., Pieper, D. H., Ramos J-L. y Palleroni, N. J. (2006). Nonmedical: Pseudomonas. Prokaryotes 6, 646-703. [ Links ]
Nagano Y., Walker J., Loughrey A., Millar C., Goldsmith C., Rooney P., Elborn S. y Moore J. (2009). Identification of airborne bacterial and fungal species in the clinical microbiology laboratory of a university teaching hospital employing ribosomal DNA (rDNA) PCR and gene sequencing techniques. Int. J. Environ. Health Res. 19, 187-199. [ Links ]
NNIS (2003). National Nosocomial Infections Surveillance, System report. Data summary from January 1992 through June 2003 Am. J. Infect. Control. 481-498. [ Links ]
Nilsson R.H., Kristiansson E., Ryberg M., Hallenberg N. y Larsson K.H. (2008). Intraspecific ITS variability in the kingdom fungi as expressed in the international sequence databases and its implications for molecular species identification. Evol. Bioinform. Online 4,193-201. [ Links ]
Nilsson R.H., Bok G., Ryberg M., Kristiansson E. y Hallenberg N. (2009). A software pipeline for processing and identification of fungal ITS sequences. Source Code Biol. Med. 4, 1. [ Links ]
Luksamijarulkul P., Ratthanakhot Y. y Vatanasombooh P. (2012). Microbial counts and particulate matter levels in indoor air samples collected from child home-care center in Bangkok, Thailand. J. Med. Assoc. Thai. 95, S161-S168. [ Links ]
Oh H.M. y Tay L. (1995). Bacteraemia caused by Rahnella aquatilis: report of two cases and review. Scand. J. Infect. Dis. 27, 79-80. [ Links ]
Ottow J.C.G. (1972). Rose bengal as a selective aid in the isolation of fungi and Actinomycetes from natural sources. Mycol. 64, 304-315. [ Links ]
Palleroni N.J. (1993). Pseudomonas classification. A new case history in the taxonomy of gram-negative bacteria. A. Van Leeuw. 64, 231-51. [ Links ]
Pasquarella C., Pitzurra O. y Savino A. (2000). The index of microbial air contamination. J. Hosp. Infect. 46, 241-56. [ Links ]
Pearson M.M., Sebaihia M., Churcher C., Quail M.A., Seshasayee A.S., Luscombe N.M., Abdellah Z., Ar-rosmith C., Atkin B., Chillingworth T., Hauser H., Jagels K., Moule S., Mungall K., Norbertczak H., Rabbinowitsch E., Walker D., Whithead S., Thomson N.R., Rather P.N., Parkhill J. y Mobley H.L. (2008). Complete genome sequence of uropathogenic Proteus mirabilis, a master of both adherence and motility. J. Bacteriol. 190, 4027-4037. [ Links ]
Rampling A., Wiseman S., Davis L., Hyett A.P., Walbridge A.N., Payne G.C. y Cornaby A.J. (2001). Evidence that hospital hygiene is important in the control of methicillin-resistant Staphylococcus aureus. J. Hosp. Infect. 49, 109-116. [ Links ]
Rose C. (1994). Bioaerosols. West. J. Med. 160, 566. [ Links ]
Rottman M., Benzerara Y., Hanau-Bercot B., Bizet C., Philippon A. y Arlet G. (2002). Chromosomal ampC genes in Enterobacter species other than Enterobac-ter cloacae, and ancestral association of the ACT-1 plasmid-encoded cephalosporinase to Enterobacter asburiae. FEMS Microbiol. Lett. 210, 87-92. [ Links ]
Samson R.A., Houbraken J., Thrane U., Frisvad J.C. y Andersen B. (2010). CBS Laboratory Manual Series 2. Food and Indoor fungi. CCentraalbureau voor Schim-melcultures, Utrecht, Países bajos. 390 pp. [ Links ]
Sarria J.C., Vidal A.M. y Kimbrough R.C. (2001). Infections caused by Kluyvera species in humans. Clin. Infect. Dis. 33, e69-e74. [ Links ]
Segal B.H., Walsh T.J., Liu J.M., Wilson J.D. y Kwon-Chung K.J. (1998). Invasive infection with Fusarium chlamydosporum in a patient with aplastic anemia. J. Clin. Microbiol. 36, 1772-1776. [ Links ]
Shen H.D., Tam M.F., Tang R.B. y Chou H. (2007). Aspergillus and Penicillium allergens: focus on proteases. Curr. Allergy Asthma Rep. 7, 351-6. [ Links ]
Thanathanee O., Enkvetchakul O., Rangsin R., Waraasawapati S., Samerpitak K. y Suwan-apichon O. (2013). Outbreak of Pythium keratitis during rainy season: a case series. Cornea 32, 199-204. [ Links ]
Thompson J.D., Gibson T.J., Plewniak F., Jeanmougin F. y Higgins D.G. (1979). The CLUSTALX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res. 25, 4876-4882. [ Links ]
Tigli G.A., Cekin Y., Baysan B.O., Ogunc D., Ongut G., Saba R., Undar L., Mutlu G. y Vural T. (2013). Evaluation of total fungal air contamination levels and efficiency of the ventilation systems used in adult haematology unit and adult stem cell transplantation unit. Afr. J. Microbiol. Res. 7, 5606-5609. [ Links ]
Tolkoff-Rubin N.E. y Rubin R.H. (1986). Urinary tract infection: significance and management. Bull. NY Acad. Med. 62, 131-148. [ Links ]
Toprak D., Soysal A., Turel O., Dal T., Ozkan O., Soyletir G. y Bakir M. (2008). Hickman catheter-related bacteremia with Kluyvera cryocrescens: a case report. Jpn. J. Infect. Dis. 61, 229-30. [ Links ]
Vartivarian S.E., Anaissie E.J. y Bodey G.P. (1993). Emerging fungal pathogens in immunocompromised patients: classification, diagnosis, and management. Clin. Infect. Dis. 17, S487-91. [ Links ]
Weill F.X., Bertrand S., Guesnier F., Baucheron S., Cloeckaert A. y Grimont P.A. (2006). Ciprofloxacin-resistant Salmonella Kentucky in travelers. Emerg. Infect. Dis. 12, 1611-1612. [ Links ]
Weisburg W.G., Barns S.M., Pelletier D.A. y Lane D.J. (1991). 16S ribosomal DNA amplification for phylogenetic study. J. Bacteriol. 173, 697-703. [ Links ]
World Health Organization (1990). Indoor air quality biological contaminants. Report on a WHO meeting. WHO Reg. Publ. Eur. Ser. 31, 1-67 [ Links ]
Whyte W., Hambraeus A., Laurell G. y Hoborn J. (1992). The relative importance of the routes and sources of wound contamination during general surgery. II. Airborne. J. Hosp. Infect. 22, 41-54. [ Links ]
Wollheim C., Guerra I.M., Conte V.D., Hoffman S.P., Schreiner F.J., Delamare A.P., Barth A.L., Echeverrigaray S. y Costa S.O. (2011). Nosocomial and community infections due to class A extended-spectrum beta-lactamase (ESBLA)-producing Escherichia coli and Klebsiella spp. in southern Brazil. Braz. J. Infect. Dis. 15, 138-143. [ Links ]














