Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista del Instituto Nacional de Enfermedades Respiratorias
versión impresa ISSN 0187-7585
Rev. Inst. Nal. Enf. Resp. Mex. vol.18 no.4 México oct./dic. 2005
Original
Detección de Mycoplasma pneumoniae mediante PCR–hibridación in vitro en niños con infección respiratoria
Mycoplasma pneumoniae detection using PCR–in vitro hybridization in children with respiratory infection
María Fernanda Escobar* María del Pilar Delgado* Carlos Jaramillo*
* Grupo de Investigación del Laboratorio de Diagnóstico Molecular, Departamento de Ciencias Biológicas. Universidad de Los Andes, Bogotá, Colombia.
Correspondencia:
Maria del Pilar Delgado
Departamento de Ciencias Biológicas, edificio J (Laboratorio 203).
Universidad de los Andes. Bogotá, Colombia.
Teléfono: (57–1) 3394949,
extensión 3761: fax: (57–1)
3394949, extensión 2817
e–mail: mdelgado@unlandes.edu.co
Trabajo recibido: 07–X–2005
Aceptado: 24–XI–2005
RESUMEN
Antecedentes: Las infecciones respiratorias agudas (IRA) son una de las causas más frecuentes de enfermedades en el mundo y pueden asociarse a complicaciones graves, entre las que la neumonía ocupa un lugar prioritario; diversos agentes virales y bacterianos se implican en su desarrollo. Recientemente Mycoplasma pneumoniae ha adquirido gran importancia ya que estudios epidemiológicos sugieren un aumento en la incidencia de enfermedades respiratorias causadas por este patógeno. Sin embargo, los laboratorios clínicos enfrentan varias dificultades para su detección haciendo necesario el estudio de nuevas técnicas moleculares.
Objetivo: Detección de M. pneumoniae mediante PCR–hibridación in vitro en niños con infección respiratoria.
Métodos: Utilizando la técnica de PCR–hibridación in vitro se analizaron 36 hisopados orofaríngeos de niños entre los 0 y 9 años de edad con infección respiratoria; 36% presentaron neumonía y el 30% bronconeumonía.
Resultados: La implementacion y utilización de la técnica de PCR–hibridación in vitro para la detección de M. pneumoniae permitió detectar su ADN en el 67% de los pacientes estudiados. De éstos, el 33% tenía diagnóstico de neumonía.
Conclusión: La técnica de PCR–hibridación in vitro demostró ser útil en la detección de M. pneumoniae en las muestras analizadas. Los resultados de este estudio evidencian que la presencia de ácidos nucleicos de este patógeno es frecuente en las muestras respiratorias analizadas y sugiere que el microorganismo puede ser un agente etiológico frecuente de neumonías atípicas y de otras patologías respiratorias.
Palabras clave: Infección respiratoria, detección, M. pneumoniae, PCR–hybridization.
ABSTRACT
Background: Acute respiratory infection is considered one of the main causes of morbidity worldwide; it can be associated to serious complications. A great number of viral and bacterial agents have been implicated in the development of the disease. Recently, importance has been given to Mycoplasma pneumoniae as a respiratory pathogen, since epidemiologic studies suggest a raise in the incidence of diseases due to this microorganism. Nevertheless, nowadays clinical laboratories deal with a variety of difficulties in the diagnosis of this agent, raising the need for other molecular methods for its detection.
Objective: The detection of M. pneumoniae in children with respiratory infection by a PCR–in vitro hybridization technique.
Methods: 36 oropharyngeal swabs from children between 0 and 9 years of age with respiratory infection were analyzed by PCR–in vitro hybridization; 36% had pneumonia and 30% bronchopneumonia.
Results: The implementation of the PCR–in vitro hybridization for the detection of M. pneumoniae in patients with respiratory infection allowed the detection of the pathogen's DNA in 67% of the patients studied. Of these, 33% had pneumonia.
Conclusion: PCR–in vitro hybridization is useful for the detection of M. pneumoniae. Our results show the frequent presence of nucleic acids of M. pneumoniae in the samples studied and suggest this microorganism can be a frequent causal agent of respiratory infection.
Key words: Respiratory infection, detection, M, pneumoniae, PCR–hybridization.
INTRODUCCIÓN
La infección respiratoria aguda (IRA) constituye uno de los mayores problemas de salud en los países en vías de desarrollo1. La neumonía es considerada la principal consecuencia de estas infecciones. La neumonía adquirida en comunidad (NAC) produce un impacto importante en los índices de morbimortalidad2,3.
Los agentes causales de IRA incluyen un amplio rango de microorganismos. En los últimos años Mycoplasma pneumoniae ha adquirido una importancia creciente como agente causal, especialmente de la neumonía en niños de edad escolar en los que se puede causar hasta 40% o más de NAC, y es responsable de aproximadamente el 18% de los casos que requieren hospitalización3–5. Estudios basados en pruebas convencionales mostraron que la prevalencia de neumonía por M. pneumoniae en Colombia varía entre 3 y 12.5%6,7.
M. pneumoniae causa manifestaciones clínicas no patognomónicas, similares a las de otras infecciones causadas por distintos patógenos, situación que dificulta el diagnóstico y tratamiento de las enfermedades, incrementando las posibilidades de complicaciones y muerte, especialmente en niños menores de cinco años y adultos de alto riesgo.
Así, en Colombia y en otros países se hace necesario el adecuado manejo y la utilización racional de las técnicas de laboratorio, tanto tradicionales como nuevas que, se encuentran disponibles para el diagnóstico etiológico6,7, aunque es difícil implementar pruebas rápidas, confiables, específicas, sensibles y reproducibles; por tanto, el diagnóstico de estas afecciones es eminentemente clínico sin determinación del agente causal8. Además, las pruebas más utilizadas son poco específicas (títulos de crioaglutininas) o dispendiosas (cultivo)3; sin embargo, las técnicas moleculares para el diagnóstico de la infección respiratoria son altamente sensibles y específicas, permitiendo una detección rápida y confiable9,10.
Objetivo. Detectar M. pneumoniae en hisopados orofaríngeos mediante la técnica de PCR–hibridación in vitro para contribuir a la implementación de estrategias diagnósticas que permitan un mejor conocimiento de la situación real de las infecciones respiratorias causadas por él, y el establecimiento de pruebas útiles para un diagnóstico preciso que permita una terapéutica específica.
MATERIAL Y MÉTODOS
Población de estudio
Treinta y seis pacientes con diagnóstico de infección respiratoria de acuerdo con los criterios establecidos por la OMS; las muestras fueron obtenidas como parte de un proyecto de Vigilancia epidemiológica de la gripe y otros virus respiratorios, en el cual se incluyeron a varias instituciones hospitalarias de Bogotá DC, Colombia.
Especímenes
Se tomaron exudados orofaríngeos mediante un hisopo Virocult® (Medical Wire & Equipment Co. Ltd, Corsham, Wilts., England) dentro de las primeras 96 horas de evolución del proceso infeccioso y fueron conservados a –70°C. Las muestras atemperadas se recuperaron del hisopo en un 1.5 mL de medio de infusión cerebro–corazón suplementado con antibiótico y antimicótico y agitadas en vortex.
Extracción del ADN
Doscientos µl de muestra (hisopado orofaríngeo) fueron transferidos a un tubo estéril y procesados de acuerdo con el protocolo establecido en el Kit QIAamp® DNA Mini Kit (QIAGEN®), donde se incluyó una lisis con proteinasa K y una posterior purificación de ADN por medio de una columna de afinidad. El ADN obtenido fue mantenido en congelación a –20°C hasta su uso en la PCR.
Ensayo de PCR–hibridación in vitro
Se realizó la prueba OligoDetect®–Mycoplasma pneumoniae (Chemicon), con una mezcla de iniciadores que amplificaron una secuencia de 143 pb del gen ATPasa de M. pneumoniae. Cada reacción tenía un volumen final de 25 µl que contenía una concentración de 200 µM de dATP, dGTP, dTTP y dCTP, 2.5 U de Taq polimerasa, 3 mM de MgCI2, 0.3µM de la mezcla de primers y buffer 1X. Después de una desnaturalización inicial por 1 minuto a 94°C, se realizaron 40 ciclos de desnaturalización por 1 minuto a 94°C, anillaje a 45°C por 30 segundos y elongación a 72°C por 1 minuto. Posteriormente se realizó una extensión final por 8 minutos a 72°C en un ter–mociclador Cycler (Bio–Rad).
Como control positivo, en cada reacción de PCR, se utilizó el control de amplificación incluido en el kit. Asimismo, se utilizó agua bidestilada estéril como blanco de reacción en cada montaje realizado. El producto del control positivo se visualizó como una banda de 100 pb, luego de una electroforesis por 1 hora y media en geles de agarosa al 1.5% teñidos con bromuro de etidio. Todas las muestras fueron evaluadas para descartar una posible inhibición de la reacción de PCR, mediante la amplificación del gen de la β–globulina humana utilizando los iniciadores PCO3 y PCO4, lo que resultaba en un producto de 110 pb11.
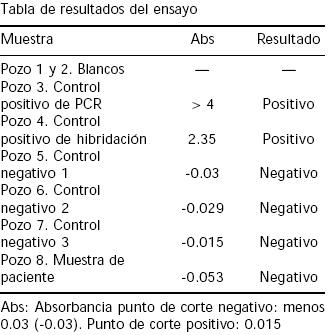
La prueba de hibridación in vitro diseñada para detectar la región amplificada del gen de la ATPasa, consistió en una denaturación bajo condiciones alcalinas seguida de una hibridación, cuyos resultados eran determinados mediante la absorbancia de los controles y las muestras.
En cada reacción de hibridación se incluyeron controles positivos y negativos. Los dos controles positivos incluidos correspondieron a un control de la reacción de hibridación, proporcionado por el kit comercial y un segundo control, constituido por el control positivo del PCR. Como controles negativos se utilizaron el blanco de reacción de PCR y de tres a cuatro controles negativos en cada prueba. El blanco de reacción de hibridación constaba de todos los reactivos utilizados en la prueba, menos los productos de amplificación o controles.
La absorbancia de cada muestra se midió en modo dual en un lector Microplate Reader Benchman (Bio–Rad), usando dos filtros de longitud de onda de 450 nm y 650 nm. Los puntos de corte para las muestras positivas y negativas fueron calculados de acuerdo con las recomendaciones del fabricante.
Inmunofluorescencia indirecta
Con el fin de descartar la posibilidad de virus como agentes etiológicos, se realizó una prueba de inmunofluorescencia para la detección de los siete virus respiratorios detectados con mayor frecuencia en nuestro medio. En vista de la negatividad de los especímenes y el incremento sugerido por algunos estudios epidemiológicos en la frecuencia de infección por otros agentes, entre ellos M. pneumoniae, se procedió a investigar la presencia de este microorganismo.
RESULTADOS
La población de estudio estuvo conformada por 36 individuos; de ellos, 36% presentaron un diagnóstico clínico de neumonía en sus distintas clasificaciones: NAC, por aspiración, multilobular, atípica o viral. De la misma manera se analizaron muestras de pacientes con diagnósticos de bronconeumonía, bronquiolitis, bronquitis, síndrome coquelochoide, síndrome broncoobstructivo y neumonitis, que pueden ser ocasionadas por M. pneumoniae u otros patógenos. Los pacientes incluidos tenían de 0 a 9 años, el 83% hasta 3 años (Tabla I).
La prueba de inmunofluorescencia (Respiratory Panel 1 Viral Screening & Identification IFA Kit, Light Diagnosis), aplicada a todas las muestras utilizadas en el estudio resultó negativa para virus del sincicio respiratorio, virus de la influenza A y B, virus de la parainfluenza tipo 1, 2 y 3 y adenovirus.
Tras la amplificación de un fragmento del gen de la ATPasa de M. pneumoniae, un ensayo de hibridación in vitro realizado en forma consecutiva permitió la detección de los productos de amplificación obtenidos en 24 de las 36 muestras analizadas. La Figura 1 muestra algunos de los resultados en la prueba de PCR–hibridación in vitro.
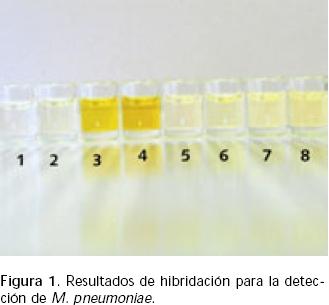
De los 24 pacientes que resultaron positivos para la prueba de PCR–hibridación in vitro, 20 (83%) se encontraron en el rango de menos de 0 a 3 años, 3 tenían entre 4 y 6 años (12.5%), y 1 entre 7 y 9 años. Al 50% se les había diagnosticado neumonía, neumonitis o bronconeumonía viral (bronconeumonía viral, 25% y neumonía viral, 21 %). NAC, 8% y bronquiolitis, 21 % (Tabla I).
Los síntomas y signos de los pacientes positivos versus los pacientes negativos fueron muy similares: Tos, rinorrea, fiebre y dificultad respiratoria fueron descritos en todos los pacientes, aunque el vómito fue más frecuente en los negativos en la prueba de PCR.
DISCUSIÓN
Los síntomas y signos de las infecciones respiratorias no son patognomónicos de un agente etiológico determinado3; los virus son los responsables más frecuentes en los niños, por lo que deben ser considerados como posibles causales de las mismas. En los casos en que se evidencie su ausencia por medio de una prueba que detecte los virus o algunos de sus antígenos, deben usarse otros métodos para establecer la etiología específica.
En los últimos 15 años las técnicas moleculares, como el PCR, han permitido evidenciar algunos microorganismos en diferentes tipos de muestras. Estudios moleculares recientemente realizados en escolares con diagnóstico de IRA han permitido establecer que, además de los virus, las infecciones por las denominadas "bacterias atípicas" como M. pneumoniae se presentan en una proporción significativa de casos, contrario a lo que antes se pensaba10,12. Hoy en día, la incidencia de M. pneumoniae es subestimada debido a que las técnicas utilizadas en el diagnóstico de M. pneumoniae son poco específicas o dispendiosas12,13.
Con base en lo anterior, es necesario desarrollar técnicas diagnósticas sensibles, específicas, rápidas y económicas que permitan precisar el agente etiológico e implementar un tratamiento adecuado.
La valoración de la técnica de PCR–hibridación in vitro en la detección de M. pneumoniae fue la base de este trabajo. Tras la aplicación del método, usando exudados orofaríngeos, se observó que 24 de 36 pacientes resultaron positivos (67%). En varios estudios se ha reportado que M. pneumoniae puede causar del 5 al 50% de las infecciones respiratorias en niños10. Probablemente los resultados encontrados se debieron a la inclusión de pacientes que tenían una alta probabilidad de infección por este patógeno y/o que, el momento de la toma de muestras era una época de gran afluencia o un pico de actividad del M. pneumoniae, descrito como un agente etiológico muy común en niños de 5 a 15 años de edad. Aunque éste no es un estudio epidemiológico, la elevada frecuencia que encontramos entre los 0 y los 4 años sugiere que se deben realizar estudios para determinar la importancia del patógeno en niños de estas edades, los cuales son considerados dentro del grupo de alto riesgo para padecer complicaciones.
Todos los pacientes incluidos en el estudio tenían diagnóstico clínico de infección respiratoria y más del 30% de los pacientes positivos para M. pneumoniae, mediante el PCR–hibridación in vitro, diagnóstico de neumonía, lo que podría sugerir una alta prevalencia de este agente en pacientes que la padecen.
Los resultados de nuestro estudio hacen evidente que las técnicas de detección son fundamentales para distinguir entre infecciones virales y bacterianas, sobre todo en pacientes que padecen complicaciones como la neumonía. Algunas de las ventajas de utilizar técnicas moleculares son la confiabilidad y rapidez; su aplicación arroja resultados en un intervalo de tiempo corto si se compara con el cultivo y algunas pruebas serológicas que utilizan sueros pareados. En el estudio se podían obtener resultados en un par de horas. Adicionalmente, en la literatura se ha descrito que la prueba de PCR–hibridación in vitro es más sensible que el PCR simple9.
Durante el presente estudio todas las extracciones resultaron positivas para la β–globulina utilizada como control interno de inhibición de la reacción, permitiendo inferir que el ADN era calidad PCR y que los hisopados nasofaríngeos son muestras adecuadas para este tipo de pruebas.
LIMITACIONES DEL ESTUDIO
Este estudio descriptivo involucró el análisis de muestras respiratorias provenientes de un proyecto de vigilancia centinela para la detección de virus respiratorios; portante, no se incluyó una población control, sino solamente una de pacientes seleccionados con base en un criterio clínico. En este sentido, el estudio presenta una limitación pues a través de los resultados obtenidos no es posible realizar cálculos de sensibilidad y especificidad de la prueba, razón por la cual no incluimos esto como parte de los objetivos del mismo.
Nuestros resultados, plantean la necesidad de realizar estudios adicionales en diferentes períodos de tiempo, incluyendo un grupo control y pruebas que apoyen el diagnóstico, como el cultivo, las crioglutininas y la detección de anticuerpos tipo IgM e IgG (a través de serologías pareadas), con el fin de obtener datos que permitan estimar la prevalencia real de M. pneumoniae en nuestro medio y establecer si la presentación del agente se puede asociar a una época definida o es constante.
CONCLUSIONES
La aplicación de la técnica molecular usada, PCR–hibridación in vitro, demostró ser de gran utilidad para la detección de M. pneumoniae en hisopados orofaríngeos, ya que la presencia de ácidos nucleicos de M. pneumoniae es frecuente en las muestras respiratorias estudiadas. El presente estudio no pretendió evaluar la técnica de PCR como un método diagnóstico, sino como una herramienta de ayuda para la detección de M. pneumoniae debido a que la sintomatología y el diagnóstico clínico no son patognomónicos de esta infección.
Agradecimientos
A los participantes del proyecto: Vigilancia Centinela de Virus Respiratorios–año 2003, y miembros del Laboratorio de Diagnóstico Molecular. Universidad de los Andes. Bogotá, Colombia.
A Aventis Pasteur S.A, y al Fondo de Investigaciones de la Facultad de Ciencias. Universidad de los Andes. Bogotá, Colombia.
REFERENCIAS
1. Rosete DP, Archundia FJ, Cabello C, Manajarrez ME. Patogenia de las infecciones respiratorias por virus. Rev Inst Nal de Enf Resp Mex 2002:15:239–254. [ Links ]
2. Mclntosh K. Community–acquired pneumonia in children. N Engl J Med 2002:346:429–437. [ Links ]
3. Waites KB. New concepts of Mycoplasma pneumoniae infections in children. Pediatr Pulmonol 2003:36:267–278. [ Links ]
4. Ferwerda A, Moll HA, de Groot R. Respiratory tract infections by Mycoplasma pneumoniae in children: a review of diagnostic and therapeutic measures. Eur J Pediatr 2001:160:483–491. [ Links ]
5. Heath PT. Epidemiology and bacteriology of bacterial pneumonias. Paediatr Respir Rev 2000:1:4–7. [ Links ]
6. Martínez O. Neumonía por Mycoplasma pneumoniae en el Hospital de la Misericordia. Actual Pediatr 1993:3:166–170. [ Links ]
7. Trujillo H, Robledo J, Díaz FJ, et al. Neumonía por Mycoplasma pneumoniae en 107 niños de Medellín. Actual Pediatr 1998:8:20–24. [ Links ]
8. Gupta SK, Sarosi GA. The role of a typical pathogens in community– pneumonia. Med Clin North Am 2001:85:1349–1361. [ Links ]
9. Loens K, leven M, Ursi D, et al. Detection of Mycoplasma pneumoniae by real–time nucleic acid sequence–based amplification. J Clin Microbiol 2003:41:4448–4450. [ Links ]
10. Waris ME, Toikka P, Saarinen T, et al. Diagnosis of Mycoplasma pneumoniae pneumonia in children. J Clin Microbiol 1998:36:3155–3159. [ Links ]
11. Saiki RK, Scharf S, Falooma F, et al. Enzymatic amplification of beta–globin genomic sequences and restriction sites analysis for diagnosis of sickle cell anemia. Science 1985:230:1350–1354. [ Links ]
12. Honeybourne D. Community–acquired pneumonia in ambulatory patients: relative importance of atypical pathogens. Int J Antimicrob Agents 2001:18 Su–ppl:57–61. [ Links ]
13. Ursi D, leven M, Noordhoek GT, Ritzier M, Zandleven H, Altwegg M. An interlaboratory comparison for detection of Mycoplasma pneumoniae in respiratory samples by polymerase chain reaction. J Microbiol Methods 2003:53:289–294. [ Links ]














