Introducción
Previo al presente reporte se llevó a cabo una amplia investigación sobre tres razas de maíz de la Altiplanicie Mexicana: Chalqueño, Cónico y Cónico Norteño, con el objetivo de encontrar patrones de distribución geográfica y racial de nudos cromosómicos que pudieran conducir a conocer la caracterización de cada raza estudiada y proponer el origen de dichas razas, como es sugerido por McClintock et al. (1981). La investigación fue publicada como libro, donde se encuentran todos los antecedentes, los materiales utilizados, los datos obtenidos y los detalles del análisis de dichos datos. En los párrafos siguientes se hace la condensación de los resultados más importantes del libro de Kato et al. (2016).
Los nudos cromosómicos son estructuras heterocromáticas esféricas u ovoides de tamaño variable que se observan en sitios fijos en los cromosomas paquiténicos de la primera división meiótica en el género Zea (teocintle y maíz). En las poblaciones de maíz se han encontrado en conjunto 21 posiciones de los nudo; los teocintles anuales mexicanos han mostrado 34 posiciones con la excepción de cinco (3C2, 4C2, 5C1, 7C, y 9C) que son terminales; en Zea diploperennis se observan 17 posiciones terminales; en los teocintles anuales guatemaltecos se han encontrado 31 posiciones (18 en los de Huehuetenango y 13 en los del sur de Guatemala), todos terminales; esto es la mejor evidencia que se tiene de que el teocintle anual mexicano es el ancestro del maíz cultivado (Kato, 1976; Kato y López, 1990; Kato y Sánchez, 2002).
Desde hace varias décadas se sabe que los cromosomas están estructurados por cromatina, que es una cadena en serie de nucleosomas (ADN e histonas) y no sólo por ADN; también se conoce que los nudos cromosómicos no sólo varían en tamaño sino que cuentan con una organización compleja que les confiere funcionalidad, cosistente en heterocromatina con segmentos repetitivos de 180 pb repetidos en serie en largas cadenas compactadas durante todo el ciclo celular (heterocromatina estructural) (Peacock et al., 1981); sin embargo, es mucho más compleja la constitución de esa heterocromatina, ya que también se encuentra entremezclada otra serie repetitiva de 350 pb (TR-1); además, se ha encontrado que diferentes nudos en distintas poblaciones de maíz pueden tener la secuencia 180 pb como predominante; en otras, la predominancia es de la secuencia 350 pb y, aún en otros casos las dos secuencias están presentes en varias proporciones formando grandes cadenas entremezcladas. Esta complejidad todavía se convierte en algo más complejo por la presencia de diferentes retrotransposones (elementos clase I que mediante moléculas de ARN llevan a cabo la transposición) formando secuencias repetitivas también de diversas longitudes y entremezcladas con cadenas de las secuencias de 180 y 350 pb (Ananiev et al. 1998a; 1998b).
La complejidad de los nudos no termina ahí, ya que otras investigaciones han mostrado que los cromosomas y, por tanto, la heterocromatina, es aún muy complicada en su estructura y organización, las moléculas de histonas H3 y H4 de los nucleosomas de la cromatina son metiladas, acetiladas, fosforiladas, etc. en diversas frecuencias y combinaciones, lo que provoca la compactación de la cromatina en diferentes grados. Con estas modificaciones de la cromatina y asociaciones posteriores con otras histonas cromosómicas, como la HP1, la configuración tridimensional de la heterocromatina puede variar, lo que afecta la expresión del genoma (variación epigenética) (Dillon, 2004; Grewal y Jia, 2007; Grewal y Moazed, 2003).
Los retrotransposones están sujetos a un probable silenciamiento mediante un proceso de metilación dirigida por ARN, que a su vez modifica las histonas que caracterizan el estado heterocromático: ausencia de la transcripción e inactivación de la transposición. Debido a que los elementos de transposición consisten en ADN repetitivo que puede causar aberraciones cromosómicas, se ha propuesto que el proceso de metilación modifica y reorganiza la cromatina para inactivar retrotransposones, reduciendo de esta manera el impacto de estos elementos sobre el genoma hospedero; sin embargo, no todos los retrotransposones son metilados, lo que sugiere que el proceso de inactivación da oportunidad a que algunos retrotransposones contribuyan a la adaptación (domesticación molecular) de las plantas, creando nuevos reguladores (promotores) de los genes. Los retrotransposones conforman el 78 % de la fracción repetitiva, constituyendo gran parte del genoma del maíz, la selección de los retrotransposones con el tiempo puede dar oportunidad para que ocurran modificaciones en las secuencias de ADN favorables en la expresión génica (SanMiguel y Vite, 2009). Lo antes descrito y explicado ha demostrado que la heterocromatina no es un ADN inactivo o de desperdicio, sino todo lo contrario, constituye una gran porción activa de los genomas contribuyendo a la expresión génica mediante mecanismos que afectan la estructura y la actividad de los elementos transponibles, que a su vez, actúan en forma diversa sobre los genes, causando lo que se denomina variación epigenética. De esta forma, los nudos cromosómicos, constituidos por heterocromatina y retrotransposones, son moduladores de la expresión genética del genoma del maíz y así pueden ser motivo de selección tanto natural como artificial y ser capaces de formar diversos patrones de distribución geográfica y racial, explicando los patrones encontrados por McClintock et al. (1981), lo que permitió formular y proponer la teoría multicéntrica del origen y diversificación del maíz (Kato et al., 2009).
Se han postulado muchas teorías sobre el origen del maíz, pero actualmente prevalecen solamente dos: la teoría unicéntrica y la multicéntrica (Kato et al., 2009). La teoría unicéntrica del origen del maíz de Matsuoka et al. (2002), aun cuando acepta que el ancestro del maíz es el teocintle, se basó en datos de frecuencias de microsatélites de una muestra pequeña de poblaciones del continente americano (193 accesiones), y además, con un número muy limitado de plantas por población de maíz y de teocintle (una planta por población), postula que el maíz fue originado y domesticado en una región entre los estados actuales de Michoacán, México y Guerrero mediante un solo evento (no especificado) a partir del teocintle Z. mays ssp. parviglumis. El problema de esta teoría es que considera tácitamente que el genoma del maíz está estructurado solamente por ADN y no por cromatina (ADN más histonas) como actualmente se sabe (Allis et al., 2007), y se analizó una muestra tan pequeña (una planta por accesión) que difícilmente puede explicar la gran variación genética racial del maíz actual en México y en el mundo, si el origen y evolución posterior del maíz se inició a partir de unas cuantas plantas formadas originalmente y se propone que la restante variación racial fue adquirida por hibridación con el teocintle (Matsuoka et al., 2002).
Hasta el presente no ha habido una propuesta de la forma en que se originó la gran variación racial existente en México, aparte de la de Wellhausen et al. (1951), que considera que las razas Palomero Toluqueño, Arrocillo Amarillo, Chapalote y Nal Tel son primitivas, con las cuales formó una genealogía consistente en una serie de hibridaciones hipotéticas entre ellas y con las resultantes de esas hibridaciones. Esta genealogía es aceptada aún en la actualidad (Ortega et al., 2013).
No habiendo otra propuesta genealógica de las razas de maíz de México y considerando la teoría multicéntrica de McClintock et al. (1981), se planteó que un estudio más amplio (con más accesiones por raza y mayor número de plantas analizadas por accesión) sería posible encontrar patrones de distribución geográfica y racial que indiquen la forma en que pudo originarse cada una de las razas y es lo aquí se muestra con los resultados más relevantes obtenidos de tres razas, Chalqueño, Cónico y Cónico Norteño, que fue publicado como libro (Kato et al., 2016). El objetivo del presente documento es dar mayor difusión a los resultados de ese libro de una forma condensada.
Materiales y métodos
Material genético
Para este estudio se utilizaron 15 accesiones de la raza Chalqueño, 22 de Cónico y 19 de Cónico Norteño. La semilla de estas accesiones fue proporcionada por el Banco de Maíz del CIMMYT. Las accesiones fueron aquellas registradas como representantes típicos de cada una de las razas en el catálogo del Banco de Maíz del CIMMYT.
Siembra en campo y fijación de muestras
La siembra se realizó en el Campo Experimental del Campus Montecillo del Colegio de Postgraduados en Montecillo, Estado de México. De las plantas obtenidas se utilizaron 20 espigas de plantas individuales por accesión, que fueron fijadas en una mezcla de alcohol etílico 96 % y ácido acético glacial 3:1 v/v por 48 horas y conservadas en alcohol etílico 70 % bajo refrigeración. Las preparaciones y las observaciones citológicas se llevaron a cabo de la misma manera que en McClintock et al. (1981).
Análisis de nudos cromosómicos
Para el análisis de los datos de nudos cromosómicos el idiograma del genoma del maíz se dividió en cuatro partes (Figura 1), de tal manera que se pudieran hacer comparaciones entre las frecuencias de diferentes grupos de nudos tomados de dos en dos de cada una de las accesiones estudiadas de las tres razas (e.g. cromosomas largos vs. cromosomas cortos, brazos largos vs. brazos cortos de todo el genoma, brazos largos vs. brazos cortos de cromosomas largos, brazos largos vs. brazos cortos de cromosomas cortos, etc). También se llevaron a cabo comparaciones entre frecuencias de los tamaños de nudos (grande, mediano, y pequeño) de distintas posiciones de nudo individuales de todos los cromosomas (1C1 vs 1L1, 2L1 vs 3L1, 9C vs 9L2, etc.). De esta manera, fue posible ver si las diferentes comparaciones llegaban a formar distintos patrones de distribución geográfica o raciales que permitieran visualizar la manera en que las frecuencias de distintas agrupaciones de tipos de nudo y de posiciones específicas en los cromosomas revelaban la caracterización de cada raza (Kato et al., 2016).
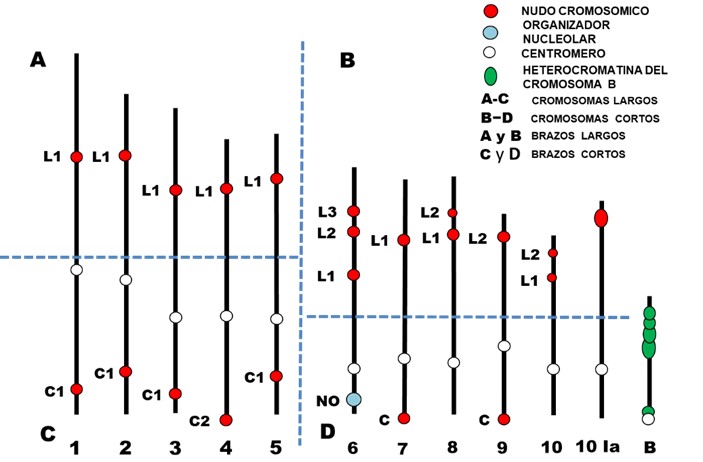
Figura 1 División del idiograma del género Zea mostrando los diferentes grupos de nudos cromosómicos: cromosomas largos y cortos, brazos largos y cortos de cromosomas largos, brazos largos y cortos de cromosomas cortos.
Esta metodología ha permitido relacionar positivamente algunas razas con pocos datos (Sánchez-González et al., 1998).
Resultados
Caracterización de las razas
Si se considera la división en grupos del genoma total (Figura 1) y se selecciona la de cromosomas largos (1, 2, 3, 4 y 5) y la de cromosomas cortos (6, 7, 8, 9 y 10), y sus frecuencias de nudos grandes se comparan entre ellos, se obtiene el resultado de la Figura 2, donde se observa que los nudos grandes se concentran en las accesiones de la raza Cónico Norteño en la parte superior derecha de frecuencias altas del espacio cartesiano, con promedio de 40 % en los brazos largos y de 25 % en los brazos cortos. y en la parte inferior izquierda se localiza un grupo de siete accesiones de Cónico (Hgo.92, Pue.32, Pue.55, Méx.3, Méx.102, Méx.207 y Tlax.2) con relativamente pocos nudos grandes en promedio (8 % en brazos largos y 2.5 % en brazos cortos) y que aquí se denominó como Cónico Mesa Central; en posición intermedia entre estos dos grupos se encuentran las accesiones restantes de la raza Cónico, que tienen menor frecuencia de nudos grandes (30 % en cromosomas largos y 12 % en cromosomas cortos), mezcladas con las de las accesiones de Chalqueño que tienen nudos grandes en frecuencias similares. Ahora, si en la misma comparación de cromosomas largos vs. cromosomas cortos se consideran los nudos medianos se obtiene que las frecuencias de tales nudos medianos de las accesiones de Cónico y de Chalqueño son similares (10 % en los cromosomas largos y 8 % en los cortos) y se localizan mezcladas; las accesiones de Cónico Norteño y de Cónico Mesa Central ahora están en la región de menores frecuencias (Figura 3); si la misma comparación se hace con las frecuencias de nudos pequeños, el patrón que resulta es que todas las accesiones de Chalqueño se posicionan en la región cartesiana de mayores frecuencias y las accesiones de Cónico Norteño y de Cónico se presentan en la parte cartesiana de menores frecuencias (Figura 4).

Figura 2 Asociación entre frecuencias globales (%) de nudos de tamaño grande en cromosomas largos vs. cromosomas cortos de los genomas de cada una de las accesiones de las razas de maíz Chalqueño, Cónico y Cónico Norteño de la Altiplanicie Mexicana.
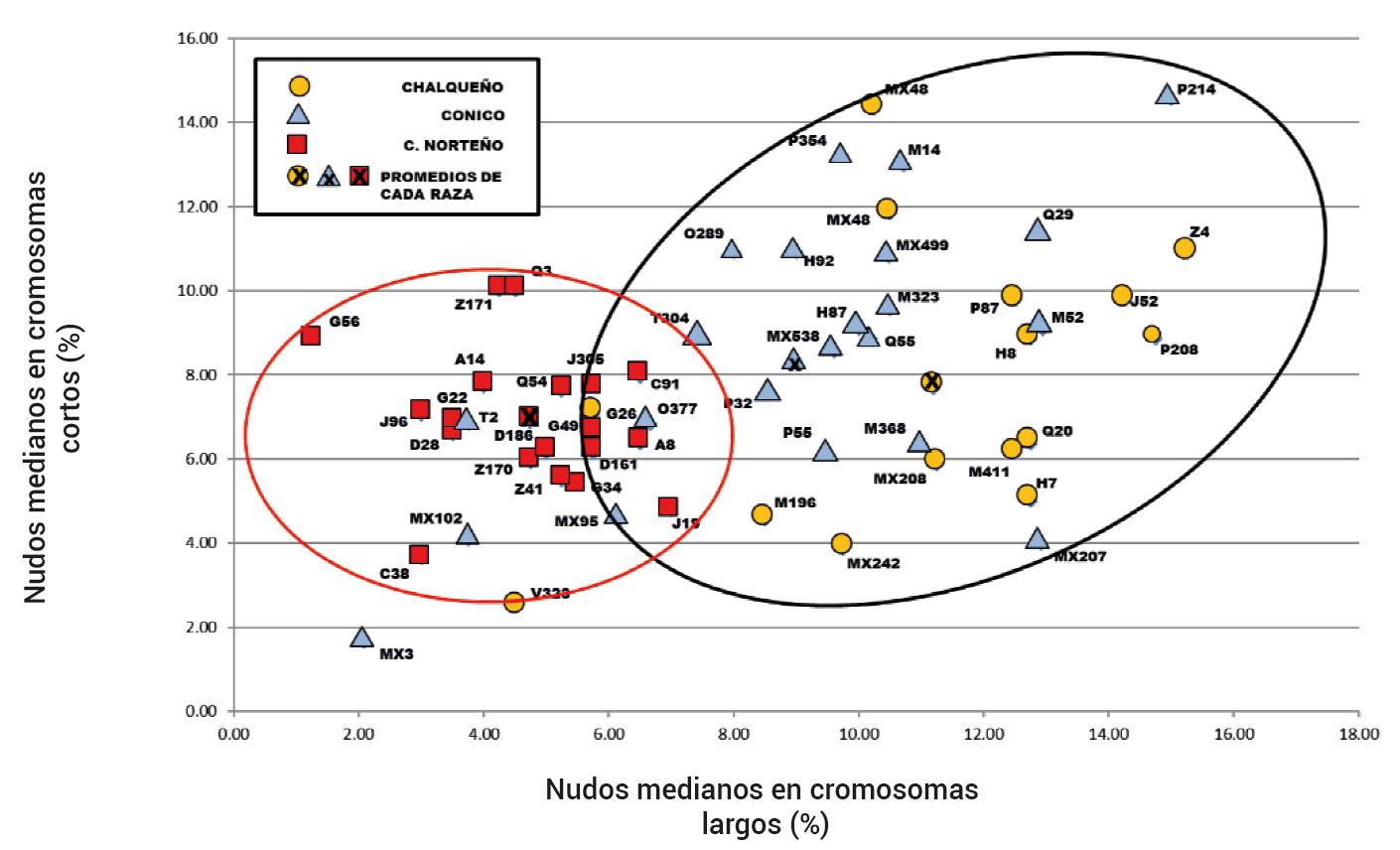
Figura 3 Asociación entre frecuencias globales (%) de nudos de tamaño mediano en cromosomas largos vs. cromosomas cortos de los genomas de cada una de las accesiones de las razas de maíz Chalqueño, Cónico y Cónico Norteño de la Altiplanicie Mexicana.
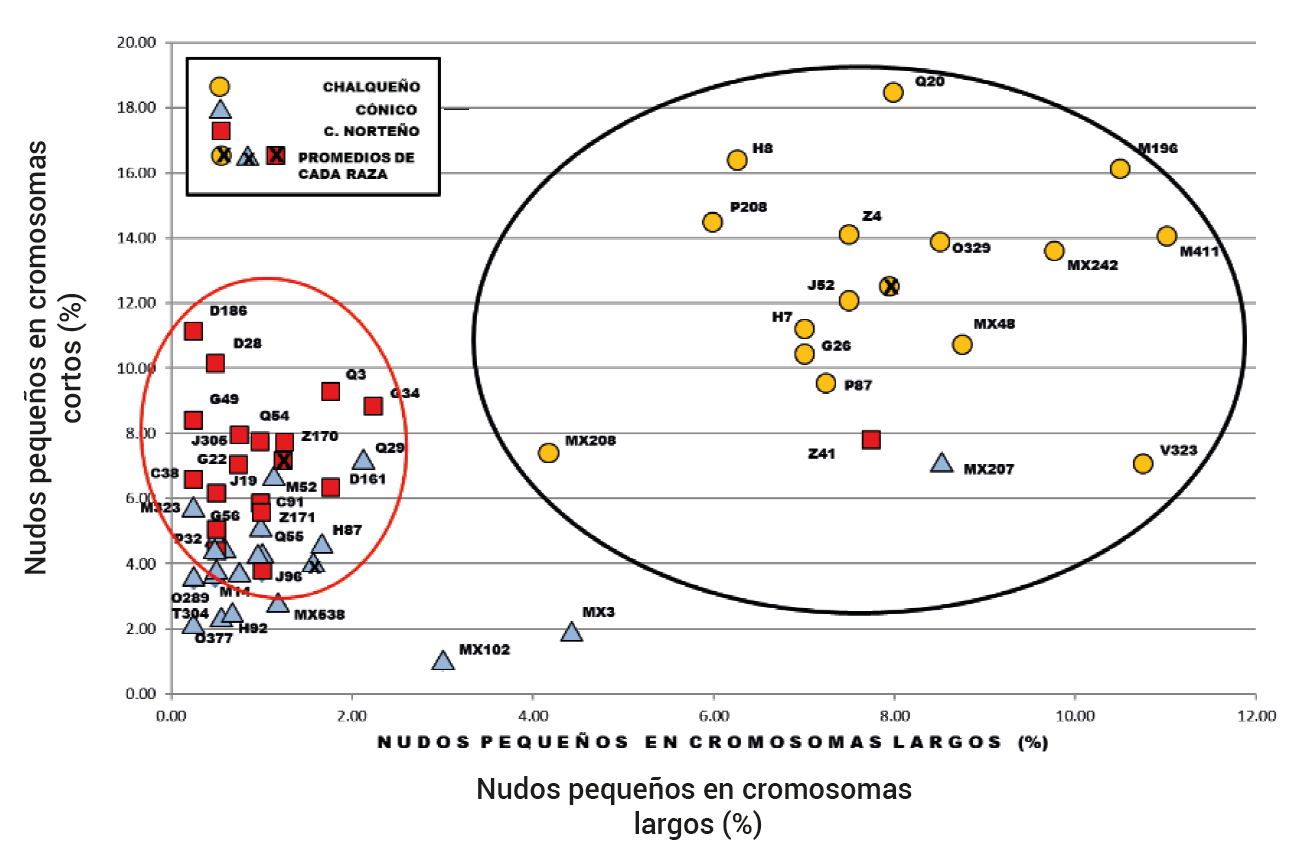
Figura 4 Asociación entre frecuencias globales (%) de nudos de tamaño pequeño en cromosomas largos vs. cromosomas cortos de los genomas de cada una de las accesiones de las razas Chalqueño, Cónico y Cónico Norteño de la Altiplanicie Mexicana
Resumiendo, la caracterización de las tres razas es la siguiente: a) la raza Cónico Norteño se caracteriza porque sus accesiones tienen las mayores frecuencias de nudos grandes y comparativamente menores frecuencias de nudos medianos y pequeños que las razas Cónico y Chalqueño; b) la raza Cónico se caracteriza porque sus accesiones tienen en general una menor frecuencia de nudos grandes que Cónico Norteño y esas frecuencias las comparte con las accesiones de Chalqueño; por el contrario las frecuencias de nudos medianos son similares a las accesiones de Chalqueño pero mayor que las de Cónico Norteño; c) la raza Chalqueño se caracteriza porque tiene mayor frecuencia de nudos pequeños que las otras razas en estudio; d) las siete acciesones de lo que se llamó Cónico Mesa Central se caracterizan por mostrar siempre menores frecuencias de los tres tamaños de nudos en las tres comparaciones; posiblemente podría considerarse a este grupo como una sub-raza de la raza Cónico.
Los patrones de distribución racial obtenidos en el grupo de cromosomas largos vs. cromosomas cortos del genoma completo se repiten al hacer comparaciones similares en los otros grupos; es decir, brazos largos vs. brazos cortos de cromosomas largos, brazos largos vs. brazos cortos de cromosomas cortos y también haciendo comparaciones entre loci individuales; por ejemplo, 1L1 vs. 2L1, 2L1 vs. 3L1, etc; sin embargo, en muchos casos de estas últimas comparaciones la fidelidad en las repeticiones se pierde bastante, lógicamente a causa de la reducción de los datos comparados.
Origen de las tres razas
Con la información generada y con ayuda de información sobre el origen multicéntrico del maíz obtenido por McClintock et al. (1981) fue posible concebir la genealogía de las razas Chalqueño, Cónico, y Cónico Norteño (Figura 5); esto sugiere, en primer lugar, que los nudos cromosómicos son estructuras funcionales que mediante sus componentes variables (heterocromatina y retrotransposones) pueden actuar como moduladores de la expresión de los genes. Si esto es aceptable, entonces los nudos están sujetos a la selección natural y a la artificial, lo que sugiere que están contribuyendo a la evolución de las poblaciones del género Zea. Esto es posible, ya que en la actualidad se conoce que los cromosomas están estructurados por la cromatina (ADN más histonas, nucleosomas) y no solamente por ADN; es decir, ahora lo que controla la genética y la evolución es la epigenética (Allis et al., 2007).
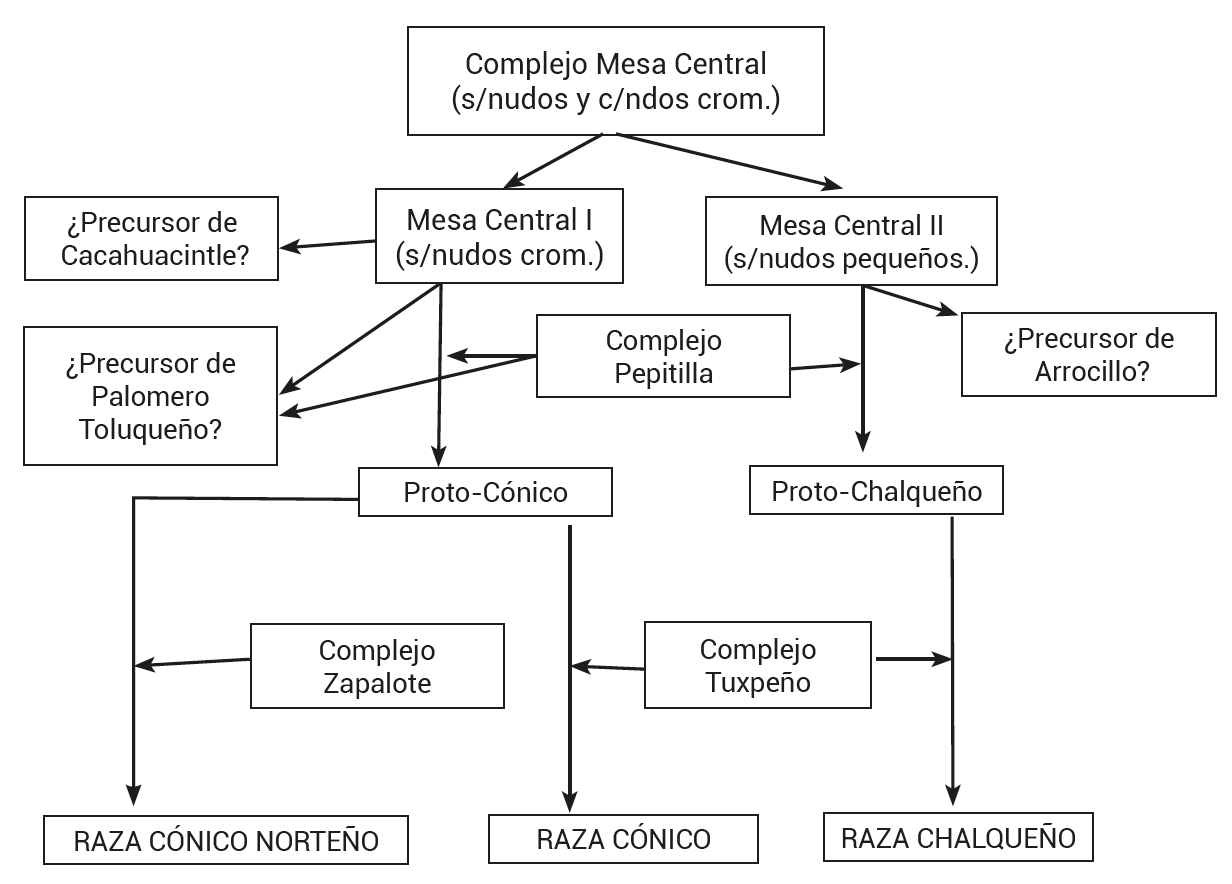
Figura 5 Genealogía propuesta con base en la constitución de nudos cromosómicos de accesiones de las razas de maíz Chalqueño, Cónico y Cónico Norteño de la Altiplanicie Mexicana.
Con el nudo grande el patrón consiste en que las accesiones de Cónico Norteño se localizan en la región de frecuencias altas, las accesiones de Cónico Mesa Central en la parte opuesta de bajas frecuencias, y entre los dos grupos las accesiones de Cónico y Chalqueño generalmente entremezcladas. En el caso de los nudos medianos, las accesiones de Cónico Mesa Central, junto con las de Cónico Norteño, se distribuyen en la parte de bajas frecuencias y las de Cónico y Chalqueño en las de altas frecuencias, con tendencia a entremezclarse entre ellas; con los nudos pequeños las accesiones de Cónico Mesa Central, Cónico y Cónico Norteño tienden a distribuirse sobrepuestas en el espacio de bajas frecuencias y, ahora, con los nudos pequeños las accesiones de Chalqueño se dispersan en la región de frecuencias mayores y las de Cónico Norteño y todas las de Cónico, incluidas las de Cónico Mesa Central, se localizan juntas en la parte de bajas frecuencias. Estas interpretaciones se confirman con el análisis de las comparaciones entre frecuencias de nudos de diferentes tamaños y localizados en posiciones especificas (loci) en los cromosomas; consecuentemente, todo indica que las conclusiones generales a que se ha llegado posiblemente son las correctas.
De acuerdo con McClintock (1978) y McClintock et al. (1981), en el Altiplano Mexicano existió un complejo germoplásmico que contenía cromosomas básicamente sin nudos cromosómicos y estos nudos eran de tamaño pequeño (Complejo Mesa Central). Este germoplasma pudo haber formado dos grupos de poblaciones: 1) con cromosomas totalmente sin los nudos pequeños (Mesa Central I) y 2) con cromosomas con nudos pequeños (Mesa Central II). Mesa Central I posiblemente dio origen al precursor de la actual raza Cacahuacintle y Mesa Central II dio origen al precursor de la actual raza Arrocillo Amarillo. Actualmente se tiene información de que dos accesiones de Palomero Toluqueño (Méx. 210 y 211) difieren en su contenido de nudos, Méx. 210 posee buen número de dichos nudos; en cambio, Méx. 211 contiene muy pocos (McClintock et al. (1981), esto sugiere que Mesa Central I pudo originar al precursor de la raza Palomero Toluqueño por introgresión del Complejo Pepitilla. El hecho de que Cónico y Chalqueño poseen frecuencias similares de nudos grandes puede explicarse si los germoplasmas Mesa Central I y II fueron influenciados por el Complejo Pepitilla creando al Proto-Cónico y al Proto-Chalqueño. El Proto-Cónico derivó en las actuales razas Cónico y Cónico Norteño porque Cónico tiene adicionalmente más nudos medianos, derivados de la influencia del Complejo Tuxpeño, mientras que Cónico Norteño contiene más nudos grandes derivados de la introgresión del Complejo Zapalote; por el otro lado, Mesa Central II obtuvo nudos grandes por introgresión del Complejo Pepitilla, creando al Proto-Chalqueño que, a su vez, adquirió nudos medianos del Complejo Tuxpeño resultando la raza Chalqueño actual (Figura 5).
Discusión General
Considerando los resultados de McClintock et al. (1981), los nudos cromosómicos grandes llegaron a la Altiplanicie Mexicana a partir de dos puntos diferentes; primero, por una corriente migratoria desde la región de Oaxaca-Chiapas del complejo Zapalote a lo largo de la costa del Pacífico hasta la frontera norte de México con Estados Unidos, de cuya parte intermedia, posiblemente a la altura de Jalisco y Michoacán, se originó un movimiento migratorio hacia la parte norteña de Zacatecas, Durango y Chihuahua de la Altiplanicie Mexicana; la segunda posibilidad de contribución de nudos grandes a los maíces primigenios de la altiplanicie es la del complejo Pepitilla, que se domesticó en el sur de la Mesa Central en la región de altura media de Morelos, sur de Puebla, suroeste del Estado de México y norte de Guerrero, germoplasma que emigró hacia el norte y el occidente de México. De esta manera, lo antes expresado confirma una vez más que la idea de buscar patrones de distribución de nudos cromosómicos es una manera muy efectiva para conocer muchos aspectos sobre la evolución del maíz y de sus razas; además, con el avance en el conocimiento de la estructura molecular de los cromosomas y, en particular de la heterocromatina, los nudos cromosómicos ahora no sólo son simples marcadores citológicos sin ninguna función genética, sino por el contrario, resultan ser tan importantes como lo son el conjunto de genes que se localizan en la eucromatina y la interacción entre ellos en la expresión del genoma (Ananiev et al., 1998a; 1998b; Bilinski, et al., 2018; González et al., 2013; González y Poggio, 2021; Llaca et al., 2011; Peacock et al., 1981; SanMiguel y Vitte, 2009); además, Bilinski et al. (2018) encontraron que en el género Zea el tamaño genómico muestra una correlación negativa con el incremento en altitud del cultivo y, adicionalmente, González et al. (2013) y González y Poggio (2021), usando la coloración con DAPI para determinar el número de nudos cromosómicos de las plantas, factor principal del tamaño genómico y, con la técnica de FISH han podido caracterizar poblaciones de maíz de varias razas del noroeste de Argentina y Bolivia, y teocintle de México y Centroamérica. El hecho de que la heterocromatina constitutiva en el género Zea se encuentre distribuida en puntos específicos de los diez cromosomas de su genoma es muy afortunado, porque de esta manera es posible analizar de muchas formas, en detalle, diversos aspectos de las propiedades del genoma y poder llegar a conocer la manera en que han evolucionado y siguen evolucionando las poblaciones de estas plantas; ésto hace posible que una caracterización más completa pueda lograrse combinando métodos moleculares y citogenéticos, asociando los estudios de nudos cromosómicos, tal como los realizados para el presente estudio y los realizados por González et al. (2013) y González y Poggio (2021), lo que puede resultar un tanto más preciso, ya que los primeros toman en cuenta la variación en tamaño de los nudos y la variación de sus componentes: nudos grandes más heterocromatina y retrotransposones, nudos pequeños menos contenido de estos compuestos; además, el estudio citológico considera la posición de los nudos en los cromosomas.
El hecho de que la presentación gráfica de los datos en los espacios determinados por dos ejes que representan frecuencias, tanto de grupos de nudos de diferentes tamaños como de nudos presentes en posiciones específicas en distintos cromosomas, resulta en patrones de distribución particulares de las accesiones de cada una de las razas estudiadas, proporcionando las indicaciones básicas con las que se pudo determinar las probables secuencias de eventos que dieron origen a esas razas; otro aspecto que se observó es que el mismo patrón se repite cuando se consideran, tanto grupos de nudos como de nudos en posiciones específicas. Por otro lado, un determinado nudo representativo de una raza (e.g., el nudo grande en Cónico Norteño o el pequeño en la raza Chalqueño) en sus diferentes accesiones se encuentra con una gran variación en frecuencias, lo que causa que se forme una amplia dispersión de esas accrsiones; asimismo, las accesiones de una raza dada muchas veces tienden a quedar separadas como grupo de las accesiones de las otras razas. Estos resultados generales son de mucho interés e importantes por dos razones: una es que resulta la pregunta ¿qué es lo que provoca dicha variación en las accesiones? y la segunda, ¿qué puede significar esa dispersión? En las investigaciones que condujeron al descubrimiento de los elementos de control (ahora conocidos como transposones), McClintock (1951) también encontró evidencias de que estos elementos de control provienen de la heterocromatina. En la actualidad, se conoce que la estructura de los nudos puede variar a causa de modificaciones de sus componentes; primero, la variación de las proporciones entre las secuencias 180 y 350 pb del ADN repetitivo y el grado de metilación de estas secuencias; la metilación de las histonas en los nucleosomas de la cromatina; variación en el número y la clase de retrotransposones presentes, que fue descrita con mayor amplitud en el capítulo introductorio de este documento (Ananiev et al., 1998a; 1998b; Grewal y Jia, 2007; Grewal y Moazed, 2003; Lamb et al., 2007; Lisch, 2009; Llaca et al., 2011; Peacock et al., 1981; SanMiguel y Vitte, 2009; Xiong et al., 2005); entonces, es clara la posibilidad de que las accesiones de una raza pueden tener nudos con diferente estructura del ADN que lo forma, inclusive si los nudos son del mismo tamaño; de esta forma, si la variación estructural le confiere también variación funcional; es decir, una actividad para controlar o modular la expresión genética del genoma, de tal manera que los nudos pueden adquirir, y así participar en los procesos de adaptación de las poblaciones y, por ende, en los cambios evolutivos dentro de la especie Zea mays, así se explicaría la variación en frecuencias de nudos dentro y entre accesiones y su dispersión dentro de una raza o entre las de diferentes razas.
Las proporciones de diferentes nudos en la mayoría de las posiciones de nudo de los diferentes cromosomas del genoma de cada una de las accesiones de las tres razas estudiadas es tal que, cuando se comparan de dos en dos, forman o tienden a formar los mismos patrones de distribución de accesiones que cuando se hacen comparaciones entre las frecuencias de nudos de distintos tamaños entre diferentes agrupaciones de cromosomas o sus brazos; esto indica que el proceso de diversificación se lleva a cabo primero en las poblaciones y después en las razas en que la heterocromatina de los nudos y los genes de la eucromatina interactúan armónicamente de forma holística; siendo esto así, lógicamente la selección natural, junto con la artificial, actuarían también en concordancia sobre los cambios fenotípicos, resultado de la expresión epigenética del genoma. Entonces, la diferenciación de poblaciones y creación de razas, y aún de especies, no es cuestión de acción de unos cuantos genes, sino de la interacción de todos los componentes de un genoma, los genes que codifican proteínas y los segmentos heterocromáticos que contienen retrotransposones.
Desde hace ya tres décadas esta concepción había sido adelantada por McClintock en ocasión de la recepción del premio Nobel, “En el futuro indudablemente la atención estará centrada en el genoma, y con gran apreciación de su significado como un órgano muy sensible de la célula, que controla las actividades genómicas y corrige errores comunes, que siente los eventos inusuales y no esperados, y responde a ellos, frecuentemente reestructurando al genoma” (McClintock, 1984). En la actualidad, la conclusión a que se llegó en este párrafo tendría como fundamento la definición del concepto de un fenómeno epigenético “como un cambio en el fenotipo que es heredable pero no involucra mutación en el ADN” (Gottschling, 2007), lo que significaría que estos estudios de nudos cromosómicos son buenos ejemplos de la aplicación de los conceptos epigenéticos; aunque ya se tiene avanzado el desarrollo de la epigenética, los estudios futuros serán cruciales para entender y explicar los procesos evolutivos de forma más precisa. Así, será posible conocer mejor las relaciones epigenéticas entre las plantas dentro de poblaciones y entre poblaciones dentro de razas, y finalmente, la genética entre las diferentes razas. Este conocimiento será muy importante en las decisiones de programación de actividades por los fitomejoradores.
Conclusiones
Con datos de nudos cromosómicos de las razas Chalqueño, Cónico, y Cónico Norteño se ha logrado caracterizar cada una de las razas de forma tal que las tres razas pueden distinguirse claramente una de otra. Con base en esta caracterización y con ayuda de los complejos Tuxpeño, Pepitilla y Zapalote de estudios precedentes fue posible proponer el origen genético de cada una de las tres razas estudiadas.











 nova página do texto(beta)
nova página do texto(beta)


