INTRODUCCIÓN
El Centro Nacional para la Información Biotecnológica (NCBI; National Center for Biotechnology Information) es un repositorio que pertenece a la Biblioteca Nacional de Medicina de los Estados Unidos de América (NLM; National Library of Medicine). Éste fue creado en 1988 para desarrollar sistemas de información en materia de biología molecular (Jenuth, 2000; Sayers et al., 2020) y mantiene la base de datos de GenBank, que contiene secuencias de ácidos nucleicos y proteínas (Benson et al., 2012) que a su vez, recibe información mediante la colaboración del Banco de Datos de ADN de Japón (DDBJ; DNA Data Bank of Japan) (Ogasawara et al., 2020) y la base de datos de secuencias de nucleótidos del Laboratorio Europeo de Biología Molecular (EMBL-EBI; The European Bioinformatics Institute). Por otra parte, el NCBI proporciona distintos sistemas de recuperación de información y recursos computacionales para el análisis de muchos otros tipos de sistemas biológicos, que se complementa con contribuciones de la comunidad científica en general.
Las bases de datos biológicas-moleculares a menudo contienen relaciones entre registros basadas en inferencias computacionales de similitud; con menos frecuencia, registran explícitamente datos que derivan de manera experimental (e.g. enlaces entre secuencias consideradas homólogas en bases de datos de proteínas y nucleótidos, así como genes que interactúan en una ruta metabólica) (Benson et al., 2012; Geer et al., 2010). Debido a la gran cantidad disponible de estos repositorios, la integración de herramientas para estudios moleculares de uso común, por ejemplo el portal de reagrupación de recursos denominado “Entrez” perteneciente al NCBI, se realiza caso por caso (Maglott et al., 2005; Sayers et al., 2009).
A través de lo anterior se lleva a cabo un intercambio de información para todo el conjunto de repositorios, integrando sus datos en las bibliotecas existentes, como es el caso de PubMed, cuya base de datos comprende más de 33 millones de citas de literatura biomédica de Medline, revistas de ciencias biológicas y libros en línea, además de que las citas suelen incluir enlaces a contenido de texto completo y sitios web de editores. Asimismo, PubChem (otra base de datos digna de mencionar) es considerada la colección más grande del mundo de información en materia de química de libre acceso. De esta manera, centralizar y vincular las bases de datos de biosistemas aumenta potencialmente su utilidad.
Tomando en cuenta lo anterior, es importante mencionar que el estudio de los recursos fitogenéticos no se excluye de la mayoría de las disciplinas bioinformáticas, pues las herramientas disponibles en el NCBI son capaces de llevar a cabo estudios in silico, como por ejemplo la identificación de regiones genómicas conservadas (marcadores moleculares) para la clasificación de especímenes vegetales (Jorrin-Novo, 2020; Morgante y Salamini, 2003; Rensink y Buell, 2005; Ricaño-Rodríguez et al., 2019); o bien, evaluar la calidad de la secuenciación de genomas en especies de plantas utilizando etiquetas de secuencia expresada (Shangguan et al., 2013).
El Cuadro 1 muestra la estadística de registros en el NCBI hasta septiembre de 2022, relacionados con el campo de la fitogenética. La Figura 1 resume las principales herramientas disponibles en el repositorio para el estudio genético-molecular de las especies (incluyendo el reino vegetal) (NCBI Resource Coordinators, 2015; 2016; 2018). Cabe destacar que el acceso a los recursos e información del NCBI es en línea y disponible a la comunidad científica.
Cuadro 1 Estadística de registros (Entrez) en el NCBI relacionados con recursos fitogenéticos. Actualizada a septiembre 2022.
| Base de datos | Registros | Descripción |
|---|---|---|
| Literatura | ||
| Catálogo NLM | 14,933 | Índice de colecciones NLM |
| Libros | 9,833 | Libros e informes |
| PubMed Central | 875,509 | Artículos de revistas de texto completo |
| PubMed | 1,153,559 | Resúmenes/citas científicos y médicos |
| Genes | ||
| Perfiles de expresión génica | 3,545,349 | Expresión génica y perfiles moleculares |
| Genes | 7,294,554 | Recopilación sobre loci genéticos |
| Colecciones de secuencias de ADN | 72,865 | Conjuntos de secuencias de estudios filogenéticos y de poblaciones |
| Grupos de datos de expresión génica | 225,929 | Estudios sobre genómica funcional |
| Proteínas | ||
| Dominios conservados | 2,806 | Dominios proteicos conservados |
| Grupos de proteínas idénticas | 18,526,704 | Secuencias de proteínas agrupadas por identidad |
| Estructuras | 7,898 | Estructuras biomoleculares in silico |
| Proteínas | 30,107,259 | Secuencias proteicas |
| Genomas | ||
| Biocolecciones | 16 | Colecciones físicas de biodepositorios |
| Genomas | 1,192 | Proyectos de secuenciación de genomas de organismos |
| Ensamblajes | 2,468 | Información sobre ensamblajes genómicos |
| Bioproyectos | 102,976 | Proyectos biológicos que proporcionan datos a NCBI |
| Biomuestras | 1,387,820 | Descripciones de materiales de origen biológico |
| Nucleótidos | 93,102,533 | Secuencias de ADN y ARN |
| Archivos de lectura de secuencia (SRA) | 1,570,076 | Archivo de lectura de secuencias de ADN y ARN de alto rendimiento |

Figura 1 Principales herramientas disponibles en el NCBI para el estudio genético-molecular de las especies, incluyendo el Reino Plantae. El repositorio BioSystems se retiró del NCBI en marzo de 2022. La herramienta de Protein incluye visores gráficos. (NCBI Resource Coordinators, 2015; 2016; 2018).
A la luz de las consideraciones anteriores, este trabajo tiene como objetivo recopilar algunas de las características generales más destacables del NCBI, tomando como base organizacional del documento elementos como el sistema Entrez, fuentes de información y actualización de literatura incluyendo la base de datos de taxonomía, gestión de metadatos y expresión genética, gestión de colecciones de secuencias nucleotídicas, procesamiento de secuencias genómicas y proteicas. Asimismo, se presentan algunos ejemplos de investigaciones en materia de fitogenética que emplearían herramientas computacionales pertenecientes al repositorio, las cuales incluyen anotaciones genómicas derivadas de secuenciaciones parciales y masivas, generación y organización de metadatos, estudios de expresión genética y transcriptómica, búsquedas de alineaciones nucleotídicas locales y globales para la identificación molecular de las especies, así como estructuración y modelación en 3D de proteínas, por mencionar algunos ejemplos. De la misma manera, se citan tutoriales enfocados a la ejecución de estos algoritmos, los cuales se encuentran disponibles de manera detallada en la página de inicio correspondiente del NCBI, al igual que su respectivo manual del usuario.
El sistema Entrez; reagrupación de bases de datos y accesos conjuntos
Entrez es un sistema integrado de reagrupación de bases de datos que brinda acceso a un conjunto diverso de 35 repositorios, los cuales contienen más de 3,000 millones de registros (Sayers et al., 2020; Trumbly, 2022). Los enlaces al portal web generalizado se proporcionan en la página de búsqueda global de Entrez. Por mencionar algunos ejemplos representativos de especies vegetales, tan sólo para el caso de Solanum lycopersicum y Arabidopsis thaliana se encuentra información registrada en 25 y 28 repositorios, respectivamente. Actualmente existe un manual de ayuda que permite conocer las utilidades de programación del sistema en mención.
Hasta septiembre de 2022, este motor de búsqueda reagrupó más de 1,153,725 documentos académicos relacionados con plantas a través de PubMed. En el apartado de genómica (Genomes) existen por lo menos 2,845 proyectos de ensamblajes (Assembly) de genomas de distintas especies de plantas y microorganismos relacionados metabólicamente, e.g. Oryza officinalis (GCA_008326285.1), Secale cereale (GCA_902687465.1) y Prunus yedoensis (GCA_005406145.1).
Asimismo, en PubMed se encuentran diversas publicaciones referentes a secuenciaciones de genomas completos y parciales (incluyendo shotguns, fragmentación y reagrupación de ADN), al igual que la generación de líneas transgénicas de especies vegetales resistentes al estrés abiótico, que en conjunto son de interés biotecnológico, agrícola y económico-social (e.g. A. thaliana, Vitis vinifera, Sorghum bicolor, Zea mays, Glycine max, Theobroma cacao, Phoenix dactylifera, Solanum tuberosum, Brassica rapa, Cannabis sativa, Prunus persica, Citrus aurantifolia y Allium sativum) (Michael y Jackson, 2013; Ricroch et al. 2022).
En este sentido, existe un repositorio independiente al NCBI denominado PLAZA que funge como punto de acceso para la genómica comparativa de plantas, ya que éste centraliza los datos genómicos producidos por diferentes iniciativas de secuenciación (Van Bel et al., 2022). En la Figura 2 se muestra una línea de tiempo que reagrupa algunas de las principales especies vegetales cuyos genomas han sido secuenciados, con base en el número de lecturas (contig), hasta septiembre de 2022. Cabe destacar que se resaltan los genomas de los cultivos alimenticios, frutales o agroindustriales más importantes; asimismo, el incremento representativo de especies estudiadas se debe en gran medida a los recursos bioinformáticos disponibles en la actualidad, como la secuenciación de nueva generación, los genotipados por secuenciación, así como el desarrollo de herramientas y una mayor disponibilidad de bases de datos a través del NCBI.
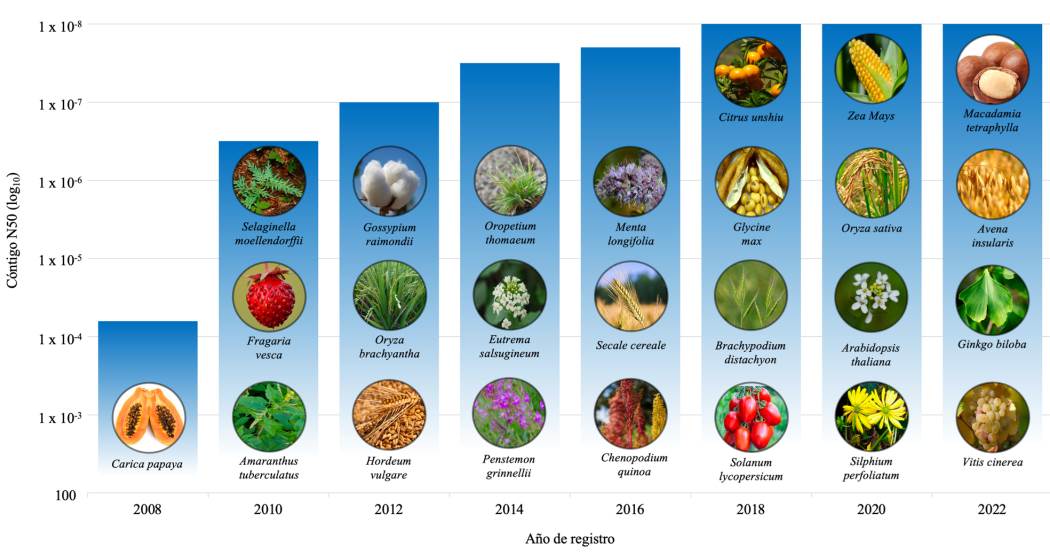
Figura 2 Línea de tiempo representativa (hasta septiembre de 2022) de las principales especies de origen vegetal cuyos genomas han sido secuenciados y ensamblados, y que fueron depositados en el NCBI. La gráfica muestra el año de registro (a partir de 2008), así como el número en aumento de lecturas (contig) N50 (log10) en cada secuenciación.
En materia de fitogenética, también es posible encontrar más de 103,000 bioproyectos (BioProject, recopilación de datos biológicos relacionados con una sola iniciativa provenientes de una organización o consorcio, lo que proporciona a los usuarios un lugar único para encontrar enlaces a los diversos tipos de datos generados); igualmente, se han registrado cientos de miles de biomuestras (BioSample, base de datos que contiene descripciones de materiales de origen biológico utilizados en ensayos experimentales), incluyendo archivos de lectura de secuencia (SRA: Sequence Read Archive, repositorio de datos de secuenciación de alto rendimiento, cuyo acervo acepta información de todas las ramas de la vida, así como estudios metagenómicos y ambientales) (Cuadro 1).
También, hasta septiembre de 2022, se encuentran depositadas en el NCBI más de 93 millones de secuencias de nucleótidos representadas en su mayoría por moléculas de ADN, ARNm y miRNA, así como algunas biocolecciones [Biocollections, conjunto de metadatos para colecciones de cultivos, museos, herbarios y otras colecciones de historia natural, incluidos los códigos de colección e institución de Darwin Core (Wieczorek et al., 2012), al igual que fórmulas de URL para mapear identificaciones de especímenes en páginas web dentro del sitio respectivo].
En el Cuadro 2 se muestran algunas de las biocolecciones globales más importantes de plantas y microorganismos directamente relacionados, que han sido depositadas en el NCBI. De la misma manera, existen millones de perfiles de expresión génica (Gene Expression Omnibus), grupos de datos de expresión génica (PopSets, colección de secuencias de ADN relacionadas con estudios poblacionales, filogenéticos, mutacionales y de ecosistemas), al igual que secuencias de proteínas caracterizadas en su mayoría hipotéticamente (NCBI Resource Coordinators, 2015; 2016; 2018; Sayers et al., 2020) (Cuadro 1).
Cuadro 2 Estadística de registros de biocolecciones en el NCBI relacionadas con recursos fitogenéticos (septiembre 2022).
| Institución | Tipo de colección | Calificador | ID |
|---|---|---|---|
| Colección de Plantas con Semillas Parásitas del Oeste de Virginia | Herbario | Comprobante de muestra | 5612 |
| Instituto de Plantas Medicinales ACECR de Irán | Herbario | Comprobante de muestra | 8725 |
| Centro de Investigación de Plantas Medicinales de la India | Herbario | Comprobante de muestra | 8053 |
| Colección Internacional de Microorganismos de Plantas de Nueva Zelanda | Cepario | Cepario | 3871 |
| Instituto Central de Plantas Medicinales y Aromáticas de la India | Herbario | Comprobante de muestra | 4439 |
| Instituto de Investigaciones de Plantas Medicinales, Departamento de Ciencias Médicas de Tailandia | Herbario | Comprobante de muestra | 7374 |
| Instituto de Investigación de Plantas Raras del río Yangtze, provincia de Hubei, China | Herbario | Comprobante de muestra | 9122 |
| Herbario de Plantas Vasculares Kathryn Kalmbach, Jardín Botánico de Denver | Herbario | Comprobante de muestra | 4578 |
| Instituto de Investigación de Plantas Medicinales y Aromáticas de toda Rusia | Herbario | Comprobante de muestra | 4219 |
| Laboratorio de Yunnan para la Conservación de Plantas Forestales Raras, Amenazadas y Endémicas | Herbario | Comprobante de muestra | 7323 |
| Universidad Estatal Mecynikov de Odessa, Departamento de Morfología y Sistemática de Plantas | Herbario | Comprobante de muestra | 4925 |
| Estación de Investigación de Plantas Medicinales del Instituto de Agroecología y Economía de los Recursos Naturales de Ucrania | Herbario | Comprobante de muestra | 8983 |
| Instituto de Bioquímica y Fisiología de Plantas y Microorganismos de la Academia Rusa de Ciencias | Cepario | Cepario | 7877 |
| Instituto Botánico de la Academia de Ciencias de Tayikistán, Departamento de Flora y Sistemática de Plantas Superiores | Herbario | Comprobante de muestra | 4343 |
| Colección de Plantas del Museo de Historia Natural de Londres | Herbario | Comprobante de muestra | 107,858 |
| Colección de Plantas Vasculares del Museo de Historia Natural de París | Herbario | Comprobante de muestra | 107,341 |
Fuentes de datos y actualización de literatura del NCBI
Las bases de datos y recursos del NCBI están organizados en siete áreas conceptuales: 1) literatura, 2) genomas, 3) variación, 4) salud, genes y expresión génica, 5) nucleótidos y proteínas, 6) moléculas pequeñas y 7) ensayos biológicos, las cuales dan origen a los 35 repositorios respectivos. Cada área conceptual comienza con un capítulo de descripción general que proporciona un marco contextual para los recursos discutidos bajo ese concepto. La descripción general va seguida de capítulos separados que cubren bases de datos o recursos individuales. Para más información respecto al apartado en mención es posible consultar una guía de literatura respectiva del NCBI (NCBI Resource Coordinators, 2015; 2016; 2018).
El repositorio genera esquemas de colaboración que involucra a investigadores de todo el mundo, además de consorcios científicos particulares y gubernamentales, incluyendo a miles de entidades académicas que pertenecen principalmente a universidades públicas; a manera de ejemplo, el NCBI colabora con el repositorio ENA (European Nucleotide Archive del EMBL) (Amid et al., 2020), el cual es una plataforma abierta y compatible para la gestión, intercambio e integración de archivos, así como difusión de datos de secuencias de nucleótidos.
Algunos ejemplos de otros repositorios externos colaboradores que también recopilan información sobre biomoléculas de origen vegetal son los siguientes: UniProt (recurso de libre acceso de secuencias de proteínas e información funcional), RNAcentral (colección de secuencias de ncRNA), EBIMgnify (base de datos metagenómicos e interacciones con microbiomas) y ArrayExpress (almacenamiento de datos de experimentos de genómica funcional de alto rendimiento).
Todos los recursos anteriores se basan en los servicios y contenido de ENA; igualmente, el NCBI colabora con el Banco de Datos de ADN de Japón y la Colaboración Internacional de Bases de Datos de Secuencias de Nucleótidos, los cuales cubren un gran espectro de lecturas sin procesar a través de alineaciones y ensamblajes, al igual que anotaciones funcionales. Estos repositorios son enriquecidos con información contextual vinculada a muestras biológicas y diseños experimentales mediante la International Nucleotide Sequence Database Collection (INSDC) (Arita et al., 2021).
De manera complementaria, existen otros recursos informáticos importantes que gestionan colecciones de literatura dentro de las bases de datos del NCBI; por ejemplo, Bookshelf (acceso gratuito en línea a libros y documentos sobre ciencias de la vida y atención médica), MeSH (Medical Subject Headings, tesauro de vocabulario controlado por NLM utilizado para indexar artículos para PubMed) y PCM (PubMed Central, archivo de texto gratuito de literatura de revistas en materia de biomedicina y ciencias de la vida perteneciente a la Biblioteca Nacional de Medicina y el Instituto Nacional de la Salud) (Sayers et al., 2020).
Base de datos taxonómica
La base de datos de taxonomía del NCBI, denominada Taxonomy, comprende un conjunto de clasificaciones y nomenclaturas curadas (organización e integración) de todos los organismos que forman parte de las bases de datos de secuencias públicas. Ésto representa actualmente alrededor del 10 % de las especies descritas en el planeta. El repositorio se encuentra estandarizado mediante la INSDC, que comprende GenBank, ENA (EMBL) y bases de datos del DDBJ. Cabe resaltar que Taxonomy es un centro de organización central para muchos de los recursos del NCBI que proporciona un medio para agrupar elementos dentro de otros dominios del sitio web (Federhen, 2012). La base de datos taxonómica sirve como un importante punto de entrada al sistema Entrez para aquellos usuarios que desean conocer la información disponible sobre un taxón en particular; desde el nivel de especie hasta género, familia, orden o superior, según los niveles de jerarquía referidos.
Muchos de los dominios de Entrez (secuencia, estructura, genes, genomas, literatura, etc.) están indexados por taxonomía en el campo de búsqueda [organismo], y estos índices apoyan vínculos recíprocos entre la taxonomía y otros dominios. Todo lo anterior se detalla en los manuales de ayuda NBK25501 y NBK54428 correspondientes (NCBI, 2010; NCBI Taxonomy Help, 2011). Las consultas de taxonomía Entrez se guardan en MyNCBI y el usuario puede registrarse para recibir actualizaciones periódicas por correo electrónico, toda vez que algo nuevo en Entrez satisfaga la consulta; por ejemplo, se puede registrar la consulta [propiedad específica] y solicitar la recepción de un correo electrónico de manera periódica, con la lista de especies que han aparecido en las bases de datos de secuencia por primera vez en la última semana.
Registro y gestión de metadatos complementarios y perfiles de expresión genética (transcriptómica)
Como se mencionó con anterioridad, el NCBI es líder en el campo de la bioinformática y se enfoca principalmente en estudios computacionales para dilucidar fenómenos biológicos (Jenuth, 2000; Sayers et al., 2020). Existen conjuntos de metadatos que invitan a los usuarios a complementar los registros de sus investigaciones; por ejemplo, las biocolecciones, bioproyectos y biomuestras, ésto a través de la reagrupación de datos de rutas metabólicas empleando el recurso PubChem Pathways. Por otra parte, el repositorio GEO DataSets reagrupa datos de expresión génica, así como registros originales de series y plataformas en el repositorio Gene Expression Omnibus (GEO).
Los registros de conjuntos de datos contienen recursos adicionales, incluidas herramientas para la generación de grupos (clusters) y consultas de expresiones diferenciales, e.g. GEO Profiles (repositorio que almacena perfiles de expresión génica individuales de datos seleccionados en GEO). Geo Profiles permite la búsqueda de perfiles específicos de interés en función de la anotación de genes o características de perfil calculadas previamente.
En la actualidad, existe un sinnúmero de antecedentes en relación con estudios genómicos en plantas, cuyas investigaciones involucraron en gran parte estudios de expresión génica que se valieron de los algoritmos anteriores; por ejemplo, Krzyszton y Kufel (2022) desarrollaron un perfilado de ARNnc (no codificante) mediante secuenciaciones de alto rendimiento en A. thaliana; el resultado principal demostró que la alteración de una enzima glucolítica enolasa llamada LOS2 (baja expresión del gen 2 de responsabilidad osmótica) provoca respuestas de defensa constitutiva o autoinmunidad en la especie sujeta a estudio. Los datos anteriores se encuentran registrados en el repositorio de bioproyectos (PRJNA715469) correspondiente.
Otro ejemplo es el trabajo realizado por Garighan et al. (2021), quienes identificaron ARN pequeños expresados diferencialmente en embriones de manzana; a través de ello, revelaron el papel potencial de un miRNA denominado Mir159-MYB durante el periodo de latencia. El proyecto se encuentra igualmente registrado (PRJNA784097) en las colecciones del NCBI.
Recientemente, Bouissil et al. (2022) hicieron uso de este tipo de repositorios para organizar perfiles de expresión genética derivados de factores de inducción de proteínas de defensa y resistencia de la palmera datilera a Fusarium oxysporum f. sp. albedinis, en respuesta a alginato extraído de Bifurcaria bifurcata. En este proyecto se incluyeron también las anotaciones funcionales de los péptidos respectivos.
Bases de datos genómicos y gestores gráficos
Hoy en día se desarrollan mejoras constantes para la búsqueda de secuencias genómicas mediante la introducción de conjuntos de datos (Datasets), un recurso que permite a los usuarios recopilar fácilmente contenido de todas las bases de datos del NCBI. Los Datasets organizan la información a través de interfaces web distintas, así como de líneas de comandos para descargarlos posteriormente en paquetes de archivos estructurados. Así, los conjuntos de datos admiten consultas de genomas y genes en una amplia gama de grupos taxonómicos. En este sentido, los usuarios que requieren utilizar datos genómicos pueden ensamblar secuencias de genomas, transcriptomas y proteínas, así como anotaciones funcionales contenidas en paquetes computacionales. Estos paquetes incluyen también informes de metadatos enriquecidos, ya que la interfaz web de Datasets permite estudiar genomas a través de reconstrucciones filogenéticas y, elegir cualquier conjunto de secuencias eucariotas y procariotas depositadas en la base de datos de NCBI Assembly. La interfaz de programación de estas aplicaciones brinda acceso también a genomas virales y respalda las búsquedas con identificadores taxonómicos o accesiones de ensamblaje (NCBI Resource Coordinators, 2015; 2016; 2018).
Para el caso de las embriofitas, cuyos especímenes se clasifican como plantas verdes antecesoras de todos los especímenes terrestres (phylum Streptophita), se han agrupado, hasta septiembre de 2022, un poco más de 7,300,000 genes relacionados con cientos de procesos metabólicos distintos, desde mecanismos fitopatológicos de defensa hasta identificación de haplotipos y polimorfismos de nucleótidos únicos (SNPs) (Jones y Dangl, 2006; Ricaño-Rodríguez et al., 2018; 2019).
En el último año se ha mejorado la capacidad de descarga de datos dentro del visor gráfico de las bases de datos de nucleótidos y proteínas del NCBI, a través de NCBI Sequence Viewer. Como se mencionó con anterioridad, es posible descargar secuencias de genes y datos de anotación funcional, inclusive de SNPs (https:// www.ncbi.nlm.nih.gov/snp/). Los usuarios también pueden copiar cadenas cortas de datos de secuencias directamente al portapapeles y descargar segmentos más grandes en formato de archivo FASTA (formato de texto que representa secuencias de nucleótidos o aminoácidos mediante códigos de una sola letra).
Con respecto al estudio estructural genómico,‒incluida la traducción a proteínas, estas herramientas también proporcionan datos referentes a clasificaciones de dominios conservados (CDD), así como número de exones e intrones en secuencias nucleotídicas. En el caso de alineaciones tipo SRA o BLAST, los algoritmos identifican cualquier dato no alineado, incluidas inserciones y colas 5′ y 3′. Las herramientas en mención son útiles en estudios de genotipeo por secuenciación; por ejemplo, a través del estudio de regiones sumamente conservadas de especímenes vegetales (e.g. Theobroma cacao L. y Aegilops umbellulata) se determinan variaciones genotípicas a nivel polimórfico, así como el mapeo de genes de resistencia a fitopatógenos, como la roya (Edae et al., 2016; Lima et al., 2009; Ricaño-Rodríguez et al., 2019); ésto permite a los investigadores dilucidar más a fondo las características genéticas distintivas de grupos de individuos que pertenecen a un mismo phylum.
Visor de datos genómicos
El navegador de datos genómicos insignia del NCBI, denominado Genome Data Viewer (GDV), es una interfaz que integra un motor gráfico con un sólido algoritmo de búsqueda/recuperación/análisis de datos. Para respaldar mejor los análisis y solventar las necesidades de investigación de cada usuario en particular, se han lanzado numerosas mejoras de los aspectos técnicos de dicha herramienta (NCBI Resource Coordinators, 2018).
El genoma de Zea mays (B73) es uno de los conjuntos de datos más completos registrados en GDV. A través del visor gráfico se observan reconstrucciones filogenéticas diversas de esta especie. Mediante los hipervínculos disponibles relacionados con el proyecto anterior (RefSeq accession GCF_902167141.1) se puede estudiar la mayoría de sus genes y transcritos correspondientes (incluyendo anotaciones), así como consultar a detalle su exoma y loci respectivos. Estos estudios se complementan con la herramienta RefSeq (conjunto completo, integrado, no redundante y bien anotado de secuencias de referencia que incluyen genómica, transcriptómica y proteómica). Las mejoras adicionales del visor gráfico también permiten la navegación agrupando por ensamblajes y regiones cromosómicas.
Gestión y procesamiento de secuencias genómicas
En el NCBI existe un recurso informático denominado “Gene” que incluye datos sobre perfiles de expresión genética de diversas especies, éstos van desde representaciones gráficas de la expresión de cada gen integrada en su propia página de informe completo, hasta la descarga de conjuntos de secuencias nucleotídicas para su procesamiento. Gene integra información de una amplia gama de especies y un solo registro incluye nomenclatura, secuencias de referencia, mapas, vías metabólicas, variaciones y fenotipos, así como enlaces a recursos específicos del genoma y loci de especímenes de todos los reinos (Brown et al., 2015).
Los perfiles de expresión son complementos útiles de funciones genéticas ya caracterizadas, y también medios potenciales para estudiar la función de genes descubiertos de manera reciente (Ozsolak y Milos, 2011). Constantemente se realizan actualizaciones para generar resúmenes de texto que acompañen al perfil de expresión de cada gen en cuestión, y que estos datos sean indexados dentro del sistema de consulta de Entrez. Los perfiles de expresión se calculan a partir de alineaciones de secuencias de ARN generadas por la canalización de cada genoma anotado dentro del NCBI. Dicho proceso selecciona conjuntos de datos disponibles públicamente en formatos SRA (Fagerberg et al., 2014).
Después de alinear las lecturas de una muestra con la secuencia genómica, para cada gen se calcula la cobertura de lectura (en comparación con todos los exones anotados), y se normaliza a todas las lecturas alineadas con el genoma de referencia, empleándolas para derivar lecturas por kb por cada millón de éstas situadas a través de la secuencia diana. Los datos de las réplicas biológicas dentro del mismo proyecto de SRA se promedian y se presentan con su respectiva desviación estándar. Es importante mencionar que los niveles de expresión de diferentes proyectos de SRA se informan de manera independiente.
Colecciones de secuencias nucleotídicas y reconstrucciones de grupos homólogos
Existen dos herramientas básicas en el NCBI denominadas HomoloGene y Nucleotide que emplean un sistema automatizado para construir grupos de homología putativa, a partir de conjuntos de genes completos de una amplia gama de especies eucariotas; por ejemplo, a través de los recursos anteriores Hosmani et al. (2019) estudiaron las características del genoma nuclear del tomate (S. lycopersicum) mediante la secuenciación de fragmentos digeridos con EcoRI submetilados. Por otra parte, el “proyecto genoma de la naranja dulce de China” (The draft genome of sweet orange) (Citrus sinensis) (Xu et al., 2013) consistió en secuenciar los cromosomas de esta especie (bioproyecto PRJNA86123) y estudiarlos posteriormente a través de HomoloGene y Nucleotide.
En la actualidad se pretende que los usuarios tengan acceso a una colección de secuencias genómicas provenientes de varias fuentes, incluidas GenBank, RefSeq, TPA, Third Party Anotation (base de datos diseñada para capturar resultados experimentales o inferenciales por parte de terceros que derivaron de los datos primarios de GenBank) y PDB, Protein Data Bank (Berman et al., 2000).
Herramienta básica de búsqueda de alineación local (BLAST)
La herramienta básica de búsqueda de alineación local (BLAST, Basic Local Alignment Search Tool) es un algoritmo informático de comparación y alineación de secuencias nucleotídicas (i.e. ADN, ARN y aminoácidos) que se utiliza a través de una interfaz web, o bien, como una herramienta independiente para comparar y analizar la consulta de otro usuario con una base de datos de secuencias previas (Altschul et al., 1990; 1997).
BLAST funciona bajo un enfoque heurístico que identifica coincidencias entre dos secuencias, e intenta iniciar alineaciones desde estos puntos de partida. Además de realizar alineaciones, BLAST proporciona información estadística sobre cada trabajo llevado a cabo; por ejemplo, sugiere un valor “esperado” o tasa de falsos positivos, y de manera general, este recurso es empleado para la búsqueda de probables genes homólogos. Dicha herramienta usa el algoritmo “Smith-Waterman” (programación dinámica optimizada con matrices de substitución) (Smith y Waterman, 1981) para realizar las alineaciones correspondientes. BLAST es una herramienta utilizada en múltiples campos de las ciencias naturales, incluyendo el estudio de la fitogenética.
En la página de inicio de BLAST se enumeran las diferentes búsquedas por naturaleza realizadas, i.e. nucleótidos, transcritos y genomas. Cuando se envía una consulta al servidor del NCBI, ya sea como una secuencia en formato FASTA o como un identificador de secuencia, e.g. número de acceso de GenBank, la búsqueda se envía al servidor BLAST y se devuelve un identificador de solicitud denominado RID. La consulta y los resultados correspondientes se almacenan en un formato estructurado hasta 24 h después de que se emite un RID. Este identificador reagrupa la búsqueda y permite ver los resultados en varios formatos, que incluyen el informe BLAST, una tabla de resultados simplificada, archivos XML (Extensible Markup Languaje, Lenguaje de Mercado Extensible) y ASN.1 (Abstract Syntax Notation one, Notación Sintáctica Abstracta uno) (Madden, 2002).
Las nuevas bases de datos BLAST están disponibles en un sitio FTP (File Transfer Protocol, Protocolo de Transferencia de Archivos) del NCBI, así como en los proveedores de nube de GCP (Google Cloud Plataform, Plataforma de Nube de Google) y AWS (Amazon Web Services, Servicios de Nube de Amazon). En este sentido, los tres sitios ofrecen las mismas 23 bases de datos y éstas poseen desde una colección de betacoronavirus, hasta genomas representativos de Ref-Seq pertenecientes a organismos eucariotas, procariotas y otras familias de virus.
De manera paralela se han desarrollado herramientas complementarias como Primer-BLAST (Ye et al., 2012), la cual diseña cebadores comunes para grupos de secuencias de ADN muy similares; ésto permite que los investigadores realicen tareas específicas, como amplificar múltiples variantes de transcripción para un solo gen, o detectar un grupo de cepas bacterianas filogenéticamente relacionadas (McGinnis y Madden, 2004); asimismo, el apartado de análisis de secuencias del NCBI cuenta con un conjunto de recursos sumamente importantes que complementan las tareas anteriores, i.e. BLAST (Stand alone), FTP: BLAST Databases, BLAST: microbial Genomes, BLAST RefSeqGene, COBALT, Genome Remaping Services, Genome Workbench, Multiple Sequence Alignment Viewer, Open Reading Frame Finder (ORF Finder), ProSplign y Tree viewer (NCBI Resource Coordinators, 2015; 2016; 2018).
Bases de datos de proteínas y herramientas para estudios proteómicos
En 2014 el NCBI introdujo el “Informe de Proteínas Idénticas” en su base de datos para corroborar las relaciones entre las secuencias de aminoácidos existentes (número de acceso de proteínas no redundantes), así como el conjunto de secuencias de dominios conservados de nucleótidos individuales (NCBI Resource Coordinators, 2015). En la actualidad, estos informes se han mejorado y recopilado en un nuevo recurso denominado Identical Protein Groups; Grupos de Proteínas Idénticas. Este recurso, abreviado IPG, incluye todas las secuencias de proteínas registradas en el NCBI y PDB, con enlaces a secuencias de transcripción de nucleótidos de GenBank y RefSeq.
Por otra parte, hace dos décadas se creó un consorcio denominado UniProt, el cual surge de la colaboración entre el Instituto Suizo de Bioinformática (SIB), el Instituto Europeo de Bioinformática (EBI) y el Recurso de Información sobre Proteínas (PIR). Swiss-Prot, junto con TrEMBL (traducción automática de EMBL), se unieron con PIR para generar la “UniProt Knowledgebase” (UniProtKB), el catálogo de proteínas más importante del mundo. En materia de proteómica vegetal existen antecedentes de proyectos que emplearon las bases de datos anteriores, incluyendo desde estudios de fitopatología hasta aspectos nutrimentales concernientes a los seres humanos (Shewry y Lucas, 1997; Yagami, 2002).
Recientemente, el NCBI lanzó una versión actualizada (2.19.0) de un software denominado iCn3D (Wang et al., 2020), una interfaz gráfica de estructuración molecular tridimensional (3D) que se ejecuta directamente en los navegadores web. Las vistas interactivas de iCn3D están integradas en las páginas de resumen de la estructura de la base de datos de modelado molecular (Molecular Modeling DataBase) del NCBI, e iCn3D visualiza los resultados de las comparaciones de estructuras 3D calculadas por un algoritmo llamado VAST+ (Vector Alignment Search Tool), que consiste en un software que se enfoca principalmente en la búsqueda de estructuras macromoleculares que tengan una unidad biológica similar, en lugar de aquellas que son similares a nivel de una molécula proteica individual o con dominios tridimensionales, así como las alineaciones de secuencias con estructuras por pares calculadas mediante BLAST. iCn3D muestra simultáneamente estructuras 3D, esquemas de interacción 2D, alineaciones y secuencias de proteínas/nucleótidos, así como anotaciones de sitios funcionales y huellas de dominios conservados.
Por otra parte, la base de datos Structure alberga más de 7,890 estructuras tridimensionales de macromoléculas de plantas, su conformación computacional proporciona una gran cantidad de información sobre la función biológica e historia evolutiva de las especies, y se pueden usar para examinar relaciones secuencia-estructura-función, así como interacciones y sitios activos moleculares. Entre las estructuras más interesantes en materia de proteómica vegetal se pueden mencionar ejemplos como proteínas relacionadas con fotosistemas (Amunts et al., 2010), supercomplejos de cloroplastos PSI-NDH (Shen et al., 2022) y proteínas ribosomales L30e (Halic et al., 2005). Asimismo, es posible observar estructuras cristalográficas de ribosomas mitocondriales, hidrolasas, glucanasas, fosfatasas, fitocromos y aldehído deshidrogenasas, entre otras (NCBI Resource Coordinators, 2015; 2016; 2018).
En la Figura 3 se muestra un ejemplo de una estructura molecular compuesta por dos elementos de una proteína Argonauta tipo 1 de A. thaliana (Argonaute protein, Gen AGO1), la cual es visualizada con la mayoría de sus elementos constitutivos a través de la interfaz del algoritmo Structure. Las proteínas argonautas desempeñan un papel importante en la regulación génica en el núcleo celular; debido a ello, estas biomoléculas son capaces de interferir en procesos epigenéticos durante la transcripción (Arribas-Hernández et al., 2016).

Figura 3 Estructura cristalográfica de la proteína argonauta Ago1 dominio medio (MID) de Arabidopsis thaliana observada a través de la interfaz del algoritmo Structure del NCBI. La estructura izquierda se encuentra representada por 147 residuos de aminoácidos, iniciando con la serina. Se muestran siete sitios de interacción (SO4, agua y sitios de anclaje a hebras de ARN 5´ guías). La estructura derecha representa el dominio tipo PIWI de la proteína (dominio encontrado en proteínas involucradas en el silenciamiento de ARN). Este fenómeno se refiere a un grupo de mecanismos de silenciamiento de genes relacionados mediados por moléculas cortas de ARN, que incluyen siARN, miARN y ARN guía relacionados con heterocromatina (Madej et al., 2014; NCBI Resource Coordinators, 2015; 2016; 2018).
Otras características recientemente agregadas a esta base de datos incluyen la visualización extendida de redes de interacción 2D entre proteínas y ligandos, u otras proteínas; también se lleva a cabo la visualización de potenciales electrostáticos calculados por el método Delphi (Li et al., 2012) y la visualización de la ubicación de bicapas de membrana en relación con estructuras de proteínas transmembranales (Lomize et al., 2012). iCn3D está disponible en https://github.com/ncbi/icn3d y sus características más novedosas actualizadas se pueden consultar en la página de la galería correspondiente. La herramienta anterior suele utilizarse de manera sinérgica con la base de datos de dominios conservados, la cual es un recurso imprescindible para la anotación de unidades funcionales en proteínas, su colección de modelos de dominios incluye un conjunto seleccionado por el NCBI, que utiliza estructuras 3D para proporcionar información sobre las relaciones de secuencia/estructura/función (Geer et al., 2002).
CONCLUSIONES
Uno de los objetivos principales del NCBI es el desarrollo constante de nuevas tecnologías de la información, lo cual conlleva a un mejor entendimiento del origen y consecuentes procesos genético-moleculares de las especies, incluyendo las plantas; asimismo, permite la descripción de nuevas hipótesis como un reto a resolver para las generaciones presentes y futuras. Dentro del grupo de las plantas (Reino: Plantae), diversas bases de datos y herramientas del repositorio en mención que en su mayoría han sido previamente descritas permiten una comprensión más profunda de la naturaleza de miles de especímenes de interés agro-biotecnológico, medicinal, industrial y hasta biocultural para la sociedad, así como para la comunidad científica, lo que incrementa su potencial de aprovechamiento. Teniendo en cuenta tales consideraciones, es importante destacar la necesidad de una mayor incursión en el tema en el cual se utilicen las herramientas bioinformáticas disponibles para el desarrollo de las ciencias ómicas de manera prioritaria, ya que este universo de conocimiento se actualiza de manera constante debido a la diaria generación de datos y recursos bioinformáticos, principalmente aplicados al campo de las ciencias naturales.











 nueva página del texto (beta)
nueva página del texto (beta)


