INTRODUCCIÓN
El incremento del rendimiento de grano en trigo (Triticum aestivum) es uno de los objetivos más desafiantes en los programas de mejoramiento genético, debido a la compleja arquitectura genética, a la baja heredabilidad y por la influencia de interacciones ambientales en todas las etapas de crecimiento de la planta (Li et al., 2019; Simmonds et al., 2014). Dada la complejidad del rendimiento, éste se puede disectar en componentes, los cuales son el número de espigas por unidad de superficie, número de granos por espiga y peso de mil granos. Estos caracteres tienen mayor heredabilidad que el carácter rendimiento y son relativamente fáciles de seleccionar en los programas de mejoramiento (Li et al., 2019).
La detección de un locus de carácter cuantitativo (QTL) con efecto en los componentes del rendimiento amplía las oportunidades para desarrollar marcadores diagnóstico que puedan utilizarse en la selección asistida por marcadores moleculares y en la piramidación de genes, permitiendo aumentar las ganancias genéticas para rendimiento (Sehgal et al., 2019).
Sehgal et al. (2019) reportaron el QTL HAP-6A-13 en el cromosoma 6A, el cual estaba asociado de manera significativa con el peso de mil granos (R 2 del marcador = 0.007) en trigo harinero, el QTL se detectó mediante un estudio de asociación del genoma completo (GWAS - genome-wide association study) basado en haplotipos. En ese estudio se utilizaron 4302 líneas de trigo que comprendieron cinco ensayos de rendimiento del programa de mejoramiento de trigo harinero del Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT) y 8443 marcadores moleculares del tipo genotipado por secuenciación (GBS-genotyping by sequencing). El haplotipo favorable del QTL HAP-6A-13 contribuyó con un incremento de hasta 2.6 g en muestras de mil granos.
Existen varias metodologías para validar el efecto de un QTL, una opción es la creación de líneas casi isogénicas (NIL), las cuales son un conjunto de líneas efectivas para estudiar los efectos fenotípicos atribuibles a un QTL, ya que el fondo genético, caracteres morfológicos y fenológicos que comúnmente influyen en las evaluaciones fenotípicas de los caracteres cuantitativos, son idénticos y sólo difieren en algunos loci específicos (Pumphrey et al., 2007).
Las familias endogámicas heterogéneas (HIF - heterogeneous inbred families) son un tipo especial de NIL, se obtienen al cruzar dos progenitores distintos y realizar autopolinizaciones durante varias generaciones; posteriormente, se detectan líneas segregantes heterocigóticas para la región de un QTL a verificar, estas líneas identificadas se autopolinizan una vez más para obtener una segregación mendeliana del QTL, y finalmente se compara el fenotipo de las líneas segregadas (Tuinstra et al., 1997).
Los objetivos de esta investigación fueron diseñar marcadores moleculares tipo KASP (kompetitive allele specific PCR) para el QTL HAP-6A-13, verificar la asociación de los marcadores con el peso de mil granos mediante GWAS en nuevas poblaciones de mejoramiento, validar el efecto del QTL en peso de mil granos empleando HIF y determinar si hay efecto en rendimiento, longitud, ancho y grosor del grano.
MATERIALES Y MÉTODOS
Diseño de marcadores KASP
Se diseñaron iniciadores de tipo KASP para los polimorfismos C/T y A/G (Cuadro 1) a partir de las secuencias GBS de los marcadores S6A_481437887 y S6A_481437894 que formaban el haplotipo del QTL HAP-6A-13 reportado por Sehgal et al. (2019). El diseño de los KASP se realizó utilizando el software PolyMarker (Ramirez-Gonzalez et al., 2015), mismo que se puede acceder de manera gratuita en: http://polymarker.tgac.ac.uk/.
Cuadro 1 Iniciadores KASP utilizados.
| SNP | Alelo favorable | Secuencia de iniciadores directos alelo-específicos | Alelo común | |
|---|---|---|---|---|
| C/T | T | ctgttctcagcccaacatgtc | ctgttctcagcccaacatgtt | ggttaggtgggctgcagg |
| A/G | G | tctcagcccaacatgtctacacaa | tctcagcccaacatgtctacacag | gaggggttaggtgggctgc |
La secuencia de los marcadores S6A_481437887 y S6A_481437894 es:
CGGGATGCTCTGCTTCTCTCTGTTCTCAGCCCAACATGT[C/T]TACACA[A/G]CCCACCTGCA
Un ensayo KASP tiene dos iniciadores directos alelo-específicos (un iniciador para cada SNP de cada alelo), un iniciador reverso común para ambos alelos y dos oligos, uno marcado con el fluoróforo FAM y otro con el HEX, cada oligo es complementario a la secuencia terminal de cada uno de los iniciadores de los alelos a evaluar.
Genotipado con los marcadores KASP
Se genotiparon tres colecciones de trigo harinero que forman parte de ensayos internacionales de rendimiento (EYT-elite yield trial) del CIMMYT y contienen líneas avanzadas de mejoramiento. Los ensayos EYT2013-14, EYT2014-15 y EYT2015-16 estuvieron formados por 983, 942 y 829 genotipos respectivamente. La extracción de ADN y la amplificación por PCR se realizó como lo describen Dreisigacker et al. (2016). Los ciclos de PCR se realizaron a una temperatura inicial de desnaturalización de 95 °C durante 15 min, seguido por 10 ciclos con rampa de temperatura (95 °C durante 20 s, 65 °C durante 25 s inicialmente y disminuyendo en 1 °C en cada ciclo posterior, seguido de 30 ciclos de alineamiento (95 °C durante 10 s y 57 °C durante 60 s). No fue necesario un paso de extensión porque los amplicones esperados tuvieron un tamaño de 50 pares de bases.
Las placas de PCR fueron leídas en PHERAstar Plus (BMG LABTECH, Ortenberg, Alemania) y las lecturas de genotipado se analizaron en el software KlusterCaller TM (LGC Genomics, Teddington, Reino Unido). El genotipado KASP de los EYT se comparó con los resultados previos del genotipado reportado por Sehgal et al. (2019).
Validación de marcadores KASP mediante GWAS
La asociación de los marcadores KASP diseñados se probó con el peso de mil granos en cuatro colecciones de trigo harinero, mediante GWAS. Se incluyeron los EYT descritos anteriormente y el panel WAMI (wheat association mapping initiative). WAMI es un conjunto diverso de trigos harineros de primavera que consta de 287 líneas avanzadas liberadas por el CIMMYT (Lopes et al., 2015).
Los paneles de validación EYT2013-14, EYT2014-15 y EYT2015-16 se genotiparon anteriormente usando el método de genotipado por secuenciación (GBS-genotyping by sequencing) como lo describen Poland et al. (2012) y Rutkoski et al. (2016), los datos fueron proporcionados por Sehgal et al. (2019). El panel WAMI fue genotipado con Illumina iSelect 90K SNP Assay, los datos fueron proporcionados por Sukumaran et al. (2015). A los resultados de secuenciación se les adicionó el genotipado KASP con los dos marcadores del Cuadro 1, el haplotipo fue formado al unir los resultados de los dos SNP como un solo marcador. Los marcadores con una frecuencia de alelos menores a 0.05 fueron eliminados. En el Cuadro 2 se muestra el total de individuos y número de marcadores finales utilizados en los GWAS.
Cuadro 2 Colecciones de trigo utilizadas en la validación de marcadores KASP.
| Colecciones de trigo | Número de individuos | Número de marcadores |
|---|---|---|
| EYT2013-14 | 983 | 13,770 |
| EYT2014-15 | 942 | 13,768 |
| EYT2015-16 | 829 | 13,768 |
| WAMI | 287 | 15,740 |
El fenotipado de EYT y WAMI se realizó en el Campo Experimental Norman E. Borlaug (CENEB), Ciudad Obregón, Sonora (noreste de México). El cultivo se realizó en condiciones de riego. Los EYT forman parte de los ensayos de rendimiento de trigo harinero de CIMMYT en sus respectivos años (2013, 2014 y 2015) y fueron evaluados en un diseño α-látice con tres repeticiones, la unidad experimental consistió de surcos de 2.8 m sembrados a doble hilera. Sehgal et al. (2019, 2020) proporcionan amplia información acerca de las evaluaciones fenotípicas de los EYT.
Los ensayos WAMI fueron sembrados en los años 2010, 2011 y 2013 en un diseño experimental α-látice con dos repeticiones, la unidad experimental consistió de surcos de 2 m sembrados a doble hilera. Lopes et al. (2015) proporcionan información adicional acerca de las evaluaciones fenotípicas de WAMI.
En EYT se evaluó el peso de mil granos (g) y en WAMI peso de mil granos y rendimiento (g m -2 ), siguiendo la metodología de Pask et al. (2013). De los valores fenotípicos evaluados en WAMI se obtuvieron las mejores estimaciones lineales insesgadas (BLUPs-best linear unbiased predictions), mientras que en EYT se obtuvieron las medias ajustadas como se describe en Sehgal et al. (2017).
El mapeo de asociación se realizó en el software TASSEL 5.0 (Bradbury et al., 2007) https://tassel.bitbucket.io/. El efecto de la estructura de la población se trató mediante análisis de componentes principales, se emplearon los primeros tres componentes principales, los cuales se determinaron mediante la librería factoextra de R (Kassambara y Mundt, 2017), los componentes principales fueron incluidos en el modelo lineal generalizado (GLM).
Validación del efecto del QTL en familias endogámicas heterogéneas
Se construyeron 10 familias endogamicas HIF (Cuadro 3) siguiendo el método de Tuinstra et al. (1997). En la primera siembra se utilizaron semillas de generación F8, de 10 cruzas de trigo harinero de primavera, desarrollado en el CIMMYT y se identificaron como generación HIF1:9. La siembra se realizó en macetas bajo condiciones de invernadero y todas las plantas fueron genotipadas con los marcadores KASP diseñados, para identificar plantas segregantes en el QTL HAP-6A-13. Se seleccionó una planta heterocigota de cada cruza y se autopolinizó. La siguiente generación (HIF2:10) fue sembrada en condiciones similares a la siembra anterior, y dentro de cada familia se seleccionaron las plantas homocigotas para el QTL HAP-6A-13. En las dos generaciones posteriores se realizaron las evaluaciones fenotípicas en campo, HIF3:11 fue evaluada en El Batán, Estado de México (centro de México) y HIF4:12 fue evaluada en Ciudad Obregón, Sonora (noreste de México), en ambos ambientes hubo un manejo similar del cultivo.
Cuadro 3 Pedigrí de las familias endogámicas heterogéneas evaluadas.
| HIF | Haplotipo | Pedigrí |
|---|---|---|
| HIF1 | CA (−) | BAJ #1/3/TRCH/SRTU//KACHU |
| TG (+) | BAJ #1/3/TRCH/SRTU//KACHU | |
| HIF2 | CA (−) | SAUAL/3/KAUZ/PASTOR//PBW343/4/TRCH/SRTU//KACHU/5/SAUAL//PI 610750/ATTILA/3/SAUAL |
| TG (+) | SAUAL/3/KAUZ/PASTOR//PBW343/4/TRCH/SRTU//KACHU/5/SAUAL//PI 610750/ATTILA/3/SAUAL | |
| HIF3 | CA (−) | CROC_1/AE.SQUARROSA (205)//BORL95/3/PRL/SARA//TSI/VEE#5/4/FRET2/6/MTRWA92.161/PRINIA/5/SERI*3//RL6010/4*YR/3/PASTOR/4/BAV92 |
| TG (+) | CROC_1/AE.SQUARROSA (205)//BORL95/3/PRL/SARA//TSI/VEE#5/4/FRET2/6/MTRWA92.161/PRINIA/5/SERI*3//RL6010/4*YR/3/PASTOR/4/BAV92 | |
| HIF4 | CA (−) | PRL/2*PASTOR//SUNSTATE/3/MUNAL #1/4/OTUS//PRL/2*PASTOR |
| TG (+) | PRL/2*PASTOR//SUNSTATE/3/MUNAL #1/4/OTUS//PRL/2*PASTOR | |
| HIF5 | CA (−) | HEILO//MILAN/MUNIA/8/2*NG8201/KAUZ/4/SHA7//PRL/VEE#6/3/FASAN/5/MILAN/KAUZ/6/ACHYUTA/7/PBW343*2/KUKUNA |
| TG (+) | HEILO//MILAN/MUNIA/8/2*NG8201/KAUZ/4/SHA7//PRL/VEE#6/3/FASAN/5/MILAN/KAUZ/6/ACHYUTA/7/PBW343*2/KUKUNA | |
| HIF6 | CA (−) | SOKOLL//W15.92/WBLL1/3/BAVIS |
| TG (+) | SOKOLL//W15.92/WBLL1/3/BAVIS | |
| HIF7 | CA (−) | SAUAL/3/KAUZ/PASTOR//PBW343/4/TRCH/SRTU//KACHU |
| TG (+) | SAUAL/3/KAUZ/PASTOR//PBW343/4/TRCH/SRTU//KACHU | |
| HIF8 | CA (−) | TRCH/SRTU//KACHU*2/3/KINGBIRD #1 |
| TG (+) | TRCH/SRTU//KACHU*2/3/KINGBIRD #1 | |
| HIF9 | CA (−) | SUP152/KACHU/3/TRCH/SRTU//KACHU |
| TG (+) | SUP152/KACHU/3/TRCH/SRTU//KACHU | |
| HIF10 | CA (−) | BL3264*2/PAURAQ |
| TG (+) | BL3264*2/PAURAQ |
La generación HIF3:11 fue sembrada en campo abierto el 3 de junio de 2020 en la Estación Experimental El Batán (CIMMYT), Texcoco, Estado de México, bajo un diseño experimental de bloques completos al azar con dos repeticiones. En cada bloque se aleatorizaron 20 unidades experimentales, las cuales son producto de las 10 HIF (para cada familia existía una NIL con el alelo favorable TG y otra con el alelo desfavorable CA). La unidad experimental fue un surco de 2 m sembrado a doble hilera. El cultivo se manejó en condiciones de riego. La cosecha se realizó de forma manual y la trilla mecánicamente.
La generación HIF4:12 fue sembrada en el campo Experimental Norman E. Borlaug (CENEB), Ciudad Obregón, Sonora. La siembra se realizó el 17 de diciembre de 2020, la unidad experimental fue un surco de 1.5 m sembrado a doble hilera. El diseño experimental, manejo del cultivo, cosecha y trilla fueron similares a lo descrito para la generación HIF3:11.
El rendimiento, peso de mil granos, longitud, ancho y grosor de grano, se midieron como se describe en Pask et al. (2013).
Para estudiar simultáneamente el efecto de las familias y los haplotipos sobre las variables fenotípicas rendimiento, peso de mil granos, longitud, ancho y grosor de grano, se utilizó el siguiente modelo:
Donde yijr es una observación de la variable fenotípica estudiada, μ es la media general, τ r el efecto del r-ésimo bloque con r = 1,2; α i el efecto del nivel i en el factor familia con i =1,..10; β j el efecto del nivel j en el factor haplotipo con j = 1,2; (αβ) ij el efecto de la interacción entre ambos factores; los factores y su interacción se consideraron como efectos fijos, en ε ijr las desviaciones aleatorias alrededor de las medias, las cuales asumen una distribución normal independiente e idéntica con media 0 y varianza σ 2 .
Bajo las pruebas de hipótesis del análisis de varianza de dos factores, se pudo concluir que las medias de las variables fenotípicas estudiadas difirieron significativamente (P ≤ 0.05) en al menos dos de las medias de los niveles de cada factor; con esta conclusión se aplicó la prueba de Tukey (P ≤ 0.05) para discernir cuales de las diferencias de los valores promedio de los haplotipos en cada familia fueron significativamente distintos. Para obtener los resultados de estos análisis se usó el programa R (R Core Team, 2020).
RESULTADOS Y DISCUSIÓN
Diseño de marcadores moleculares
Se desarrollaron y verificaron dos marcadores KASP (Cuadro 1), a partir de secuencias GBS reportadas en Sehgal et al. (2019). Los ensayos KASP pudieron diferenciar genotipos en grupos separados, como se esperaba (Figura 1). Al comparar el genotipado GBS contra KASP se obtuvo una coincidencia del 96 % para el SNP C/T y 95 % para el SNP A/G.
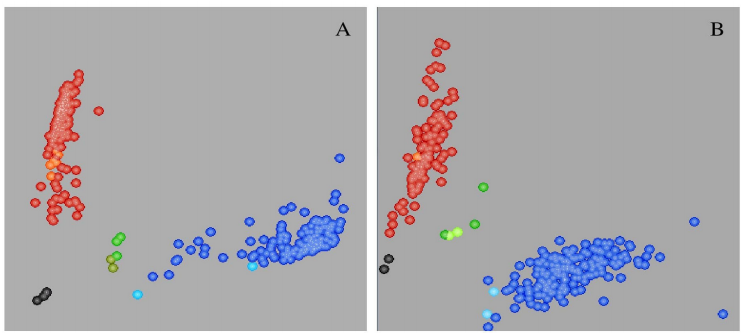
Figura 1 Gráficos de dispersión para ensayos KASP mostrando el agrupamiento de genotipos de trigo en los ejes X (FAM) y Y (HEX). A) ensayo KASP para SNP C/T, B) ensayo KASP para SNP A/G. Puntos de color rojo corresponden a genotipos con el alelo HEX, puntos en color azul corresponden a genotipos con el alelo FAM, puntos de color verde corresponden a genotipos heterocigotos y puntos de color negro corresponden a controles sin ADN.
Las diferencias en el genotipado correspondieron principalmente a los resultados reportados como heterocigotos, lo cual puede deberse a limitantes de la tecnología GBS, como la poca profundidad de secuenciación y la secuenciación multiplexada o por la heterocigosidad residual dentro y entre los lotes de semillas, la cual no es tan infrecuente (Dreisigacker et al., 2020). En cuanto a datos faltantes, el genotipado KASP tuvo un porcentaje de datos perdidos del 6 %, mientras que en el GBS fue del 59 %.
Los dos métodos de genotipado tienen resultados similares para los polimorfismos C/T y A/G, ésto prueba que los marcadores KASP amplifican el mismo locus que GBS. El buen desempeño de los ensayos KASP los convierte en herramientas útiles en la selección asistida, y al ser una plataforma de genotipado de alto rendimiento, los ensayos KASP pueden utilizarse para analizar un gran conjunto de accesiones de trigo.
Los polimorfismos C/T y A/G generaron los haplotipos TG, CA y CG que se encontraron en frecuencias de 30.3, 62.6 y 6.9 % respectivamente en el panel WAMI y frecuencias de 47.8, 32.4 y 19.7 % respectivamente en los EYT analizados. Considerando que WAMI está formado por líneas liberadas desde hace 35 años y los EYT contienen líneas más recientes, se demuestra la existencia de selección indirecta para el haplotipo favorable TG.
Estudio de GWAS
En el mapeo de asociación, los dos marcadores KASP mostraron asociación significativa con el peso de mil granos en las cuatro colecciones de trigo analizadas (Cuadro 4) y, al analizar los dos marcadores como un haplotipo, se obtuvieron asociaciones más significativas que los análisis de marcador individual, ésto fue así en seis de los siete análisis. La variación fenotípica explicada por el marcador (R 2 ) osciló entre 4.6 y 5.9 % en las cuatro colecciones de trigo analizadas.
Cuadro 4 Resumen de resultados de GWAS con marcadores individuales y haplotipos para validar la asociación de los marcadores KASP con el peso de mil granos en cuatro colecciones de trigo.
| Colección de trigo | P-valor | Marcador-R2 | |
|---|---|---|---|
| Marcador KASP C/T | EYT2013-14 | 2.70E-08 | 0.03670 |
| EYT2014-15 | 5.91E-04 | 0.01512 | |
| EYT2015-16 | 0.0055 | 0.00927 | |
| WAMI 2010 | 1.92E-04 | 0.03413 | |
| WAMI 2011 | 6.94E-04 | 0.03188 | |
| WAMI 2013 | 0.00929 | 0.02013 | |
| Marcador KASP A/G | EYT2013-14 | 7.64E-09 | 0.03873 |
| EYT2014-15 | 3.95E-09 | 0.03506 | |
| EYT2015-16 | 1.07E-08 | 0.03923 | |
| WAMI 2010 | 7.39E-06 | 0.05922 | |
| WAMI 2011 | 6.57E-05 | 0.05450 | |
| WAMI 2013 | 4.17E-04 | 0.04708 | |
| Haplotipo | EYT2013-14 | 3.95E-10 | 0.05077 |
| EYT2014-15 | 1.90E-10 | 0.04661 | |
| EYT2015-16 | 4.87E-09 | 0.04841 | |
| WAMI 2010 | 8.95E-06 | 0.05902 | |
| WAMI 2011 | 1.18E-04 | 0.05177 | |
| WAMI 2013 | 3.51E-04 | 0.04850 |
Debido al bajo R 2 del marcador (menos de 10 %), se considera como un QTL menor para peso de grano y, dado que el QTL tuvo significancia estadística en las cuatro colecciones de trigo analizadas, se sugiere que el QTL HAP-6A-13 es estable; por lo tanto, ofrece potencial de ser usado para la piramidación de alelos favorables a través de selección asistida por marcadores.
La asociación de los marcadores KASP con el rendimiento sólo se analizó en WAMI, el análisis basado en haplotipos mostró significancia estadística en los años 2010 y 2011, no así en 2013, el valor promedio de R 2 del marcador para estos tres años fue de 2 % (Cuadro 5). Los resultados obtenidos mostraron un comportamiento propio de los QTL menores, los cuales tienen un efecto pequeño en el fenotipo y son afectados por las interacciones ambientales y epistáticas al punto de enmascarar su efecto (Zhang et al., 2016). A pesar de las limitaciones de los QTL menores, éstos pueden usarse en conjunto con otros QTL para producir de manera aditiva o sinérgica incrementos en peso de grano y rendimiento.
Cuadro 5 Resumen de resultados de GWAS con marcadores individuales y haplotipos para validar la asociación de los marcadores KASP con rendimiento en WAMI.
| Año de cultivo | P-valor | Marcador_R2 | |
|---|---|---|---|
| Marcador KASP C/T | 2010 | 3.57E-04 | 0.04059 |
| 2011 | 0.00301 | 0.02652 | |
| 2013 | 0.24788 | 0.00466 | |
| Marcador KASP A/G | 2010 | 1.79E-04 | 0.05491 |
| 2011 | 0.01197 | 0.02710 | |
| 2013 | 0.07567 | 0.01831 | |
| Haplotipos | 2010 | 2.07E-04 | 0.05396 |
| 2011 | 0.00734 | 0.03038 | |
| 2013 | 0.30965 | 0.00846 |
La mayor significancia de los análisis basados en haplotipos comparados con los marcadores individuales puede deberse a que los haplotipos tienen el potencial de estar en mayor desequilibrio de ligamiento con las variantes causales que los SNP individuales (Sehgal y Dreisigacker, 2019).
Validación en familias endogámicas heterogéneas
El análisis de varianza reveló efectos significativos (P ≤ 0.05) del ambiente y de familia para los caracteres rendimiento, peso de mil granos, ancho y grosor de grano (Cuadro 6). El genotipo y el ambiente fueron los factores más importantes que explicaron la variación encontrada. El efecto del ambiente fue particularmente alto para rendimiento (96 %) y ancho de grano (51.9 %). El efecto de familia se atribuye al fondo genético, los mayores efectos del fondo genético fueron para largo de grano (82.4 %) y peso de mil granos (66 %), lo que demuestra que el fondo genético está afectando el comportamiento del QTL HAP-6A-13.
Cuadro 6 Efecto de ambiente, familia, haplotipo y sus interacciones, expresados como porcentajes de la suma total de cuadrados del ANOVA.
| Fuente de variación | Ren | PMG | LG | AG | GG |
|---|---|---|---|---|---|
| Ambiente | 96.00 ** | 21.23 ** | 0.27 | 51.91 ** | 3.16 ** |
| Familia | 0.47 | 66.09 ** | 82.45 ** | 39.76 ** | 0.82 ** |
| Haplotipo | 0.07 | 0.34 | 4.91 ** | 0.11 | 0 |
| Ambiente × Repetición | 0.19 | 0.59 * | 0.02 | 0.08 | 0 |
| Ambiente × Familia | 0.47 | 2.91 * | 4.28 ** | 2.91 ** | 0.04 |
| Ambiente × Haplotipo | 0.07 | 0.00 | 0.88 ** | 0.00 | 0 |
| Familia × Haplotipo | 0.29 | 3.09 * | 2.28 * | 2.16 ** | 0.03 |
| Ambiente × Familia × Haplotipo | 0.29 | 0.78 | 1.31 | 0.37 | 0.02 |
| Residual | 2.14 | 4.96 | 3.58 | 2.70 | 0.12 |
Ren: rendimiento, PMG: peso de mil granos, LG: longitud de grano, AG: ancho de grano, GG: grosor de grano. Los asteriscos indican la significancia (*: P ≤ 0.05, **: P ≤ 0.01).
La comparación de medias para los caracteres fenotípicos mostró un efecto significativo (P ≤ 0.05) del haplotipo TG en peso de mil granos en la familia HIF 3 y en longitud de grano en las familias HIF 3, HIF 5 y HIF 6, y no hubo un efecto significativo de algún haplotipo para rendimiento, ancho y grosor de grano (Cuadro 7).
Cuadro 7 Comparación de medias de rendimiento (Ren), peso de mil granos (PMG), longitud de grano (LG), ancho de grano (AG) y grosor de grano (GG) en 10 HIF portadoras de los haplotipos TG y CA del QTL HAP-6A-13 evaluadas en los ambientes Batán y Ciudad Obregón, Mexico.
| Familia | Ambiente | Haplotipo | Ren (g) | PMG (g) | LG (mm) | AG (mm) | GG (mm) |
|---|---|---|---|---|---|---|---|
| HIF 1 | Batán | CA (-) | 849 | 43.75 | 6.4 | 3.52 | 3.38 |
| TG (+) | 918.5 | 44.9 | 6.54 | 3.51 | 3.38 | ||
| Obregón | CA (-) | 2070 | 42.8 | 6.54 | 3.35 | 3.01 | |
| TG (+) | 2034 | 42.3 | 6.5 | 3.3 | 3.01 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 2 | Batán | CA (-) | 798.5 | 41.4 | 6.36 | 3.45 | 3.35 |
| TG (+) | 742.5 | 40.6 | 6.49 | 3.43 | 3.28 | ||
| Obregón | CA (-) | 2015 | 38.25 | 6.29 | 3.28 | 2.96 | |
| TG (+) | 1932 | 40.35 | 6.41 | 3.31 | 2.97 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 3 | Batán | CA (-) | 957.5 | 39.4 | 6.26 | 3.39 | 3.24 |
| TG (+) | 921 | 44.8 | 6.42 | 3.52 | 3.34 | ||
| Obregón | CA (-) | 2130 | 35.8 | 6.38 | 3.09 | 2.82 | |
| TG (+) | 2054 | 38.6 | 6.53 | 3.15 | 2.87 | ||
| Efecto del QTL | ns | 10.9 % * | 2.45 % * | ns | ns | ||
| HIF 4 | Batán | CA (-) | 987.5 | 49.75 | 6.69 | 3.65 | 3.38 |
| TG (+) | 890.5 | 51.55 | 6.72 | 3.7 | 3.43 | ||
| Obregón | CA (-) | 1883 | 43.75 | 6.76 | 3.32 | 3 | |
| TG (+) | 1932 | 45.65 | 6.77 | 3.4 | 3 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 5 | Batán | CA (-) | 1037.5 | 44.5 | 6.54 | 3.47 | 3.24 |
| TG (+) | 886.5 | 45.15 | 6.74 | 3.47 | 3.24 | ||
| Obregón | CA (-) | 2030 | 40.05 | 6.67 | 3.15 | 2.92 | |
| TG (+) | 2029 | 40.05 | 6.73 | 3.08 | 2.84 | ||
| Efecto del QTL | ns | ns | 1.96 % * | ns | ns | ||
| HIF 6 | Batán | CA (-) | 977 | 35 | 6.48 | 3.2 | 3.09 |
| TG (+) | 830 | 36.35 | 6.62 | 3.28 | 3.08 | ||
| Obregón | CA (-) | 1889 | 33.5 | 6.54 | 2.89 | 2.63 | |
| TG (+) | 1926 | 32.65 | 6.67 | 2.97 | 2.57 | ||
| Efecto del QTL | ns | ns | 2.07 % * | ns | ns | ||
| HIF 7 | Batán | CA (-) | 1037 | 42.35 | 6.51 | 3.43 | 3.27 |
| TG (+) | 927 | 39.55 | 6.53 | 3.36 | 3.2 | ||
| Obregón | CA (-) | 2022 | 35.5 | 6.43 | 3.1 | 2.87 | |
| TG (+) | 1850 | 34.3 | 6.47 | 3.06 | 2.82 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 8 | Batán | CA (-) | 821.5 | 44.4 | 6.41 | 3.42 | 3.38 |
| TG (+) | 865 | 43.2 | 6.53 | 3.39 | 3.3 | ||
| Obregón | CA (-) | 1933 | 39.9 | 6.63 | 3.21 | 2.87 | |
| TG (+) | 2022 | 38.95 | 6.52 | 3.13 | 2.89 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 9 | Batán | CA (-) | 915.5 | 38.15 | 6.22 | 3.33 | 3.15 |
| TG (+) | 1064 | 39.8 | 6.28 | 3.37 | 3.16 | ||
| Obregón | CA (-) | 2017 | 33.25 | 6.13 | 3.06 | 2.78 | |
| TG (+) | 1920 | 35.1 | 6.17 | 3.15 | 2.88 | ||
| Efecto del QTL | ns | ns | ns | ns | ns | ||
| HIF 10 | Batán | CA (-) | 978 | 48.35 | 6.68 | 3.6 | 3.37 |
| TG (+) | 824.5 | 47.1 | 6.79 | 3.58 | 3.29 | ||
| Obregón | CA (-) | 2140 | 41.95 | 6.71 | 3.3 | 2.94 | |
| TG (+) | 1903 | 41.75 | 6.75 | 3.32 | 2.96 | ||
| Efecto del QTL | ns | ns | ns | ns | ns |
Los asteriscos indican las diferencias significativas entre haplotipos dentro de una misma familia (*: P ≤ 0.05, **: P ≤ 0.01), ns: no significativo. El rendimiento se expresa como gramos obtenidos de la unidad experimental.
El haplotipo TG confirió superioridad en peso de mil granos en las familias HIF 3 en los ambientes del centro y noreste de México (P ≤ 0.05), donde dicho haplotipo TG confirió incrementos del 10.9 %.
Los genotipos portadores del haplotipo TG mostraron un efecto superior en longitud del grano en las 10 familias cuando se cultivaron en el centro de México y en ocho familias cuando se cultivaron en el noreste de México, aunque sólo hubo significancia estadística (P ≤ 0.05) en las familias HIF 3, HIF 5 y HIF 6, donde el haplotipo TG confirió incrementos del 2.45, 1.96 y 2.07 % respectivamente.
Estudios anteriores de QTLs que controlan longitud del grano han reportado incrementos similares a los de este estudio para longitud de grano; por ejemplo, Guan et al. (2019) construyeron NIL para el QTL QTgw.cau-4A.3, el cual se había reportado como controlador del tamaño del grano; las NIL fueron evaluadas en tres ambientes durante dos años, el alelo favorable de este QTL confirió incrementos en longitud de grano desde 0.74 hasta 3.87 %. En otra investigación realizada para validar el QTL QTgw.caas-5B mediante NIL el alelo favorable (5B+) confirió incrementos de 2 a 10 % en longitud de grano (Zhao et al., 2021).
Previo a este estudio, sólo se había analizado la asociación del QTL HAP-6A-13 con el peso de mil granos; con los nuevos datos y análisis realizados en esta investigación se añade que el QTL también afecta la longitud de grano. Estudios posteriores para el QTL HAP-6A-13 pueden incluir GWAS de caracteres morfométricos para validar el efecto en longitud de grano.
La asociación significativa encontrada mediante GWAS para peso de grano y rendimiento, pero con falta de significancia (P > 0.05), al comparar líneas isogénicas se demuestra que el estudio de QTLs menores que afectan caracteres complejos sigue siendo un reto para la genética cuantitativa. El desempeño inestable de los QTLs menores son resultado de las interacciones del QTL con el ambiente y del fondo genético, el cual propicia interacciones complejas con otros QTLs.
CONCLUSIONES
El diseño de ensayos KASP a partir de secuencias GBS fue exitoso, los resultados de ambos métodos de genotipado fueron comparados y se obtuvieron altas similitudes, KASP tuvo mejor desempeño en el genotipado de individuos heterocigotos y tuvo menor cantidad de datos perdidos. HAP-6A-13 es un QTL menor, que afecta el peso de mil granos a través del incremento en longitud de grano, su expresión está sujeta al fondo genético, interacciones ambientales e interacciones entre genotipo-ambiente. Para el aprovechamiento del QTL HAP-6A-13 en un programa de mejoramiento genético se deben evaluar las combinaciones posibles con otros alelos favorables para peso de grano y rendimiento y seleccionar sólo las combinaciones que actúan de manera sinérgica para la piramidación de alelos favorables.











 nueva página del texto (beta)
nueva página del texto (beta)


