Introducción
Candidatus Liberibacter solanacearum (CLs) es un endosimbionte secundario en Bactericera cockerelli Sulc (Bc) y ha sido asociado a tres de las enfermedades más importantes de solanáceas en México: “Zebra Chip” (ZC) en papa (Solanum tuberosum L.) (Munyaneza et al., 2009a), “Brotes cloróticos” (BC) en chile (Capsicum annuum L.) (Munyaneza et al., 2009b) y “Permanente del Tomate” (PT) (Melgoza et al., 2018; Munyaneza et al., 2009c). Hasta la fecha se han reportado nueve haplotipos de cepas de CLs: A, B, C, D, E, F, G, H y U. Los haplotipos A, B, F y G son transmitidos por B. cockerelli en América y el haplotipo A ha sido también descrito en Nueva Zelanda. Los haplotipos C, D y E se han reportado en Europa, los haplotipos D y E en África del Norte, mientras que los haplotipos H y U han sido descritos en Finlandia (Haapalainen et al., 2019).
El PT fue reportado por primera vez en tomate (Solanum lycopersicum L.) en la zona del Bajío, México en 1984 (Garzón-Tiznado et al., 2009) y está considerada como una de las enfermedades más importantes de este cultivo, ya que ha reducido la superficie de siembra de esta hortaliza en un 45 % en México (Jirón-Rojas et al., 2016). Cuando el PT ocurre durante las primeras cuatro semanas de floración las pérdidas varían del 75 al 98 %. Sus principales síntomas son clorosis apical, hojas quebradizas y encorvadas, foliolos con márgenes de color morado, oscurecimiento del tejido vascular en la base del tallo y la raíz, aborto de flor, amarillamiento foliar y finalmente muerte de la planta. Es una enfermedad que inicialmente se relacionaba con organismos tipo fitoplasmas (Garzón-Tiznado et al., 2009); sin embargo, estudios posteriores la describen como Candidatus Liberibacter solanacearum (CLs) (Liefting et al., 2009); así mismo, se ha determinado la relación entre esta bacteria y el insecto vector B. cockerelli (Garzón-Tiznado et al., 2009).
En la actualidad, el control de las enfermedades transmitidas por psílidos se basa principalmente en aplicaciones de plaguicidas; sin embargo, esta alternativa no es lo suficientemente eficiente al considerar la alta densidad en la que se encuentran los psílidos y el corto periodo de acceso a la inoculación requerida para inducir enfermedades (Garzón-Tiznado et al., 2009; Greenway y Rondon, 2018); además, las frecuentes aplicaciones de insecticidas tienen impactos negativos en el ambiente, la salud humana y contribuyen al desarrollo de resistencia en las poblaciones de los vectores (Rashidi et al., 2017). Por lo anterior, el uso de plantas resistentes se ha propuesto como un método complementario y una alternativa sustentable de largo plazo para el manejo de Candidatus Liberibacter solanacearum (CL) (Cooper y Bamberg, 2016); sin embargo, a la fecha no se han encontraron reportes de resistencia a CLs en cultivares comerciales de tomate (Solanum spp.). Por lo anterior, los objetivos de la presente investigación fueron analizar la respuesta de genotipos de tomate ante la infección con Candidatus Liberibacter solanacearum y estudiar la diversidad genética entre ellos.
Materiales y métodos
Material genético
Los genotipos de tomate evaluados fueron cuatro cultivares comerciales tipo saladette de crecimiento indeterminado: Río Grande®, Moctezuma® (Harris Moran), La Roca® (Nirit Seeds) y DRK 2180® (Seminis); un cultivar tipo bola con crecimiento indeterminado, Bonny Best, un cultivar comercial tipo Marmande, con fruto multilocular aplanado y crecimiento indeterminado y la línea avanzada UAS 2016, obtenida de una colecta local de tomate silvestre (Solanum lycopersicum var. cerasiforme), la cual presentó previamente un buen comportamiento ante la presencia de CLs en condiciones de campo abierto y que al cruzarse inicialmente con la variedad de polinización libre Hayslip produjo una F1 susceptible; el comportamiento de tolerancia se registró en una selección individual de la generación segregante en F2, lo que indicó que el carácter de tolerancia a CLs está contenido en al menos un gen recesivo que debió estar de forma homocigota. El genotipo Río Grande®, en este ensayo, se utilizó como control susceptible.
Sitio experimental y manejo agronómico
El ensayo agronómico se llevó a cabo en un sitio experimental ubicado en la localidad de Culiacancito, Sinaloa (24° 48’ 56.53” N y 107° 32’ 2.16” O). La siembra de los genotipos se realizó en enero del 2017 en condiciones de invernadero, con el empleo de semilleros de poliestireno de 200 cavidades, con sustrato comercial tipo peat-moss y previamente desinfestadas con hipoclorito de sodio 5 %. La plántula se regó diariamente con agua corriente y cada segundo día se adicionó solución nutritiva Steiner al 50 %; además, se realizaron tratamientos preventivos contra hongos mediante aplicaciones del fungicida clorotalonil (7.5 g L-1) y contra insectos con el insecticida cipermetrina (2.0 mL L-1). Una vez que brotó la tercera hoja verdadera las plantas se trasplantaron a macetas y se realizaron actividades de fertirrigación con riego por goteo. La fertilización consistió en una dosis final de 7-2-2 g (N-P-K) por planta con productos comerciales hidrosolubles: Ultrasol inicial (15-30-15), Urea (46 % de N) y Ultrasol Multipropósito (18-18-18). La nutrición se complementó con tres aplicaciones de fertilizante foliar líquido (Bayfolán Forte®) para aportar elementos menores (Leyva-Mir et al., 2013).
Ensayo de resistencia
Para establecer la colonia del vector B. cockerelli, y simultáneamente la fuente de inóculo de CLs con la finalidad de realizar el ensayo de resistencia, se recolectaron insectos en campo abierto con el apoyo de un succionador entomológico a partir de plantas de tomate que manifestaban síntomas de la enfermedad PT que se describen en este documento. Los insectos, previo al establecimiento de la colonia, se identificaron de acuerdo con su descripción morfológica (Marín-Jarillo et al., 1995). Estos insectos se transfirieron a plantas sanas de tomate y chile para su alimentación, contenidas en jaulas entomológicas de madera y cubiertas con una malla calibre 40 hilos por cm2 (40 × 40 × 60 cm de longitud, anchura y altura). Las jaulas entomológicas se mantuvieron en invernadero a 28 ± 2 °C, lo que permitió el desarrollo de nuevas generaciones de insectos y la manifestación de los síntomas del PT en un total final de 50 plantas, hasta alcanzar la población necesaria del vector para llevar a cabo el ensayo de transmisión de la bacteria (Rotbi et al., 2010). El ensayo de resistencia a CLs se realizó bajo un diseño de bloques completos al azar con cuatro repeticiones. Cada repetición incluyó 10 plantas por genotipo, en total 280 plantas, a las cuales se realizaron siete observaciones, una cada semana. La transmisión de la bacteria a las plantas por medio del insecto vector B. cockerelli se realizó cuando éstas tenían tres hojas verdaderas. Se colocaron 20 insectos adultos en cada planta y los insectos se contuvieron con un recipiente de plástico. El tiempo de alimentación del insecto en la planta fue de una semana y posteriormente éstos fueron eliminados con insecticida Imidachloprid asperjado a una dosis de 4 mL L-1 de agua.
Detección de CLs
La identidad de la bacteria CLs se realizó en los insectos y en las plantas con base en el protocolo descrito por Melgoza et al. (2018), mediante la amplificación por PCR de un fragmento del gen 16S de ARNr. Para ello, se extrajo el ADN de adultos del insecto y del tejido vegetal de plantas de tomate sanas y enfermas, con base en la metodología de extracción de ADN descrita por Doyle y Doyle (1990). El ADN extraído de plantas de tomate sanas fue utilizado como control negativo. Las muestras de ADN se almacenaron en refrigeración a una temperatura de 4 ºC hasta su uso en el ensayo de PCR.
Para la identificación de CLs, las muestras de ADN fueron analizadas con el empleo del par de oligonucleótidos específicos OA2/OI2c (5’-GCGCTTATTTTTAATAGGAGCGGCA-3’/5’-GCCTCGCGACTTCGCAACCCAT-3’), los cuales amplifican un fragmento predicho de 1168 pb (Contreras-Rendón et al., 2019; Munyaneza et al., 2009c). Las reacciones de amplificación se realizaron en un volumen final de 25 µL, que contenían 3 mM de MgCl2, 0.2 mM de dNTPs, 0.25 µM de cada oligonucleótido, 1 U de Taq ADN polimerasa (Invitrogen, Carlsbad, California, EUA) y 50 ng de ADN. Las condiciones de amplificación fueron: desnaturalización inicial de 3 min a 94 ºC, seguida de 35 ciclos de desnaturalización por 30 s a 94 ºC, alineamiento por 30 s a 60 ºC y extensión por 1 min a 72 ºC, seguidos de una extensión final de 10 min a 72 ºC (Melgoza et al., 2018).
Los fragmentos amplificados se analizaron en agarosa 1 % teñidos con colorante Gel red (BIOTIUM) y se observaron en un fotodocumentador Chemidoc™ XRS (Bio-Rad, Hercules, California, EUA). Los amplicones predichos se purificaron con el empleo del kit comercial UltraClean 15 DNA Purification Kit (Mo Bio Laboratories Inc., Solana Beach California, EUA) y posteriormente fueron secuenciados en la Unidad de Síntesis y Secuenciación del Instituto de Biotecnología de la UNAM. La identidad de la bacteria, presente en el insecto y en el tejido vegetal de plantas de tomate, se obtuvo a partir de un alineamiento BLAST entre las secuencias obtenidas en el presente estudio y las secuencias reportadas para CLs en la base de datos del NCBI.
Evaluación del nivel de daño de CLs a plantas de tomate
Las plantas de tomate inoculadas con CLs por insectos se mantuvieron en observación hasta que aparecieron los primeros síntomas de la enfermedad del PT, y a partir de ese momento se inició el proceso de registro de los síntomas observados en la planta. Los parámetros se establecieron con base en el síndrome descrito para la enfermedad del PT (Figura 1A); para ello se diseñó una escala acumulativa que varió de 1 (planta marchita) a 10 (planta sin síntomas aparentes), cuyos valores progresivos representaron el avance de la enfermedad (Figura 1B). El registro de los valores se realizó cada semana a partir de la aparición de los primeros síntomas. Las temperaturas promedio máximas y mínimas durante el ensayo fueron 32.4 y 15.3 °C, respectivamente. El análisis de datos se realizó con el empleo del paquete estadístico Minitab® 17 Statistical Software (State College, Pennsylvania, EUA), donde se realizó análisis de varianza y para la comparación de medias se aplicó la prueba de Tukey (P ≤ 0.05).
| Calificación | Síntomas |
| 1 | Planta marchita |
| 2 | Hojas marchitas |
| 3 | Clorosis general |
| 4 | Aborto de flor |
| 5 | Deformación foliar |
| 6 | Achaparramiento |
| 7 | Amarillamiento en hojas jóvenes |
| 8 | Epinastia |
| 9 | Clorosis en el ápice |
| 10 | Planta sin síntomas aparentes |
Análisis de variabilidad genética
Con la finalidad de conocer la variabilidad genética entre y dentro de los siete genotipos de tomate se realizó un ensayo para analizar la diversidad alélica; para ello se revisaron 15 marcadores microsatélites (SSR), previamente reportados para el genoma de tomate y seleccionados de la base de datos Solanaceae Genomic Network (SolGenomics) (http://www.sgn.cornell.edu/).
El ADN genómico se extrajo a partir de hojas jóvenes de 15 plantas de cada uno de los genotipos de tomate, de acuerdo con lo descrito por Doyle y Doyle (1990). La amplificación de los microsatélites se llevó a cabo en reacciones con un volumen final de 15 µL, las cuales contenían 2 mM de MgCl2, 0.1 mM de dNTPs, 0.4 µM de cada oligonucleótido, 1 U de Taq ADN polimerasa (Invitrogen, Carlsbad, California, EUA) y 50 ng de ADN. Las amplificaciones se realizaron en un termociclador C1000™ (Bio-Rad, Hercules, California, EUA), con las siguientes condiciones: una desnaturalización inicial de 5 min a 94 ºC, seguida de 30 ciclos de desnaturalización por 30 s a 94 ºC, alineamiento por 30 s a una temperatura entre 42 y 59 ºC, dependiendo del marcador (Cuadro 2), una extensión por 1.5 min a 72 ºC, seguidos de una extensión final de 5 min a 72 ºC.
Cuadro 1 Descripción de la ubicación, número, tamaño y contenido de información polimórfica de 15 microsatélites empleados para estimar la diversidad genética de siete genotipos de tomate.
| Marcador | Secuencia repetida | Cr | TA (º C) | No. de alelos | Tamaño de alelos (pb) | CIP |
| LEE8 (1) | (TA)5 | 1 | 54 | 4 | 152-182 | 0.55 |
| OCT3 (1) | (TAT)15-1(TGT)4 | 11 | 55 | 4 | 155-174 | 0.65 |
| OCT8 (1) | (CCCCA)4 | 12 | 55 | 5 | 163-447 | 0.64 |
| SSR20 (4) | (GAA)8 | 12 | 55 | 4 | 126-194 | 0.53 |
| SSR594 (4) | (TCT)8 | 8 | 55 | 4 | 295-333 | 0.64 |
| OTA11(3) | (TA)20 | 9 | 54 | 4 | 237-279 | 0.62 |
| OTA8 (1) | (CT)8-1(AT)4 | 9 | 54 | 4 | 254-286 | 0.65 |
| SSR66 (1) | (ATA)8 | 6 | 55 | 4 | 168-240 | 0.26 |
| TMS60 (2) | (AT)13(CA)23(AT)2 | 7 | 46 | 3 | 153-221 | 0.35 |
| TMS29 (2) | (CT)3C14(CT)23 | 8 | 54 | 4 | 115-391 | 0.37 |
| EST253712 (1) | (AT)14 | 6 | 52 | 3 | 112-136 | 0.45 |
| LEEF1A (1) | (TA)8(ATA)9 | 2 | 55 | 4 | 128-263 | 0.52 |
| OTA13 (1) | (TAT)15-1(TGT)4 | 11 | 55 | 4 | 332-409 | 0.55 |
| SSR117 (4) | (TC)11 | 1 | 59 | 5 | 124-318 | 0.7 |
| SSR26 (4) | (CGG)6 | 2 | 46 | 3 | 162-185 | 0.43 |
| Promedio | - | - | - | 3.93 | - | 0.53 |
Cr: cromosoma, TA: temperatura de alineamiento, pb: pares de bases, CIP: contenido de información polimórfica. (1) Smulders et al. (1997), (2) Areshchenkova y Ganal (1999), (3) He et al. (2003), (4) Ranc et al. (2008).
Cuadro 2 Diversidad Genética dentro de los siete genotipos de tomate.
| Genotipo | Número de alelos por locus | Diversidad genética (Hs) |
| UAS 2016 | 2.4 | 0.39 |
| Río Grande® | 2.8 | 0.43 |
| La Roca® | 2.5 | 0.39 |
| DRK 2180® | 2.5 | 0.44 |
| Bonny Best® | 2.3 | 0.36 |
| Moctezuma® | 2.5 | 0.40 |
| Marmande | 2.3 | 0.41 |
| Diversidad genética total | 3.9 | 0.58 |
Los productos amplificados fueron separados por electroforesis en geles de poliacrilamida 15 % (p/v), teñidos con bromuro de etidio (1 µg mL-1) y se visualizaron bajo luz UV en un fotodocumentador Chemidoc™ XRS (Bio-Rad, Hercules, California, EUA). Las imágenes digitalizadas de los geles se analizaron por medio del software Gel Analyzer. La presencia y el tamaño de los fragmentos se estimaron comparándolos con el marcador de peso molecular de 50 pb (Jena Bioscience, Jena, Alemania).
El análisis de datos se realizó con el programa Power Marker ver. 3.25 (Liu y Muse, 2005). Se utilizaron las frecuencias alélicas para calcular el contenido de información polimórfica (CIP) de cada SSR y la diversidad genética (Hs) en cada uno de los genotipos. Para determinar la contribución de cada uno de los alelos a la variabilidad genética total se realizó un análisis de componentes principales. El análisis se realizó a partir de las frecuencias alélicas de los marcadores utilizando el programa Stata 11.1 (StataCorp, College Station, Texas, EUA). La distancia genética entre los genotipos se estimó usando el coeficiente de Nei (1972) y las relaciones genéticas entre los mismos se establecieron mediante análisis de agrupamiento por promedios aritméticos (UPGMA). El dendograma generado se visualizó mediante el software NTSYS a partir de la matriz de distancia genética (Zhou et al., 2015).
Resultados y discusión
Ensayo de resistencia a CLs
La presencia de CLs en las plantas de la fuente de inóculo y en la colonia de insectos se confirmó por PCR (Figura 2) y secuenciación del amplicón predicho del gen 16S. En la Figura 2 se puede observar el marcador molecular de 1 kb y posteriormente, en el carril 1, el fragmento de 1168 pb, correspondiente al control positivo amplificado a partir de una planta enferma. En el carril 2 se confirma la amplificación del fragmento predicho a partir del ADN del insecto adulto de B. cockerelli. En el carril 3 se corrobora la presencia del fragmento predicho en una planta de tomate con síntomas de PT. Finalmente, el carril 4 se trata del control negativo correspondiente a una planta de tomate sin síntomas de PT.

Figura 2 Amplicones del gen 16S de CLs. M1Kb marcador de peso molecular de 1 Kb. El carril 1 corresponde al control positivo, planta de tomate con síntomas de PT, el carril 2 a un insecto adulto de B. cockerelli, el carril 3 a una planta de tomate con síntomas de PT posterior a la transmisión por insecto de CLs, el carril 4 al control negativo, planta de tomate sin síntomas de PT.
El fragmento amplificado en el carril 3 fue secuenciado y la secuencia obtenida fue alineada con la base de datos del GenBank, que arrojó una identidad entre el 99.66 y 100 % con secuencias de CLs reportadas. La secuencia de este ensayo fue registrada en el GenBank bajo el número de accesión MK620852; adicionalmente, mediante un alineamiento BLAST se determinó que ésta corresponde al haplotipo A de CLs, ya que no presentaron las mutaciones T/G232, G/A466 y T/C601 características del haplotipo B en el gen 16S de CLs (Contreras-Rendón et al., 2019).
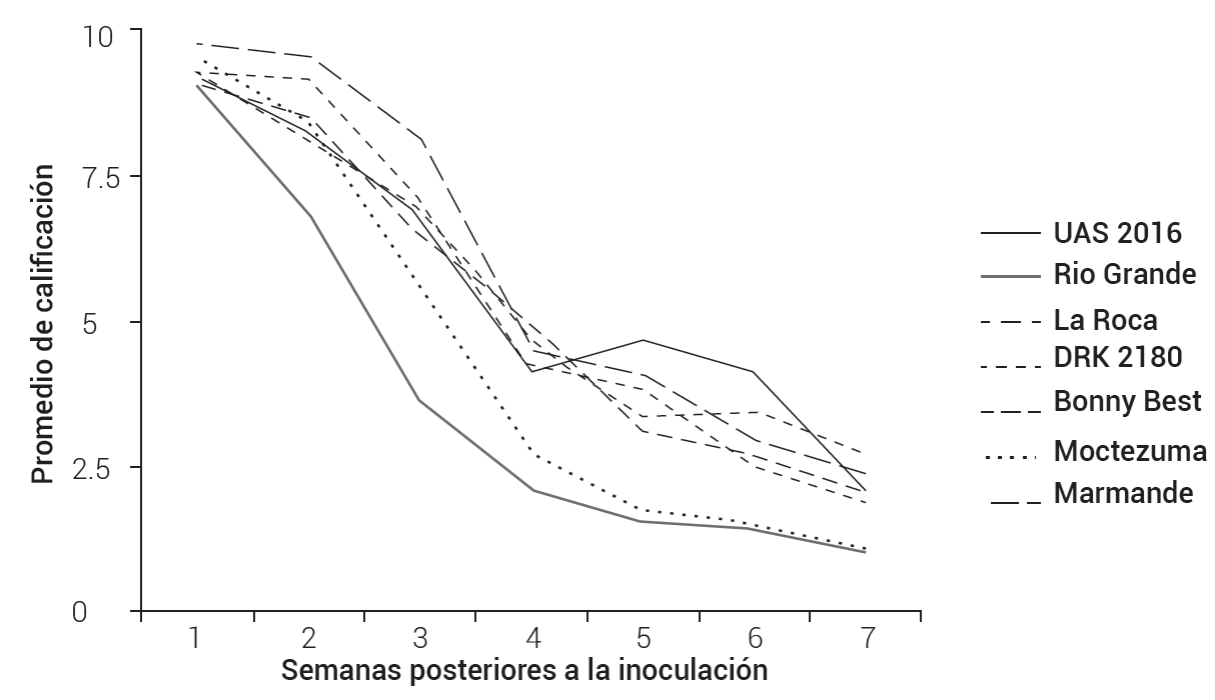
Los síntomas de la enfermedad “Permanente del Tomate” se presentaron en los siete genotipos estudiados tres semanas posteriores al inicio de la inoculación y se registró el avance de los síntomas durante siete semanas. El análisis de los datos de las calificaciones registradas en el ensayo verificó los supuestos de normalidad de los errores (P = 0.823) y varianzas iguales (P = 0.437). Los bloques no afectaron el experimento (P = 0.455), mientras que los genotipos sí manifestaron diferencias entre ellos (P ≤ 0.001).
La comparación de medias reveló diferencias significativas entre la respuesta al PT de los cultivares UAS 2016, La Roca®, DRK 2180®, Bonny Best® y Marmande en comparación con el control susceptible Río Grande®. UAS 2016 y Marmande presentaron una respuesta a CLs significativamente mayor que Río Grande® y Moctezuma® (Figura 3).
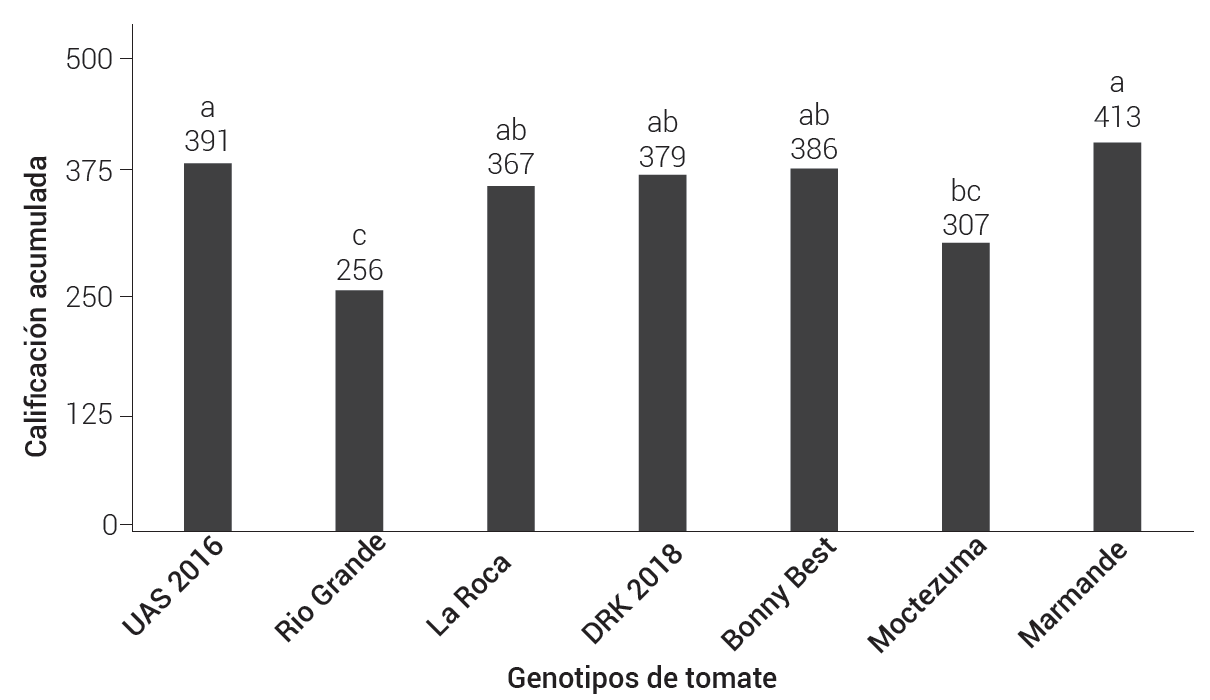
Figura 3 Calificaciones acumuladas del efecto de CLs en siete genotipos de tomate durante siete semanas. Letras diferentes en las barras indican diferencias estadísticas significativas (Tukey, P ≤ 0.05, DSH = 37.45).
Los valores de Río Grande® descendieron con rapidez, Moctezuma® y DRK 2180® mostraron un comportamiento similar al de Río Grande® al iniciar el descenso en calificación en la segunda semana, lo que indicó mayor susceptibilidad a CLs. Los genotipos UAS 2016, La Roca®, Bonny Best® y Marmande descendieron paulatinamente durante las primeras tres semanas y en la cuarta semana mostraron aceleración en el declive. UAS 2016 fue la única que reflejó una recuperación en la quinta y sexta semana, lo que se puede interpretar como mejor defensa contra el efecto del patógeno, aunque regresó a su descenso en la séptima semana cuando las temperaturas fueron superiores a 32 ºC, lo que afectó la floración y el desarrollo de las plantas, sobre todo a las de crecimiento determinado como UAS 2016 (Figura 4). Por lo anterior, se suspendió el registro de la respuesta de los genotipos ante el PT en la octava semana para evitar que el efecto de la temperatura enmascarara los resultados.
El descenso rápido en la calificación reflejó la severa afectación que presentaron los genotipos de tomate ante la enfermedad del PT. Lo anterior podría deberse a que la inoculación de la bacteria se realizó en una edad fenológica temprana; esto es, en plantas con una fenología correspondiente a tres hojas verdaderas, aptas para trasplantarse (Mejía et al., 1993). Garzón-Tiznado et al. (2009) indicaron que cuando se inoculan plantas jóvenes con menos de seis hojas verdaderas, los daños son más severos que al inocular posteriormente; sin embargo, se decidió la inoculación temprana para reducir el riesgo de escape al efecto del patógeno en etapas tardías; no obstante, ésto limitó la toma de datos en etapas más tardías del desarrollo de la planta por el incremento de la temperatura antes mencionado.
Marmande y UAS 2016 fueron los genotipos que presentaron menor sensibilidad al patógeno, sin diferencia significativa entre ellos, aunque tuvieron comportamientos diferentes en el desarrollo de la enfermedad. Marmande mostró una resistencia superior a UAS 2016 durante las primeras tres semanas; sin embargo, UAS 2016 mostró una recuperación y mayor tolerancia al desarrollo de síntomas en la quinta y sexta semana (Figura 4).
Variabilidad genética en siete genotipos de tomate
Para determinar la variabilidad genética se analizaron 15 marcadores moleculares SSR polimórficos en los genotipos estudiados. Con base en estos marcadores se detectó un total de 59 alelos, el número de alelos por locus varió de 3 a 5, con un promedio de 3.93 (Cuadro 1). Estos valores son similares a los reportados por Mazzucato et al. (2010) en un estudio sobre la variabilidad genética de 25 genotipos de tomate endémicos de Italia y por Zhou et al. (2015) en 50 genotipos de tomate provenientes del Centro de Recursos Genéticos de Tomate (TGRC, Davis, California, EUA), aunque superiores al valor 2.72 reportado por Choudhary et al. (2018) en 28 genotipos de tomate de la India.
El contenido de información polimórfica (CIP) es un parámetro de relevancia en el estudio de la variabilidad genética, ya que estima el poder discriminatorio de un marcador y considera el número y frecuencia relativa de los alelos de un locus en la población. Un CIP mayor a 0.5 señala que los loci analizados son altamente polimórficos, valores entre 0.25 y 0.5 indican que son moderadamente informativos, mientras que CIP menores a 0.25 son no informativos (Ceballos et al., 2017). El contenido de información polimórfica promedio obtenido con los marcadores de este estudio fue de 0.53 (Cuadro 1), valor similar al 0.49 registrado por Zhou et al. (2015), pero mayor al 0.39 informado por Choudhary et al. (2018). Ceballos et al. (2017) documentaron un CIP de 0.63, obtenido en tomate tipo Cherry. Por lo anterior, se consideró que el valor de CIP que se obtuvo a partir de los marcadores utilizados en el presente estudio fue suficientemente informativo para desarrollar el análisis de variabilidad genética (Hs), donde se determinó que el valor más alto fue de 0.44 y correspondió al genotipo DRK 2180®. Por otro lado, los genotipos de tomate con variabilidad genética menor fueron Bonny Best®, con valor de 0.36, en segundo lugar UAS 2016 y La Roca® con 0.39. Los genotipos Moctezuma®, Marmande y Río Grande® tuvieron valores de 0.40, 0.41 y 0.43, respectivamente. En términos generales, los genotipos estudiados mostraron valores altos de variabilidad genética. Esta variabilidad se ve reflejada en el error aleatorio (78 % de la varianza total) mostrado en el análisis estadístico; es por ello, que para futuros estudios se recomienda usar semillas de variedades de tomate más homogéneas genéticamente, para incrementar la probabilidad de separar las medias de las calificaciones acumuladas de los síntomas correspondientes al PT entre las variedades estudiadas. Los valores de variabilidad genética de los siete genotipos utilizados fueron muy similares entre ellos. El valor de diversidad genética total fue de 0.58. El coeficiente de diferenciación génica (GST) entre poblaciones fue de 0.30, lo que indica que el 30 % de la variación total se debe a diferencias entre las accesiones (Cuadro 2).
El análisis de componentes principales esclareció la contribución de cada alelo. Los primeros dos componentes principales explicaron el 45 % de la variación total, el primer componente aportó el 25 % de la variación mientras que el segundo explicó el 20 %. Para el PC1 el alelo de mayor impacto fue el amplificado con el marcador OTA8 de 254 pb, seguido por los alelos amplificados por los marcadores OCT8 (367 pb), LEE8 (172 pb) y SSR20 (157 pb); mientras que para el PC2 los alelos de mayor contribución fueron EST25 (136 pb), OTA13 (371 pb), OCT8 (334 pb) y OTA13 (409 pb), lo que sugiere que los marcadores OTA8, OCT8, EST25 y OTA13 son candidatos para ser utilizados posteriormente en análisis de asociación entre la variabilidad genética de genotipos de tomate con los niveles de tolerancia al patógeno.
Análisis de agrupamiento
Los valores de distancia variaron de 0.24 (Río Grande®-DRK 2180®) a 0.54 (Bonny Best®-Marmande), con un promedio de 0.42. El análisis de agrupamiento por promedios aritméticos (UPGMA) generó un dendrograma donde se representan de forma gráfica las relaciones genéticas entre los genotipos (Figura 5). En el dendrograma se observa la separación de los genotipos en dos grupos principales. En uno de los grupos se separó el genotipo Marmande y en la segunda agrupación se asociaron los demás genotipos en dos subclados. El subclado IA se conformó por UAS 2016 y Bonny Best®. El subclado IB se integró con DRK 2180®, Río Grande®, La Roca® y Moctezuma®. Este análisis de agrupamiento no mostró relación con el nivel de resistencia a CLs presentado en el ensayo agronómico previamente descrito; sin embargo, se identificó una posible correspondencia entre el resultado del agrupamiento y la característica forma de fruto de los genotipos estudiados. El genotipo Marmande produce un fruto irregular con forma de globo aplanado arriñonado, UAS 2016 produce un fruto tipo “bola” similar a Bonny Best®, mientras que DRK 2180®, Río Grande®, La Roca® y Moctezuma® producen fruto tipo saladette.

Figura 5 Agrupamiento de los siete genotipos de tomate construido por el método de promedios aritméticos (UPGMA) basado en la distancia de Nei (1972).
Conclusiones
Los genotipos de tomate Marmande y UAS 2016 presentaron menores valores de daño por Candidatus Liberibacter solanacearum, seguidos por Bonny Best®, DRK 2180® y La Roca®; la mayor susceptibilidad la presentaron Moctezuma® y el control Río Grande®. El agrupamiento de las distancias genéticas entre los genotipos evaluados no se asoció con la respuesta a CLs. Los genotipos de tomate analizados mostraron valores altos de variabilidad genética y muy similares entre ellos. Bonny Best®, La Roca® y UAS 2016 presentaron la menor diversidad genética.











 nueva página del texto (beta)
nueva página del texto (beta)