INTRODUCCIÓN
Colletotrichum truncatum (Schwein.) es un hongo fitopatógeno causante de antracnosis en diversos hospedantes, entre los cuales se encuentran papaya (Carica papaya L.), chícharo (Pisum sativum L.), haba (Vicia faba L.), lenteja (Lens culinaris Medik.), chile (Capsicum annuum L.), garbanzo (Cicer arietinum L.), cacahuate (Arachis hypogaea L.) y frijol (Phaseolus vulgaris L.) (Latunde-Dada y Lucas, 2007; O’Connell et al., 1993; Pring et al., 1995; Ranathunge et al., 2012; Tapia-Tussell et al., 2008;). En el caso del cultivo de la papaya, C. truncatum produce pérdidas de hasta 50 % y es su principal enfermedad en el periodo de poscosecha; C. truncatum es uno de los agentes causales de antracnosis y su incidencia es de 40 % en frutos de papaya (Tapia-Tussell et al., 2008; Torres-Calzada et al., 2012); además, en México existen otras especies de Colletotrichum que también causan antracnosis como C. coccodes, que afecta la producción de los cultivos de papa (Solanum tuberosum L.) y tomate (Solanum lycopersicum L.) (Nieto-Angel et al., 2019).
Durante la patogénesis, C. truncatum utiliza procesos de infección distintos para ocasionar antracnosis en sus hospedantes. Durante la infección se presentan eventos de adhesión, germinación, penetración, estado biótrofo o estado necrótrofo, o ambos (De Silva et al., 2017). En otras especies de Colletotrichum se han estudiado genes que codifican proteínas mediadoras de la patogénesis como quitina sintasa (CHS1) y β-1-3 glucano sintasa (GLS1), implicadas en la síntesis de quitina y β-glucanos, respectivamente (O´Connell et al., 2012).
La quitina es un polímero de N-acetilglucosamina que representa el principal componente de la pared celular fúngica, le brinda rigidez y es esencial para el crecimiento vegetativo del hongo (Liu et al., 2017). La síntesis de quitina se lleva a cabo por la enzima glicosil transferasa, denominada quitina sintasa (CHS1), la cual es primordial para la producción de estructuras infectivas y la colonización en el hospedante, puesto que se requiere sintetizar pared celular para producir apresorios durante la penetración, hifas de infección durante el estado necrótrofo, así como conidios dentro de acérvulos durante la reproducción y dispersión (Werner et al., 2007).
Otro componente esencial de la pared celular fúngica es β-1-3 glucano, polisacárido en cuya síntesis está involucrada la enzima β-1-3 glucano sintasa (GLS1) (Bowman y Free, 2006; Fesel y Zuccaro, 2016). En hongos patógenos, la regulación de la formación de polímeros de pared celular es importante, así como el cambio estructural durante la morfogénesis relacionada con el proceso de infección; GLS1 se requiere para dar rigidez a la pared celular del apresorio, la formación de hifas en células del hospedante y el establecimiento de comunicación y compatibilidad en la interacción planta-patógeno (Oliveira-García y Deising, 2013).
Por otra parte, la cutinasa (CUT1) es una esterasa que hidroliza los monómeros de cutina de la cutícula del hospedante, lo que desencadena señales moleculares para la formación del apresorio durante el proceso de penetración; además, las cutinasas también están implicadas en la adhesión y germinación de conidios (Vidhyasekaran, 2008). Una etapa del proceso de infección es la penetración, en la que en el extremo apical de los tubos germinativos se forman apresorios que facilitan su adhesión y penetración al hospedante por medio de acción mecánica y lisis enzimática (Kubo et al., 2000). Se han realizado estudios histológicos del proceso de infección de C. truncatum en distintos hospedantes (Ranathunge et al., 2012; Rojo-Báez et al., 2016); sin embargo, la información de la expresión de genes de patogenicidad de esta especie es escasa. El objetivo de este estudio fue determinar el nivel de expresión de los genes CHS1, GLS1 y CUT1 en C. truncatum en su proceso de infección a través de tejido foliar en papaya Maradol y su relación con la formación de estructuras de infección como apresorios, hifas infectivas y acérvulos durante el desarrollo del patógeno.
MATERIALES Y MÉTODOS
Material biológico
La cepa fúngica utilizada en este estudio fue C. truncatum CCM, una cepa previamente caracterizada (ID NCBI: KF147902). El material vegetal consistió de 50 hojas sanas completamente desarrolladas, escindidas de plantas de papaya Maradol de ocho meses de edad en etapa de madurez fisiológica o fructificación, cultivadas en Balbuena, Navolato, Sinaloa con coordenadas 24° 44’ 58” LN 107° 32’ 28” LO y clima cálido. Las hojas se trasladaron al Laboratorio de Fitopatología del Centro de Investigación en Alimentación y Desarrollo, Culiacán; se lavaron con agua corriente, se secaron, se desinfectaron con etanol 70 % y se colocaron en cámara húmeda (bolsas de plástico de 50 × 80 cm con papel absorbente humedecido con agua destilada) a 25 °C y 80 % de humedad relativa; después, se asperjaron 30 mL de una suspensión de esporas de C. truncatum a una concentración de 1 × 106 esporas mL-1 en haz y envés de las hojas de papaya. La inoculación se realizó con extremo cuidado para evitar daños físicos al tejido (Auyong et al., 2015).
Los muestreos de tejido vegetal se realizaron a las 0, 2, 6, 16, 20, 24, 48, 60, 72, 96 y 120 h después de inoculación (hdi). Las muestras se congelaron en nitrógeno líquido y se almacenaron a -80 °C en un ultracongelador (Revco, Thermo Scientific, Waltham, Massachusetts, EUA) para su uso posterior. El proceso se realizó por triplicado.
Extracción de ácidos nucleicos
El ARN total obtenido de las muestras de tejido vegetal desde 0 hasta 120 hdi se extrajo utilizando el reactivo de Trizol (Tri Reagent, Sigma, USA), como lo recomienda el fabricante; la concentración y pureza del ARN total se determinó espectrofotométricamente (NanoDrop, Thermo Scientific, Waltham, Massachusetts, EUA) y su integridad se evaluó mediante electroforesis en gel de agarosa en condiciones desnaturalizantes. Se eliminó el ADN genómico presente en las muestras de ARN mediante DNasa I (Invitrogen, Carlsbad, California, EUA), siguiendo las instrucciones del fabricante. Después, se realizó la transcripción reversa de 1000 ng de ARN total en un volumen de reacción de 20 µL usando 100 U de SuperScript III First-Strand Synthesis System (Invitrogen, Carlsbad, California, EUA) con los iniciadores Oligo DT y Primer H proporcionados en el kit, siguiendo las instrucciones del fabricante. El ADN complementario (ADNc), obtenido a partir de ARN total, se cuantificó mediante espectrofotometría (NanoDrop, Thermo Scientific, Waltham, Massachusetts, EUA) y se diluyó en agua nanopura (Invitrogen, Carlsbad, California, EUA) hasta obtener una concentración de 125 ng µL-1.
Expresión génica relativa
Se emplearon iniciadores previamente reportados para el gen CUT1 y el gen endógeno Tef (Auyong et al., 2015; Fang y Bidochka, 2006). Se diseñaron los iniciadores para los genes CHS1 y GLS1; primeramente se amplificó la región genómica CHS1 de C. truncatum de acuerdo con los iniciadores reportados por Jiang et al. (2014); así mismo, se amplificó la región genómica GLSs1 de C. truncatum, de acuerdo con los iniciadores reportados por Oliveira-García y Deising (2013) a partir de ADN de cultivo puro extraído de acuerdo con Zelaya-Molina et al. (2011); posteriormente, ambos amplicones se secuenciaron y con base en esta secuencia se diseñaron los iniciadores para los genes CHS1 y GLS1 utilizados en ensayos de qPCR, mediante el software PrimerBlast (https://www.ncbi.nlm.nih.gov/tools/primer-blast/); tanto los iniciadores directos como los inversos se diseñaron sobre la región del exón del gen (Cuadro 1) (NCBI, 2018). Los iniciadores fueron sintetizados por la empresa Sigma-Aldrich® a una concentración de 50 nmol y purificados por desalación. Como análisis confirmatorio de especificidad, se alinearon las secuencias de los iniciadores diseñados CHS1 y GLS1 utilizando el software ClustalW (http://www.genome.jp/tools/clustalw/). Para comprobar la eficiencia de cada par de iniciadores durante la amplificación, a partir de 500 ng µL-1 del ADNc obtenido de cada tratamiento, se prepararon siete diluciones seriadas (1:2) (500, 250, 125, 62.5, 31.2, 15.62 y 7.81 ng µL-1).
Cuadro 1. Iniciadores para qPCR empleados en el estudio.
| Nombre | Secuencia 5´- 3´ | Referencia | Amplicón (pb) |
|---|---|---|---|
| Tef-F | AGGACGACAAGACTCACATC | Fang y Bidochka (2006) | 268 |
| Tef-R | GTTCAGCGGCTTCCTTCTC | ||
| CUT1-F | TCTGTTCGGCTACACCAAGAA | Auyong et al. (2015) | 136 |
| CUT1-R | CAAGAAATGCGCCGGCAGGAT | ||
| CHS1-F | GCAGCAGGTCAACAACAAGG | Este estudio | 102 |
| CHS1-R | TGCTTGGGGATCAAGGTGAC | ||
| GLS1-F | ACGAAAGTCCGCATCCACTT | Este estudio | 133 |
| GLS1-R | ATCTTCACGGACCTAGGCGA |
Las mezclas de reacción consistieron en 5 µL de SYBR Green Master Mix (Bio-Rad, Hercules, California, EUA), 300 nM del iniciador endógeno (Tef) o 750 nM de cada iniciador de los genes de interés (CUT1, CHS1 y GLS1) y 125 ng µL-1 de ADNc molde. Se usaron las siguientes condiciones de amplificación: precalentamiento 95 °C 5 min, seguido de 40 ciclos de 95 °C 30 s; 55.7 °C 30 s (Tef), 62.9 °C 30 s (CUT1), 63 °C 30 s (CSH1), 57.5 °C 30 s (GLS1); 72 °C 30 s. Al finalizar se realizó una curva de disociación con intervalo de 60 a 95 °C de los amplicones generados en cada reacción para la identificación de cada fragmento de ADN esperado de acuerdo con la temperatura de fusión (Tm); se realizaron tres réplicas técnicas por cada réplica biológica.
Para cada gen se realizó una gráfica semi-logarítmica Ct versus la dilución. La eficiencia de un par de iniciadores se calculó usando la fórmula E = 10(-1/pendiente) - 1; una pendiente de -3.321 indica 100% de eficiencia (Bustin et al., 2009). Los pares de iniciadores con un nivel de eficiencia de 90 a 110 % se eligieron para el análisis de expresión génica.
El análisis de expresión se realizó mediante la plataforma de detección en tiempo real CFX96 (Bio-Rad, Hercules, California, EUA) para cuantificar la expresión de los genes de interés: CUT1, CHS1 y GLS1, así como del gen endógeno Tef. Los niveles de expresión relativos de los genes de interés se normalizaron contra el gen endógeno y la cantidad relativa del producto amplificado con respecto al control (0 hdi) se calculó mediante el método 2-ΔΔCt (Livak y Schmittgen, 2001).
Diseño Experimental
Con los datos se realizó un análisis de varianza (ANOVA) de una sola vía, con un solo factor (tiempo en hdi), se utilizaron 11 niveles (2, 6, 16, 20, 24, 48, 60, 72, 96 y 120 hdi) así como 0 hdi como control; se realizaron 2 réplicas biológicas con 3 réplicas técnicas y la unidad experimental consistió en una hoja de papaya. La variable respuesta fue el nivel de expresión relativa de los genes (CHS1, GLS1 y CUT1). Se utilizó el programa estadístico Minitab 17; las medias se compararon con la prueba de Dunnet (P ≤ 0.05).
RESULTADOS Y DISCUSIÓN
En este estudio se determinaron los niveles de expresión relativa de tres genes relacionados a patogenicidad, CUT1, GLS1 y CHS1, los cuales codifican, respectivamente, para las enzimas cutinasa, β-1,3 glucano sintasa y quitina sintasa en C. truncatum, durante su interacción curso temporal con hojas de papaya Maradol. Para ello, se emplearon los iniciadores Tef y CUT1, previamente reportados, y se diseñaron los iniciadores CHS1 y GLS1. Posteriormente, se evaluó la especificidad de cada par de iniciadores para los genes de interés (CUT1, GLS1, CHS1) y el gen endógeno (Tef), obteniendo valores de eficiencia de 97 % para el gen CUT1, 106 % para el gen GLS1 y 95 % para el gen CHS1 (Figura 1), los cuales se ubican entre los valores de 90 a 110 % descritos como adecuados por diversos autores (Brookman-Amissah et al., 2015; Bustin et al., 2009). La especificidad de los amplicones se comprobó a partir de la curva de disociación de las muestras de ADN analizadas, al obtener un único amplicón con temperatura de fusión específica para el fragmento predicho (Figura 2).
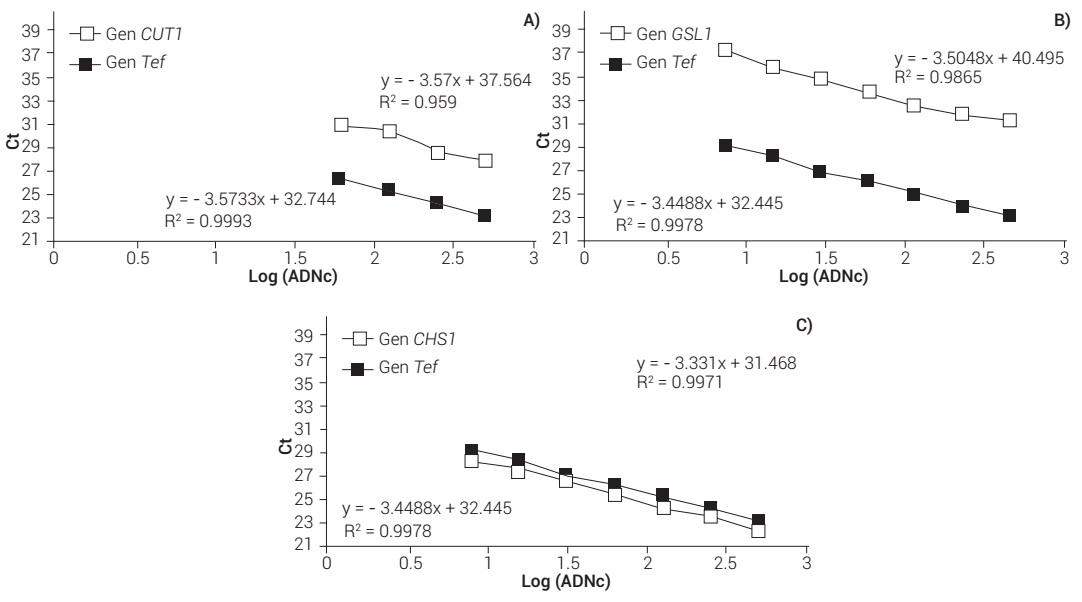
Figura 1. Eficiencia de los genes de C. truncatum con respecto al gen endógeno (Tef). A) Gen CUT1, B) Gen GLS1, C) Gen CHS1.
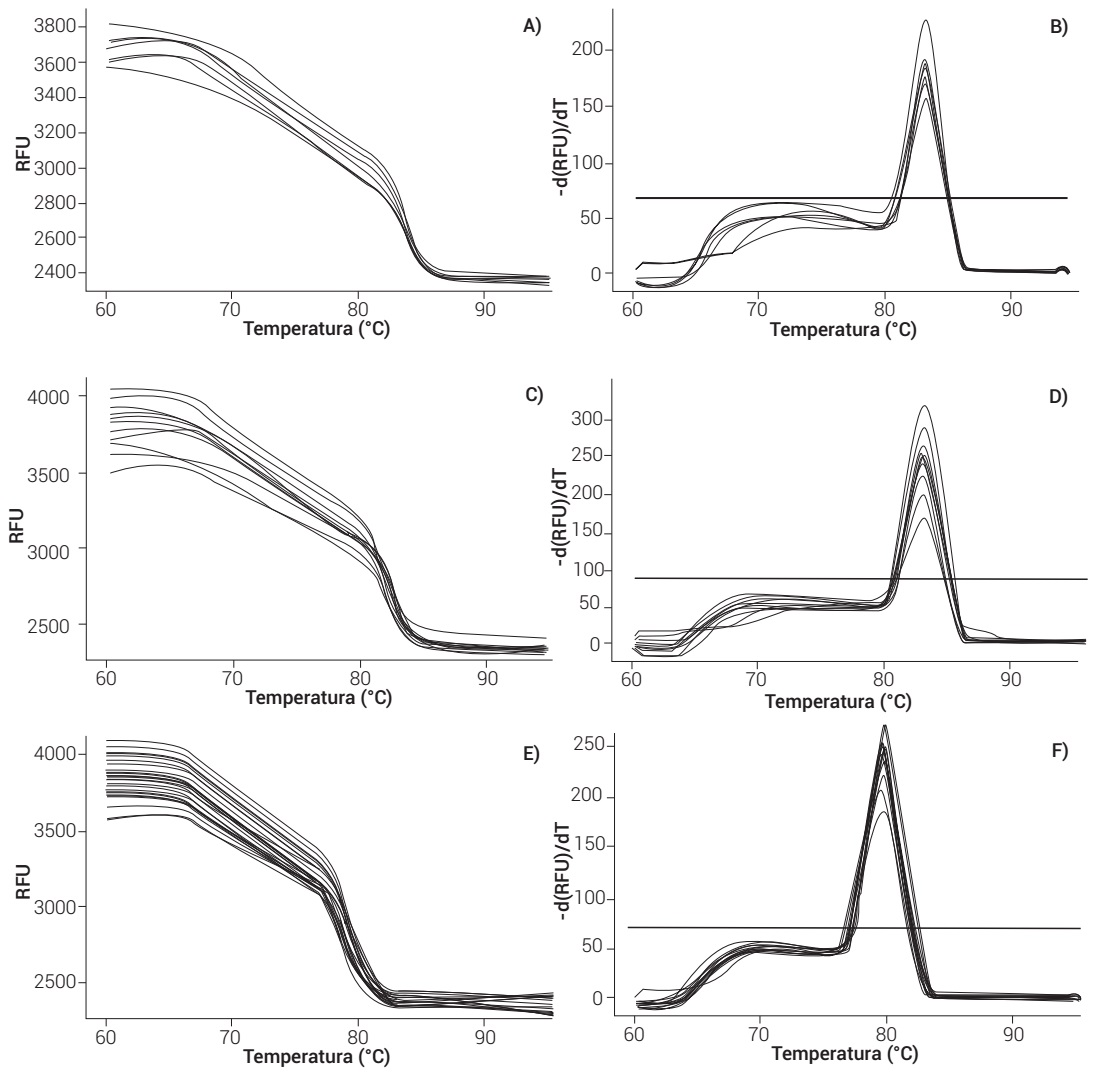
Figura 2. Curva de disociación y amplicón con temperatura de fusión específica de C. truncatum para los genes CUT1 (A-B), GLS1 (C-D) y CHS1 (E-F).
Expresión del gen CUT1
La expresión del gen CUT1 de C. truncatum se detectó a partir de las 20 hdi. El nivel de expresión del gen fue en aumento desde las 20 hasta las 120 hdi; a las 120 hdi se observó un incremento significativo en los niveles de expresión relativa (835 veces más que el control) (Figura 3).

Figura 3. Expresión relativa del gen CUT1 de C. truncatum a diferentes horas después de inoculación con respecto al control (0 hdi). Expresión relativa normalizada con el gen endógeno Tef. Letras diferentes en cada barra indican diferencias estadísticas (P ≤ 0.05).
La detección de la expresión de CUT1 en hojas de papaya Maradol a partir de las 20 hdi puede deberse al inicio de la penetración de C. truncatum en este hospedante. La expresión del gen CUT1 facilita la entrada del patógeno en sus hospedantes (Auyong et al., 2015; Liang et al. 2018; Villafana y Rampersad, 2020).
La expresión de este gen coincide con las etapas de germinación y formación de apresorios (etapa de penetración) durante el proceso de infección de C. truncatum en hoja de papaya Maradol (Rojo-Báez et al., 2016). A pesar de que en el presente estudio se detectó la expresión del gen a partir de las 20 hdi, Auyong et al. (2015) y Rao y Nandineni (2017) reportaron la expresión del gen cutinasa en la interacción C. truncatum-chile durante las primeras horas de infección (6 a 24 hdi), etapa en que C. truncatum emplea estas enzimas para la penetración en hojas de chile.
La posterior sobreexpresión de CUT1 a las 120 hdi (estado necrótrofo) posiblemente es debida a que C. truncatum rompe la cutícula de su hospedante en la etapa de reproducción y dispersión durante el proceso de infección. Los antecedentes sobre la medición de los niveles de expresión de cutinasa durante el estado necrótrofo del proceso de infección son escasos debido a que esta enzima se asocia principalmente con los eventos iniciales (adhesión, germinación y penetración); no obstante, en este estudio se observó una notoria elevación del nivel de transcrito de este gen al final del proceso de patogénesis. De acuerdo con Rojo-Báez et al. (2016), durante el estado necrótrofo del proceso de infección de C. truncatum en hojas de papaya se observó la formación de conidios dentro de acérvulos, los cuales emergieron de la cutícula del hospedante entre las 96 y las 148 hdi, coincidiendo con el incremento aquí observado de la expresión del gen CUT1. Durante esta etapa el patógeno se desarrolló y se reprodujo para finalmente dispersarse. En el estado necrótrofo puede requerirse la síntesis de cutinasa, ya que los acérvulos de Colletotrichum spp. contienen una matriz mucilaginosa que envuelve a los conidios, los cuales segregan cutinasa extracelular para romper la cutícula de su hospedante, facilitando el proceso de dispersión del patógeno (Bergstrom y Nicholson, 2000).
Expresión del gen GLS1
La expresión del gen GLS1 de C. truncatum se indujo fuertemente de forma temprana durante las primeras horas de contacto con el hospedante, siendo las 2 hdi la etapa con mayor expresión (58 veces más con respecto al control), después disminuyó su expresión de 16 a 96 hdi y finalmente, volvió a sufrir un aumento en la expresión a las 120 hdi (38 veces más) durante el estado patogénico, con respecto al control (Figura 4).
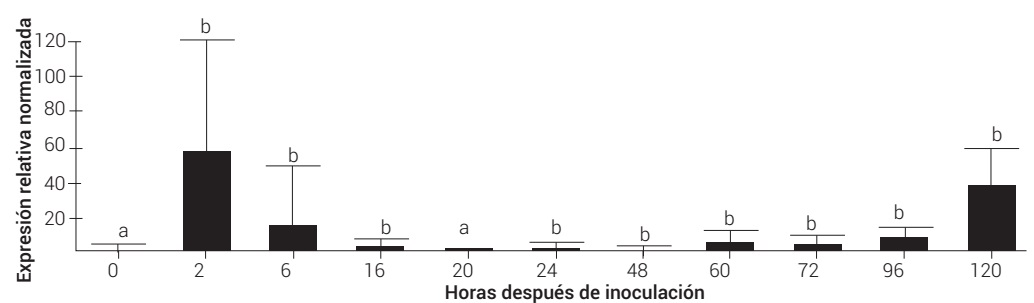
Figura 4. Expresión relativa del gen GLS1 de C. truncatum a diferentes horas después de inoculación con respecto al control (0 hdi). Expresión relativa normalizada con el gen endógeno Tef. Letras diferentes en cada barra indican diferencias estadísticas (P ≤ 0.05).
La expresión del gen GLS1 en hojas de papaya Maradol inició en etapa temprana, recuperó los niveles normales y se incrementó a las 120 hdi, lo que indica que la síntesis de β-1-3 glucanos se inhibió en etapas intermedias de la patogénesis y se activó días después de la infección inicial. En este estudio se detectó la expresión relativa del gen GLS1 a sólo 2 h de contacto con su hospedante. En contraste, Oliveira-García y Deising (2013) detectaron en maíz la expresión del gen GLS1 de C. graminicola hasta las 12 hdi, atribuyendo su expresión a la formación de estructuras como apresorios e hifas de infección.
En este estudio, la expresión de GLS1 observada durante las últimas horas de infección (120 hdi) sugiere que el patógeno también requiere de la producción de β-1-3 glucanos durante el estado necrótrofo, probablemente para la producción de estructuras de infección como hifas de infección. De acuerdo con Rojo-Báez et al. (2016), durante el estado necrótrofo del proceso de infección de C. truncatum en hojas de papaya Maradol, se observó la colonización del patógeno mediante hifas secundarias de infección, así como la formación de conidios dentro de acérvulos de las 96 a las 148 hdi. Los resultados de este estudio coinciden con ese proceso, ya que se indujo la expresión el gen GLS1 de manera significativa entre las 96 y 120 hdi, coincidiendo con la etapa necrótrofa del proceso de patogénesis.
Expresión del gen CHS1
La expresión del gen CHS1 de C. truncatum se indujo de forma significativa durante las primeras horas de infección (2-6 hdi), 4.5 y 2.2 veces más con respecto al control (0 hdi), respectivamente; a partir de las 16 hdi no se encontró inducción de la expresión con respecto al control (Figura 5).

Figura 5. Expresión relativa del gen CHS1 de C. truncatum a diferentes horas después de inoculación con respecto al control (0 hdi). Expresión relativa normalizada con el gen endógeno Tef. Letras diferentes en cada barra indican diferencias estadísticas (P ≤ 0.05).
El gen CHS1 usualmente se utiliza como marcador molecular para la identificación de especies de Colletotrichum (Hyde et al., 2014); sin embargo, también se ha relacionado con patogenicidad en especies fúngicas. En este estudio, la expresión del gen CHS1 de C. truncatum en hojas de papaya Maradol se indujo en su máxima expresión a las 2 hdi; ésto puede deberse a que la quitina, como componente estructural del hongo, participa en los eventos de morfogénesis de las estructuras de infección durante el proceso de patógenesis. De acuerdo con Kong et al. (2012), el gen CHS1 de Magnaporthe oryzae se expresó durante los eventos iniciales de la infección en hojas de arroz, destacando la importancia de la expresión del gen CHS1 en la conidiogénesis como evento esencial del ciclo natural de infección.
En contraste con los resultados aquí obtenidos, Werner et al. (2007) reportaron la inducción del gen CHS1 de C. graminicola a las 48 hdi (etapa de formación de apresorios) y 72 hdi (estado necrótrofo) durante el proceso de infección en hoja de maíz, siendo mayor la expresión durante el estado necrótrofo. En este estudio la expresión del gen CHS1 disminuyó conforme avanzaba la infección, siendo importante su expresión durante las primeras horas de contacto con el hospedante.
CONCLUSIONES
Los niveles de expresión de los genes CUT1, GLS1 y CHS1 de C. truncatum se indujeron diferencialmente durante su interacción curso temporal con hojas de papaya Maradol. La expresión del gen CUT1 fue incrementando hasta llegar a su máxima expresión durante la etapa tardía del proceso de infección; además, el gen GLS1 tuvo su máxima expresión durante la etapa temprana y tardía; por su parte, el gen CHS1 aumentó su expresión al inicio del proceso de patogénesis. Esta expresión génica es congruente con la producción de estructuras de infección y desarrollo del ciclo de vida del patógeno previamente descritos.











 nueva página del texto (beta)
nueva página del texto (beta)


