Introducción
La transición de flor a fruto (TFF) es un proceso crítico en la evolución y desarrollo de la mayoría de las angiospermas. Dicha transición está definida bajo un modelo general que considera dos eventos que ocurren de manera simultánea: (1) La polinización, que es el contacto del polen con el estigma de una flor y (2) la fecundación, que es la unión de los gametos para la formación de un embrión (Lord y Russell, 2002). Bajo este supuesto, se considera que la estructura del óvulo y el saco embrionario se han desarrollado completamente cuando los granos de polen germinan en el estigma, de modo que el tubo polínico se alarga y algunas horas después de la polinización ocurre la unión de las células espermáticas con la ovocélula, es decir, la fecundación (Lord y Russell, 2002).
Sin embargo, algunos grupos de los órdenes Fagales y Asparagales presentan una serie de modificaciones, tanto estructurales como funcionales, en el perianto y el ovario durante la TFF, que se conocen como síndrome post-polinización (SPP) (O’Neill y Nadeau, 1997). La familia Orchidaceae es uno de los grupos más representativos del SPP, en donde en la mayoría de las especies el desarrollo de gametófitos femeninos está ausente o incompleto antes de la polinización. En estos casos, el contacto del polen con el estigma es la señal que activa el mecanismo hormonal que permite el desarrollo de los óvulos de manera sincrónica a la elongación del tubo polínico hasta su fecundación, la cual puede ocurrir entre 20 y 70ddp (Chen y Fang, 2016; O’Neill y Nadeau,1997). Dicho sistema de fecundación es altamente sensible a cambios ambientales a través de la modificación de las señales hormonales (principalmente de auxinas y etileno), que sincronizan el desarrollo de los óvulos y el crecimiento de los tubos polínicos (Chen y Fang, 2016; O’Neill, 1997). Se sabe que el ácido indolacético (AIA) tiene un papel central en los procesos de polinización, fecundación, inducción y maduración del fruto (Pattison et al., 2014). A través de las fluctuaciones en la concentración de la hormona se regula el encendido y apagado de genes correspondientes a las familias: Factores de Respuesta a Auxinas (ARF), Auxina/Ácido indol-3- acético (Aux/IAA), así como las Proteína de Respuesta de Inhibición de Transporte 1 (TIR1) (Jayasinghege et al., 2019; Li et al., 2016).
En especies con sistema de fecundación modelo, como Arabidopsis thaliana L. Heynh (Brassicaceae), Cucurbita pepo L. (Cucurbitaceae), Musa acuminata Colla (Musaceae) y Solanum lycopersicum L. (Solanaceae), se ha planteado un modelo para la explicación de la TFF, que describe la relación entre los niveles de expresión de los genes ARF8, IAA25 y TIR1 y el comportamiento de las proteínas ARF8, IAA25 y TIR1 durante dos etapas del desarrollo: (1) la pre-polinización y (2) la polinización-fecundación (Calderon-Villalobos et al., 2010; Goetz et al., 2007; Hu et al., 2015; Pomares-Viciana et al., 2017; Wu et al., 2012).
Durante la pre-polinización se describe un ambiente de baja concentración de auxinas en el tejido del ovario, relacionado con la sobre-expresión de los genes ARF8, TIR1 y algunos Aux/IAA (Goetz et al., 2007; Pomares-Viciana et al., 2017; Wu et al., 2012); mientras que las proteínas ARF8 e IAA25 conforman un heterodímero que impide que ARF8 participe como factor de transcripción de los genes de respuesta a auxinas (Calderon-Villalobos et al., 2010). Por otra parte, el complejo de ubiquitinación SCFTIR1/AFB, que integra a los receptores nucleares TIR1/AFB con las proteínas ASK1, CUL1 y RBX, no está formado (Calderon-Villalobos et al., 2010).
Durante la polinización se describe un aumento en las concentraciones de auxinas en el ovario, lo cual disminuye los niveles de expresión de ARF8 y TIR1, mientras que la expresión de IAA25 se incrementa (Goetz et al., 2007; Jayasinghege et al., 2019; Wu et al., 2012). En este momento se forma el complejo de ubiquitinación SCFTIR1/AFB, TIR1 funciona como receptor de AIA, unión que permite acoplar a la proteína IAA25 al complejo y romper el heterodímero que formaba con ARF8, para que RBX1 coloque las marcas de ubiquitina sobre IAA25 necesarias para su posterior degradación vía proteasoma 26S (Calderon-Villalobos et al., 2010; Pomares-Viciana et al., 2017; Wu et al., 2012). Durante este proceso, la separación del heterodímero permite que ARF8 quede libre para participar como factor de transcripción de genes de respuesta a auxinas (Li et al., 2016).
Se ha observado que los niveles de expresión genética de ARF8, IAA25 y TIR1 están influenciados por diversas fuentes de variación, como el estado de desarrollo y la variación intraespecífica (Hu et al., 2015; Pomares-Viciana et al., 2017). En cuanto al estado de desarrollo, se ha identificado en especies como M. acuminata, S. lycopersicum y C. pepo que los niveles de expresión de los genes ARF8, IAA25 y TIR1 en tejidos del ovario presentan un patrón de sobrexpresión durante la pre-polinización (Hu et al., 2015; Pomares-Viciana et al., 2017; Wu et al., 2012). Esta sobreexpresión disminuye durante la polinización en M. acuminata, S. Lycopersicum, C. pepo y Pisum sativum L. (Fabaceae) (Cui et al., 2014; Goetz et al., 2006; Jayasinghege et al., 2019). Por esta razón, se ha sugerido que el comportamiento a lo largo del tiempo de los genes ARF8, IAA25 y TIR1 es determinante en el desarrollo y maduración de los óvulos, así como en el mecanismo de formación de los frutos (Calderon-Villalobos et al., 2010; Pomares-Viciana et al., 2017; Wu et al., 2012).
En cuanto al efecto de la variación intraespecífica, se ha demostrado que la expresión del gen ARF8 puede variar entre individuos de una misma especie, como se describe para C. pepo, donde se asocia la sobrexpresión de ARF8 con variedades no partenocárpicas y la subexpresión con variedades partenocárpicas (Pomares-Viciana et al., 2017). Además, en M. acuminata se ha relacionado la subexpresión de dicho gen con variedades caracterizadas por la alta calidad de producción y la sobrexpresión con variedades resistentes al estrés abiótico (Hu et al., 2015). Por otro lado, se ha demostrado que la expresión de TIR1 varía entre individuos partenocárpicos de distintos cultivares de C. sativus, por lo que se sugiere que existe un mecanismo distinto de formación del fruto entre individuos de una especie (Cui et al., 2014). En este sentido, se conoce con detalle que la TFF en plantas modelo responde a un mecanismo hormonal que involucra cambios en los niveles de expresión de las familias genéticas como ARF, Aux/IAA y TIR1; sin embargo, aún se desconoce su funcionamiento en plantas con síndrome post-polinización como las orquídeas (Li et al., 2016; O’Neill y Nadeau, 1997). Particularmente en Vanilla planifolia Andrews (Orchidaceae) se ha planteado que el metabolismo de auxinas está relacionado con la respuesta diferencial en la interrupción de la TFF (definida comercialmente como ’caída prematura de frutos’), entre el genotipo silvestre CH-I, considerado como ‘tolerante’ debido a las altas tasas de éxito reproductivo, y el genotipo comercial CH-VI, reconocido como ‘susceptible’ debido al bajo porcentaje de éxito en las flores polinizadas (Salazar-Rojas et al., 2016).
Por lo anterior, y dado que no se conocen con detalle los procesos moleculares que dirigen la TFF en organismos con SPP, se planteó como objetivo analizar los patrones de expresión diferencial de los genes ARF8, IAA25 y TIR1 durante los 45ddp, críticos para el desarrollo y maduración de las flores y estructuras reproductivas en dos genotipos contrastantes de Vanilla planifolia Andrews.
Materiales y métodos
Colecta del material biológico
Flores de los genotipos CH-I y CH-VI de V. planifolia se polinizaron manualmente y se colectaron durante siete momentos del desarrollo: 1) en el botón floral previo a la antesis (1dap), 2) en la antesis sin polinizar (As/p) y 3) polinizada (Ap), 4) en la flor polinizada un día después (1ddp), 5) 10 días después (10ddp), 6) 25 días después (25ddp) y 7) 45 días después (45ddp). El material biológico se almacenó en la solución de preservación RNAlater® (SIGMA) y/o en N2 líquido hasta su procesamiento.
Extracción de ARN total
El gineceo se separó y se trituró con N2 líquido. Posteriormente se realizó la extracción de ARN total, con TRIzol Reagent®, siguiendo las instrucciones del fabricante. Al ARN obtenido se le aplicó un tratamiento con ADNasa I, para eliminar remanentes de ADN genómico. El ARN se precipitó adicionando 20 µl de acetato de sodio 3 M y 60 µl de etanol frío al 100 % durante 12 h a -80 °C; posteriormente se removió el etanol y la pastilla se resuspendió en 20 µl de agua libre de nucleasas. Finalmente, se verificó la integridad del ARN total mediante electroforesis en gel de agarosa al 1.5 %.
Síntesis de ADNc
La reacción de síntesis de ADNc se realizó con la enzima SuperScript™ III Reverse Transcriptase de Invitrogen® y oligodT, de acuerdo con las especificaciones del fabricante, en un termociclador BIO-RAD T100 Thermal Cycler, con un ciclo inicial de 42 °C durante 60 min y 70 °C por 10 min.
qPCR
Para amplificar los genes ARF8, IAA25-like y TIR1 de V. planifolia se diseñaron pares de iniciadores (Cuadro 1) en el programa Geneious 9 ®, con una longitud de 21 pb, Tm de 60 °C, 50 % de GC y un producto de amplificación de 80-120 bases, a partir de secuencias de V. planifolia, disponibles en la base de datos de nucleótidos GenBank: VpARF8 (MK850820.1), VpIAA25-like (MK850819.1), VpTIR1 (MK850821) y VpACT (KF513175.1) (Benson et al., 2008). Los productos de amplificación se validaron mediante PCR de punto final y electroforesis en gel de agarosa al 1.2 %.
Cuadro 1 Iniciadores específicos para VpARF8, VpIAA25-like, VpTIR1 y VpACT con características para qPCR.
| Nombre del gen | Directo (5’-3’) | Reverso (5’-3’) | Accesión NCBI |
| VpARF8 | GAGCTAGGGCAGATGTTTGGT | GGTCGTCTCCAAGAAGAAGCA | (MK850820.1) |
| VpIAA25-like | TCTCCTCTACGAAGACCACGA | GCCTCTTGACCGAAGAAAGGA | (MK850819.1) |
| VpTIR1 | TCGATTACTGGCAAGGAGGATG | ATGTAGAGCTTCTCAACAGGGC | (MK850821) |
| VpACT | GGGTTACTCCTTTACGACCACA | GCTGCTCTTCGCTGTCTCAA | (KF513175.1) |
La reacción de qPCR se realizó mediante el kit Maxima SYBR Green/ROX qPCR Master Mix ajustado a 10 µl con 1 µl de cDNA por reacción, en el termociclador StepOne™ Real-Time PCR System con el siguiente programa: ciclo inicial de 95 °C durante 10 min, seguido por 45 ciclos de 95 °C durante 15 seg, 60 °C durante 1 min, 95 °C durante 15 seg, 60 °C durante 1 min y 95 °C durante 15 seg. La expresión genética se calculó por triplicado de acuerdo con la fórmula E = Peff (-ΔCt) (Pfaffl, 2004), donde Peff es la primera eficiencia calculada utilizando el programa LinRegPCR v3.0.0. Los niveles de expresión se indican como expresión relativa (ER) comparados con el gen control (House keeping) actina.
Análisis estadístico
Se consideró a las fases del desarrollo del fruto (siete fases) y el genotipo (dos genotipos) como fuentes de variación. Los datos para cada factor fueron analizados mediante un modelo equivalente a un diseño de dos factores completamente al azar balanceado. La comparación de medias entre genotipos, fases del desarrollo y la interacción entre genotipos por fase del desarrollo se realizó mediante una ANOVA de dos factores en el programa SAS (SAS, 2004). Además, se realizó un análisis de conglomerados tipo Heatmap, para conocer el perfil global de expresión relativa de los genes VpARF8, IAA25-like y VpTIR1 en las siete fases del desarrollo y en los dos genotipos (CH-I y CH-VI). Se trabajó en línea con la aplicación Heatmapper/Expression HeatMap mediante el método de medición de distancias euclidianas como medida de distancia y método de agrupamiento de enlace promedio (Babicki et al., 2016).
Resultados y discusión
Análisis de expresión diferencial de los genes VpARF8, VpIAA25-like y VpTIR1
Se observaron diferencias significativas en los niveles de expresión de VpIAA25-like únicamente entre genotipos, mientras que la expresión de los genes VpARF8 y VpTIR1 presentó diferencias significativas entre fases de desarrollo y en todos los genes analizados resultó significativa la interacción entre genotipos y fases de desarrollo (P <0.0001) (Cuadro 2).
Cuadro 2 Medias y coeficiente de variación de la expresión relativa (ER) de los genes VpARF8, VpIAA25-like y VpTIR1 de los genotipos CH-I y CH-VI de V. planifolia.
| Genes | Media (ER) | Coeficiente de variación | Cuadrados medios | ||
| Genotipo | Fase del desarrollo | Genotipo*Fase del desarrollo | |||
| VpARF8 | 0.413 | 60.989 | 0.020NS | 1.686*** | 1.314*** |
| VpIAA25-like | 1.220 | 12.069 | 0.497*** | 0.071NS | 0.307*** |
| VpTIR1 | 1.447 | 51.831 | 0.280NS | 3.368*** | 18.15*** |
NS: valores estadísticamente no significativos; ***: diferencia significativa (P < 0.0001).
Al respecto, se ha documentado que entre genotipos de C. pepo existe diferencia en los niveles de expresión de ARF8 y TIR1 (Pomares-Viciana et al., 2017). Mientras que la variación en la expresión del gen IAA25 se ha asociado principalmente con cambios durante la TFF (Wu et al., 2012). En V. planifolia se observó un patrón de expresión similar únicamente cuando se analiza la interacción entre genotipos y fases de desarrollo, debido a que cada genotipo presentó un patrón de expresión relativa distinto en cada etapa del desarrollo (Cuadro 2).
Perfil de expresión de los genes VpARF8, VpIAA25-like y VpTIR1
El agrupamiento superior presentó horizontalmente tres grupos (A), explicados a partir del patrón de expresión de los genes VpARF8, VpIAA25 y VpTIR1 (Figura 1). El grupo A1 integró el patrón más alto de expresión de los genes VpIAA25 y VpTIR1 observado en el genotipo CH-I (Figura 1). El grupo A2 describe un patrón de expresión bajo para el gen VpARF8 en el genotipo CH-I. El grupo A3 representó niveles de expresión medios-altos para los tres genes en genotipo CH-VI (Figura 1). El agrupamiento vertical integró tres perfiles de expresión (P), de acuerdo con los patrones de similitud de las fases de desarrollo (Figura 1). P1 agrupó a las fases del desarrollo 45ddp, 1ddp y 25ddp (Figura 1). P2 reunió como a un patrón de expresión genética las fases del desarrollo Ap, As/p y 10ddp. El perfil 3, correspondiente a 1dap, presentó un patrón de expresión único entre las distintas fases de desarrollo evaluadas, donde las diferencias más evidentes se registraron en el genotipo CH-I, el cual presentó los valores más bajos de expresión para los tres genes analizados.
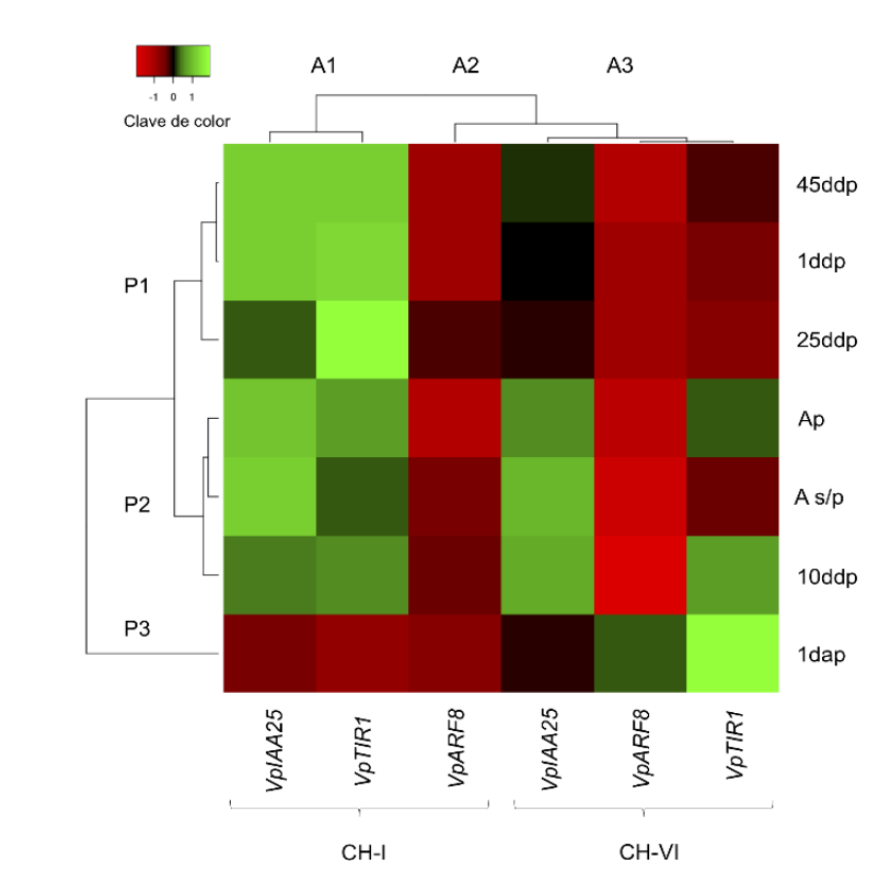
Figura 1 Dendrograma Heatmap de los niveles ER para los genes VpARF8, VpIAA25-like y VpTIR1 en dos genotipos de V. planifolia CH-I (‘tolerante’) y CH-VI (‘susceptible’), durante siete fases del desarrollo. Análisis realizado mediante el método de medición de distancias euclidianas y método de agrupamiento por enlace promedio. Fases de desarrollo: un día antes de la polinización (1dap), botón floral en antesis sin polinizar (As/p), botón floral en antesis después de ser polinizado (Ap), un día después de la polinización (1ddp), diez días después de la polinización (10ddp), 25 días después de la polinización (25ddp) y 45 días después de la polinización (45ddp).
A partir de la relación observada, entre agrupamientos y perfiles mediante el análisis de Heatmap se identificó, en ambos genotipos, un patrón de similitud en el nivel de expresión de los genes VpARF8, VpIAA25-like y VpTIR1, un día después de la polinización (1ddp) y durante los 45ddp. Particularmente, en el genotipo CH-I se observó que la regulación del complejo ARF8-IAA25-like-TIR1 es prácticamente igual en la etapa de polinización y en la de fecundación (Figura 1).
Las plantas con SPP, como la familia Orchidaceae, presentan un desfase temporal entre las etapas de polinización y fecundación (Mayer et al., 2011; O’Neill, 1997). Este desfase se confirmó en los ovarios de los dos genotipos de V. planifolia a partir de los perfiles de ER de VpARF8, VpIAA25-like y VpTIR1; así como de la observación de preparaciones histológicas (Figuras 2A1 y 2B1).
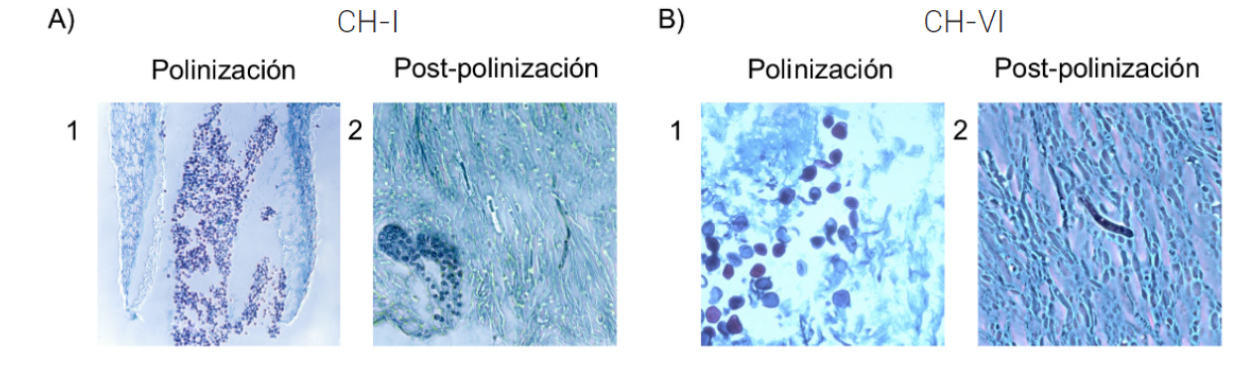
Figura 2 Preparaciones histológicas de la cavidad estigmática. Germinación de polinios y tubos polínicos durante la polinización y post-polinización de los dos genotipos: A) Genotipos CH-I en la etapa de polinización (1) y post-polinización (2). B) Genotipo CH-VI en la etapa de polinización (1) y post-polinización (2).
A partir de la descripción del genotipo CH-I se reconocieron cuatro fluctuaciones significativas en la expresión de los tres genes, que sugieren la existencia de cuatro etapas en el síndrome post-polinización: (1) pre-polinización, (2) polinización, (3) post-polinización y (4) fecundación (Figura 3):

Figura 3 Niveles de expresión relativa (ER) para los genes VpARF8, VpIAA25-like y VpTIR1 en los genotipos CH-I y CH-VI en diferentes fases del desarrollo: un día antes de la polinización (1dap), en la antesis sin polinizar (As/p), la antesis una vez polinizada la flor (Ap), un día después de la polinización (1ddp), diez días después de la polinización (10ddp), 25 días después de la polinización (25ddp) y 45 días después de la polinización (45ddp). En la figura se muestran cortes macroscópicos de ovario (2, 4 y 6), proyecciones placentarias (1 y 3), la célula madre de la megaespora (5 y B7) y estados iniciales de semilla (A7).
(1) Pre-polinización (1dap-As/p). En esta etapa no se identificaron óvulos formados, solamente proyecciones placentarias en estado inmaduro Figura 3A1), así como niveles marginales de expresión de los genes VpARF8, VpIAA25-like y VpTIR1 Figura 3A). Esta información contrasta con los trabajos de Goetz et al. (2007), Jayasinghege et al. (2019) y Wu et al. (2012), quienes mencionan que los niveles altos de transcritos de los genes ARF8, IAA25 y TIR1 son característicos de la etapa de pre-polinización en plantas modelo que presentan óvulos funcionales en el momento de antesis.
(2) Polinización (Ap-1ddp). Justo en el momento en que el polen toca el estigma se presentó una disminución a niveles basales de expresión de VpARF8 y un aumento de expresión en VpIAA25-like y VpTIR1, particularmente en 1ddp (Figura 3). Dicho patrón de expresión coincide con el comportamiento de los niveles de expresión de TIR1, IAA25 y ARF registrados durante la etapa de polinización en A. thaliana, Pisum sativum, S. lycopersicum, C. pepo y Cucumis sativus L. (Cui et al., 2014; Goetz et al., 2007; Jayasinghege et al., 2019; Pomares-Viciana et al., 2017; Wu et al., 2012). En esta etapa se observó la germinación de los polinios en el estigma de ambos genotipos (Figuras 2A1 y 2B1) y en la pared interna del ovario, además de un incremento en el número de proyecciones placentarias alargadas (Figura 3A3).
(3) Post-polinización (10ddp-25ddp). En esta etapa se distinguió un aumento en la expresión de VpARF8 y una disminución de VpIAA25-like y VpTIR1 durante los 10ddp, seguido de un incremento en la expresión de VpIAA25-like y VpTIR1 a los 25ddp Figura 3A). Durante esta etapa se observó el desarrollo de tubos polínicos y tejido de transmisión en el estigma (Figura 2A2), así como la proliferación de las proyecciones placentarias y la diferenciación de sus extremos apicales, donde se distingue claramente la célula madre de la megaespora (Figuras 3A5 y 3A6). Esto coincide con lo descrito para Pisum sativum por Jayasinghege et al. (2019), quienes plantean que el aumento en la ER de TIR1 es característico de los momentos previos a la fecundación.
(4) Fecundación (45ddp). En esta etapa se registró una disminución en la expresión de VpARF8 y VpTIR1, al mismo tiempo que se observó un incremento en la expresión de VpIAA25-like ((Fig. 3A. Además, las paredes internas del ovario se han diferenciado completamente y se distinguen estados iniciales de semillas (Figura 3A7). Al respecto, se ha descrito previamente en C. pepo, A. thaliana y S. lycopersicum que existe una disminución en la expresión de ARF8 y TIR1, justo cuando comienza el crecimiento y desarrollo del fruto (Goetz et al., 2006; Pomares-Viciana et al., 2017).
En el caso del genotipo CH-VI se observó un patrón de expresión contrastante respecto al del genotipo CH-I. En éste se distinguió que en la fase de pre-polinización el genotipo CH-VI inicia con niveles de expresión marcadamente altos de TIR1 en 1dap y después de la polinización se identifica un patrón de disminución en los tres genes hasta los 45ddp (Figura 3B). Este comportamiento también se percibe en el tejido interno del ovario, donde después de la polinización inicia la proliferación de las proyecciones placentarias, pero su diferenciación se realiza de forma lenta y progresiva (Figura 3B1 y Figura 3B3). Posteriormente se incrementa la cantidad de células madre de la megaespora y se observa la presencia de tejido de transmisión y tubos polínicos; sin embargo, los óvulos aún no están diferenciados (Figura 3B5), lo cual sugiere que hasta los 45ddp el tejido del genotipo CH-VI se mantiene aún dentro de la fase post-polinización.
Modelo de expresión de los genes VpARF8, VpIAA25-like y VpTIR1 durante la transición de flor a fruto en V. planifolia
A partir de la integración de los resultados observados en los dos genotipos de V. planifolia respecto a la fluctuación en la expresión de los genes VpARF8, VpIAA25-like y VpTIR1 y del conjunto de señales que inducen la formación del fruto en especies modelo con óvulos formados en el momento de antesis (Calderon-Villalobos et al., 2010; Goetz et al., 2007; Pomares-Viciana et al., 2017; Wu et al., 2012) se plantea la siguiente propuesta para explicar el proceso de TFF en condiciones normales de desarrollo durante el SPP de V. planifolia:
1) La primera etapa corresponde a pre-polinización (1dap-As/p), donde las altas concentraciones de VpARF8, VpIAA25-like y VpTIR1 sugieren la presencia de bajas concentraciones de auxinas en el botón floral, la heterodimerización de la proteína VpARF8 con VpIAA25-like, que impide la transcripción de genes de respuesta a auxinas y la inducción de óvulos, por lo que en esta etapa no existen óvulos formados (Figura 4A y Figura 3 A1) ((Goetz et al., 2006; Pomares-Viciana et al., 2017; Wu et al., 2012).

Figura 4 Modelo de transición de flor a fruto en V. planifolia basado en los resultados obtenidos en el genotipo CH-I y en los trabajos de Calderon-Villalobos et al. (2010), Goetz et al. (2007) y Pandolfini et al. (2007). A) Pre-polinización: 1dap-As/p. B) Polinización: Ap-1ddp. C) Post-polinización: 10ddp-25ddp. D) Fecundación: 45ddp.
2) Cuando ocurre la polinización aumenta la concentración de auxinas exógenas que provienen del polen (O’Neill, 1997), lo cual disminuye la expresión de transcritos de VpARF8 y aumenta la expresión de VpIAA25-like y VpTIR1. Durante antesis y el día posterior a la polinización, el complejo SCFTIR1/AFB recluta a la proteína VpIAA25-like uniéndose al receptor VpTIR1, mientras que RBX1 podría ubiquitinar a la proteína VpIAA25-like para su degradación vía proteosoma 26S, lo cual permite que la proteína VpARF8 quede libre y active los genes de inducción, desarrollo y maduración de óvulos (Figura 4B) (Calderon-Villalobos et al., 2010). Posteriormente, durante la etapa de post-polinización disminuye la concentración de auxinas en el tejido, aumenta la expresión de VpARF8, mientras disminuye la expresión de VpTIR1 y VpIAA25-like (Figura 4C) y la heterodimerización de las proteínas VpARF8 con VpIAA25-like impiden la transcripción de genes de inducción para el desarrollo de fruto (Figura 4C) (Calderon-Villalobos et al., 2010; Goetz et al., 2006).
3) Finalmente, durante la etapa de fecundación se incrementa la concentración de auxinas endógenas (por biosíntesis en óvulos maduros) y exógenas (provenientes del polen) (Goetz et al., 2007; Sundberg y Østergaard, 2009). Posteriormente se fertilizan los óvulos y con ello disminuye la expresión de VpARF8, se aumentan los niveles de VpIAA25-like y VpTIR1 alcanza su mayor expresión y disminuye hacia los 45ddp (Figura 4D). Por esto se asume que el complejo SCFTIR1/AFB recluta a la proteína VpIAA25-like uniéndose al receptor VpTIR1, mientras que RBX1 podría ubiquitinar a la proteína VpIAA25-like para su degradación vía proteosoma 26S, lo cual permite que la proteína VpARF8 quede libre y active los genes de inducción, desarrollo y maduración de frutos (Calderon-Villalobos et al., 2010; Goetz et al., 2007) (Figura 4D).
Conclusiones
La expresión temporal de los genes VpARF8, VpIAA25-like y VpTIR1 presentó un efecto genotipo dependiente durante el síndrome post-polinización en V. planifolia. El genotipo CH-I ('tolerante a caída de fruto') presentó cambios en los niveles de expresión de los tres genes durante las etapas del desarrollo, tuvo un desarrollo acelerado de las proyecciones placentarias que derivó en la conformación inicial de la semilla. El genotipo CH-VI ('susceptible') mantuvo niveles bajos de expresión y un desarrollo lento del ovario. En vainilla, lo que comercialmente se reconoce como caída prematura de frutos corresponde a ‘caída prematura de flor’.











 nueva página del texto (beta)
nueva página del texto (beta)


