INTRODUCCIÓN
Xylella fastidiosa es un grupo heterogéneo de bacterias Gram negativas, limitadas al xilema de las plantas, con capacidad de infectar diversas especies de plantas herbáceas, arbustivas o leñosas, cultivadas o adventicias (EFSA, 2018; Marco-Noales et al., 2017) y de ocasionar numerosas enfermedades, entre las que destacan la enfermedad de Pierce en vid (Vitis vinifera), phony en durazno (Prunus persica), clorosis variegada de los cítricos en naranjo dulce (Citrus sinensis), decaimiento súbito del olivo en olivo (Olea europaea) y quemadura de la hoja de la rosa laurel (Nerium oleander) (Baldi y La Porta, 2017). Aunque algunas de las especies de plantas desarrollan síntomas y otras no, todas ellas son de importancia por el papel que juegan en la epidemiología de las enfermedades que ocasiona la bacteria (IPPC, 2018).
Actualmente, con base en el tipo de secuencia (Sequence Type, ST) obtenido mediante el genotipado de secuencias multilocus (Multilocus Sequence Typing, MLST) (Yuan et al., 2010), los aislados de X. fastidiosa se clasifican en las subespecies X. fastidiosa subsp. fastidiosa, X. fastidiosa subsp. multiplex, X. fastidiosa subsp. pauca y X. fastidiosa subsp. sandyi (Baldi y La Porta, 2017), pero independientemente del genotipo, X. fastidiosa se transmite de forma persistente por insectos vectores especializados considerados como los principales responsables de la dispersión natural de la bacteria (Morente y Fereres, 2017).
En 2005 se detectó por primera vez la presencia de X. fastidiosa en el municipio de Parras, Coahuila, por lo que se ha implementado la Campaña Nacional Contra la Enfermedad de Pierce para el manejo de la enfermedad y sus vectores, así como medidas estratégicas para reducir y evitar la dispersión de la bacteria hacia zonas libres de la enfermedad (SENASICA, 2019); sin embargo, a pesar de que durante el periodo de 2016 a 2018 en el municipio de Parras se registró un aumento en la superficie sembrada con un incremento del 64 % en la producción, el rendimiento por unidad de superficie mostró una reducción del 17 % respecto a lo registrado en los seis años anteriores a este periodo (SIAP, 2019).
Lo anterior indica que aún con la implementación de la campaña, es posible que la enfermedad de Pierce continúe afectando los rendimientos por unidad de superficie de la producción de uva en el estado (SENASICA, 2017; Com. Pers.)1, debido a que X. fastidiosa es un factor limitante para la producción de vid y que las zonas donde se siembra este cultivo están rodeadas por árboles frutales y forestales, además de plantas silvestres que podrían ser hospedantes de la bacteria. Por lo tanto, el objetivo de la presente investigación fue identificar la subespecie de la bacteria presente en Parras, Coahuila, y determinar su asociación con diferentes especies de plantas cultivadas y silvestres, a fin de contribuir al entendimiento de la epidemiología y manejo de las enfermedades ocasionadas por X. fastidiosa en el municipio.
MATERIALES Y MÉTODOS
Recolecta de muestras
Las muestras de plantas cultivadas y silvestres se recolectaron en los meses de junio, agosto, septiembre y octubre de 2018 en los predios La Encantada (4 ha) y San Lorenzo (10 ha) del municipio de Parras, Coahuila (25° 26’ 13’’ latitud N, 102° 11’ 01’’ longitud O). Se obtuvieron 83 muestras correspondientes a diferentes géneros y especies de árboles frutales (n = 36), que incluyeron vid, árboles forestales (n = 4), plantas ornamentales (n = 2) y plantas silvestres (n = 41). Se muestrearon secciones de tallo con cinco a 20 hojas de plantas de vid durante la etapa de fructificación, de cinco a 10 hojas con peciolo de árboles frutales y forestales y de plantas ornamentales.
De plantas silvestres se obtuvieron ejemplares completos en etapa vegetativa y floración, ubicadas entre las hileras o adyacentes a los viñedos o cercanas a cuerpos de agua. Algunas de las muestras presentaban síntomas de clorosis, marchitez o escaldadura (quemadura de tejido). Estas muestras se colocaron entre toallas de papel secante, se guardaron en bolsas de plástico con cierre hermético y se almacenaron en frío hasta su análisis en el laboratorio. La identificación de plantas silvestres se hizo tomando como referencia el catálogo de Malezas de México (http://www.conabio.gob.mx/malezasdemexico/2inicio/homemalezas-mexico.htm).
En el mes de agosto en el predio de San Lorenzo se instaló por 4 días una trampa de panel amarillo en la vegetación silvestre aledaña a un viñedo, donde se capturaron seis ejemplares de insectos para determinar la presencia de vectores de X. fastidiosa. La detección de la bacteria se realizó después de la identificación de los insectos con la clave taxonómica de Young (1968).
Detección de Xylella fastidiosa
Extracción de DNA total
La extracción se llevó a cabo a partir de peciolos y nervaduras centrales de cinco a 10 hojas de frutales, forestales y ornamentales; de 0.5 a 0.8 g de hojas de plantas silvestres y de la cabeza (sin ojos) de insectos (Loconsole et al., 2014). El tejido se colocó en morteros estériles y se maceró con nitrógeno líquido hasta obtener un polvo fino. Se depositaron 0.5 g del macerado en un tubo estéril (2.0 mL, Eppendorf®), se adicionaron 1.5 mL de CTAB 2 % (20.0 g de bromuro de hexadecil-trimetil-amonio, 82.0 g de NaCl, 2.42 g de tris base, 1.46 g de EDTA en 1 L de agua destilada) y 100 µL de dodecil sulfato sódico 10 %, se mezcló en un vórtex por 60 s, se incubó a 80 °C por 20 min y 5 min a temperatura ambiente y se centrifugó a 13,000 × g por 15 min. Posteriormente, 1 mL del extracto se transfirió a un tubo estéril (2.0 mL, Eppendorf®), se adicionaron 500 µL de una mezcla de fenol:cloroformo:alcohol isoamílico (25:24:1, Sigma-Aldrich®), se mezcló por inversión y se centrifugó a 13,000 × g por 15 min. Se transfirieron 600 µL del sobrenadante a un tubo estéril y se adicionó medio volumen de etanol:metanol:ácido acético glacial (9:3:1, Sigma-Aldrich®), se mezcló por inversión y se centrifugó a 13,000 × g por 10 min.
El DNA se precipitó con 500 μL de una mezcla fría de etanol:agua:metanol:acetato de sodio 3 M (30:9:5:1, Sigma-Aldrich®) y 500 μL de etanol frío 70 % (SigmaAldrich®) y se centrifugó a 13,000 × g por 5 min. La pastilla de DNA se secó a temperatura ambiente por 1 h y se resuspendió en 50 μL de agua grado biología molecular (Sigma-Aldrich®). La pureza y cantidad del DNA se estimó por espectrofotometría en un Nanodrop 2000 (Thermo Scientific®, Waltham, Massachusetts, USA) previo a las amplificaciones por la reacción en cadena de la polimerasa (PCR).
PCR-punto final
Se amplificaron las secuencias parciales de los genes gyrB y HL con los iniciadores XYgyr499/XYgyr907 y HL5/ HL6, respectivamente (Francis et al., 2006; Rodrigues et al., 2003). La mezcla de reacción se preparó en un volumen final de 25 µL con 2.5 µL de buffer de PCR 10X, 0.75 µL de MgCl2 (50 mM), 0.5 µL de mezcla de dNTP´s (10 mM) 1 µL por iniciador (10 µM), 0.3 µL de Taq DNA polimerasa (5 U µL-1, Invitrogen®, USA), 5 µL de DNA (100 ng µL-1) y 13.95 μL de agua. Para los controles se prepararon mezclas con DNA de la cepa ATCC® 35879TM de X. fastidiosa subsp. fastidiosa para el positivo y para los negativos se utilizó el DNA de una planta de vid sana y otro con agua (sin DNA molde).
Los productos de amplificación se separaron por electroforesis en geles de agarosa 1.5 %, se consideró una muestra positiva cuando la banda de DNA del tamaño esperado se visualizó en un fotodocumentador (Gel Doc EZ Imager, BioRad®, Hercules, California, EUA). Los amplicones correspondientes a los genes gyrB (429 pares de bases, pb) y HL (221 pb) se secuenciaron en un equipo Genetic Analyzer 3130 (Applied Biosystems®, Foster City, California, EUA) mediante la química BigDye Terminator v 3.1. Las secuencias sentido (F) y antisentido (R) se analizaron y editaron con el programa BioEdit® v 7.0.9.0 (Hall, 1999). Las secuencias consenso se compararon con las secuencias depositadas en el GenBank NCBI (National Center for Biotechnology Information) con el programa BLASTn (Basic Local Alignment Search Tool nucleotide, https://blast.ncbi.nlm.nih.gov/Blast.cgi).
PCR-tiempo real
Se amplificó la secuencia parcial del gen 16S rRNA con los iniciadores XF-F/XF-R y la sonda XF-P Taqman (Harper et al., 2010). La mezcla de reacción se preparó en un volumen final de 22 µL que consistió en 2.2 µL de buffer de PCR 10×, 1.76 µL de MgCl2 (50 mM), 0.6 µL de mezcla de dNTP´s (10 mM), 0.6 µL por iniciador (10 µM), 0.4 µL de Taq DNA polimerasa (5 U µL-1), 0.1 µL de sonda (10 µM), 4 µL de DNA (80 ng µL-1) y 11.64 µL de agua grado biología molecular. Para los controles se prepararon mezclas como se indicó anteriormente. La reacción se estableció en un sistema de PCR-tiempo real CFX96TM (BioRad®, Hercules, California, EUA). Se consideró una muestra positiva cuando la fluorescencia alcanzó valores de Cq < 35.00.
Identificación de la subespecie de X. fastidiosa
Se amplificaron secuencias parciales de los genes MLST de mantenimiento: leuA, petC, malF, cysG, holC, nuoL, gltT y del gen de adherencia a superficies pilU (Harris y Balci, 2015; Yuan et al., 2010). La mezcla de reacción se preparó en un volumen final de 25 µL y consistió en 2.5 µL de buffer de PCR 10X, 0.75 µL de MgCl2 (50 mM), 0.5 µL de mezcla de dNTP´s (10 mM), 1 µL por iniciador (10 µM), 0.3 µL de Taq DNA polimerasa (5 U µL-1), 5 µL de DNA y 13.95 μL de agua grado biología molecular. Las bandas de DNA esperadas se visualizaron en un fotodocumentador como se indicó anteriormente.
Para obtener el perfil alélico se secuenció el fragmento amplificado de una muestra positiva de Baccharis sp., Carya sp., Celtis pallida, Cydonia sp., Ficus carica, Olea europaea, Phragmites sp., Rubus sp., Salsola kali, Tribullus terrestris y un pasto (familia Poaceae), así como de tres muestras de V. vinifera y el fragmento de un insecto positivo. Las secuencias F y R de cada gen se analizaron, editaron y ensamblaron con el programa BioEdit® v. 7.0.9.0 (Hall, 1999). Las secuencias consenso se compararon con las secuencias de las subespecies de X. fastidiosa de la base de datos Xylella fastidiosa PubMLST Database (https://pubmlst.org/xfastidiosa/). A cada alelo de un gen se le asignó un número y con la combinación de números generada se determinó el tipo de secuencia (ST).
Análisis filogenético
Se elaboraron dos árboles filogenéticos de X. fastidiosa con el método de máxima verosimilitud. Para el primer árbol se utilizaron 30 secuencias concatenadas de los siete genes MLST, de acuerdo con el orden indicado en Xylella fastidiosa PubMLST Database; para ésto, las secuencias se editaron previamente y se alinearon con el ClustalW incluido en el programa Molecular Evolutionary Genetics Analysis (MEGA 6) (Tamura et al., 2013). El segundo árbol se construyó con 12 secuencias de X. fastidiosa subsp. fastidiosa correspondientes al gen pilU; posteriormente, se determinó la confiabilidad de los clados con 1000 repeticiones bootstraps (Tamura, 1992). En ambos árboles se utilizó a X. taiwanensis como grupo externo.
RESULTADOS Y DISCUSIÓN
Ocurrencia de X. fastidiosa
De 83 muestras en estudio, se detectaron 22 positivas a X. fastidiosa con los iniciadores XYgyr499/XYgyr907 y HL5/HL6. En los frutales de Carya sp. (nogal), Cydonia sp. (membrillo), F. carica (higuera), O. europaea (olivo) y V. vinifera (vid) se identificaron por secuenciación 14 muestras positivas, y en las especies silvestres de Baccharis sp. (jarilla), C. pallida (acebuche), Phragmites sp. (carrizo gigante), Rubus sp. (mora silvestre), S. kali. (rodadera), T. terrestris (torito) y un pasto (familia Poaceae) se identificaron ocho muestras positivas para la bacteria. Del total de muestras sólo 12 resultaron positivas a X. fastidiosa cuando se analizaron con los iniciadores XF-F/XF-R y la sonda XF-P Taqman. Estas muestras correspondieron a Carya sp., Cydonia sp., F. carica, Phragmites sp. y V. vinifera (Cuadros 1 y 2). En el caso de los insectos capturados pertenecientes a especies de Aphis sp. (n = 1), Chrysoperla sp. (n = 2), Empoasca sp. (n = 1) y Homalodisca sp. (n = 2) se detectó con los iniciadores XYgyr499/XYgyr907, HL5/HL6, XF-F/XF-R y la sonda XF-P Taqman un solo insecto de Homalodisca positivo a X. fastidiosa (Cuadro 1). Las especies del género Homalodisca se han considerado entre los principales vectores de X. fastidiosa (Morente y Fereres, 2017), por lo que estudios futuros que incluyan mayor número de colectas (espacio-temporal) son necesarios para dilucidar el papel que pudieran tener insectos de este género en la dispersión de X. fastidiosa en la región.
Cuadro 1 Detección por PCR de Xylella fastidiosa en plantas cultivadas y silvestres e insectos en el predio San Lorenzo, municipio de Parras, Coahuila.
| Especie | Muestra (M)† | Iniciadores†† | ||
|---|---|---|---|---|
| XYgyr499/907 | HL5/HL6 | XF-F/XF-R | ||
| Salsola kali | M3 | + | + | - |
| Celtis pallida | M2 | + | + | - |
| Pasto Poaceae¶ | M55 | + | + | - |
| Tribullus terrestris | M57 | + | + | - |
| Rubus sp. | M11, M63 | + | + | - |
| Aphis sp. | I’5 | - | - | - |
| Empoasca sp. | I’6 | - | - | - |
| Homalodisca sp. | I’1, I’2 | + / - | + / - | + / - |
| Chrysoperla sp. | I’3, I’4 | - | - | - |
| Vitis vinifera | M52 | + | + | + |
| Vitis vinifera | M53, M54 | - | - | - |
| Pasto Apocynaceae | M51 | - | - | - |
| Vitis vinifera | M50 | + | + | + |
| Vitis vinifera | M56, M58 | - | ||
| Vitis sp. | De M74 a M83 | - | - | - |
| Carya sp. | M60, M61 | + / - | + / - | + / - |
| Lepidium sp. | M59 | - | - | - |
| Setaria sp. | M64 | - | - | - |
| Vitis vinifera | M71, M72, M73 | - | - | - |
| Verbena sp. | M69 | - | ||
| Vitis vinifera | M65, M66, M67 | + | + | + |
| Vitis vinifera | M68 | - | - | - |
| Lagerstroemia indica | M62 | - | - | - |
| Lavandula sp. | M5 | - | - | - |
| Vitis vinifera | M1, M14, M15 | - | - | - |
| Vitis sp. | M6, M7 | - | - | - |
| Malva sylvestris | M4 | - | - | - |
| Vitis vinifera | M20, M24 | - | - | - |
| Pasto (Poaceae) | M38, M39, M41 | - | - | - |
| Taraxacum officinale | M37 | - | - | - |
| Malva sylvestris | M21 | - | - | - |
| Portulaca sp. | M36 | - | - | - |
| Setaria sp. | M40 | - | - | - |
| Eucalyptus sp. | M23, M42 | - | - | - |
| Olea europaea | M10, M12, M16, M17 | + | + | + |
| Olea europaea | M13 | - | - | - |
| Cynodon sp. | M25 | - | - | - |
†Muestra organizada en orden ascendente de acuerdo con su posición en el predio (georreferenciación de 25.48440 a 25.51815 N, y de 102.17508 a 102.20222 O), ††Muestras positiva por iniciador (+, en negrita) o negativa (-), ¶ Familia.
Cuadro 2 Detección por PCR de Xylella fastidiosa en plantas cultivadas y silvestres en el predio La Encantada, municipio de Parras, Coahuila.
| Especie | Muestra (M)† | Iniciadores†† | ||
|---|---|---|---|---|
| XYgyr499/907 | HL5/HL6 | XF-F/XF-R | ||
| Baccharis sp. | M10’A | + | + | - |
| Cyperus sp. | M6’A | - | - | - |
| Cynodon sp. | M1’A, M7’A | - | - | - |
| Phragmites sp. | M3’A | + | + | + |
| Pasto (Poaceae)¶ | M2’A, M4’A, M5’A | - | - | - |
| Rumex sp. | M8’A, M9’A | - | - | - |
| Cupressus sp. | M18, M19 | - | - | - |
| Cydonia sp. | M2, M70 | + | + | + |
| Malus domestica | M9, M44, M45 | - | - | - |
| Prunus armeniaca | M46, M47 | - | - | - |
| Persea americana | M8 | - | - | - |
| Populus sp. | M43 | - | - | - |
| Ficus carica | M48, M49 | + | + | + |
†Muestra organizada en orden ascendente de acuerdo con su posición en el predio (georreferenciación de 25.48751 a 25.49183 N, y de 102.17304 a 102.17658 O), ††Muestras positiva por iniciador (+, en negrita), o negativa (-), ¶ Familia
Las secuencias de los productos de amplificación de las muestras positivas de plantas y las del insecto Homalodisca sp. se alinearon con las secuencias de los genes gyrB y HL de X. fastidiosa CP006740.1 y CP002165, respectivamente, con un porcentaje de identidad mayor a 97 %, excepto las de las muestras de Cydonia sp. (M2), F. carica (M48 y M49) y Rubus sp. (M11 y M63) (Cuadro 4). Es posible que el gen HL de estas muestras no estuviera bien amplificado o secuenciado (Baldi y La Porta, 2017).
En este estudio se registró casi el doble de muestras de especies frutales (39 %) que de plantas silvestres (20 %) positivas a X. fastidiosa, lo que sugiere que la presencia de la bacteria en el municipio de Parras, Coahuila puede deberse a introducciones de material propagativo de frutales infectados provenientes de otras regiones. En México, la mayor parte del material propagativo de V. vinifera proviene de los Estados Unidos de América, país que reporta desde 1880 la presencia de X. fastidiosa (Nunney et al., 2010) y con quien se tiene el mayor intercambio de este material vegetal. El movimiento de material propagativo mediado por personas es uno de los principales factores responsables de la introducción de X. fastidiosa en áreas libres de la bacteria y de su aparición en nuevas especies de plantas hospedantes (Sicard et al., 2018).
Se observaron síntomas similares a los ocasionados por X. fastidiosa (Appel et al., 2010) sólo en las muestras de las siguientes especies: en Cydonia sp. y Phragmites sp. se observó escaldadura en el ápice de las hojas, y en O. europaea, escaldadura en el ápice de las hojas y marchitez en las ramas, similares al síndrome de decaimiento súbito del olivo (Loconsole et al., 2014) (Figura 1). En V. vinifera se detectaron plantas con clorosis, marchitez y defoliación prematura, escaldadura de la hoja, pasificación de frutos, síntoma de cerillo e islas verdes. Estos resultados indican que los viñedos están rodeados por especies de árboles perennes y de plantas silvestres infectadas, a partir de las cuales los insectos vectores pueden adquirir a X. fastidiosa, debido a que la dispersión de la bacteria generalmente se origina desde fuentes externas que sirven como alimento a los insectos vectores hacia el interior de los viñedos (Purcell y Saunders, 1999).
Con excepción de Phragmites sp., las plantas silvestres positivas a X. fastidiosa, pero con síntomas no visibles, pudieron estar actuando sólo como reservorios que contribuyen al mantenimiento y dispersión de la bacteria (Sicard et al., 2018). Los aislados de X. fastidiosa que ocasionan la enfermedad de Pierce en V. vinifera pueden también infectar a otras especies de plantas sin causar enfermedad debido a que en plantas con ausencia de síntomas las poblaciones de la bacteria declinan con el tiempo, tienen poca capacidad para multiplicarse y moverse sistémicamente en comparación con las de plantas sintomáticas (Purcell y Saunders, 1999); sin embargo, es necesario establecer estudios con los postulados de Koch para definir el papel de las plantas silvestres estudiadas en la región.
Por otro lado, las especies leñosas que permiten la reproducción de X. fastidiosa desarrollan síntomas típicos de enfermedad (Appel et al., 2010). En el caso de Cydonia sp. se observaron síntomas de escaldadura similares a los ocasionados por X. fastidiosa en otras especies vegetales (Harris et al., 2014), por lo que se propone a Cydonia como un hospedante de X. fastidiosa y candidata a integrarse en la lista de la Autoridad Europea de Seguridad Alimentaria (EFSA, 2018). Posiblemente, la ausencia de síntomas en Carya sp. y F. carica se deba a una baja reproducción de la bacteria en el tejido de estas plantas, o bien, se trata de infecciones latentes como ocurre en árboles de roble rojo (Quercus rubra) y roble escarlata (Q. palustris) que no desarrollan síntomas cuando son atacados por la bacteria (Harris et al., 2014).
Identificación de la subespecie
En general, las muestras positivas de frutales (n = 14) y silvestres (n = 8) y la del insecto Homalodisca sp., amplificadas y secuenciadas con los siete genes MLST y el gen pilU, correspondieron a X. fastidiosa subsp. fastidiosa (Cuadro 3). Las secuencias de las amplificaciones mostraron identidad con el alelo 1, por lo que se les agrupó en la secuencia tipo ST1, la cual está relacionada con X. fastidiosa subsp. fastidiosa de V. vinifera, originaria de los Estados Unidos de América. Con excepción de la secuencia de la muestra de F. carica, se registraron diferencias en las secuencias de los genes cysG y nuoL de las muestras cuando se compararon con las depositadas en la base de datos Xylella fastidiosa PubMLST Database (Cuadro 3). Las sustituciones encontradas en dichos genes proponen la aparición de nuevos alelos en los aislamientos de X. fastidiosa subsp. fastidiosa presentes en el municipio de Parras, tomando en cuenta que al existir cambios en alguna de las bases de la secuencia de algún locus se considera como un nuevo alelo (Scally et al., 2005). Estudios futuros que impliquen la obtención de cultivos axénicos de la bacteria y la secuenciación de genes en los que se observan variaciones son requeridos para confirmar la aparición de nuevas secuencias ST.
Cuadro 3 Perfil alélico de subespecie y secuencia tipo (ST) de Xylella fastidiosa presente en el municipio de Parras, Coahuila.
| Predio / Muestra† | Gen multilocus | Subespecie | ST | |||||||
|---|---|---|---|---|---|---|---|---|---|---|
| leuA | petC | malF | cysG†† | holC | nuoL†† | gltT | pilU | |||
| Predio San Lorenzo | ||||||||||
| Silvestres: Salsola kali, M3 | 1¶ | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | 1 | fastidiosa | 1 |
| Celtis pallida, M22 | 1 | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | 1 | fastidiosa | 1 |
| Pasto Poaceae, M55 | 1 | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | - | fastidiosa | 1 |
| Tribullus terrestris, M57 | 1 | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | - | fastidiosa | 1 |
| Rubus sp., M63 | 1 | 1 | 1 | 62C=G | 1 | 1 | 1 | 1 | fastidiosa | 1 |
| Insecto: Homalodisca sp., I’1 | 1 | 1 | 1 | 1 | 1 | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| Frutales: Vitis vinifera, M52 | 1 | 1 | 1 | 1 | - | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| M50 | 1 | 1 | 1 | 1 | 1 | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| Carya sp., M60 | 1 | 1 | 1 | 1 | - | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| Vitis vinifera, M65 | 1 | 1 | 1 | 1 | 1 | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| Olea europaea, M10 | 1 | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | 1 | fastidiosa | 1 |
| Predio La Encantada | ||||||||||
| Silvestres: Baccharis sp., M10’A | 1 | 1 | 1 | 62C=G | 1 | 495A=C 497G=A | 1 | - | fastidiosa | 1 |
| Phragmites sp., M3’A | 1 | 1 | 1 | 1 | - | 241G=A 460T=C | 1 | 1 | fastidiosa | 1 |
| Frutales: Cydonia sp., M70 | 1 | 1 | - | 1 | 1 | 1 | 1 | 1 | fastidiosa | 1 |
| Ficus carica, M48 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | fastidiosa | 1 |
†La muestra se presenta en orden ascendente de acuerdo con la posición geográfica en la que se tomó (georreferenciación de 25.48751 a 25.49183 N, y de 102.17304 a 102.17658 O). ††Gen con sustitución en alelo 1. ¶Alelo 1. El símbolo negativo (-) indica no amplificación de la banda esperada.
Análisis filogenético
El árbol filogenético de X. fastidiosa construido con las secuencias concatenadas de los siete genes MLST obtenidas en este estudio, que incluyen 30 ST, mostró que los aislamientos presentes en el municipio de Parras se agrupan con las secuencias ST1, ST2 y ST3, identificadas como X. fastidiosa subsp. fastidiosa (Yuan et al., 2010), lo que confirma la presencia de esta subespecie en el municipio (Figura 2). Similarmente, el árbol filogenético del gen pilU mostró la agrupación de todas las secuencias obtenidas en el presente estudio en un clado, el de la subespecie fastidiosa, y las secuencias de las subespecies pauca y multiplex, y la de X. taiwanensis en clados diferentes (Figura 3).
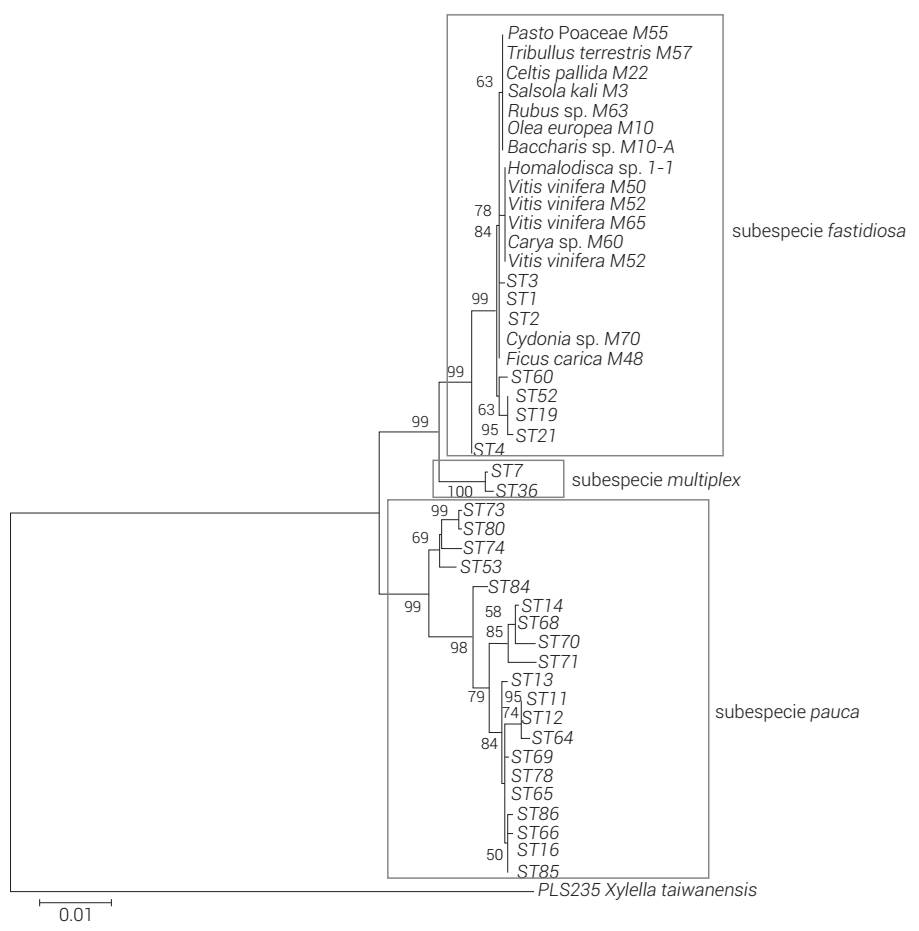
Figura 2 Árbol filogenético de Xylella fastidiosa construido con 30 secuencias Tipo (ST) con el método de máxima verosimilitud. El árbol se generó a partir de una matriz concatenada con siete genes MLST procedente de 15 muestras obtenidas del municipio de Parras, Coahuila. La barra de la escala indica el número de sustituciones por sitio.

Figura 3 Árbol filogenético de Xylella fastidiosa construido con el método de máxima verosimilitud con las secuencias del gen pilU de 12 muestras obtenidas en el municipio de Parras, Coahuila. La barra de la escala define el número de sustituciones por sitio. En rectángulo se indican las secuencias obtenidas en la presente investigación.
Las secuencias consenso se agruparon con la secuencia ST1 (alelo 1) del gen pilU del aislamiento PD0001 de X. fastidiosa subsp. fastidiosa. Así mismo, se determinó la presencia del alelo 1 del gen pilU en V. vinifera identificado previamente en plantaciones de vid infectadas con la bacteria en EUA (Yuan et al., 2010). Las diferencias entre las subespecies de X. fastidiosa están en concordancia con la variabilidad mostrada por la bacteria que tiene amplio espectro de hospedantes (Scally et al., 2005). Las secuencias de los genes de este estudio se depositaron en la base de datos del GenBank-NCBI con los números de accesión gyrB y HL indicados en el Cuadro 4; así mismo, las de los genes MLST y pilU con los números de accesión leuA, MT674718-MT674732; petC, MT674747-MT674761; malF, MT674733-MT674746; cysG, MT674691-MT674705; holC, MT674706-MT674717; nuoL, MT674762-MT674776; gltT, MT674789-MT674803 y pilU, MT674777-MT674788.
Cuadro 4 Porcentaje de máxima identidad (MI)† y número de accesión de secuencias obtenidas en el presente estudio.
| Hospedante | Muestra | Gen gyrB | Gen HL | ||
|---|---|---|---|---|---|
| % de MI | Número de Accesión | % de MI | Número de Accesión | ||
| Carya sp. | M60 | 98.38 | MN853301 | 99.54 | MN853308 |
| Cydonia sp. | M2 | 99.07 | MN735934 | 96.52 | MN755928 |
| M70 | 99.30 | MN755922 | 99.55 | MN853304 | |
| Ficus carica | M48 | 99.53 | MN735935 | 84.62 | MN853309 |
| M49 | 99.30 | MN755910 | 84.62 | MN853310 | |
| Olea europaea | M10 | 99.07 | MN755923 | 99.55 | MN755933 |
| M12 | 98.83 | MN755924 | 97.80 | MN853305 | |
| M16 | 98.83 | MN755925 | 99.53 | MN853306 | |
| Vitis vinifera | M50 | 99.53 | MN755915 | 98.82 | MN755932 |
| M52 | 99.53 | MN755916 | 100.00 | MN755931 | |
| M65 | 99.53 | MN755917 | 99.54 | MN853311 | |
| M66 | 99.53 | MN755913 | 99.53 | MN755930 | |
| M67 | 99.30 | MN755912 | 99.54 | MN755929 | |
| Baccharis sp. | M10’A | 99.06 | MN755914 | 99.54 | MN853312 |
| Celtis pallida | M22 | 97.92 | MN755919 | 98.65 | MN853313 |
| Phragmites sp. | M3’A | 99.53 | MN755921 | 99.05 | MN853315 |
| Pasto (Poaceae) | M55 | 97.46 | MN853302 | 98.23 | MN853314 |
| Rubus sp. | M11 | 97.26 | MN755908 | 96.92 | MN853316 |
| M63 | 99.53 | MN755909 | 96.91 | MN853317 | |
| Salsola kali | M3 | 99.07 | MN755927 | 98.65 | MN853318 |
| Tribullus terrestris | M57 | 99.07 | MN853303 | 99.54 | MN853319 |
| Homalodisca sp. | I’1 | 99.53 | MN755918 | 99.52 | MN853320 |
†Genes gyrB, y HL de X. fastidiosa CP006740.1 y CP002165, respectivamente. ††Número, Banco NCBI
CONCLUSIONES
Xylella fastidiosa subsp. fastidiosa se encuentra establecida en el municipio de Parras, Coahuila y está asociada con las especies frutales Carya sp., Cydonia sp., Ficus carica, Olea europaea y Vitis vinifera, y las especies silvestres de Baccharis sp., Celtis pallida, Phragmites sp., Rubus sp., Salsola kali., Tribullus terrestris y un pasto (familia Poaceae). Este documento constituye el primer reporte de X. fastidiosa subsp. fastidiosa en Cydonia sp., la cual se propone como nuevo hospedante de la bacteria.











 nueva página del texto (beta)
nueva página del texto (beta)



