Introducción
México es considerado centro de origen, domesticación y diversificación del maíz (Zea mays L.) (Matsuoka et al., 2002), donde se han identificado 68 razas nativas (Caballero-García et al., 2019). La variabilidad morfológica de los frutos de maíz es aprovechada para la elaboración de una amplia gama de alimentos. El patrimonio ecogeográfico y cultural de México ha sido factor clave en la generación de una amplia variabilidad de maíces, los cuales difieren en su morfología, genética y ecología, lo que ha conducido a su diferenciación en grupo y a su reconocimiento como razas (Wellhausen et al., 1951). La diversidad genética está relacionada con factores geográficos, ecológicos y culturales, mismos que permiten obtener genotipos mejorados. Esta diversidad puede ser evaluada con marcadores moleculares basados en secuencias simples repetidas de ADN (SSR).
Existen antecedentes de análisis de la diversidad genética mediante el uso de marcadores moleculares. En América Latina y El Caribe se evaluaron 194 poblaciones correspondientes a 131 razas de maíces locales empleando 28 marcadores SSR; los agrupamientos resultantes corresponden a las regiones de México, tierras bajas de Mesoamérica y Los Andes (Bedoya et al., 2017). Aci et al. (2013) caracterizaron 18 marcadores SSR en 15 poblaciones nativas de Argelia, en las cuales encontraron una diversidad génica total (He) de 0.57 y 5.8 alelos por locus (APL). En México, en la región noroeste se analizaron 28 poblaciones de maíces nativos con 20 SSR, se encontraron 6.1 APL y una He de 0.72 (Pineda-Hidalgo et al., 2013); Vega-Álvarez et al. (2017) registraron 20.9 APL con 31 SSR en 107 poblaciones de nueve razas de maíz; Herrera-Saucedo et al. (2019) en ocho razas norteñas reportaron 18.3 APL con 31 SSR; en maíces tropicales se encontraron nueve APL y una He de 0.58 (González et al., 2013). Para Valles Altos, Rocandio-Rodríguez et al. (2014) analizaron 31 SSR en 107 poblaciones y reportaron 20.5 APL.
Conocer la diversidad genética de las razas de maíz permite identificar alelos favorables para el mejoramiento genético; dicha diversidad debe apoyarse de una descripción fenotípica para identificar genotipos de interés. Los estudios de diversidad genética son de utilidad para conocer patrones de diversidad regional y poder formular estrategias integrales de conservación in situ, que permita a los cultivos evolucionar en respuesta a los factores ambientales adversos (Perales y Golicher, 2014). El objetivo del presente estudio fue caracterizar una población representativa de cada una de 25 razas de maíz para conocer su diversidad genética y contribuir al conocimiento sobre la diversidad de los maíces mexicanos para ayudar a plantear estrategias de mejoramiento y conservación.
Materiales y métodos
Material genético
Se analizaron 25 poblaciones de sendas razas de maíz de México del Banco de Germoplasma del Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT). La elección de las razas se determinó mediante la clasificación de Wellhausen et al. (1951) y en función de la pureza racial de la colección del CIMMYT (Cuadro 1). Los genotipos se sembraron en charolas de germinación con sustrato Peat Moss (Cosmopeat®) para facilitar el crecimiento de plántulas. Se evaluaron 10 individuos por cada población.
Cuadro 1 Datos geográficos de las 25 razas de maíz bajo estudio con marcadores moleculares SSR.
| Raza | Accesión | Altitud (msnm) | Latitud N | Longitud O |
|---|---|---|---|---|
| Indígenas Antiguas | ||||
| Arrocillo | Pueb-778 | 1978 | 19º 48’ 46.5” | 97º 22’ 52.4” |
| Chapalote | Sina-2 | 46 | 24º 49’ 27.8” | 107º 27’ 36.8” |
| Nal-Tel | Camp-103 | 14 | 19º 46’ 59” | 90º 37’ 00” |
| Palomero Toluqueño | Mexi-5 | 2597 | 19º 17’ 10.2” | 99º 34’ 15.1” |
| Exóticas precolombinas | ||||
| Cacahuacintle | Pueb-552 | 2568 | 19º 01’ 43” | 97º 27’ 37” |
| Harinoso de Ocho | Chis-151 | 1566 | 16º 06’ 59” | 91º 55’ 59.9” |
| Olotón | Chis-238 | 1961 | 16º 42’ 52.23” | 92º 27’ 28.76” |
| Mestizas prehistóricas | ||||
| Comiteco | Chis-24 | 736 | 15º 41’ 59” | 92º 13’ 00” |
| Cónico | Mexi-3 | 2676 | 19º 16’ 59” | 99º 39’ 00” |
| Jala | Naya-6 | 1099 | 21º 06’ 39.1” | 104º 26’ 59.8” |
| Olotillo | Chis-599 | 1060 | 17º 03’ 36” | 92º 43’ 11.99” |
| Pepitilla | More-22 | 1338 | 18º 40’ 59” | 98º 47’ 59” |
| Reventador | Naya-39 | 10 | 21º 56’ 59.9” | 105º 16’ 59.9” |
| Tabloncillo | Jali-43 | 1402 | 19º 58’ 00” | 104º 16’ 00” |
| Tehua | Chis-228 | 1087 | 15º 23’ 17.44” | 92º 11’ 25.8” |
| Tepecintle | Chis-58 | 33 | 15º 25’ 14.27” | 92º 53’ 58.25” |
| Tuxpeño | Vera-39 | 17 | 20º 28’ 48” | 97º 5’ 51.52” |
| Vandeño | Chis-30 | 42 | 15º 26’ 10.54” | 92º 56’ 17.16” |
| Zapalote Chico | Oaxa-448 | 48 | 16º 25’ 59” | 95º 1’ 00” |
| Zapalote Grande | Chis-236 | 21 | 16º 16’ 59” | 94º 10’ 59.9” |
| Modernas incipientes | ||||
| Bolita | Oaxa-180 | 1165 | ||
| Celaya | Guan-69 | 1786 | 20º 30’ 00” | 100º 58’ 59.9” |
| Chalqueño | Mexi-718 | 2400 | 19º 50’ 6.9” | 99º 13’ 3” |
| No bien definidas | ||||
| Dulcillo del Noroeste | Sina-79 | 140 | 26º 18’ 00” | 108º 34’ 00” |
| Serrano de Jalisco | Jali-173 | 2301 | 20º 08’ 34.87” | 103º 43’ 23.1” |
Procedimiento de laboratorio
Se usaron hojas de plántulas para la extracción de ADN mediante el método CTAB 2X con 1 % de polivinilpirrolidona (PVP). La calidad del ADN se confirmó con electroforesis en gel de agarosa 0.8 % (p/v), que se corrió en amortiguador Tris-Acetato-EDTA (TAE 0.5 X) (Tris-Base, ácido acético y EDTA 0.5 M, pH 8.0) (Ven Lee y Bahaman, 2012). El gel fue teñido con bromuro de etidio 0.1 % (p/v) y se visualizó en un fotodocumentador KODAK EDAS-290 (Kodak Ltd., Rochester, Nueva York, EUA). Se cuantificó el ADN con un espectrofotómetro GENESYS 10uv (Thermo Fisher Scientific, Waltham, Massachusetts, EUA). El ADN para la PCR tuvo una concentración de 10 ng µL-1. Se usaron 10 pares de iniciadores SSR de la base de datos genéticos y genómicos para maíz Maize Genetics and Genomic Database (https://www.maizegdb.org) mediante el criterio de patrón de geles para diferenciar alelos (Cuadro 2).
Cuadro 2 Características de los 10 pares de iniciadores SSR y posición del marcador en el cromosoma (Bin).
| Locus | Repetición | Iniciador hacia adelante/iniciador en reversa | Núm. de Bin |
|---|---|---|---|
| umc-1886 | (CG)8 | GTTTGACAGCACAAGTGCAAGAAA/GAGGTGGACATTGGACAACACC | 3.02 |
| umc-1113 | (CACAG)5 | ATCATGCGTCATCACTCTCAGAAC/GCTGGAGCTAGCTGTAGTGTAGCA | 10.02 |
| umc-1377 | (TAATA)4 | GACTTAGTGCCAGCTCAGATCCAG/GATATTGCTGTCTTTTGCTACGGC | 8.03 |
| umc-1656 | (CGGT)7 | AGTTTTGACCGCGCAAAAGTTA/GTACGAGCAGGCCATTAACCC | 6.02 |
| umc-2180 | (GGCC)4 | ATCAGCATCGATAGCGAAGAAAGA/ATTGCTACTAGGGTTGTTGTTGCC | 10.03 |
| umc-1071 | (TACGA)5 | AGGAAGACACGAGAGACACCGTAG/GTGGTTGTCGAGTTCGTCGTATT | 1.01 |
| umc-1143 | AAAAT | CGTGGTGGGATGCTATCCTTT/GACACTAGCAATGTTCAAAACCCC | 6.00 |
| umc-1369 | (AAAAAC)4 | TTCCAGCACTAACTTACAGCAACG/AGATATGCGTATGGCTCTTGTTGG | --- |
| umc-1698 | (AAC)5 | GTGTCGTGTTGGGAGAACATGAG/TAACTACTACACCACTCGCGCAAA | 3.04 |
| umc-1638 | (CTCCGG)5 | AGGTGACCTCGACGTCCTACG/GAGGGGAACAAAGACTTGACGTT | 8.09 |
La amplificación por PCR se llevó a cabo en un termociclador Techne® TC-512 (Bibby Scientific, Vernon Hills, Illinois, EUA). Las condiciones de reacción fueron: un ciclo de desnaturalización inicial a 94 ºC durante 3 min; 30 ciclos con temperaturas de 94 ºC por 1 min, 55 ºC por 1 min y 72 ºC por 2 min; finalmente se dio un ciclo de extensión a 72 ºC durante 10 min. Los fragmentos se separaron mediante electroforesis en geles de agarosa 2 % (p/v) a 100 V durante 1.5 h. En la tinción y visualización de bandas se emplearon las mismas condiciones descritas para verificar la calidad del ADN (Ven Lee y Bahaman, 2012).
Recolección y análisis de datos
La codificación de cada locus se realizó en función del peso molecular de las bandas para formar la matriz de datos. A la banda de más alto peso molecular le correspondió el 1 y así sucesivamente. Un individuo con una sola banda se codifica 1-1; es decir, un homocigoto para el alelo 1. Las codificaciones 1-2, 1-3 y 2-3 serían ejemplos de loci heterocigotos.
Una vez conformada la matriz, se empleó el programa GeneAlEx versión 6.5 (Peakall y Smouse, 2012) para obtener las distancias genéticas con el coeficiente Dice y el índice de diversidad génica esperada (He) de Nei (1987). Con la misma matriz se efectuó un análisis de coordenadas principales (ACoP) con el método de covarianza estandarizada y análisis de varianza molecular con los estadísticos R, se asumieron modelos de mutación paso a paso. Ambos análisis se realizaron con 1000 permutas (Peakall y Smouse, 2012).
Resultados y discusión
En el ACoP, las dos primeras coordenadas explicaron 60.29 % de la variación total (Figura 1). La primera coordenada explicó el 42.11 % de la variación y la segunda el 18.18 %. Con respecto a la coordenada principal 1, en los cuadrantes A y B se ubicaron las poblaciones de maíz que por lo general se distribuyen en el sur y occidente de México, clasificadas como Mestizas Prehistóricas y generalmente son de ciclo corto, con excepción de Jala y Comiteco (Wellhausen et al., 1951); en los cuadrantes C y D se aglomeraron las poblaciones de maíz de las razas indígenas antiguas descritas por Wellhausen et al. (1951). En relación con la coordenada principal 2, en los cuadrantes B y C se ubicaron principalmente poblaciones de maíz de las razas con distribución en el sur y occidente de México, mientras que en los cuadrantes A y D se localizaron las poblaciones propias del centro y norte de México, de acuerdo con descripciones de Sánchez et al. (2000) y Wellhausen et al. (1951).
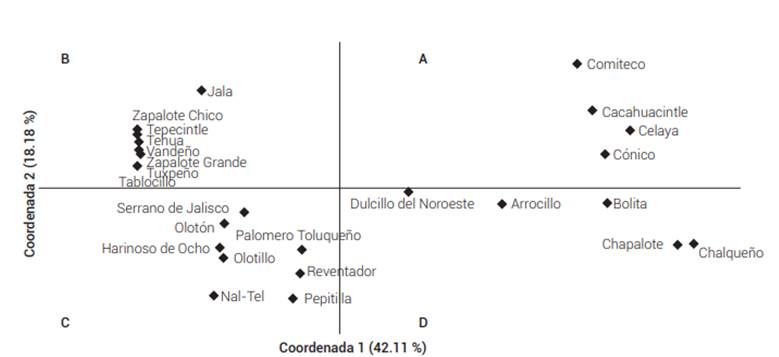
Figura 1 Representación gráfica de las dos primeras coordenadas principales para 25 razas mexicanas de maíz con marcadores moleculares SSR; las letras A, B, C y D indican los cuadrantes.
Se encontró un promedio de 2.62 APL; el número efectivo de alelos fue de 1.93 y el valor de He fue de 0.44. Rocandio-Rodríguez et al. (2014) encontraron 20.52 APL; Vega-Álvarez et al. (2017) registraron 20.9 APL, Herrera-Saucedo et al. (2019) 18.3 APL. mientras que González et al. (2013) encontraron 9 APL y Pineda-Hidalgo et al. (2013) 6.1 APL en ocho razas de maíz; A diferencia del presente estudio, los trabajos antes citados emplearon secuenciadores automáticos, los cuales pueden detectar un mayor número de APL al poder discriminarlos hasta por un nucleótido individual.
Las poblaciones de maíz de las razas con los niveles más altos de diversidad genética fueron Tabloncillo (0.53), Serrano de Jalisco (0.53), Tuxpeño (0.52), Zapalote Grande (0.51), Vandeño (0.5), Tehua (0.49), Bolita (0.49), Olotillo (0.49), Zapalote Chico (0.48), Tepecintle (0.48), Pepitilla (0.46) y Olotón (0.46); las poblaciones de maíces de las razas con niveles intermedios de diversidad fueron Harinoso de Ocho (0.43), Palomero Toluqueño (0.42), Cónico (0.42), Jala (0.41), Nal-Tel (0.42) y Arrocillo Amarillo (0.41), mientras que las poblaciones de maíces de las razas con los niveles más bajos de diversidad genética fueron Reventador (0.39), Chapalote (0.39), Cacahuacintle (0.37), Celaya (0.31), Dulcillo del Noroeste (0.36), Comiteco (0.33) y Chalqueño (0.30).
La población de la raza Chalqueño (Mexi-718) utilizada en el presente estudio es una población de grano color amarillo, la cual aparece como población representativa en la colección del CIMMYT, pero algunas de estas poblaciones se reportan en bajas frecuencias, lo que puede explicar su baja diversidad genética (Herrera-Cabrera et al., 2013). La mayor parte de razas con baja diversidad son destinadas para usos especiales, que requieren alta calidad de grano, los agricultores les practican una rigurosa selección de semilla y su distribución geográfica es restringida (González et al., 2013). Perales y Golicher (2014) mapearon la diversidad de las razas de maíz mexicanas en los últimos 60 años en función de la composición racial y no encontraron cambios significativos indicativos de una disminución en la diversidad de los maíces nativos.
Respecto al análisis de varianza molecular, el índice FST tuvo un valor de 0.427 (P ≤ 0.001), lo que indica alta diferenciación genética inter-poblacional, con una variación del 43 % entre las poblaciones. Se encontró un 19 % de variación intra-poblacional y 38 % entre individuos. Rocandio-Rodríguez et al. (2014) reportaron una variación intra-poblacional de 76.3 %, indicativo de que usaron una gran cantidad de poblaciones pertenecientes a la misma raza, se evaluaron más individuos y por ende obtuvieron altos niveles de variación intra-poblacional. González et al. (2013) encontraron 23.18 % de variación intra-poblacional, pero con menor número de APL (9). El número de poblaciones, la cantidad de marcadores moleculares y el número de individuos analizados influyen en la cantidad de APL, He y en la partición de la diversidad entre y dentro de poblaciones. El coeficiente de endogamia FIS de 0.333 (P ≤ 0.001) denota la presencia de una considerable deficiencia de individuos heterocigotos, a pesar de la naturaleza alógama del maíz.
Las razas de maíz con adaptación restringida y cultivadas en pequeñas extensiones de terreno pueden experimentar procesos de endogamia y deriva genética, tal y como lo indica el valor RIT de 0.618 (P ≤ 0.001). El flujo génico (Nm) fue de 0.336 individuos por generación, lo que sugiere que las razas están separadas geográficamente. Si el flujo génico es alto no se presenta diferenciación genética entre las poblaciones; y de ser bajo, como en este caso, se promueve que las poblaciones se diferencien genéticamente. Los niveles de diversidad genética más altos fueron para la población de maíz correspondiente a Tabloncillo (Jali-43) y a la población de Serrano de Jalisco (Jali-173), con un valor de 0.53 para ambas. Los niveles más bajos de diversidad correspondieron a las poblaciones de Comiteco (0.33) y Chalqueño (0.30). El número de individuos migrantes por generación entre las poblaciones fue menor de uno, lo que explicó el alto grado de diferenciación entre las poblaciones. La mayor parte de la variación genética fue inter-poblacional con 43 %.
Tanto la población de la raza Tabloncillo como la de Serrano de Jalisco mostraron la más alta diversidad génica; por otra parte, la mayor parte de la variación genética fue entre las poblaciones correspondientes a las razas de maíz debido a la metodología empleada en este estudio. En relación con el ACoP, las distancias genéticas entre las poblaciones de las razas de maíz permiten visualizarlas y agruparlas parcialmente en concordancia con el estudio de Wellhausen et al. (1951).











 text new page (beta)
text new page (beta)


