INTRODUCCIÓN
Agave tequilana Weber var. azul es un cultivo de gran relevancia económica y cultural en México, ya que es la única variedad permitida como materia prima para la producción del tequila, producto de exportación con denominación de origen (Cedeño, 1995; Dalton, 2005; Secretaría de Economía, 2012). Por su manejo agronómico, esta variedad de características morfológicas bien diferenciadas, muestra alta homogeneidad genética en campo (Díaz-Martínez et al., 2012; Trejo et al., 2018). Los agaves o magueyes pertenecen a la familia Asparagaceae, subfamilia Agavoideae (APG III, 2009) y conforman un género amplio constituido por 210 especies; 159 de ellas se encuentran en México y 119 son endémicas, por lo que el país se considera su centro de origen y diversidad (García-Mendoza et al., 2017; García-Mendoza y Galván, 1995). Además de su utilidad para la producción de bebidas alcohólicas, los agaves se han aprovechado desde épocas prehispánicas de múltiples formas (Colunga-GarcíaMarín y May-Pat, 1993; Davidson y Ortiz De Montellano, 1983; Gentry, 1982). Actualmente, a nivel internacional existe interés en los agaves por su potencial para la producción de bioenergía en zonas áridas (Davis et al., 2011; Stewart, 2015). Entre las alternativas para el aprovechamiento integral de los agaves se considera la extracción de metabolitos secundarios bioactivos (López-Romero et al., 2018).
Estudios farmacológicos con extractos de Agave muestran su potencial como anticancerígenos, antiinflamatorios, antihipertensivos, antiulcerogénicos, inmunomoduladores, antioxidantes, antibacterianos, antifúngicos y antiparasíticos (López-Romero et al., 2018; Pereira et al., 2017; Santos-Zea et al., 2012; Sidana et al., 2016); también han mostrado potencial de uso en la industria agrícola por sus efectos como molusquicida, larvicida, insecticida y fitotóxico (Abdel-Gawad et al., 2015; Herbert-Doctor et al., 2016). Las actividades biológicas y farmacológicas de los extractos de agaves derivan de compuestos bioactivos como flavonoides, terpenos, esteroles, fenoles y otros (López-Romero et al., 2018; Santos-Zea et al., 2012); sin embargo, la mayor parte de la bioactividad de los extractos de Agave ha sido atribuida a las saponinas. Sidana et al. (2016) compilaron las estructuras de 141 saponinas aisladas a partir de plantas, hojas, savia, rizomas, flores o callos in vitro de diferentes especies de Agave; enumerando sus actividades biológicas.
Las saponinas (del latin sapo o jabón) son metabolitos secundarios con propiedades surfactantes producidos por las plantas para defenderse contra patógenos y plagas (Sahu et al., 2008; Wink, 1999). Las saponinas poseen gran diversidad estructural y están conformadas por una parte polar glucosídica y una apolar (aglicona). Su parte glucosídica está constituida por oligosacáridos lineales o ramificados. Se clasifican según la estructura de su aglicona en triterpénicas o esteroidales (Vincken et al., 2007). Las saponinas se producen a través de la ruta de biosíntesis del escualeno-cicloartenol-sitosterol mediante reacciones de hidroxilación, oxidación, glicosilación y ciclación. Aunque algunas de las enzimas involucradas en la biosíntesis de las saponinas esteroidales han sido caracterizadas, la mayoría no se han estudiado, por lo que la ruta biosintética no está claramente establecida (Upadhyay et al., 2018). Identificar cada una de las enzimas involucradas y estudiar la regulación de la ruta biosintética es un área de interés actual.
Los agaves producen saponinas esteroidales tipo furostanol o espirostano, clasificadas según la estructura unida al C26 de la aglicona. Las saponinas tipo furostanol son precursoras de las del tipo espirostano. Se ha propuesto que, en el último paso de la biosíntesis, la unidad de glucosa unida al hidroxilo del C26 del furostanol glucósido es escindida mediante hidrólisis por la furostanol glicósido 26-O-β-glucosidasa (F26G) para formar el glicósido tipo espirostano (Upadhyay et al., 2018). La única F26G nativa que ha sido purificada a homogeneidad y cuya actividad específica se ha demostrado es la furostanol glicósido 26-O-β-glucosidasa aislada a partir de rizomas de Costus speciosus (Inoue y Ebizuka, 1996). Dos F26G recombinantes producidas en E.coli a partir de DNAs complementarios (cDNAs) de C. speciosus y Dioscorea esculenta han sido caracterizadas (Inoue et al., 1996; Nakayasu et al., 2015). En este trabajo se reportan ESTs (Expressed Sequence Tags) obtenidos a partir de cDNAs nativos de diferentes tejidos de A. tequilana Weber var. azul, potencialmente codificantes de tres diferentes secuencias similares a F26G; se determinó su expresión diferencial en tejidos vegetativos detectada con iniciadores específicos, así como protocolos de qPCR para su detección cuantitativa.
MATERIALES Y MÉTODOS
Identificación de ESTs
La base de datos de secuencias ESTs AgaveDB del Colegio de Postgraduados-Campeche (MartínezHernández et al., 2007) fue consultada para identificar ESTs cuyo análisis de BLASTX contra la base de datos no redundante (nr) de NCBI (https://blast.ncbi.nlm.nih. gov/Blast.cgi) indicara homología con enzimas tipo F26G (Cuadro 1). AgaveDB contiene información de 35,000 ESTs secuenciados por Sanger (Simpson et al., 2011) a partir de bibliotecas de cDNAs expresados en hojas, piña, raíz, meristemo vegetativo, meristemo floral, anteras, ovarios y sitios de emergencia de bulbilos en el tallo floral (Figura 1) (Martínez-Hernández et al., 2010).
Cuadro 1. Hits del BLASTX de ESTs de A. tequilana contra base de datos nr.
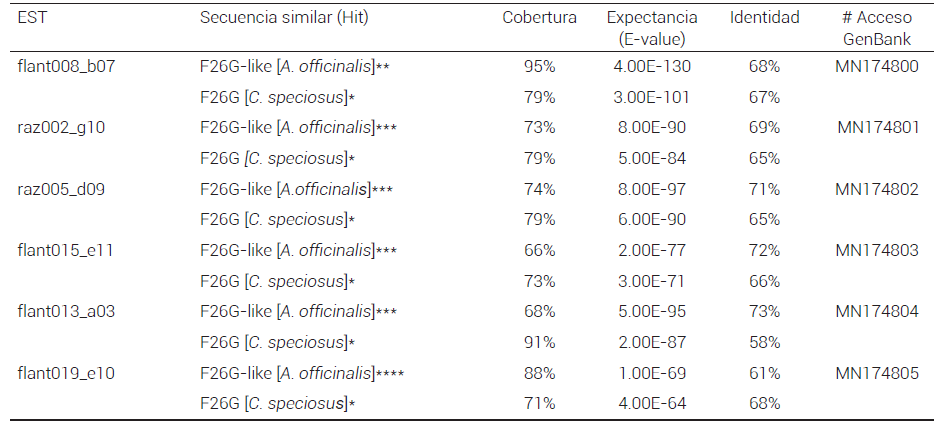
Código de acceso del hit en NCBI: * Q42707.1, ** XP_020250593.1, *** XP_020246472.1, **** XP_008807567.2
Identificación de clonas con cDNAs completos
Para identificar cDNAs completos, los ESTs se alinearon con ClustalOmega (https://www.ebi.ac.uk/Tools/msa/ clustalo/) contra la región 5’ de las secuencias codificantes (CDS) de la F26G de C. speciosus y Asparagus officinalis, para visualizar los alineamientos con BioEdit. Los posibles ATGs se identificaron traduciendo los ESTs con ExPASy. Para verificar el tamaño de los cDNAs clonados, los plásmidos extraídos según el método de lisis alcalina (Zhou et al., 1990) se digirieron con la enzima BsrGI (Cat. No. R0575S, BioLabs) para escindir los cDNAs del vector pDONR222 donde se encuentran clonados (MartínezHernández et al., 2010). Los productos de la digestión se analizaron mediante electroforesis en gel de agarosa al 1%. Los tamaños de los fragmentos se determinaron usando como referencia un marcador de 1kb (Invitrogen Cat. 15615-016).
Detección de la expresión por RT-PCR
Los oligonucleótidos usados como iniciadores específicos de la reacción en cadena de la polimerasa (PCR) se diseñaron para cada tipo de cDNA mediante el programa Primer design de NCBI dirigiéndolos a las zonas diferenciales ubicadas en el extremo 5’ de los ESTs (Figura 2) y se sintetizaron por Invitrogen. La secuencia de los iniciadores diseñados y para el gen de actina se muestran en el Cuadro 2. Los plásmidos de las clonas que se utilizaron para los ensayos de especificidad y para las curvas estándar del PCR cuantitativo (qPCR) se purificaron con el QIAprep Spin Miniprep kit (Qiagen Cat. 27104) y se cuantificaron en un espectrofotómetro (Nanodrop 1000 Thermo Scientific; Waltham, Massachusetts, EUA). Para las curvas estándar se prepararon diluciones seriales 1:10 de cada plásmido por triplicado, incluyendo concentraciones de 1 ng a 0.1 pg. La síntesis de cDNA para el RT-PCR punto final se llevó a cabo conforme Martínez-Hernández et al. (2010) mediante la transcriptasa reversa Superscript III (Invitrogen Cat. 18080085) y 160 ng de mRNA.
Cuantificación por qPCR
Las reacciones de PCR, tanto en punto final como para el protocolo de qPCR, se realizaron en un termociclador iQ5 BioRad con la enzima IQ SYBRGreen Supermix (BioRad Cat. 170-8880) y 20 pmoles de iniciadores por reacción. Los programas de PCR consistieron de un ciclo inicial de desnaturalización a 95 ºC por 5 min, seguido de ciclos de amplificación de 30 s a 95 ºC, 30 s a la temperatura de alineamiento (Ta) de cada iniciador conforme el Cuadro 2, 30 s a 72 ºC y un ciclo de extensión final a 72 ºC por 7 min. Se realizaron 25 ciclos para la prueba de especificidad o 35 ciclos para el RT-PCR punto final y la curva estándar. Los ciclos umbral (Ct) y las temperaturas de disociación (Tm) de los productos se determinaron con el iQ5 Optical System Software 2.0. Controles negativos sin DNA molde se realizaron en paralelo. El tamaño de los productos se verificó mediante electroforesis en gel de agarosa al 1.2 % usando marcadores de peso molecular de 100 y 50 pb (Invitrogen Cat. 15628-050 y 10416014)
Las curvas estándar para el qPCR se construyeron conforme a lo recomendado para realizar cuantificación absoluta (Heid et al., 1996), los Ct se graficaron con respecto al logaritmo en base 10 de las distintas concentraciones de plásmido utilizadas. Cada valor de Ct representó la media aritmética de los triplicados de cada concentración. Se graficó la desviación estándar de cada punto. Se generó la recta de regresión por el método de los mínimos cuadrados y se calculó el coeficiente de determinación (R2 ).
RESULTADOS Y DISCUSIÓN
Identificación de ESTs similiares a F26G
Mediante la búsqueda de ESTs en la base de datos AgaveDB por BLASTX se identificó a 46 ESTs de A. tequilana Weber var. azul similares a la furostanol glicósido 26-O-βglucosidasa (F26G) de C. speciosus, codificante de la única enzima nativa para la que ha sido demostrada su actividad. En la Figura 1 se muestra la distribución de los 46 EST conforme a los tejidos a partir de los cuales fueron clonados y secuenciados. El 50 % de los ESTs similares a F26G provienen de anteras. Otra cantidad importante de ESTs provienen de ovarios y raíces. Considerando que de cada biblioteca se secuenció una cantidad similar de ESTs (Simpson et al., 2011), la proporción diferente de ESTs similares a F26G encontrados entre bibliotecas es un referente indirecto del nivel de su expresión en el tejido a partir del cual se construyó cada biblioteca. Estos resultados concuerdan en que tanto la flor como la raíz son órganos productores de saponinas en agaves (Pérez et al., 2013; Sidana et al., 2016); sin embargo, se encontraron relativamente pocos ESTs en hojas, órgano a partir del cual se han extraído la mayoría de las saponinas de Agave (Sidana et al., 2016). Análisis de expresión de las enzimas involucradas en la biosíntesis de saponinas, como la F26G, en diversos tejidos de Agave permitirán verificar cuáles órganos producen saponinas y cuáles las acumulan, así como las condiciones medioambientales que regulan su producción.
cDNAs codificantes de distintas enzimas similares a F26G
Los 46 ESTs se alinearon y compararon contra CDS de C. speciosus, identificándose seis distintos ESTs que alinearon hacia el extremo 5’ de los CDS de referencia (Figura 2), lo que sugiere que provienen de cDNAs que se clonaron completos, lo que indica que la transcriptasa inversa alcanzó la región 5’ del transcrito. Cuatro ESTs provienen de anteras (flant008_b07, flant015_e11, flant013_a03 y flant019_e10) y dos de raíz (raz002_g10 y raz005_d09). Estos seis ESTs fueron comparados mediante BLASTX contra nr (Cuadro 1). Todos mostraron mayor similitud a la enzima similar a F26G deducida a partir de secuencias genómicas de A. officinalis que a la F26G de C. speciosus, lo cual es congruente con que A. tequilana pertenece a la familia de las Asparagaceae. La expectancia (E-value) de los ESTs contra las secuencias similares identificadas (hits) es altamente significativa, pero el porcentaje de identidad es relativamente bajo, por lo que se consideran similares a F26G. La cobertura fue en promedio del 70 %, ya que los ESTs divergen de sus hits en el extremo 5’.
En el alineamiento de la Figura 2 se muestra que los primeros 250 nucleótidos (nt) de la región 5’ es una zona variable entre los seis ESTs de A. tequilana y los CDS de referencia; sin embargo, a partir de ese punto presentan clara similitud, la cual se mantiene a todo lo largo de la secuencia de los ESTs (750-870 nt). Se requiere secuenciar el lado 3’ de los cDNAs clonados para completar el alineamiento. En la figura se muestran los posibles ATGs, lo que sugiere que la región variable incluye los UTRs 5’ y el extremo N-terminal de las proteínas codificadas.

Figura 2. Alineamiento de ESTs de A. tequilana ubicados hacia el extremo 5’ del CDS de la F26G de Costus speciosus y Asparagus officinalis. Los recuadros a color señalan el sitio blanco de los iniciadores específicos para las clonas flant008_b07 (verde), raz002_g10 (negro) y flant015_e11 (rosa). Los codones de inicio de los CDS de referencia, así como los posibles ATGs de las enzimas similares a F26G de A. tequilana se resaltan en color azul.
En el alineamiento también se puede observar que las secuencias de A. tequilana son similares entre sí, pero hay mayor similitud entre las dos secuencias de raíz o entre tres secuencias de anteras, mientras que la secuencia flant008_ b07 muestra mayor similitud con A. officinalis. Lo anterior sugiere que hay al menos tres distintos tipos de F26G en A. tequilana. En A. officinalis y Musa acuminata hay dos genes de F26G y en Phoenix dactylifera se identificaron nueve loci de F26G por lo que, aunque en C. speciosus solo se caracterizó una F26G, es factible la existencia de una familia de F26G en Agave.
Para determinar si el tamaño de los cDNAs insertados en las clonas a partir de las cuales se secuenciaron estos seis ESTs coinciden con la longitud del CDS de la F26G de C. speciosus, fueron escindidos con la enzima BsrGI del vector donde se encuentran clonados. En la Figura 3 se muestran los fragmentos de 2.5 Kb que corresponden al vector sin inserto y el resto de los fragmentos que conforman el cDNA liberado, seccionado por cortes internos con la enzima. Cinco de las seis clonas liberaron fragmentos que en total suman aproximadamente 1900 pb, tamaño coincidente con la longitud del CDS de la F26G de C. speciosus más los UTRs, lo que indica que son cDNAs que se clonaron completos. En el caso de la clona flant008_b07, la más divergente en el alineamiento de la Figura 2, el cDNA es más pequeño (1500 pb), por lo que podría estar incompleto o tener una N-terminal más corta.

Figura 3. Digestión de clonas de A. tequilana liberando cDNAs similares a F26G. Los plásmidos fueron digeridos con BsrGI. Carriles1 y 2: flant008_b07, 3 y 4: raz002_g10, 5 y 6: raz005_d09, 7 y 8: flant015_e11, 9 y 10: flant013_a03, 11 y 12: flant019_e10. Carriles 1, 3, 5, 7, 9, 11: DNA plasmídico sin digerir. Carriles 2, 4, 6, 8, 10 y 12: plásmidos digeridos. 1 kb: marcador de peso molecular.
Las enzimas similares a F26G de A. tequilana
Para estudiar la expresión de los tres tipos de F26G identificados en A. tequilana se diseñaron iniciadores específicos para reacciones de PCR dirigidos a la región 5’ variable de cada tipo de EST (Figura 2). En el Cuadro 2 se muestran las secuencias de los iniciadores, las temperaturas de alineamiento (Ta) utilizadas en el PCR y el tamaño esperado de cada producto. Para verificar su especificidad, los iniciadores se probaron contra las clonas de los tres tipos de cDNAs. En la Figura 4A se muestra que, bajo las condiciones establecidas, los iniciadoresamplifican específicamente la clona para la cual fueron diseñados, generando productos del tamaño esperado. Mediante los iniciadores específicos se detectó la expresión de los tres tipos de transcrito de F26G-like en tejidos vegetativos de A. tequilana (hoja, piña y raíz) mediante RT-PCR. En la Figura 4B se observa que, a diferencia del transcrito de actina expresado en los tres tejidos analizados, cada transcrito de F26G-like de Agave tiene un patrón de expresión distinto; así, flant008_b07, clonado de anteras, no se detecta en ninguno de los tejidos vegetativos analizados, mientras que raz002_g10 se detectó en hoja y en raíz, y flant015_e11 se detectó en los tres tejidos. Los patrones de expresión distintos concuerdan con la hipótesis de que estos ESTs representan a tres diferentes F26G de A. tequilana.

Figura 4. A) Productos de PCR de la amplificación específica de clonas que contienen cDNAs de enzimas similares a F26G de A. tequilana. Carriles 1, 4 y 7: flant008_b07; 2, 5 y 8: raz002_g10; 3, 6 y 9: flant015_e11. Amplificados con los iniciadores 2842_2_8 en carriles 1-3, 2256_4_12 en carriles 4-6 y 2988_12_14 en carriles 7-9; 100 pb y 50 pb son marcadores de peso molecular. B) Expresión diferencial de transcritos de las tres F26G similares en hoja (H), piña (P) o raíz (R) de A. tequilana detectados por RT-PCR utilizando los iniciadores específicos de (A). Actina: control de expresión constitutiva en hoja, piña y raíz.
Para evaluar la utilidad de los iniciadores específicos para realizar PCR cuantitativo se generó una curva de disociación de los productos. En la Figura 5A se muestra que los iniciadores propuestos generan un único pico de disociación a la temperatura de fusión (Tm) del producto y que los controles sin DNA no producen picos adicionales por dímeros u horquillas, por lo que cumplen con las características para ser usados en qPCR. En la Figura 5B se observa que los protocolos de qPCR utilizados permiten detectar de forma lineal de 1 ng a 0.1 pg de plásmido que contiene cada cDNA de F26G-like de A. tequilana, por lo que pueden ser aplicados con alta sensibilidad en la cuantificación absoluta de estos genes (Heid et al., 1996) y aplicarlos en la cuantificación de cDNAs sintetizados a partir de transcritos con bajos niveles de expresión durante estudios de regulación de su expresión génica.

Figura 5. Amplificación cuantitativa de clonas que contienen cDNAs de enzimas similares a F26G de A. tequilana. A) Curvas y Temperaturas de disociación (Tm) de los productos de PCR de clonas de cDNAs de F26G de A. tequilana (flant008_b07, raz002_g10 y flant015_e11) amplificadas con los iniciadores específicos. Las líneas inferiores en cada gráfica corresponden a los controles negativos sin DNA molde. Los números debajo de los picos indican la Tm en o C. B) Curva estándar de amplificación por qPCR de concentraciones seriales (de 1 ng a 0.1 pg por reacción) de cada clona, utilizando los iniciadores específicos de (A). Las barras en cada punto representan la desviación estándar de la media de los triplicados del Ciclo umbral (Ct) por concentración. Las líneas representan la recta de regresión y R2 es el coeficiente de determinación.
A pesar de los numerosos estudios que reportan estructuras químicas y actividades biológicas de saponinas esteroidales del género Agave, las enzimas que participan en su ruta de biosíntesis se desconocen y sólo algunas han sido incipientemente estudiadas en otras plantas (Upadhyay et al., 2018). En el caso de la F26G, que participa en el último paso transformando las saponinas tipo furostanol a tipo espirostano, sólo han sido caracterizadas bioquímicamente mostrando actividad específica la enzima nativa aislada a homogeneidad de C. speciosus (Inoue y Ebizuka, 1996) y dos proteínas recombinantes expresadas en E. coli (Inoue et al., 1996; Nakayasu et al., 2015).
Transcritos de Asparagus, Allium, Dracaena, Chlorophytum, Trillium y Dioscorea potencialmente involucrados en la ruta de biosíntesis de saponinas esteroidales han sido identificados a través de análisis transcriptómicos de seqRNA bajo el criterio de que un incremento en la producción de saponinas correlaciona con el aumento de esos transcritos (Upadhyay et al., 2018); así mismo, CDS de F26G-like de Asparagus, Phoenix y Musa, deducidos a partir de sus genomas, fueron identificados por su similitud con la F26G de C. speciosus; sin embargo, en todos esos casos sus cDNAs nativos no han sido clonados, no se ha demostrado la actividad de las enzimas codificadas y los estudios de regulación génica son incipientes, por lo que queda mucho por hacer para comprender el papel de esta enzima en la producción de saponinas en plantas monocotiledóneas, su papel fisiológico y su potencial biotecnológico. Los cDNAs clonados identificados en este trabajo podrán ser expresados en sistemas recombinantes para la caracterización bioquímica de las F26G de A. tequilana y los protocolos de qPCR establecidos podrán ser aplicados en la cuantificación de su expresión génica durante estudios de regulación de la biosíntesis de saponinas esteroidales por factores intrínsecos o medioambientales.
CONCLUSIONES
Se reportan por primera vez secuencias parciales de al menos tres distintos tipos de cDNAs nativos, clonados a partir de Agave tequilana Weber var. azul, con patrón de expresión tisular diferencial, similares a la furostanol glicósido 26-O-β-glucosidasa, enzima responsable de la conversión de saponinas tipo furostano al tipo espirostano durante la biosíntesis de saponinas esteroidales. Se establecieron protocolos útiles para la cuantificación de los tres tipos de enzimas similares a F26G de A. tequilana, aplicables a estudios de regulación de su expresión durante la biosíntesis de saponinas en Agave.











 nueva página del texto (beta)
nueva página del texto (beta)




