INTRODUCCIÓN
La subespecie Apis mellifera scutellata se introdujo del sur de África a Brasil en 1956 para realizar recombinaciones genéticas y generar segregantes adaptados a zonas tropicales (Kerr, 1967); sin embargo, en 26 colonias escaparon en 1957 y con ello se inició un proceso de africanización en el continente americano (Kerr, 1967; Padilla et al., 1992). La abeja africanizada se reconoce como el resultado del apareamiento, en primera instancia, de reinas de origen europeo de apiarios locales con zánganos de origen africanizado escapados del Brasil, y en segunda instancia la invasión de colmenas por reinas de origen africanizado (Clarke et al., 2002); la dispersión hacia el norte se da con esta dinámica y a finales de 1986 se detectaron los primeros enjambres en México (Winston, 1992), lo que ha provocado pérdidas económicas en la apicultura por su comportamiento altamente defensivo, migratorio y fuerte tendencia a enjambrar (Guzmán-Novoa y Page, 1994a; Seeley, 2016; Stort, 1974).
En 1984 se creó el Programa Nacional para el Control de la Abeja Africana para prevenir y controlar su diseminación en México. La estrategia que se planteó para hacer frente a la invasión de la abeja africanizada consistió en establecer trampas y su consecuente eliminación y así preservar los apiarios locales con germoplasma europeo (Cajero, 1990, Com. Pers.)1. La experiencia de más de dos décadas de dispersión mostró que las trampas fueron inefectivas y la abeja continuó su dispersión a través de territorio mexicano y en 1990 se localizó en Texas, EE. UU. en 1990 (Sugden y Williams, 1991), por lo que el programa de control de la abeja africanizada terminó siendo sólo de monitoreo de su avance en el territorio mexicano (Winston, 1992).
Una estrategia alternativa la planteó el Dr. Tarcicio Cervantes-Santana, quien en 1988 inició un programa de mejoramiento genético, el cual consideró el aprovechamiento de la variabilidad genética que representaba la incorporación de genes de abejas africanizadas. El apiario experimental del Colegio de Postgraduados, Campus Montecillo, Estado de México se integró en 1978 inicialmente con abejas reinas europeas colectadas de diferentes partes del país, para aplicar selección con criterios de sanidad y buen comportamiento apícola. En 1993 se introdujeron al apiario abejas africanizadas que habían estado sometidas a un proceso de semidomesticación mediante selección en Cárdenas, Tabasco por cuatro generaciones, para mansedumbre, producción de miel y bajo comportamiento de enjambrazón y migración (Cervantes y Cruz, 1997, Com. Pers.)2. Se criaron reinas de las mejores colmenas africanizadas seleccionadas, las cuales se aparearon a través de cruzamientos controlados con zánganos europeos mejorados provenientes de las colmenas iniciales.
Derivado de este proceso y más de 20 años de selección continua, en el Colegio de Postgraduados se cuenta con una población de abejas denominadas euroafricanas (Pérez-Sato y Cervantes-Santana, 2001), de diferente constitución a las africanizadas mencionadas por otros autores (Schneider et al., 2004), llamadas euroafricanas por la recombinación genética de abejas reinas africanizadas seleccionadas con zánganos europeos (Utrera, 1998, Com. Pers.)3.
Aunque se conoce la manera en que se construyó el acervo genético de la población, no se tiene información a nivel de ADN que respalde su naturaleza genética, por lo que es importante realizar estudios que permitan tener información confiable sobre el origen materno de este material genético. Es conveniente señalar que este apiario experimental se ha mantenido sin aplicar ningún tipo de tratamiento contra la varroasis por más de 20 años, capacidad que puede ser atribuible a la contribución del germoplasma africanizado con la mejora genética señalada.
Anteriormente, era difícil distinguir a la abeja africanizada de las subespecies europeas debido a la homogeneidad en la morfología de la especie (Gary et al., 1985); sin embargo, hoy en día existen métodos eficientes para detectar la africanización, como la amplificación de fragmentos de ADNmt, debido a que éste se hereda vía materna y revela la identidad genética de la reina a través de las obreras sin tomar en cuenta la herencia paterna (Hall y Muralidharan, 1989). Con información de morfometría, de polimorfismo isoenzimático y de ADNmt actualmente se reconocen 29 subespecies de A. mellifera, que se agrupan en cinco linajes: linaje A, incluye a las subespecies africanas; linaje M, formado por subespecies de Europa occidental; linaje C, formado por las subespecies de Europa oriental; linaje O, que abarca a las subespecies del cercano oriente, y linaje Y, que incluye a la subespecie de Etiopía (Özdil y İlhan, 2012; Rahimi, 2015; Ruttner, 1988; Valido et al., 2014).
Por lo anterior, el objetivo del presente estudio fue determinar el origen materno en el apiario mediante el análisis de las regiones intergénicas COI-COII y ND5 del ADNmt con la expectativa de que corresponde al linaje africano que se empleó en el establecimiento de la población experimental.
MATERIALES Y MÉTODOS
Sitio experimental
La investigación se realizó durante los meses de febrero a julio de 2015 en el apiario experimental del Colegio de Postgraduados, Campus Montecillo, ubicado en Texcoco, Estado de México (19º 27’ 50.01” latitud N, 98º 54’ 17.53” longitud O, 2,250 msnm). El clima predominante es templado semi-seco, con temperatura media anual de 15.9ºC, y heladas poco frecuentes en otoño-invierno.
Material genético
La población provino de cruzas efectuadas en Montecillo, Edo. de México en 1993, de abejas europeas mejoradas con abejas africanizadas que habían sido sujetas a un proceso de selección para su semidomesticación (Guzmán-Novoa, 1996; Pérez-Sato y Cervantes-Santana, 2001). Las abejas africanizadas procedieron de una población en proceso de mejoramiento genético en Cárdenas, Tabasco, México. La población de abejas africanizadas se formó con enjambres recolectados en Tabasco, Campeche y Veracruz en 1988. Esta población de enjambres fue seleccionada durante cuatro generaciones, una por año, para los caracteres de mansedumbre, sedentarismo y productividad apícola mediante la cría artificial de reinas y fecundación natural.
La población de abejas europeas, también mejoradas, se integró de la siguiente forma: 1) en 1978 se introdujeron reinas europeas fecundadas provenientes de 10 localidades de México al apiario en Chapingo, Texcoco, Edo. de México, para criar reinas hijas; 2) la progenie resultantes se fecundó en apareamiento natural para obtener la primera generación recombinante; 3) después de dos generaciones de recombinación, la población se seleccionó durante 13 generaciones (una por año) hacia características asociadas con la productividad de miel, tales como sanidad y uniformidad de postura, mediante cría artificial de reinas y con apareamiento natural; posteriormente, el apiario se trasladó de Chapingo a Montecillo, Texcoco, Edo. de México.
En 1993 se tomaron 20 reinas africanizadas seleccionadas para mansedumbre, baja enjambrazón y productividad apícola de Cárdenas, Tabasco, mismas que se introdujeron al apiario de Montecillo, integrado con colonias de abejas europeas mejoradas. De las reinas africanizadas introducidas se seleccionaron seis, cuyas colonias presentaron los mejores atributos apícolas, tales como mansedumbre, calidad de postura, desarrollo en su población y productividad de miel. De estas reinas se obtuvieron reinas hijas por cría artificial, las cuales se fecundaron en forma natural con zánganos europeos que predominaban en el tiempo referido. Las cruzas entre reinas vírgenes africanizadas con zánganos africanizados se evitaron colocando trampas en la piquera de cada colmena y destruyendo pupas de zánganos. La población resultante se mantuvo bajo selección durante tres ciclos para docilidad, calidad de postura y producción de miel. Este proceso de selección se mantuvo de manera permanente por las generaciones correspondientes a uno por año hasta el inicio de este estudio.
Análisis genético
Se eligieron 19 colmenas al azar para su valoración molecular de las más de 100 colmenas tipo jumbo que integran el apiario. Se colectaron al menos 30 obreras de cada una de las 19 colonias, las cuales se depositaron en frascos de plástico para ser conservadas a -20 ºC hasta su uso. El ADN total se extrajo del cuerpo completo de cada abeja obrera (cinco individuos por colmena) con el método CTAB (Doyle y Doyle, 1987). El análisis del ADNmt se llevó a cabo mediante la amplificación por reacción en cadena de la polimerasa (PCR) de las regiones intergénicas COI-COII y ND5, según el protocolo descrito por Garnery et al. (1992). Los iniciadores utilizados para COI-COII fueron: E2 forward 5’-GGCAGAATAAGTGCATTG-3’ y H2 reverse 5’-CAATATCATTGATGACC-3’, que amplifican un fragmento de 530 a 1230 pb (Magnus et al., 2014); para el gen ND5 los iniciadores fueron: ND5 forward 5’-CGAAATGAATAGGATACAG-3’ y ND5 reverse 5’-TTAGGATTTGGTAGAGTTGG-3’, que amplifican un fragmento de aproximadamente 822 pb (Bouga et al., 2005; Özdil y İlhan, 2012).
La mezcla de reacción de PCR se preparó en un volumen final de 15 µL, integrado por 7.86 µL de agua HPLC, 3 µL de buffer, 0.6 µL de dNTPs, 0.18 de cada iniciador, 3 µL de ADN genómico y 0.18 µL de Taq ADN polimerasa (Promega, USA). Las condiciones de amplificación de PCR para COI-COII fueron una desnaturalización inicial a 94 ºC por 4 min, 40 ciclos de 94 ºC por 1 min, 48 ºC por 1 min y 72 ºC por 1 min, y una extensión final de 72 ºC por 6 min; y para ND5 una desnaturalización inicial a 94 ºC por 5 min, 35 ciclos de 94 ºC por 45 s, 46 ºC por 1 min y 72 ºC por 1 min, y una extensión final a 72 ºC por 5 min. La amplificación de los genes se verificó en geles de agarosa 1.5 % con amortiguador 1X TAE. El producto final de PCR se purificó con ExoSAP-IT Affymetrix®. La secuenciación se realizó en ambas direcciones con un secuenciador Genetic Analyzer modelo 3230® (Applied Biosystem®, Foster City, California, EUA).
Análisis de la información
El ensamble de secuencias se obtuvo mediante el programa de cómputo BioEdit versión 7.0.5 (Hall, 1999) y éstas fueron alineadas con el procedimiento Profile Mode Muscle (Thompson et al., 1994) incluido en el programa Mega6 (Tamura et al., 2013). Las secuencias consenso fueron comparadas con las depositadas en el GenBank del National Center for Biotechnology Information (NCBI) (https://www.ncbi.nlm.nih.gov/genbank/), mediante la opción BLASTN 2.2.19 (Zhang et al., 2000). Se obtuvieron 95 secuencias en total, de las cuales 50 se utilizaron para el análisis Bayesiano.
Las regiones COI-COII y ND5 se concatenaron para realizar un análisis filogenético Bayesiano mediante el software MrBayes v.3.2.0 (Ronquist y Huelsenbeck, 2003); el número de generaciones de cadenas de Markov de Montecarlo (MCMC) fue de 3 × 106, con un muestreo cada 1000 generaciones y una desviación estándar final de 0.009608. El árbol filogenético se construyó utilizando la secuencia del abejorro (Bombus terrestris) como grupo externo, tomada de la base de datos de secuencias depositadas en el Genbank (número de accesión DQ870926.1) y como testigos se utilizaron secuencias de genomas mitocondriales de miembros de los linajes A (africano) y M (europeo): KJ601784 A. m. scutellata (A), KX870183 A. m. capensis (A), KX943034 A. m. scutellata × capensis(A), KT279826 A. m. scutellata (A), KY926883 A. m. intermisa (A), KX463941 A. m. iberiensis (M) y KY926884 A. m. mellifera (M).
RESULTADOS Y DISCUSIÓN
Presencia de polimorfismos y agrupamiento
La longitud de las secuencias concatenadas de las regiones intergénicas ND5 y COI-COII varió de 993 a 1235 pb, derivado de inserciones y deleciones en los fragmentos analizados (Cuadro 1). El análisis concatenado produjo un dendrograma que permitió separar las colmenas en estudio, con base en las secuencias analizadas y tomando como grupo externo a la secuencia de B. terrestris, lo que dio como resultado una topología de seis subclados (Figura 1), de los cuales, en cinco (I, II, III, IV, VI) se agruparon las 19 colmenas analizadas y uno (V) quedó conformado por tres testigos: A. m. iberiensis (M), A. m. intermisa (A) y A. m. mellifera (M).
Cuadro 1 Diferencia en polimorfismo en secuencias concatenadas de los espacios intergénicos COI-COII y ND5 de 19 colmenas del apiario experimental del Colegio de Postgraduados, Campus Montecillo, Estado de México.
| Subclado | No. de secuencias | Longitud de secuencias (pb) | Proporción de sitios conservados | Proporción de sitios variables | Inserciones/deleciones con respecto a A. m. scutellata KJ601784 |
|---|---|---|---|---|---|
| I | 11 | 1041 | 1039/1233 (84.2 %) | 2/1233 (0.16 %) | Deleción 191 pb |
| II | 2 | 1234 | 1232/1235 (99.7 %) | 3/1235 (0.24 %) | Inserción 4 pb |
| III | 26 | 1234 | 1232/1234 (99.8 %) | 2/1234 (0.16 %) | Deleción 2 pb |
| IV | 3 | 1252 | 1232/1234 (99.8 %) | 2/1234 (0.16 %) | Inserción 2 pb |
| VI | 12 | 993 | 1232/1234 (80.3 %) | 2/1234 (0.16 %) | Deleción 240 pb |
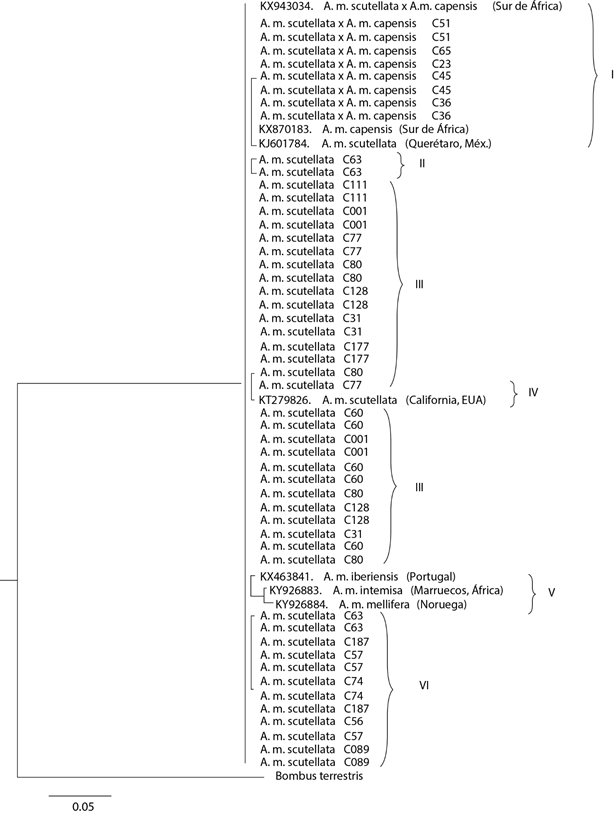
Figura 1 Árbol filogenético con inferencia bayesiana de 19 colmenas del apiario experimental del Colegio de Postgraduados, Campus Montecillo, México. El dendrograma fue construido mediante secuencias concatenadas de ND5 y COI-COII, la desviación estándar final fue de 0.009608
El subclado I agrupó secuencias de ocho ejemplares, pertenecientes a cinco colmenas (C51, C65, C23, C45 y C36), así como a las secuencias de tres testigos: el híbrido A. m. scutellata × A. m. capensis, A. m. capensis y A. m. scutellata; estos testigos fueron caracterizados como linaje A, de origen africano. Dicho grupo se distinguió por presentar una deleción de 191 pb con respecto al testigo africano A. m. scutellata (KJ601784, africanizada colectada en Querétaro, México), manteniendo el 84.2 % de los sitios conservados y dos sitios variables.
El subclado II agrupó a dos ejemplares de una colmena, C63, con una inserción de 4 pb con respecto al testigo africanizado anterior. El subclado III, agrupó a 26 ejemplares de nueve colmenas (C01, C31, C60, C77, C80, C111, C117, C128, C177). Este grupo tuvo una deleción de 2 pb y 99.8 % de los sitios conservados con relación al testigo A. m. scutellata (KJ601784, africanizada colectada en Querétaro, México). El subclado IV, agrupó a dos ejemplares, uno de cada una de dos colmenas (C80 y C77) y al testigo A. m. scutellata (KT279826, africanizada colectada en California, EE. UU.). Este último presentó 99.83 % de sitios conservados en su secuencia y una inserción de 2 pb con relación al testigo A. m. scutellata (KJ601784, africanizada colectada en Querétaro, México), de acuerdo con la base de datos del GenBank.
El subclado V agrupó a tres testigos: A. m. iberiensis (KX463941, europeo, linaje M, proveniente de Portugal), A. m. intermissa (KY926883, africano, linaje A, proveniente de Marruecos) y A. m. mellifera (KY926884, europeo, linaje M, proveniente de Noruega), que difirieron por presentar una inserción de 44 pb, una deleción de 159 pb y una deleción 163 pb, respectivamente, con respecto al testigo africanizado (KJ601784, africanizada colectada en Querétaro, México); además de las inserciones/deleciones, se detectó el 0.32, 2.11 y 2.6 % de sitios variables con respecto al testigo africanizado de referencia (Cuadro 2). El subclado VI agrupó a 12 ejemplares de ocho colmenas (C63, C187, C57, C74, C187, C56, C57, C89), las cuales presentaron una deleción de 240 pb y dos sitios variables (0.16 %) con relación al testigo A. m. scutellata de Querétaro, México.
Cuadro 2 Diferencia en polimorfismo en secuencias concatenadas de los espacios intergénico COI-COII y ND5 de testigos incluidos en el análisis filogenético.
| Testigos | Longitud (pb) | Proporción de sitios conservados | Cantidad de sitios variables(%) | Inserciones/deleciones con respecto a A. m. scutellata KJ601784 |
|---|---|---|---|---|
| A. m. capensis KX870183 | 1042 | 1038/1233 (84.18 %) | 2/1233 (0.16 %) | Deleción 191 pb |
| A. m. mellifera KY926884 | 1069 | 1035/1232 (84 %) | 33/1232 (2.6 %) | Deleción 163 pb |
| A. m. iberiensis KX463941 | 1261 | 1211/1277 (94.83 %) | 4/1277 (0.32 %) | Inserción 44 pb |
| A. m. intermisa KY926883 | 1072 | 1046/1231 (84.97 %) | 26/1231 (2.11 %) | Deleción 159 pb |
| A. m. scutellata KT279826 | 1034 | 1229/1234 (99.59 %) | 2/1234 (0.16 %) | Inserción 2 pb |
| A. m. scutellata x A. m. capensis KX943034 | 1041 | 1040/1232 (84.4 %) | 0 | Deleción 191 pb |
El subclado VI agrupó a los dos testigos del linaje europeo M, así como a un testigo africano del linaje A, pero que provino de Marruecos, en el norte de África, además de B. terrestris, como testigo de grupo externo. Este subclado se separó de manera relevante del resto de unidades de estudio, con magnitud de 0.3 unidades de tasa de sustitución por sitio, determinadas por las deleciones/inserciones, así como por la mayor diferencia en sitios variables, lo cual permite aseverar que el material de estudio presenta ADNmt de origen africano.
La mayor parte del material de estudio (14 de 19 colmenas) formó subclados con testigos africanizados colectados en México y en California; también, en la comparación con la base de datos del GenBank se corroboró la identidad con A. m. scutellata. Las cinco colmenas restantes se identificaron como híbridos de A. m. scutellata × A. m. capensis.
El haber encontrado estos dos tipos de germoplama, de A. m. scutellata y del híbrido A. m. scutellata × A. m. capensis pudiera explicarse por el hecho de que las seis reinas africanizadas con las que se formó la población euroafricana fue el resultado de la captura de 177 enjambres africanizados (Pérez-Sato y Cervantes-Santana, 2001) de lo que venía migrando desde su escape de Sao Paulo, Brasil en 1957 (Kerr, 1967; Padilla et al., 1992). Según lo descrito, el germoplasma africanizado fue colectado de enjambres en Tabasco, parte de Campeche y Veracruz entre 1987 y 1989, justo después de la entrada de la abeja A. m. scutellata a México (Moffett et al., 1987). El origen geográfico de las abejas que llegaron a Brasil fue Tabora, Tanzania, y Pretoria y Johannesburgo, Sudáfrica (Nogueira-Neto, 1964). A. m. scutellata tiene una distribución geográfica en el centro y el oeste de África, mientras que A. m. capensis se localiza al sur de Sudáfrica (Beekman et al. 2008), por lo que es probable que al menos alguna de las reinas introducidas a Brasil haya sido variante híbrida de A. m. scutellata × A. m. capensis, de Sudáfrica.
La población de estudio se formó con reinas vírgenes de origen africanizado, seleccionadas por mansedumbre y criterios apícolas, que se cruzaron con zánganos de origen europeo también seleccionados (Pérez-Sato y Cervantes-Santana, 2001). El propósito de realizar la recombinación genética de esa manera fue el aprovechamiento de atributos apícolas deseables de ambos tipos de abejas (Guzmán-Novoa et al., 2011); de las africanizadas, se buscaron atributos tales como alta postura de reinas, tolerancia a enfermedades (entre ellas varroasis), alta producción de polen y propóleo; así mismo, con la selección hacia mansedumbre, sedentarismo y otros criterios de importancia apícola, antes de la recombinación genética, se habían contrarrestado efectos negativos como alta tendencia a enjambrar, pillaje y alto comportamiento defensivo que implicaba alto costo de manejo de colmenas (Guzmán-Novoa et al., 2011); con la recombinación con zánganos europeos se trató de aprovechar la capacidad productiva de miel, mansedumbre y uniformidad de postura. El resultado ha sido favorable debido a que el comportamiento defensivo es altamente heredable (Guzmán-Novoa y Page, 1992; 1993; 1999; Stort 1975) y puede ser determinado por efectos de dominancia genética (Degrandi-Hoffman et al., 1998; Guzmán-Novoa y Page, 1994b; Guzmán-Novoa et al., 2002; Stort, 1975) y los efectos paternos hacia mansedumbre son más relevantes que los maternos (Degrandi-Hoffman et al., 1998; Guzmán-Novoa et al., 2002).
Es conocido que las abejas africanizadas (A. m. scutellata) son más resistentes a varroa (De Jong, 1997; Guzmán-Novoa et al., 1996; Mondragón et al., 2006) por su conducta higiénica y de acicalamiento, además de la baja fertilidad del ácaro en las celdas de cría de obreras (Arechavaleta-Velasco y Guzmán-Novoa, 2001; Calderón et al., 2010; Guzmán-Novoa et al., 1999; Vandame et al., 2002).
Es conveniente resaltar que, desde su establecimiento como población recombinante de abejas africanizadas con europeas y hasta la fecha, el apiario experimental del Colegio de Postgraduados es una de las pocas poblaciones experimentales que muestran tolerancia natural a varroa y se ha mantenido sin aplicar acaricidas, lo cual puede ser atribuible a la contribución del germoplasma africanizado y con capacidad productiva de miel sobresaliente como contribución del germoplasma europeo.
El hecho de haber encontrado solamente ADNmt de origen africano en el estudio parece indicar que las abejas africanizadas, en su dispersión desde el escape en Brasil en 1957 y hasta su establecimiento en México, han conservado el material genético mitocondrial de origen africano.
CONCLUSIONES
El análisis de ADNmt confirma que el linaje materno del apiario del Colegio de Postgraduados, Montecillo, Estado de México corresponde al africano (A), como se infería de antecedentes del establecimiento de dicha población. Los resultados indican que 74 % de las colmenas analizadas corresponden a la subespecie A. m. scutellata y 26 % al híbrido A. m. scutellata × A. m. capensis.











 nueva página del texto (beta)
nueva página del texto (beta)


