INTRODUCCIÓN
La roya de la hoja en trigo (Triticum aestivum), causada por Puccinia triticina, se encuentra ampliamente distribuida a nivel mundial (Bolton et al., 2008). Este patógeno reduce la disponibilidad de agua en la hoja y sustancias de almacenamiento en el grano, causando pérdidas económicas importantes por la reducción de, incluso, 50 % del rendimiento en epifitias severas (Huerta-Espino et al., 2011). La resistencia específica, otorgada por un gen de efecto mayor, frecuentemente es superada por el fitopatógeno en corto tiempo; sin embargo, se ha visto que la resistencia durable a la roya se logra mediante la interacción de genes de efectos menores (Solís-Moya et al., 2014). La identificación de los genes de efectos aditivos involucrados en la resistencia es importante porque permite desarrollar variedades con resistencia incorporada a través de mejoramiento asistido por marcadores moleculares, acortando los tiempos con respecto al mejoramiento tradicional (Collard y Mackill, 2008).
Actualmente, las herramientas moleculares se utilizan para la selección asistida en el mejoramiento genético de plantas de importancia agrícola (Collard y Mackill, 2008; Gupta et al., 1999; Kumar et al., 2009). Este método ofrece una rápida y eficiente selección de genes de interés, como se ha reportado para la resistencia en arroz (Oryza sativa), maíz (Zea mays), cebada (Hordeum vulgare), frijol (Phaseolus vulgaris), soya (Glycine max), jitomate (Solanum lycopersicum) y trigo (Chen et al., 2000; Muranty et al., 2014). En México (Gasca-González et al., 2008) estudiaron la expresión de genes por hibridación sustractiva supresiva en respuesta a infección por virus en chile (Capsicum chinenese). El análisis de genes expresados diferencialmente es importante porque incluyen los mecanismos moleculares implicados en la respuesta del organismo ante factores bióticos o abióticos, los cuales modifican el estado normal de funcionamiento de la planta, el perfil de expresión de los genes implicados y los procesos celulares que operan en el organismo (Byers et al., 2000). Una técnica ampliamente usada para aislar estos genes es la hibridación sustractiva supresiva, que consiste en hibridar dos poblaciones de ARNm (ADNc), en donde una corresponde a plantas sometidas a roya u otra condición de estudio, mientras que la segunda corresponde a plantas en condiciones normales. En esta hibridación los transcritos hibridados son removidos, mientras que los no hibridados corresponden a los transcritos expresados diferencialmente (Al-Taweel et al., 2011; Diatchenko et al., 1996).
La construcción de una biblioteca sustractiva supresiva de ADNc de trigo infectado con P. triticina es necesaria para encontrar genes de respuesta al patógeno, así como de las funciones que desempeñan cada uno de éstos en la planta e indagar si las proteínas traducidas están asociadas con el mecanismo de defensa. La expresión diferencial permite el uso de los niveles de transcripción de estos genes como marcadores moleculares de expresión para la selección de materiales resistentes a la roya de la hoja. El objetivo de esta investigación fue aislar e identificar ESTs de genes expresados durante la infección por la roya de la hoja en estado adulto, evaluar su nivel de transcripción, y explorar su posible uso como marcadores de transcripción para la selección de genotipos de trigo resistentes a la roya.
MATERIALES Y MÉTODOS
Material genético
En la construcción de la biblioteca sustractiva supresiva de ADNc se utilizó la variedad resistente de trigo Monarca F2007, la cual fue liberada por el Programa de Mejoramiento de Trigo del INIFAP para su siembra comercial en el Bajío y el Norte de México en el año 2007 (Huerta-Espino, 2011). Ésta se caracteriza porque en la etapa de plántula presenta los genes de resistencia Lr14a y Lr27+31, mientras que en planta adulta presenta los genes de resistencia durables Lr34 y Lr46, además de un gen desconocido (Huerta-Espino et al., 2011). Los materiales usados para validar los genes diferenciales y determinar perfiles de expresión como medio de selección fueron 15 líneas avanzadas F6 del Programa Nacional de Trigo del INIFAP y Monarca F2007, los cuales se sembraron en el ciclo primavera-verano del 2014. La variedad susceptible (Moroco) se inoculó en la base del tallo con una mezcla equivalente de urediniosporas de las razas de roya de la hoja MCJ/SP y MBJ/SP (2.5 mL de una solución que contenía 225,000 esporas por mililitro), que son las que prevalecen en la Región Centro de México (Huerta-Espino et al., 2002).
Colecta de material vegetal
La siembra de los genotipos de trigo se realizó en el INIFAP Campo Experimental Bajío, Celaya, Guanajuato, México. En el ciclo primavera-verano del 2013 se colectaron 3 hojas de 10 plantas de la variedad Monarca F2007 inoculadas por dispersión de esporas de roya desde una variedad susceptible y de 10 plantas sin inocular (sembradas en otra parcela), todas los cuales se encontraban en etapa de llenado de grano al momento de la colecta. Para asegurar que Monarca se encontraba bajo la presión de la roya se sembró la variedad altamente susceptible Moroco en los bordes y pasillos del cultivo, para que funcionara como inóculo natural del patógeno. El muestreo de las hojas se realizó cuando la variedad Moroco se encontraba completamente infectada por la roya.
Extracción y purificación de ARN
Se muestreó el tejido foliar de Monarca inoculada con roya y sin inocular y se congeló inmediatamente con N líquido hasta su arribo al laboratorio donde se mantuvo a -80 ºC. La extracción de ARNm se realizó de acuerdo con el método propuesto por Mishra et al. (2005), que se basa en la separación magnética de ARN poly(A)+ después de hibridarlo a un oligo dT(18) biotinilado inmovilizado en microesferas para-magnéticas ligadas a estreptavidina. La integridad del ARN se analizó por electroforesis en geles de agarosa 1.5 % en condiciones desnaturalizantes y se tiñó con bromuro de etidio (0.5 mg mL-1). Las imágenes se visualizaron con luz UV en un fotodocumentador ChemidocTM de Bio-Rad.
Aislamiento de genes diferenciales relacionados con la resistencia a la roya de la hoja
Los transcritos expresados diferencialmente se aislaron mediante el método sustractivo propuesto por Mishra et al. (2007), que requiere la hibridación de ARN poly(A)+ de la muestra inoculada con ADNc de cadena sencilla biotinilada de la muestra sin inocular. Los híbridos de ARN poly(A)+-ADNc y un exceso de ADNc se inmovilizan en las microesferas; posteriormente las microesferas se separan magnéticamente y los ARN poly(A) expresados diferencialmente (que no se unen a las microesferas) quedan en el sobrenadante. La síntesis de la primera cadena se realizó con la enzima transcriptasa reversa Super ScriptTM II (Invitrogen). Para la síntesis de la segunda cadena de transcritos diferenciales se utilizó la enzima AdvantageTM PCR (Invitrogen), usando el iniciador 5´-PCR primer IIA. Este ADN se purificó por columna (QIAquickTM, Qiagen) y el producto resultante se ligó al vector pGEM®-T Easy (Promega), el cual se utilizó para transformar células competentes DH5α (Invitrogen) por medio de choque térmico; una alícuota de 50 mL de estas células se sembraron por el método de difusión en caja Petri con medio LB adicionado con ampicilina (100 mg mL-1), 20 mL de X-gal (50 mg mL-1) y 100 mL de IPTG (0.1 M). El ADN plasmídico se extrajo de acuerdo al protocolo de Birnboim y Doly (1979). La presencia del inserto se corroboró por PCR empleando como templado el ADN plasmídico de cada una de las clonas usando los iniciadores universales T7 y SP6, los cuales flanquean el sitio de inserción del vector.
El volumen de la PCR fue de 25 mL. La mezcla de reacción se preparó con 1 mL de templado (500 ng), 15.5 mL agua, 2.5 mL buffer 10X, 1 mL MgCl2 50 mM, 1 mL dNTP´s 10 mM, 0.2 µL Taq polimerasa 1 U y 1 mL de cada primer 10 mM. El programa de amplificación constó de una desnaturalización a 95 ºC por 5 min, 25 ciclos de 95 ºC por 1 min, 60 ºC por 2 min, 72 ºC por 2 min y una fase de extensión de 72 ºC por 5 min. La secuenciación se realizó en Langebio, CINVESTAV-Unidad Irapuato. Una vez obtenidas las secuencias de las clonas se consultó la base de datos del NCBI (National Center for Biotechnology Information) con el algoritmo BLAST (Basic Local Alignment Search Tool) para encontrar semejanzas con secuencias ya reportadas. Posteriormente se diseñaron iniciadores específicos para cada una de las secuencias con la herramienta Primer BLAST del NCBI.
Validación de genes asociados y perfiles de expresión
De la misma forma que se describe en la sección anterior se cultivaron 15 líneas avanzadas con diversos niveles de respuesta a la roya de la hoja, además de la variedad Monarca F2007 (Cuadro 1). Todos estos materiales se mantuvieron bajo presión por roya. En la etapa de llenado de grano se colectó tejido foliar y se extrajo ARN total de acuerdo con el protocolo de Logemann et al. (1987). La síntesis de ADNc se realizó con la enzima transcriptasa reversa (Super ScriptTM II, Invitrogen). Un total de 16 pares de iniciadores, diseñados a partir de las clonas secuenciadas, se emplearon para determinar los perfiles de expresión (Cuadro 2). La estandarización del nivel basal de expresión se realizó mediante la amplificación del gen ribosómico constitutivo 26S, de acuerdo al procedimiento de Montero-Tavera et al. (2017).
Cuadro 1 Genealogía de 15 líneas avanzadas de trigo utilizadas para los perfiles de expresión.
| Id. | Genealogía |
|---|---|
| 156 |
|
| 157 |
|
| 223 |
|
| 305 |
|
| 306 |
|
| 328 |
|
| 334 |
|
| 337 |
|
| 344 |
|
| 357 |
|
| 358 |
|
| 367 |
|
| 381 |
|
| 394 |
|
| 399 |
|
Posteriormente los ADNc de los 15 materiales se amplificaron por PCR con las siguientes condiciones: una fase inicial de desnaturalización a 95 ºC por 5 min, seguido de 95 ºC por 1 min, 60 ºC por 2 min y 72 ºC por 2 min por el número de ciclos normalizados para cada muestra, finalmente se dio un ciclo de extensión a 72 ºC por 5 min. Los productos de PCR se visualizaron en un baño con GelRed ® y se fotodocumentaron con el equipo Gel Logic 112® de Kodak Sciences. El análisis densitométrico se realizó con el software TotalLab Quant TL120 1D v.2009.
Análisis de agrupamiento de perfiles de expresión
A partir de los análisis densitométricos de los perfiles de expresión se hicieron análisis de agrupamiento para generar dendrogramas de similitud, con el objetivo de asociar los materiales en grupos. Para el análisis se ingresaron los datos densitométricos en una matriz básica con el paquete estadístico NTSYS v2.1p y se corrió la opción de análisis multivariado, obteniendo una matriz de similitud utilizando el Coeficiente de Concordancia Simple o Simple Matching Coeficient (SMC) (Sokal y Sneath, 1963). Para el análisis de agrupamientos se utilizó el programa NTSyspc v2.1p, basado en el UPGMA (Unweighted Pair Group Method using Arithmetic Averages), con el objetivo de obtener el dendrograma.
RESULTADOS
Se generó una biblioteca de ADNc de la variedad Monarca, de la que se seleccionaron 51 ESTs como resultado de la expresión diferencial causada por P. triticina. Los tamaños de las secuencias fluctuaron de 44 a 561 pares de bases; sin embargo, para los perfiles de expresión sólo se estudiaron 15 ESTs (Cuadro 2). Los genes representados por las ESTs se encontraron registrados en las bases de datos del Genbank, aunque no siempre asociados a royas. Los genes identificados se agruparon en tres categorías: genes relacionados con estrés (16 %), infecciones por presencia de patógeno (67 %) y metabolismo (17 %). Entre los genes expresados por estrés abiótico se encuentran de respuesta a salinidad, deshidratación, sequía moderada y calor; entre los inducidos por infecciones o inoculaciones de fitopatógenos fúngicos están Fusarium graminearum (BM135418.1), F. udum (GR467847.1), Puccinia graminis (EV254283.1), P. triticina (CJ895632.1), Septoria tritici (CA744232.1), Colletotricum lindemuthianum (CB542715.1), y Erysiphe graminis (CA677987.1); finalmente están los genes relacionados con el metabolismo, como la expresión de proteínas de almacenamiento (BJ286751.1), proteínas de transferencia de lípidos no específicos (PvLTP-24) (U72765.1), y los que actúan como mecanismos de defensa ante los patógenos, como las proteínas relacionadas con patogénesis (PvPR1) (HQ541962.1) y la proteína similar a F-box (PP2-B10) (KF033248.1).
Cuadro 2 Genes de expresión diferencial entre líneas de trigo contrastantes por su resistencia a la roya de la hoja.
| ID. del gen* | Iniciadores (5´- 3´) | Descripción | Nº de acceso GenBank | Tamaño del amplicón |
|---|---|---|---|---|
| RHT-1 |
|
Inoculación con Puccinia triticina | CJ892825.1 | 120 |
| RHT-2 |
|
Transposón Sabrina | EF567062.1 | 100 |
| RHT-4 |
|
Inoculación con Erysiphe graminis | CA677987.1 | 110 |
| RHT-5 |
|
Biblioteca de ADNc de trigo infectado con Puccinia triticina | CJ934834.1 | 140 |
| RHT-9 |
|
Proteína hipotética de Phaseolus vulgaris | XM_007161532.1 | 111 |
| RHT-11 |
|
Estrés por sequía moderada | GR409424.1 | 109 |
| RHT-12 |
|
Transposón Sabrina | FN564430.1 | 152 |
| RHT-18 |
|
Biblioteca de ADNc de trigo. | CA731237.1 | 113 |
| RHT-19 |
|
Proteína relacionada a patogénesis | HQ541962.1 | 170 |
| RHT-20 |
|
Biblioteca de ADNc obtenidos en diferentes estadios de plántula bajo estrés | AK330698.1 | 80 |
| RHT-23 |
|
Proteína hipotética con función desconocida | XM_007144369 | 72 |
| RHT-24 |
|
Secuencia de hoja de plántula de trigo etiolada | CA635284.1 | 210 |
| RHT-25 |
|
Pertenece a una biblioteca de ADNc de vaina en desarrollo de frijol | CV540306.1 | 179 |
| RHT-27 |
|
Biblioteca de ADNc de trigo de la variedad Chinese Spring | AK332645.1 | 121 |
| RHT-28 |
|
Biblioteca de ADNc de cebada | AK368215.1 | 196 |
| RHT-29 |
|
ADNc de biblioteca de trigo | CJ849274.1 | 121 |
* Identificación rápida asignada a los genes obtenidos de la biblioteca.
Los niveles de expresión de los genes involucrados en la respuesta a la infección por P. triticina mostraron un comportamiento diferencial en cada genotipo en presencia del patógeno (Figura 1). En los perfiles generados para las diversas líneas de trigo se observó que algunos genes no presentaron fragmentos amplificados (RHT-1, RHT-9 y RHT-18). En otros se observó un nivel de expresión similar entre todas las líneas, según su valor de densidad, lo cual indica que se expresan de manera constante durante la infección, independientemente del genotipo analizado (RHT-12). También se identificaron genes que no presentaron un patrón determinado. Esto sugiere que cada genotipo muestra diferencias en la expresión de distintos genes, y estas divergencias pueden asociarse a la resistencia a la roya de la hoja.

Figura 1 Niveles de expresión para cada uno de los genes asociados a la resistencia a la roya de hoja en trigo. Los números en lo alto de la figura indican las diferentes líneas avanzadas de trigo y la variedad Monarca (M). Los números localizados debajo de cada fragmento corresponden al valor densitométrico de la expresión de cada gen, considerando como control de comparación relativa la intensidad del fragmento de 1650 pares de bases del marcador 1 kb Plus.
En el dendrograma generado (Figura 2) se observaron cuatro grupos definidos. En el primero se encontró a la variedad Monarca y las líneas reportadas como resistentes (357,394) y susceptibles (156,306). Lo anterior indica que presentaron un perfil de expresión similar; sin embargo, la ausencia de expresión de los genes RTH-27 y RTH-28 en las líneas 156 y 306 podría explicar su susceptibilidad; para ello se requiere hacer estudios funcionales de estos genes. El segundo grupo estuvo conformado por las líneas 223, 305, 334, 337, 344, 367, 381, 328 y 358, las cuales están identificadas como resistentes en pruebas de campo. Las líneas 157 (susceptible) y 399 (resistente) son completamente contrastantes de acuerdo con sus perfiles de expresión y los reportes en campo. Estos resultados sugieren que las diferencias en la transcripción de genes específicos y/o la combinación de ellos inducen respuestas diferenciales de resistencia o susceptibilidad ante la infección por roya.
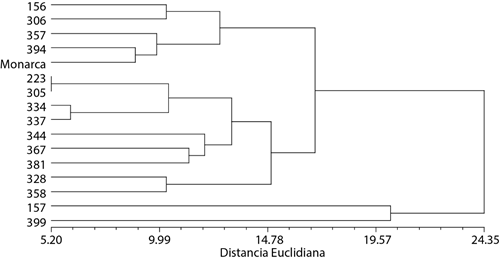
Figura 2 Dendrograma del perfil de expresión de los genes obtenidos de la biblioteca sustractiva supresiva en trigo.
Los resultados se cotejaron con los datos proporcionados por el programa de mejoramiento de trigo correspondientes al ciclo primavera-verano del año 2014. Para medir la incidencia a Puccinia triticina se tomaron cuatro lecturas y se reportan como porcentaje. La incidencia de la roya en condiciones de campo confirma la asociación entre la susceptibilidad o resistencia de los materiales con sus perfiles de expresión (Cuadro 3).
Cuadro 3 Porcentaje de incidencia de roya de la hoja en condiciones de campo.
| Línea | Lecturas | |||
|---|---|---|---|---|
| 1ra | 2da | 3ra | 4ta | |
| 156 | 5 | 30 | 30 | 60 |
| 157 | 5 | 30 | 30 | 60 |
| 223* | 10 | 30 | 30 | 40 |
| 305* | 5 | 20 | 30 | 40 |
| 306 | 1 | 20 | 30 | 40 |
| 328* | 1 | 1 | 5 | 20 |
| 334* | 1 | 5 | 10 | 20 |
| 337* | 1 | 10 | 10 | 20 |
| 344* | 1 | 10 | 10 | 20 |
| 357* | 1 | 20 | 20 | 40 |
| 358* | 1 | 20 | 20 | 30 |
| 367* | 1 | 20 | 20 | 30 |
| 381* | 1 | 1 | 1 | 5 |
| 394* | 1 | 5 | 10 | 30 |
| 399 | 1 | 10 | 10 | 20 |
*Materiales sobresalientes como resistentes con base en sus niveles de expresión.
DISCUSIÓN
La construcción de la biblioteca de ADNc permitió conocer los genes cuya síntesis de transcritos se induce cuando el trigo está sometido a la roya, e inferir su posible participación en la resistencia a este patógeno. La activación de la transcripción de los genes expresados diferencialmente es básica para la comprensión de los mecanismos moleculares que las plantas han desarrollado para defenderse de los patógenos o adversidades abióticas (Al-Taweel et al., 2011). Aunque no se conoce la función específica de un número importante de genes logramos determinar la relación que tienen con los demás. De esta manera encontramos genes relacionados con estrés y con diferentes procesos metabólicos, resultados semejantes a los de Manickavelu et al. (2010), ya que ellos también reportaron que el mayor número de genes expresados está relacionado con procesos biológicos. El gen RHT-5 tuvo mayor nivel de expresión en las líneas 328, 334, 344, 358, 367, 381 y 394, en etapa adulta.
La inducción de este gen, de acuerdo a información de la secuencia en el GenBank (datos no publicados), se ha reportado en plántulas, lo que indica que puede formar parte de un mecanismo de defensa que se activa y puede mantenerse activa en estado adulto. Igualmente, el gen RHT-4 presentó mayor expresión en las líneas 328 y 358, y se expresó en hoja de trigo 12 horas después de haberse inoculado con E. graminis (GenBank, datos no publicados) (Figura 1).
El gen RHT-11 se reportó previamente por Varshney et al. (2009) en una biblioteca de ADNc de raíz de garbanzo (Cicer arietinum). En este estudio se reporta que el genotipo utilizado (ICC 4958) es resistente a sequía moderada y se identificó un contig PAL (Fenilalanina amonio liasa) involucrado en la ruta de la biosíntesis de los fenilpropanoides en las plantas en respuesta al estrés biótico. La expresión de este gen fue más notable en las líneas 367 y 399. El gen RHT-28 codifica para una proteína que contiene un dominio Snf7 (Protein ID BAJ99418.1), cuya función es clasificar y transportar proteínas (Matsumoto et al., 2011). Las líneas 328 y 358 presentaron mayor nivel de expresión de este gen. El gen RHT19 codifica para una proteína relacionada con patogénesis en trigo en la etapa de plántula. Estas proteínas se acumulan en el tejido donde se localiza la infección, alterando el metabolismo de la planta, que se manifiesta por la aparición de lesiones necróticas. Este gen se expresó en varias líneas, tanto en susceptibles como en resistentes, lo que sugiere una respuesta generalizada de las líneas de trigo durante la patogénesis. Los retrotransposones son elementos transponibles que podrían funcionar como una respuesta adaptativa a factores de estrés (Sergeeva y Salina, 2011). Los genes RHT-2 y RHT-12 son similares al retrotransposón Sabrina, cuya función es desconocida; sin embargo, parece estar relacionado con la respuesta a la presencia del fitopatógeno.
La selección de genes de efectos menores es importante para generar nuevas variedades que tengan la capacidad de contener el ataque de P. triticina. De acuerdo con este criterio se proponen como genes blancos en el mejoramiento del trigo: al RHT-5, porque se observó en mayor nivel de expresión en las plantas resistentes, al RHT-4, porque es una proteína que se activa durante la patogénesis y a su vez podría activar otros mecanismos de defensa, al RHT-11, que potencialmente se asocia con el rompimiento de la epidermis por la liberación de las urediniosporas, al RHT-28, porque contiene un dominio conocido como Snf7 que está involucrado en la clasificación de proteínas y el transporte a la vacuola o lisosoma. La combinación de los genes es un factor importante ya que en algunos casos un gen puede incrementar la resistencia comparado con otras combinaciones.
CONCLUSIONES
La expresión de los genes en respuesta a la roya de la hoja sugiere que el metabolismo del trigo se modifica para defenderse del patógeno y adversidad ambiental. La acumulación diferencial de transcritos se relaciona con una mayor capacidad del genotipo para defenderse del ataque de la roya y la expresión de más genes, con un probable efecto menor, y con su importancia para manifestar mayor resistencia a la roya de la hoja. Las similitudes de los perfiles de expresión de genotipos resistentes con susceptibles indica que comparten un mecanismo de defensa parecido, pero la diferencia en uno o más genes (ejem. RHT-20, RHT-25, RHT-28) puede ser clave para inducir o reprimir la resistencia. Las líneas estudiadas presentan una mayor expresión en los genes RHT-2, RHT-4, RHT-5, RHT-25, RHT-28 y RHT-29 comparadas con la variedad Monarca F2007, lo que sugiere que el Programa de Trigo del INIFAP, a través de sus acciones de mejoramiento, está fijando genes en sus líneas avanzadas que confieren resistencia a la roya de la hoja. Los perfiles de expresión de estos genes empleados como marcadores de expresión tienen un uso potencial como marcadores de expresión en la selección de genotipos con mayor resistencia.











 nueva página del texto (beta)
nueva página del texto (beta)


