Introducción
Heliconia es un género que integra cerca de 220 especies (Kress et al., 2004), el cual se distribuye de manera natural desde Brasil hasta México (De Castro et al., 2007). En México la diversidad de Heliconia está representada por 14 especies (De Castro et al., 2007), las cuales habitan en condiciones de clima tropical y subtropical y de suelos ácidos a alcalinos (Ortiz-Curiel et al., 2015). Especies como H. latispatha, con alta variabilidad intraespecífica, proliferan desde Ecuador hasta el noroeste de México (Berry and Kress, 1991; De Castro et al., 2007), mientras que especies como H. uxpanapensis, endémica, (De Castro et al., 2007; Gutiérrez, 2000) están restringidas al sureste mexicano.
El estudio de la estructura del genoma mediante el análisis citogenético genera conocimiento básico para subsecuentes estudios enfocados al aprovechamiento de la diversidad. Con este tipo de estudios es posible conocer el número cromosómico y con ello el nivel de ploidía, además de detectar aneuploidías; asimismo, el análisis de la morfología de los cromosomas (tamaño absoluto y relativo, posición del centrómero, la ubicación y el número de satélites) aporta información del dinamismo cromosómico (Poggio et al., 2008). La información generada puede explicar parcialmente el polimorfismo interespecífico e intraespecífico (Alcántar, 2014).
En Heliconia, los estudios cariológicos se han centrado en especies de Centro y Sudamérica, pero no en especies nativas de México, mucho menos en endémicas. Andersson (1984), en su estudio de 29 especies originarias de Panamá, Colombia, Ecuador y Guayana Francesa, reportó que las especies estudiadas poseen un número cromosómico 2n = 2x = 24; resultados similares reportaron Guangsui et al. (2010) en ocho variedades de Heliconia. Por otra parte, en el Index to Plant Chromosome Numbers (IPCN) (Goldblatt y Johnson, 2010), de 36 especies de Heliconia enlistadas, todas presentan n = 12 cromosomas, con excepción de H. arabica Lam., que posee n = 11; por su parte, Do Nascimento et al. (2014), en H. bihai, originaria de Sudamérica, reportaron 2n = 22, mientras que Lee et al. (1994), en cultivares de H. psittacorum y Kaemwong and Eksomtramage (1998) en H. densiflora, reportaron 2n = 3x = 36.
Respecto a la morfología cromosómica, Andersson (1984) mencionó que los cromosomas son metacéntricos a submetacéntricos, con longitud de 0.7 a 1. 7 µm y sólo en algunas especies pudo observar satélites. De la misma manera, Do Nascimento et al. (2014) encontraron solamente cromosomas metacéntricos a submetacéntricos.
El estudio cromosómico en células somáticas se realiza mediante pretratamientos físicos o químicos. Una compactación moderada (prometafase) de los cromosomas hace posible localizar el centrómero, las constricciones secundarias y los satélites, con los cuales es posible determinar una serie de parámetros que otorgan una clasificación única a cada genoma (Stace, 2000).
Múltiples estudios utilizan medios químicos para el pretratamiento; no obstante, Schweizer (1973) y Lodish et al. (2013) mencionan que mediante la exposición de células somáticas a 4 ºC los microtúbulos se despolimerizan debido a que se afecta su ensamblaje y estabilidad y con ello se interrumpe el proceso mitótico. Kato (1997, 1999) en raíces de maíz (Zea mays L.), encontró que el frío como pretratamiento permitió la adecuada compactación y dispersión de los cromosomas, mientras que Mirzaghaderi (2010) utilizó agua fría en el pretratamiento de raíces de trigo (Triticum aestivum L.) y obtuvo de tres a cuatro veces más células en metafase que incluso con colchicina.
Para contribuir en el conocimiento cariológico de las especies de Heliconia nativas de México, el presente estudio tuvo por objetivo determinar el cariotipo de H. uxpanapensis (endémica), H. latispatha y H. stricta, originaria de Sudamérica.
Materiales y métodos
Material vegetal
Se estudiaron las especies H. uxpanapensis (endémica), H. latispatha y H. stricta cv. Iris, originaria de Sudamérica. El material vegetativo de las dos especies de Heliconia nativas, previo a los estudios citogenéticos, se corroboró a nivel de especie en el herbario UCAM de la Universidad Autónoma de Campeche, lugar donde se resguardan los ejemplares herborizados. Para el estudio se tomaron cinco rizomas por cada especie, cada rizoma de diferente población. El estudio cariológico se realizó en el laboratorio de citogenética del Colegio de Postgraduados, Campus Montecillo, México en los meses de enero a abril de 2017.
Preparación de las muestras
Debido a que las especies son tropicales y a que en el periodo en que se realizó el trabajo prevalece un clima frío (4 ºC ± 1), para inducir a los rizomas a la emisión de raíces se utilizó el protocolo propuesto por Do Nascimento et al. (2014), que consiste en colocarlos en baño María a 30 ºC.
Se colectaron raíces nuevas de 0.5 a 1 cm de longitud y se colocaron en tubos Eppendorf, éstos se llenaron con agua destilada y posteriormente se expusieron al frío para inducir células en metafase según la metodología de Kato (1997). Después de 8, 12, 16, 18 y 24 h se extrajeron las raíces y se fijaron en alcohol etílico y ácido acético glacial 3:1, en seguida se colocaron en refrigeración a 4 ºC. Después de 12 a 18 h las raíces se hidrolizaron por 10 min en ácido clorhídrico 1 N a 60 ºC, se sometieron a digestión enzimática con pectinasa 0.3 μM en baño María a 32 ºC por 2 h y se tiñeron con reactivo de Feulgen a 60 ºC por 2 min para favorecer la tinción. Para observar los cromosomas, un pequeño segmento del ápice de la raíz se colocó sobre el portaobjetos y se aplicó la técnica del aplastado (García, 1990). Los tiempos de exposición al frío se ajustaron hasta obtener células que permitieran visualizar sin ambigüedad la morfología de los cromosomas.
Recolección y análisis de la información
Las imágenes se capturaron con una cámara digital modelo MU-1000 (Marca AmScope, China). adaptada al microscopio. Cinco fotomicrografías de mejor resolución, de igual número de individuos por especie, se seleccionaron y procesaron con el software KaryoType (Altınordu et al., 2016) (disponible en http://mnh.scu.edu.cn/soft/blog/karyotype/). Este software, con base en la posición del centrómero en el cromosoma, calcula: 1) Longitud total = Brazo largo + Brazo corto (LT = BL + BC), 2) Índice centromérico = Brazo corto/Longitud total × 100 (IC = (BC/LT) × 100), 3) Relación de brazos = Brazo largo/Brazo corto (RB = BL/BC) y 4) Tipo de cromosoma, metacéntrico, submetacéntrico o subtelocéntrico (Levan et al., 1964). El programa proporciona también la fórmula cariotípica, la longitud del genoma haploide y el grado de asimetría de Stebbins (1971); esta última se basa en la variación intercromosomal (proporción de cromosomas grandes/pequeños) e intracromosomal (proporción de cromosomas con relación de brazos < 2:1 respecto al genoma diploide).
Resultados
El pretratamiento de las raíces con frío generó células en prometafase. El tiempo para la compactación óptima estuvo en función del genotipo; de esta manera, se requirieron 12, 13 y 14 h para H. latispatha, H. stricta y H. uxpanapensis, respectivamente. Las raíces más gruesas requirieron mayor tiempo para la óptima condensación.
En este trabajo se determinó por primera vez el cariotipo de H. uxpanapensis. Se encontró que esta especie presenta 2n = 2x = 24 cromosomas (Cuadro 1), los cuales tienen longitudes de entre 1.0 y 2.76 µm (Cuadro 2) y aun cuando los cromosomas se observaron en prometafase, con base en la clasificación de Stace (2000), éstos van de pequeños a medianos. Los satélites se observaron en el segundo par cromosómico, (Figura 1A), cuya longitud total haploide resultó de 22.21 ± 0.1 µm (Cuadro 2), mientras que la asimetría del genoma, debido a que el tamaño de los cromosomas oscila de pequeño a mediano y al reducido número de cromosomas metacéntricos, se caracteriza como asimétrico tipo 3A (Cuadro 2).
Cuadro 1 Parámetros cariotípicos del genoma haploide de tres especies de Heliconia.
| ChrH | H. uxpanapensis | H. latispatha | H. stricta | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LT (µm) | IC (%) | RB (µm) | Tipo | LT (µm) | IC (%) | RB (µm) | Tipo | LT (µm) | IC (%) | RB (µm) | Tipo | |||
| 1 | 2.76 ± 0.2 | 20.31 ± 2.2 | 3.92 ± 0.5 | st | 3.41 ± 1.2 | 16.37 ± 1.9 | 5.11 ± 0.7 | st | 3.03 ± 0.2 | 31.10 ± 4.4 | 2.21 ± 0.46 | sm | ||
| 2 | 2.33 ± 0.3 | 29.33 ± 10.1 | 2.41 ± 1.2 | sm | 2.89 ± 1.1 | 28.24 ± 10.0 | 2.54 ± 1.3 | sm | 2.58 ± 0.1 | 33.93 ± 4.6 | 1.95 ± 0.41 | sm | ||
| 3 | 2.22 ± 0.1 | 25.08 ± 2.4 | 2.99 ± 0.4 | sm | 2.60 ± 0.1 | 31.12 ± 10.3 | 2.21 ± 1.1 | sm | 2.42 ± 0.1 | 36.28 ± 6.7 | 1.76 ± 0.52 | sm | ||
| 4 | 2.12 ± 0.1 | 28.09 ± 8.9 | 2.56 ± 0.9 | sm | 2.53 ± 0.7 | 24.49 ± 7.3 | 3.08 ± 1.3 | st | 2.36 ± 0.1 | 41.09 ± 5.9 | 1.43 ± 0.37 | m | ||
| 5 | 2.03 ± 0.1 | 30.66 ± 5.9 | 2.26 ± 0.6 | sm | 2.34 ± 0.6 | 22.08 ± 4.0 | 3.53 ± 0.8 | st | 2.32 ± 0.1 | 37.39 ± 7.9 | 1.67 ± 0.63 | m | ||
| 6 | 1.97 ± 0.1 | 23.60 ± 2.2 | 3.24 ± 0.4 | st | 2.30 ± 0.6 | 28.21 ± 7.4 | 2.54 ± 0.9 | sm | 2.25 ± 0.0 | 41.61 ± 3.9 | 1.40 ± 0.22 | m | ||
| 7 | 1.91 ± 0.1 | 25.34 ± 1.9 | 2.95 ± 0.3 | sm | 2.20 ± 0.6 | 29.66 ± 12.0 | 2.37 ± 1.1 | sm | 2.12 ± 0.1 | 35.10 ± 4.4 | 1.85 ± 0.39 | sm | ||
| 8 | 1.75 ± 0.1 | 23.98 ± 5.7 | 3.17 ± 1.2 | st | 1.91 ± 0.5 | 25.64 ± 6.2 | 2.90 ± 0.8 | sm | 2.03 ± 0.2 | 42.31 ± 7.5 | 1.36 ± 0.51 | m | ||
| 9 | 1.63 ± 0.1 | 37.22 ± 8.4 | 1.69 ± 0.7 | m | 1.82 ± 0.5 | 29.29 ± 7.1 | 2.41 ± 0.7 | sm | 1.72 ± 0.1 | 37.34 ± 5.1 | 1.68 ± 0.35 | m | ||
| 10 | 1.35 ± 0.1 | 40.72 ± 7.6 | 1.46 ± 0.5 | m | 1.72 ± 0.5 | 34.04 ± 9.6 | 1.94 ± 0.7 | sm | 1.59 ± 0.1 | 40.69 ± 4.2 | 1.46 ± 0.23 | m | ||
| 11 | 1.14 ± 0.1 | 45.94 ± 1.1 | 1.18 ± 0.1 | m | 1.50 ± 0.4 | 45.72 ± 1.1 | 1.19 ± 0.1 | m | 1.51 ± 0.2 | 41.43 ± 7.1 | 1.41 ± 0.49 | m | ||
| 12 | 1.00 ± 0.1 | 43.68 ± 3.6 | 1.29 ± 0.2 | m | 1.36 ± 0.4 | 42.34 ± 2.5 | 1.36 ± 0.1 | m | 1.44 ± 0.2 | 41.96 ± 3.4 | 1.38 ± 0.21 | m | ||
ChrH: cromosoma haploide, LT: longitud total, IC: índice centromérico, RB: relación de brazos, st: subtelocéntrico, sm: submetacéntrico, m: metacéntrico.
Cuadro 2 Parámetros y fórmula del cariotipo de tres especies de Heliconia.
| Especie | 2n | LTH (µm) | Fórmula 3 cariotípica† | AC |
|---|---|---|---|---|
| H. uxpanapensis | 24 | 22.21 ± 0.1 | 2n = 2x = 8m + 10sm(2sat) + 6st | 3A |
| H. latispatha | 24 | 26.56 ± 0.6 | 2n = 2x = 4m + 14sm + 6st(2sat) | 3A |
| H. stricta | 24 | 25.37 ± 0.1 | 2n = 2x = 16m + 8sm(1sat) | 2A |
†Los datos entre paréntesis en la fórmula cariotípica indican número de satélites y tipo de cromosoma que lo porta; 2n: número cromosómico diploide;
LTH: longitud total del genoma haploide; AC: asimetría del cariotipo con basa en la variación inter e intracromosomal.
Heliconia latispatha presenta un número cromosómico 2n = 2x = 24, el tamaño de sus cromosomas oscila de pequeños a medianos y su grado de asimetría corresponde a 3A (Cuadro 1). La principal diferencia de este cariotipo con respecto al de H. uxpanapensis radica en que los satélites se localizan en diferentes cromosomas (Figura 1B).
El número cromosómico de H. stricta es 2n = 2x = 24, el cual es idéntico al de las otras dos especies estudiadas. En el presente estudio se observó la presencia de al menos un satélite en el complemento cromosómico, el cual se localiza en el brazo corto de un cromosoma submetacéntrico (Figura 1C). La longitud de los cromosomas oscila entre 1.44 y 3.03 µm (Cuadro 2), mientras que la simetría, a diferencia de las dos especies nativas, presenta mayormente cromosomas metacéntricos, por lo que presenta asimetría clase 2A.
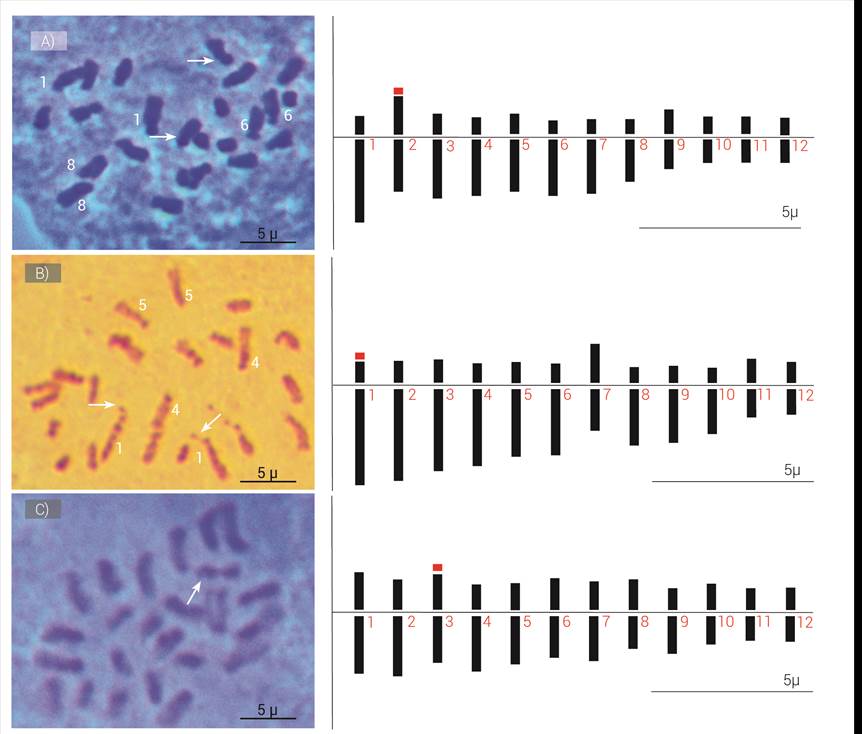
Figura 1 Cromosomas en prometafase y su respectivo ideograma. A) H. uxpanapensis, B) H. latispathay C) H. stricta. Las flechas señalan cromosomas con satélites. Los números dentro de la imagen indican cromosomas subtelocéntricos.
Discusión
El número cromosómico de H. uxpanapensis, que se identificó en este trabajo como 2n = 24, coincide con el de H. latispatha, mientras que, el número cromosómico de H. latispatha coincide con lo que obtuvo Andersson (1984) en ejemplares de la misma especie, pero originarios de Panamá y Ecuador. En a H. stricta, se observaron 2n = 24 cromosomas, como lo reportaron Guangsui et al. (2010).
Los cariotipos de H. uxpanapensis y H. latispatha presentaron similitudes en cuanto a la presencia de cromosomas metacéntricos, submetacéntricos y subtelocéntricos (Cuadro 2), además de la presencia de satélites en el brazo corto; sin embargo, la principal diferencia entre ambos es la ubicación del satélite. Estos resultados, en conjunto, son diferentes a los que encontró Andersson (1984) en las 29 especies de Centroamérica y Sudamérica; dicho autor reportó cromosomas metacéntricos y submetacéntricos únicamente; además, observó satélites en los brazos largos.
En H. stricta cv. Iris solamente se observaron cromosomas metacéntricos y submetacéntricos, lo cual coincide con los estudios de Guangsui et al. (2010); sin embargo, en el presente estudio fue posible observar el satélite en un cromosoma de cada célula observada.
Destaca la presencia de cromosomas subtelocéntricos en las especies de México aquí estudiadas, pues hasta ahora, ningún estudio ha reportado este tipo de cromosomas en especies del género Heliconia. Costa et al. (2016) en 18 genotipos de Heliconia, entre ellos triploides, corroboraron la presencia exclusiva de cromosomas metacéntricos y submetacéntricos.
Los índices de asimetría 2A de la especie H. stricta y 3A de H. uxpanapensis y H. latispatha, aducen diferencias entre cariotipos, lo cual significa que el cariotipo de H. stricta es más simétrico. Stebbins (1971), Schubert (2007) y Medeiros-Neto et al. (2017) mencionan que los cariotipos asimétricos son resultado del dinamismo cromosómico, como consecuencia de inversiones paracéntricas o pericéntricas, por lo que al final de los re-arreglos cromosómicos el centrómero puede posicionarse hacia los extremos, lo que genera cromosomas subtelocéntricos o telocéntricos.
Con base en estas afirmaciones, se deduce que el cariotipo de las especies H. uxpanapensis y H. latispatha ha sufrido re-arreglos y los factores antes citados pueden contribuir a la asimetría observada. En relación con la posición de los satélites, Schubert (2007) indica que éstos son de carácter altamente polimórfico, y su movilidad dentro del genoma obedece a factores ajenos que aún se desconocen.
Stace (2000), por su parte, afirmó que los genomas más asimétricos son producto de adaptaciones a las condiciones ambientales; en este sentido, Flores-Maya et al. (2006), en Quercus rugosa, encontraron correlación entre su cariotipo asimétrico y la adaptación que muestran a ambientes menos favorables, contrario a Q. elliptica, que solamente prospera en hábitats bien conservados y posee un cariotipo simétrico. Lo anterior puede tener cierta relación con las especies H. uxpanapensis y H. latispatha que prosperan en la zona de transición del trópico a neártico, mientras que las especies de Heliconia citadas en la literatura, entre ellas H. stricta, son originarias de zonas tropicales de los países centros de origen como Colombia y Ecuador. Estudios más intensivos sobre el cariotipo de las especies de México deben realizarse para confirmar esta aseveración.
La procedencia de los ejemplares podría ser una de las posibles causas de que se haya observado un solo cromosoma con satélite en H. stricta. Ramsaroop (1995) señala que el ahora cv. Iris se colectó en ambiente silvestre con base en el potencial comercial de su inflorescencia; observaciones en campo y estudios de laboratorio indican que este material no produce frutos debido a que presenta anormalidades en sus estructuras sexuales, entre ellas, aborto del polen y aberraciones en los pistilos y estigmas, lo cual podría estar relacionado con anormalidades cromosómicas. Swanson (1968) menciona que todos los genomas haploides tienen al menos un cromosoma con constricción secundaria, por lo que en el genoma diploide del cv. Iris es razonable esperar que se observe un segundo satélite. Con base en lo anterior, no se descarta que sea necesario realizar adecuaciones más puntuales en el pretratamiento a esta especie para observar de manera más detallada sus cromosomas.
Referente a la longitud de los cromosomas, los valores que se encontraron en el presente estudio fueron superiores a los que reportaron Andersson (1984) en las 29 especies y Guangsui et al. (2010) en ocho variedades. Debido a que la longitud es un parámetro que puede estar condicionado por el pretratamiento, en este estudio se puede atribuir al agua fría la condensación moderada de los cromosomas y con ello, la visualización de los satélites. Mirzaghaderi (2010) en su estudio sobre pretratamientos, que incluyeron medios físicos o químicos y la combinación de ambos para obtener células en metafase en variedades de trigo, encontró que el índice de metafases con agua fría fue de tres a cuatro veces superior al de otros tratamientos. Kato (1997), con raíces de maíz, encontró que el agua fría propició índices de metafases similares al uso de medios químicos, incluso al de colchicina.
Debido a que en la literatura existe información poco precisa sobre la caracterización cariotípica del género Heliconia, es necesario realizar trabajos complementarios con otras especies de México mediante la evaluación de pretratamientos que eluciden las características cromosómicas con mayor precisión.
Conclusiones
Heliconia uxpanapensis, H. latispatha y H. stricta poseen un número cromosómico 2n = 2x = 24, al igual que la mayoría de las especies de Sudamérica hasta ahora estudiadas; no obstante, las especies nativas H. uxpanapensis, H. latispatha difieren por la presencia de cromosomas subtelocéntricos, lo cual sugiere posibles rearreglos cromosómicos como resultado de un genoma en constante evolución, mientras que H. stricta presenta cromosomas metacéntricos en la mayoría de los casos.











 nueva página del texto (beta)
nueva página del texto (beta)


