Introducción
La jamaica (Hibiscus sabdariffa L.) es un cultivo cuyo potencial radica en su consumo en forma de bebida obtenida a partir de los cálices de la planta, en sus propiedades fitoquímicas y los usos farmacológicos de interés en la nutrición humana y en la industria (Sáyago-Ayerdi et al., 2007). México se encuentra entre los 10 principales países productores donde el estado de Guerrero constituye la principal zona de cultivo (SIAP-SAGARPA, 2014). En esta zona la enfermedad más importante es causada por el oomyceto P. parasitica con incidencias de hasta 40 % con afectación al cultivo en su calidad y rendimiento (Hernández y Romero, 1990).
Actualmente no existen variedades de jamaica resistentes a P. parasitica, por lo que su manejo se basa principalmente en el uso de fungicidas los cuales no han sido completamente efectivos, aunado al impacto negativo que implica su uso en el ambiente. Por ello es necesario buscar nuevas estrategias eficientes y sostenibles que se integren al manejo para el control de la enfermedad ocasionada por este oomyceto. El uso de antagonistas microbianos nativos en la rizosfera de las plantas como agentes de biocontrol y promotores de crecimiento vegetal, puede ser una alternativa para reducir el uso de productos químicos en la agricultura. La rizosfera incluye a las raíces de la planta y al suelo que las rodea, y se define como el volumen de suelo que está directamente influenciado por las propiedades físicas, químicas y biológicas de la raíz.
El término "rizobacterias" implica a un grupo de bacterias de la rizosfera que compiten en la colonización del entorno de la raíz y promueven un beneficio para la planta (Prashar et al., 2014). Se ha documentado que los patrones de exudación de un amplio rango de compuestos de carbono exudados por la raíz de las plantas, modifican las propiedades físicas y químicas del suelo que la rodea e influencian la estructura de sus comunidades bacterianas y de la rizosfera (Bais et al., 2006). Las plantas cultivadas han desarrollado una estrategia de estimular y promover la colonización de grupos específicos de microorganismos antagonistas nativos en la rizosfera, como primera línea de defensa contra patógenos del suelo (Latz et al., 2015).
Se ha demostrado que las comunidades bacterianas en la rizosfera de muchas especies de plantas producen un efecto benéfico en el crecimiento y sanidad de las mismas, directa e indirectamente, y que dichas comunidades bacterianas están fuertemente influenciadas en su composición por el suelo, especie y genotipo de la planta (Berg y Smalla, 2009) y clones (Liu et al., 2014). Las comunidades nativas son una fuente de microorganismos útiles para aplicaciones biotecnológicas de formulaciones como agentes de biocontrol o para la síntesis de nuevos compuestos bioactivos de interés en el área médica, veterinaria e industrial (Aranda et al., 2011).
La rizosfera de jamaica puede albergar poblaciones bacterianas cultivables nativas, bien adaptadas, con diversidad metabólica, alto potencial como antagonistas contra P. parasitica y promotores del crecimiento de la planta. En la actualidad se carece de conocimiento de las comunidades bacterianas en la rizosfera de este cultivo, por lo que el objetivo de esta investigación se centró en las poblaciones bacterianas cultivables nativas en la rizosfera de jamaica en diferentes localidades del estado de Guerrero con potencial antagonista contra P. parasítica, y la caracterización in vitro de su actividad antimicrobiana.
Materiales y métodos
Aislamiento de bacterias nativas de la rizosfera de Jamaica
Muestras de raíz de jamaica var. Tecoanapa se colectaron a una profundidad de 30 cm del suelo durante el ciclo de cultivo 2012, en cinco localidades del municipio de Tecoanapa, Guerrero, México: Las Ánimas (16º 57' 97" N; 099º 19' 37" O), Saucitos (16º 58' 68" N; 099º 18' 52" O), Pueblo Largo (16º 59' 59" N; 099º 15' 15" O), Santa Rosa (16º 57' 60" N; 099 17' 22" O) y Xalpatláhuac (17º 0' 41.31" N; 99º o 20' 04" O). Los estados fenológicos del cultivo en que se tomaron las muestras fueron: 1) crecimiento inicial, 2) desarrollo de botones florales y 3) floración.
Se seleccionaron al azar cinco plantas sanas, cuyas muestras de raíz se tomaron con suelo naturalmente adherido a ellas y se preparó una muestra compuesta mediante la mezcla de raíces y suelo rizosférico por localidad y estado fenológico. Se hizo un análisis físico y químico del suelo rizosférico en el laboratorio de Química de Suelos del Colegio de Postgraduados, Campus Montecillo. De cada muestra se utilizaron 2.5 g de raíces y se pusieron en 20 mL de agua destilada estéril que se mantuvieron en agitación y ultrasonido por 10 min.
Las suspensiones obtenidas fueron diluidas en serie desde 10-1 hasta 10-4 y se sembraron 100 μL en medio R2A agar (Aranda et al., 2011) e incubadas a 28 ± 1 °C por 48 h con tres repeticiones por dilución. Se cuantificó el número de colonias totales y de éstas se seleccionaron treinta morfotipos diferentes por localidad y estado fenológico, que fueron considerados representativos del total de bacterias cultivables de las que se generó una colección de 450 bacterias. El número de bacterias se expresó como log UFC g-1 de raíz, con el que se efectuó el análisis de la varianza (ANOVA) por localidad y por estado fenológico, y la prueba de comparación de medias por el método de Tukey (α = 0.05). El análisis estadístico se desarrolló con el programa SAS System para Windows versión 9.0.
Bioensayo de antagonismo in vitro contra P. Parasitica
El efecto inhibitorio de las cepas sobre una cepa virulenta de P. parasitica previamente identificada y aislada del cultivo de jamaica en Tecoanapa, Guerrero, se hizo mediante cultivo dual in vitro en medio de cultivo Waksman agar (Berg et al., 2006). En el centro de cada placa con medio de cultivo, se colocó un disco de agar (5 mm diámetro) con micelio del oomyceto. A 2.5 cm del disco se sembraron en punto cuatro bacterias y de cada una se depositaron 3 μL de suspensión bacteriana con 3.0 x 106 UFC. Las placas sembradas se incubaron a 28 ± 1 °C por 5 a 7 d. Como testigo se establecieron placas inoculadas solo con el oomyceto. Cada cepa bacteriana se evaluó con tres repeticiones, y las que registraron la mayor zona de inhibición promedio (mm) se seleccionaron como las de mayor antagonismo al oomyceto. La zona de inhibición considerada fue un área bien definida entre el margen de avance del crecimiento de micelio y las colonias bacterianas desarrolladas.
Caracterización in vitro en la producción de metabolitos
Las 18 cepas que mostraron el mayor antagonismo in vitro contra P. parasitica se caracterizaron cualitativamente por triplicado para la producción in vitro de metabolitos involucrados en la promoción de crecimiento y antagonismo, conforme a los protocolos descritos previamente por Hantsis-Zacharov y Halperin (2007) para la actividad lipolítica y proteolítica, por Schwyn y Neilands (1987) para la producción de sideróforos, por Frey-Klett et al. (2005) para ácido indol-3-acético (AIA), y por El-Yazeid et al. (2007) para solubilización de fosfato mineral.
Amplificación del gen 16S rADN, secuenciación e identificación de cepas antagonistas
La identificación de las cepas bacterianas se hizo por medio de la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) con la secuenciación del gen 16S rADN con ayuda de los iniciadores 27F y 1492R que amplifican un fragmento aproximado de 1500 pares de bases (pb). La extracción de ADN genómico, amplificación y secuenciación se hizo en Macrogen (DNA Sequency Service. Korean Biotechnology Company), y luego fueron alineadas con secuencias depositadas en el GenBank del Centro Nacional de Biotecnología de los Estados Unidos (NCBI) (http://www.ncbi.nlm.nih.gov/BLAST/).
Resultados y discusión
Densidad de poblaciones bacterianas cultivables en la rizosfera de Jamaica
Los resultados de los valores medios de las bacterias cultivables (BC) en la rizosfera de jamaica mostraron que la población de BC estuvo en intervalos de log 3.50 y 6.56 UFC g-1 de raíz, en las cinco localidades muestreadas. Estos resultados son similares con los citados por Berg et al. (2006) quienes estimaron una densidad en intervalos de log 7.63 ± 0.18 UFC g-1 de raíz en fresa (Fragaria X ananassa Duch). Los valores totales de la población bacteriana en UFC g-1 de raíz de jamaica fueron diferentes en las cinco localidades analizadas (Cuadro 1).

Medias con letras iguales no son estadísticamente diferentes. DMSH = 0.251 para comparaciones entre localidades (Tukey, 0.05).
Cuadro 1: Valores promedio de la población bacteriana total registrada en las cinco localidades del municipio de Tecoanapa, Gro. México, y comparación estadística de medias.
El estudio comparativo entre las etapas fenológicas del cultivo mostró que la población de BC en UFC g-1 de raíz en la etapa de crecimiento inicial fue mayor (P > 0.05) al promedio registrado en la etapa de desarrollo de botones florales; la variable tuvo sus valores más altos en los sitios Las Ánimas, Saucitos, Santa Rosa y Pueblo Largo, donde hubo mayor densidad de BC en la etapa de crecimiento inicial (Cuadro 2). En contraste, en Xalpatláhuac se presentó la mayor población bacteriana de raíz en la etapa de desarrollo de botones florales (Figura 1).

Medias con letras iguales no son estadísticamente diferentes. DMSH = 0.159 para comparaciones entre estados fenológicos (Tukey, 0.10).
Cuadro 2: Valores promedio de la población bacteriana total registrada en tres estados fenológicos de plantas de jamaica (Hibiscus sabdariffa L.), y comparación estadística de medias.

Figura 1: Promedios de poblaciones bacterianas totales registradas en cinco localidades del municipio de Tecoanapa, Gro. México, y su relación con tres estados fenológicos de jamaica (Hibiscus sabdariffa L.). Medias con letras iguales en cada localidad no son estadísticamente diferentes (Tukey, 0.10). Las medias solo son comparables dentro de cada localidad.
Las diferencias en densidad poblacional de las bacterias cultivables (BC) pueden ser atribuidas a las características físicas y químicas del suelo entre las localidades estudiadas, y a las características fenotípicas y genéticas (var. Tecoanapa) de la jamaica en los patrones cualitativos y cuantitativos de exudados de la rizosfera a través de las diferentes etapas fenológicas del cultivo. Rumberger et al. (2007) también observaron diferencias significativas en las poblaciones microbianas de la rizosfera en función del genotipo de manzano (Malus X domestica Borkh) y de las características físicas y químicas del suelo. Otras investigaciones han documentado que la composición de los exudados radicales es dependiente del estado fisiológico de la especie de planta y de los microorganismos que interactúan (Marques et al., 2014), y que la densidad de las poblaciones bacterianas varían entre estados fenológicos de las plantas por los cambios que ocurren en los exudados en las raíces, e incluso que la cantidad y calidad de estos exudados también es dependiente del estado fisiológico de la especie de planta (Kang et al., 2010).
Los coeficientes de correlación de Pearson entre las características físicas y químicas del suelo (Cuadro 3) y el número de UFC g-1 de raíz mostraron que Mg (r = 0.92) y P (r = 0.80) tienen una correlación significativa con los valores de UFC. En Las Ánimas el contenido de Mg y P en el suelo fue más alto que el registrado en Xalpatláhuac, y correlaciona con un incremento en la densidad de bacterias totales en la rizosfera. Estos resultados difieren de otras investigaciones debido a que el pH del suelo es uno de los factores que influencia la densidad y estructura de las comunidades bacterianas de la rizosfera (Hinsinger et al., 2009; Ibekwe et al., 2010); en este estudio se encontró que la cantidad de P y Mg en el suelo afectan significativamente la densidad de bacterias cultivables en la rizosfera de jamaica.
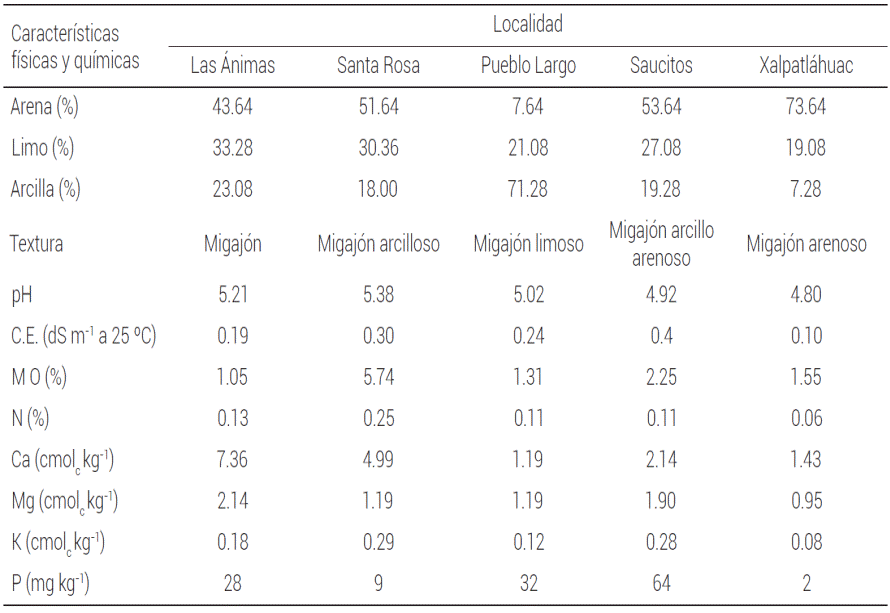
Cuadro 3: Características físicas y químicas del suelo con crecimiento de jamaica (Hibiscus sabdariffa L.) en cinco localidades del municipio de Tecoanapa, Gro. México.
Estudios previos han demostrado la influencia del P sobre la densidad y fluctuaciones de las poblaciones bacterianas en la rizosfera de otras especies de plantas (Marschner et al., 2004). Sin embargo, muy pocos estudios han aportado evidencia sobre el efecto del Mg sobre estas comunidades, por lo que la presente investigación contribuye a demostrar el papel del Mg edáfico en las poblaciones microbianas cultivables en la rizosfera.
Inhibición in vitro de P. Parasitica
De 450 aislamientos bacterianos, 72 inhibieron el crecimiento micelial in vitro de P. parasítica, y el promedio de inhibición radial (mm) indicó que 18 cepas expresaron el mayor grado de antagonismo. La distribución de estas 18 cepas por sitio fue: 33 % de las Ánimas, 27.7 % de Xalpatláhuac, 22.2 % de Santa Rosa, 11.1 % de Pueblo Largo, y 5.7 % de Saucitos. La producción de metabolitos in vitro mostró que las 18 cepas (100 %) expresaron actividad proteolítica y lipolítica; 16 cepas (88.8 %) produjeron sideróforos; 8 cepas (44.4 %) produjeron AIA; y 13 cepas (72.2 %) solubilizaron fosfatos (Cuadro 4).

+ = actividad metabólica positiva; - = actividad metabólica negativa; Tpb = tamaño en pares de bases; LIP = actividad lipolítica; PRO = actividad pro teolítica; SID = producción de sideróforos; AIA = producción de ácido indol-3-acético; FOS = solubilización de fosfato mineral.
Cuadro 4: Identificación de cepas de la rizosfera de jamaica que expresan el mayor grado de antagonismo in vitro contra P. parasitica, y caracterización en la producción de los metabolitos evaluados.
Identificación de bacterias antagonistas
El alineamiento de las secuencias de las 18 cepas bacterianas en el GenBank mostró que pertenecen a los géneros Serratia, Pseudomonas y Stenotrophomonas, con una similitud de 99 %. Todas ellas son Gram negativas dentro del phylum Proteobacteria, en las familias Enterobacteriaceae, Pseudomonadaceae y Xanthomonadaceae respectivamente. Serratia fue el taxón antagonista identificado con mayor proporción entre los sitios de muestreo con 50 % (nueve cepas), seguido de Pseudomonas 38.9 % (siete cepas) y Stenotrophomonas 11.1 % (dos cepas).
Dentro de Serratia, 88.8 % se identificaron como Serratia marcescens y 11.2 % como Serratia sp. El género Pseudomonas mostró 14.3 % identificadas como Pseudomonas fluorescens y 85.7 % Pseudomonas sp. En el caso de Stenotrophomonas las cepas no pudieron ser identificadas a nivel de especie (Cuadro 4). Los resultados mostraron que Serratia, Pseudomonas y Stenotrophomonas son las rizobacterias nativas en el cultivo de jamaica con mayor grado de antagonismo in vitro contra P. parasitica.
El género Serratia cada vez despierta más el interés como agente de control biológico, que se encuentra como saprófito en el suelo, agua y en las raíces de plantas. Particularmente, S. marcescens se considera con un alto potencial de biocontrol a oomycetos (Roberts et al., 2007), hongos (Someya et al., 2002); bacterias (Czajkowsky et al., 2012) y nematodos fitopatógenos (Almaghrabi et al., 2013). Estos resultados coinciden con los obtenidos por Queiroz y De Melo (2006), quienes reportaron que S. marcescens posee el mayor grado de antagonismo in vitro al inhibir el crecimiento micelial de P. parasitica; además tiene la capacidad de reducir en 50 % la infección de este patógeno, inducir el crecimiento de las plantas y suprimir a P. parasitica en condiciones de campo en plántulas de cítricos. S. plymuthica, es otra especie con potencial para uso en biocontrol (Weise et al., 2014).
Pseudomonas constituye uno de los grupos de bacterias más diverso y significativo del planeta. Son bacterias cosmopolitas en suelos agrícolas y con un alto grado de adaptación en el ambiente rizosférico. Miembros de este género tienen potencial como agentes de biocontrol o como promotor de crecimiento de plantas, y naturalmente intervienen en la supresión contra fitopatógenos de suelos (Selvakumar et al., 2015).
Los resultados de la eficiencia en la inhibición in vitro a P. parasitica en esta investigación están respaldados con los resultados en otras investigaciones, lo que demuestra la eficiencia en campo de Pseudomonas como agente de control biológico contra hongos del suelo (Yasmin et al., 2015), particularmente contra especies de Phytophthora (Sang et al., 2008) y bacterias fitopatógenas (Cigna et al., 2015). Diversas cepas de P. fluorescens han mostrado especial eficiencia por competencia y antagonismo en el control de fitopatógenos de raíz y alta agresividad para colonizar la rizosfera, y para inducir resistencia sistémica en la planta (Maldonado-González et al., 2015); la prevalencia de ciertas cepas de P. fluorescens en la rizosfera de especies de plantas, se ha atribuido a la presencia de carbohidratos de bajo peso molecular y ácidos orgánicos que favorecen la colonización eficiente de este grupo de bacterias (Kravchenko et al., 2003).
El género Stenotrophomonas incluye ocho especies, con predominio de la especie S. maltophilia la cual está estrechamente asociada con plantas y es considerada una excelente colonizadora de la rizosfera. Se ha encontrado en la rizosfera de cultivos como papa (Solanum tuberosum L.), nabo (Brassica rapa L.), arroz (Oryza sativa L.), orquídea (Oncidium cavendishianum L.), café (Coffea arabica L.), vid (Vitis vinifera L.) (Berg et al., 2010). Stenotrophomonas es motivo de estudios en la interacción benéfica como promotor de crecimiento y en su importancia como una nueva herramienta en la biotecnología y microbiología aplicada en la agricultura, y ha sido citado como efectivo agente de biocontrol de oomycetos (Jin et al., 2011), bacterias (Elhalag et al., 2015), y nematodos fitopatógenos (Huang et al., 2009), mediada por una eficiente producción de enzimas quitinolítcas, gluconolíticas, lipolíticas y proteolítcas.
El perfil metabólico del género Stenotrophomonas coincide con los resultados en este estudio de las cepas aisladas de la rizosfera de jamaica, ya que todas mostraron actividad proteolítica y lipolítica in vitro (Cuadro 4). Pliego et al. (2007) encontraron que Pseudomonas y Stenotrophomonas fueron los antagonistas más abundantes en las raíces de aguacate (Persea americana L.), y con alta eficiencia como agentes de biocontrol a P. cinammomi. Por su parte, Kobayashi et al. (1995) demostraron que S. marcensces y S. maltophilia fueron los antagonistas con el mayor grado de supresión del patógeno del suelo Magnaporthe poae, con niveles de supresión mayores a 70 y 50 % respectivamente.
Es de resaltar que, en esta investigación, Stenotrophomonas fue aislada solamente en la localidad de Xalpatláhuac en plantas de jamaica en la etapa de floración y no en los otros sitios de muestreo a través de diferentes estados fenológicos del cultivo. Es conocido que las características físicas y químicas del suelo, así como los cambios en los patrones de exudación en las raíces que dependen del estado fisiológico de la especie de planta, tienen efecto en la composición y abundancia relativa de poblaciones bacterianas. Lo anterior puede conducir a una selección específica de poblaciones microbianas en la rizosfera.
En el sitio de Xalpatláhuac existen diferencias principalmente en contenido de P y Mg del suelo con el resto de las localidades, lo cual podría sugerir que también son factores que promuevan una selección específica de ciertas poblaciones bacterianas, lo que permite explicar la presencia de Stenotrophomonas únicamente en ese sitio y estado fenológico de la planta. La anterior correlación se ha observado en poblaciones de Pseudomonas fluorescentes antagonistas, cuya frecuencia de aislamiento en un genotipo de maíz (Zea mays L.) fue significativamente diferente en tres localidades que difieren en características físicas y químicas del suelo y en el estado fenológico de la planta (Picard y Bosco, 2006).
La selección específica y sobrevivencia de S. maltophilia en función de la especie de la planta y de los patrones diferenciales de aminoácidos exudados en la raíz, se ha documentado en plantas como judía (Phaseolus vulgaris L.), maíz y jitomate (Solanum lycopersicum L.), que revela que la metionina favorece el crecimiento de esta bacteria (Elhalag et al., 2015). La interacción específica de las comunidades bacterianas con estos factores resalta la importancia del estudio de cepas nativas en la rizosfera y en regiones productoras de un cultivo en particular, en donde se esperaría obtener cepas con una ventaja de adaptación, colonización y competencia, por encima de otras cepas introducidas y con diferente origen y patosistema, si se pretenden utilizar como agentes de biocontrol o como biofertilizante.
Caracterización in vitro en la producción de metabolitos
La caracterización in vitro de las 18 cepas bacterianas resultó en la clasificación de seis cepas (33.3 %) (cuatro cepas de Serratia marcensces y dos de Pseudomonas sp.) que expresaron las cinco actividades evaluadas (Cuadro 4). El éxito en la supresión de patógenos del suelo se ha atribuido a la expresión de características multifuncionales del agente de biocontrol en la interacción planta-rizobacteria-patógeno. En este estudio se identificaron seis cepas de la rizosfera de jamaica que expresaron actividad lipolítica, proteolítica y producción de sideróforos, que intervienen directamente en el antagonismo y competencia, solubilización de fosfatos y producción de AIA, que tienen relevancia en la absorción de nutrimentos, promoción del crecimiento e inducción de resistencia sistémica en la planta.
Por lo anterior, estas representan cepas nativas con origen en la rizosfera de jamaica, bien adaptadas al ambiente, multifuncionales metabólicamente y con potencial para su uso como agentes de biocontrol, promotores de crecimiento o inoculantes en el cultivo de jamaica, que deberían ser evaluadas en condiciones de campo para confirmar su potencial. La presente investigación representa la primera información sobre las comunidades bacterianas cultivables nativas asociadas a la rizosfera de jamaica y su potencial antagonista contra P. parasitica.
Conclusiones
Se puede inferir que las poblaciones bacterianas presentes en la rizosfera de jamaica varían de acuerdo con la etapa fenológica de la planta y con las características nutricionales del suelo ya que estas poblaciones bacterianas son afectadas por los contenidos de P y Mg del suelo. Se identificaron 18 cepas bacterianas nativas de la rizosfera de jamaica dentro de los géneros Serratia, Pseudomonas y Stenotrophomonas que mostraron capacidad para inhibir el desarrollo in vitro de P. parasítica, y seis de ellas expresaron la producción de múltiples metabolitos involucrados en el antagonismo o promoción del crecimiento de las plantas.











 nueva página del texto (beta)
nueva página del texto (beta)


