Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista fitotecnia mexicana
versión impresa ISSN 0187-7380
Rev. fitotec. mex vol.38 no.4 Chapingo oct./dic. 2015
Artículo Científico
Modelación de la interacción genotipo X ambiente en rendimiento de híbridos de maíz blanco en ambientes múltiples
Modeling genotype X environment interaction in grain yield of white maize hybrids in multiple enviroments
Águeda Lozano-Ramírez2, Amalio Santacruz-Varela1*, Félix San-Vicente-García2, José Crossa2, Juan Burgueño2 y José D. Molina-Galán1
1 Campus Montecillo, Colegio de Postgraduados. Km 36.5 Carr. México-Texcoco. 56230, Montecillo, Texcoco, Edo. de México. * Autor para correspondencia (asvarela@colpos.mx)
2 Centro Internacional de Mejoramiento de Maíz y Trigo. Km 45 Carr. México-Veracruz. 56237, El Batán, Texcoco, Edo. de México.
Recibido: 1 de Abril del 2014
Aceptado: 1 de Junio del 2015
Resumen
Los programas de fitomejoramiento enfocados a la obtención de genotipos con mayor rendimiento y estables en una amplia gama de condiciones ambientales enfrentan factores ambientales que enmascaran el potencial de los genotipos. La interacción genotipo × ambiente (G × A) puede hacer que esta predicción no sea precisa. El objetivo de este estudio fue modelar la interacción G × A mediante distintos modelos estadísticos, en un grupo de híbridos de maíz (Zea mays L.) en ambientes de clima tropical. Se evaluaron 29 híbridos de endospermo blanco en 15 ambientes de América tropical, con un diseño alfa-látice. La variable rendimiento se analizó primero con un análisis combinado de varianza. Posteriormente se aplicaron los métodos de efectos principales aditivos e interacción (AMMI) y de regresión en los sitios (SREG) con el modelo de factores analítico (FA), para estudiar la interacción G × A y definir ambientes que discriminen mejor a los genotipos, y que permitan el agrupamiento de ambiente y genotipos. El análisis AMMI señaló a una localidad de Guatemala, una de México y una de Nicaragua como las de mayor interacción, identificó cuatro mega-ambientes, y definió al híbrido más estable y con buen rendimiento. El método SREG FA resultó un buen predictor porque permitió identificar cuatro subgrupos y agrupó ambientes de diferentes países con características semejantes.
Palabras clave: Zea mays, G×A, AMMI y SREG FA.
Abstract
Plant breeding programs aimed at obtaining genotypes with high grain yield and stable in a wide range of environmental conditions face environmental factors that mask potential genotypes. The Genotype x Environment interaction (G × E) might cause the suitability of predicted genotypes to a particular environment to be inaccurate. This study modelled the G × E interaction using different statistical models in a group of hybrids of maize (Zea mays L.) evaluated in tropical environments. Twenty-nine white-endosperm hybrids were evaluated in 15 environments of tropical America, with an alpha-lattice design. Grain yield was first analyzed with a combined analysis of variance. Subsequently, the additive main effect and multiplicative interaction (AMMI) and the site regression (SREG) with analytic factors (FA) model were applied to study and model G × E and to define environments that best discriminate genotypes and allow the grouping of environments and genotypes. The AMMI method pointed out a locality from Guatemala, one from México and one from Nicaragua as the ones with highest G × E; generated four mega-environments; and defined the most stable and good-yielding hybrid. The SREG FA method proved a good predictor since it allowed the identification of four subgroups and grouped environments of different countries with similar features.
Key words: Zea mays, G×E, AMMI, SREG FA.
INTRODUCCIÓN
Un reto en los programas de fitomejoramiento genético es obtener genotipos con rendimientos mayores, pero con frecuencia el potencial de rendimiento es enmascarado por la interacción genotipo por ambiente (G × A). Gordón-Mendoza et al. (2006) mencionan que la interacción G × A ocurre cuando los genotipos responden de manera diferente a las variantes del ambiente. Acerca de la importancia de la interacción G × A, Gauch y Zobel (1997) explican que "si no hubiera interacción, en una sola variedad de maíz (Zea mays L.) o cualquier otro cultivo se podría alcanzar aquella que rendiría más en todo el mundo, y además el ensayo de cultivos se podría realizar en una sola ubicación y proporcionar resultados universales". Por su parte, Sprague y Eberhart (1977) recomiendan aumentar el número de ambientes para evaluación de los ensayos si la interacción G × A se debe a factores impredecibles del ambiente.
El mejorador de plantas tiene que encarar estos problemas para así obtener un genotipo más cercano al ideal. Se pueden utilizar varios modelos para interpretar la respuesta de los genotipos a través de ambiente y para estudiar e interpretar la interacción G × A en experimentos agrícolas, entre ellos los modelos lineales, bilineales y lineal-bilineal. Los modelos de efectos fijos lineal-bilineal, como el SREG (Crossa y Cornelius, 1997) y el de efectos principales aditivos e interacción multiplicativo (AMMI) (Gauch, 1988; Gauch y Zobel, 1997) se utilizan para el estudio de los patrones de respuesta genotípica a través de ambientes.
En las últimas décadas ha habido un desarrollo acelerado de los modelos estadísticos y biométricos para el estudio y disección de la G × A. En su revisión de los principales modelos estadísticos para la G × A, Crossa et al. (2006) mencionan los modelos que incorporan marcadores moleculares o covariables ambientales, así como información de pedigrí entre los genotipos, para explicar mejor el fenómeno de la interacción G × A. Claramente, los modelos antes propuestos consideraban sólo efectos fijos pero los actuales asumen efectos aleatorios que permiten una mejor modelación de la interacción.
Modelo básico de análisis de la varianza para ensayos de genotipos en ambientes múltiples
Los modelos individual y combinado que explican el comportamiento de cualquier genotipo en los diferentes ambientes para un diseño en látice de acuerdo con Martínez (1988), son:
Modelo lineal individual:

donde: yilp = valor del genotipo i en el bloque l en la repetición p; μ = media general de todas las observaciones; rp = efecto de la repetición p; βl(p) = efecto del bloque l dentro de la repetición p; τi = efecto del i-ésimo genotipo; εilp = error experimental.
Modelo lineal combinado:
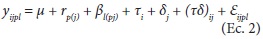
donde: yijpl comportamiento medio del genotipo i en ambiente j en la repetición p en el bloque l; μ = media general; rp(j) = efecto de la repetición p dentro del ambiente j; βl(pj) = efecto del bloque l dentro de la repetición p en el ambiente j; τi = efecto del i-ésimo genotipo; δj = efecto del j-ésimo ambiente; (τδ)ij = efecto de la interacción (G × A) del i-ésimo genotipo en el j-ésimo ambiente; εijpl es el error aleatorio no observable que se asume homogéneo con distribuciones normales e independientes, o sea, NID (0, I σ2) (σ2 = varianza del error aleatorio).
Modelo AMMI
El modelo AMMI se define como el modelo de efectos principales aditivos e interacción multiplicativa (Gauch, 2006; Glaz y Kang, 2008). El análisis AMMI se basa en la Ecuación (2), pero en el que el término de la interacción G×A se separa en componentes, de acuerdo con el siguiente modelo lineal:

donde los términos son los mismos de la Ecuación 2, pero la interacción G × A está dada por la suma de los términos multiplicativos ∑t(n=1) λnαinγjn. Los parámetros a ser estimados, aparte de la varianza del error aleatorio (σ2ε), y los efectos principales del genotipo y ambientes, son los siguientes: λn es el parámetro de escala o valor singular del n-ésimo componente multiplicativo, cuyos valores ordenados en forma decreciente quedan λn ≥ λ2 ≥... ≥ λt; αin es el vector singular izquierdo del n-ésimo componente y representa sensibilidades de los genotipos a ciertos factores ambientales hipotéticos dados por el vector singular derecho, γjn del n-ésimo componente del ambiente asociados a λn. Los αin y los γjn están sujetos a restricciones de normalización ∑i α2in = ∑j γ2jn = 1 y a restricciones de ortogonalidad ∑i αin αin, ∑j γjn γjn = 0 para n ≠ n' (Crossa y Cornelius, 2000).
Modelo de regresión en los sitios (SREG)
Del modelo AMMI es posible estimar el efecto de genotipos combinado con el efecto de la interacción (G + G × A), de tal forma de generar el modelo llamado de regresión en sitios (SREG). El componente G + G × A (GGA) puede ser considerado de diferentes formas, por ejemplo como un efecto fijo aditivo de genotipo anidado en ambiente, o como un efecto fijo pero con términos multiplicativos que es el que se expresa en la Ecuación 4. También puede ser considerado como un efecto aleatorio, lo que da lugar a diversas formas de modelar la estructura de covarianzas de GGA. Para el caso de efecto fijo multiplicativo, el modelo se expresa de la siguiente forma:

donde: λn = valor singular para el componente; αin = vectores propios asociados con los genotipos para cada componente principal; γjn = vectores propios asociados con los ambientes para cada componente. Al igual que el modelo AMMI, la interpretación del componente GGA se puede realizar a través del denominado "biplot" GGE (mismo que GGA, por sus siglas en inglés) (Yan et al., 2000; Yan y Hunt, 2001).
Modelo lineal mixto y estructura de covarianza del modelo de factores analíticos
El modelo lineal mixto básico para analizar genotipos en ambientes en repeticiones con bloques, en su forma SREG con expresión matricial, es el siguiente:

donde X es la matriz de incidencia para los efectos fijos ambientales de repeticiones, Zr y Zg son matrices de incidencia de los efectos aleatorios de bloques dentro de repeticiones y ambientes y el efecto de genotipos dentro de ambientes respectivamente. Este último incluye el efecto principal del genotipo más la interacción genotipo-ambiente (GGA). El vector b es de efectos fijos de ambientes y repeticiones, y los vectores r, g y e contienen los efectos aleatorios de bloques anidados en repeticiones y ambientes, genotipos anidados en ambientes y el error experimental.
Al considerar el efecto GGA, se supone que g~NID(0, Σgga) y es posible modelar Σgga de diversas formas. Un modelo adecuado por parsimonioso y que a la vez permite incluir información genética en el componente GGA, es el que considera una estructura separable en donde Σgga = (Γ' Γ + Ψ)⨂Σg (Crossa et al., 2006, Burgueño et al., 2008), donde (Γ' Γ + Ψ) corresponde al modelo de factores analíticos con Γ de dimensión, txJ son los vectores propios, y Ψ es una matriz diagonal de dimensión J cuyos elementos son varianzas residuales de cada ambiente; Σg es la matriz de varianzas y covarianzas de genotipos, que también puede ser modelada o puede considerarse conocida y sustituirla por la matriz de parentesco o cualquier otra matriz de relaciones entre genotipos (Crossa et al., 2006) y ⨂ representa el producto Kronecker.
El modelo de factores analíticos (FA) es una estructura que modela varianzas-covarianzas del modelo mixto de forma parsimoniosa pues usa únicamente (J + 1)t comparado con un modelo no estructurado de varianzas-covarianzas que necesita [J(J-1)/2] + J parámetros. La estructura FA puede expresarse también como una regresión lineal múltiple del i-ésimo genotipo en el j-ésimo ambiente en función lineal de variables latentes xik con coeficientes δjk para k = 1,2…,t más un residual ηij y µj como la media general de los genotipos. Para dos factores latentes puede escribirse como:

donde ȳij puede ser la media, la media ajustada o el BLUP del genotipo i en el ambiente j.
La matriz Γ contiene los coeficientes δjk, que son equivalentes a los vectores propios de ambientes a partir de los cuales se obtienen los escores de los genotipos para construir el gráfico "biplot". La matriz Γ debe ser rotada para obtener una solución de componentes principales (Smith et al., 2002), lo que resulta en el mismo gráfico como el obtenido con el modelo SREG de efectos fijos cuando los datos están balanceados (Burgueño et al., 2008).
En el presente trabajo se plantea como objetivo modelar la interacción del rendimiento de 29 híbridos de maíz de endospermo blanco en distintos ambientes de clima tropical, mediante el modelo AMMI y el modelo SREG con FA, visualizando el agrupamiento de genotipos y ambientes.
MATERIALES Y MÉTODOS
Material genético y ubicación de los experimentos
Se evaluaron 29 híbridos trilineales de endospermo blanco, dos con alta calidad de proteína (QPM), y 27 normales de maíz provenientes de líneas élite desarrolladas en el sub-programa Trópicos Bajos del Programa Global de Maíz del Centro Internacional de Mejoramiento de Maíz y Trigo (Cuadro 1). Los ensayos se establecieron en 15 localidades, tres de ellas en México y el resto distribuidas en 12 países de América Central, Norte de Sud-América (Cuadro 2), durante junio-diciembre de 2012.
Diseño y unidad experimental
El diseño experimental utilizado en todas las localidades fue de alfa-látice (Patterson y Williams, 1976) con tres repeticiones. La unidad experimental estuvo constituida por dos surcos de 5 m de longitud y 0.80 m de ancho, y una separación entre plantas de 0.20 m, para una densidad de población de 60 mil plantas por hectárea.
Variable de respuesta y análisis estadístico
Se evaluó el rendimiento, expresado en toneladas por hectárea, ajustado a 12.25 % de humedad y con un factor de desgrane de 80 %. Se realizó el análisis individual y combinado a través de ambientes con el modelo básico de las Ecuaciones 1 y 2, respectivamente. Del análisis individual solo se extrajeron los promedios de los genotipos a través de localidades. Posteriormente se realizó un análisis de la interacción G × A cuando el análisis de varianza determinó significancia estadística para este efecto, mediante análisis AMMI (Ecuación 3) de componentes principales aditivos de la interacción G × A (Gauch, 2006; Glaz y Kang, 2008); y el análisis SREG FA (Ecuación 5).
Los resultados obtenidos por descomposición del valor singular de ambos modelos, para reducir la direccionalidad de las variables en componentes principales, se plasmaron en un gráfico "biplot". Para el método AMMI se realizó la prueba de Gollob (1968) para determinar la significancia de cada componente de este modelo. Adicionalmente, para el SREG FA se realizó un análisis de conglomerados con los factores obtenidos, mediante distancias Euclidianas y el método de ligamiento de Ward. Los análisis estadísticos individual, combinado y AMMI fueron realizados con el paquete estadístico SAS (Statistical Analysis System), versión 9.1. El análisis SREG FA se realizó con el paquete estadístico ASReml (Gilmour et al., 2002).
RESULTADOS Y DISCUSIÓN
Análisis de varianza
En el Cuadro 3 se presentan los valores promedios de rendimiento de los híbridos evaluados en cada localidad, los cuales variaron desde 1.4 t ha-1 (Sabana, Escuintla, Guatemala) hasta 11.14 t ha-1 (Tlaltizapán, Morelos, México). Los valores de la diferencia mínima significativa fueron en general bajos, inferiores a 1.88 en los distintos ambientes, con excepción de Tocorón, Aragua, Venezuela (2.84 t ha-1) y de San Gilberto, Chinandenga, Nicaragua (3.06 t ha-1), lo cual lleva a la inclusión de un mayor número de híbridos dentro de un agrupamiento. Los coeficientes de variación presentaron valores desde 6.7 % (Cotaxtla, Veracruz, México) hasta 19.5 % (Tocorón, Aragua), con excepción de Sabana, Escuintla donde se obtuvo un valor de 31.1 %. Todas las localidades presentaron un coeficiente de variación por debajo de 19.5 %, excepto Sabana, Escuintla (31.1 %), lo cual es indicativo de buen control ambiental y de la precisión de la técnica experimental utilizada.
En el Cuadro 4 se presenta el análisis de varianza combinado de ambientes para la variable rendimiento de grano (t ha-1), el cual muestra que existen diferencias significativas (P < 0.01) entre ambientes, genotipos y G×A. La interacción significativa indica que los genotipos no se comportaron de igual manera ante las variaciones ambientales de las diferentes localidades y, por ende, existen buenos materiales para unos ambientes pero que no responden bien en otros.
Método AMMI
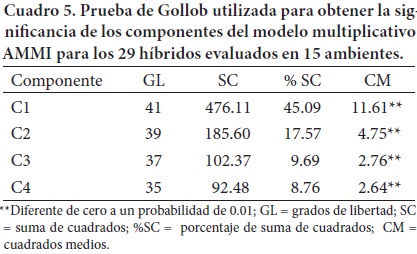
En el Cuadro 5 se muestra la prueba de Gollob utilizada para obtener la significancia de los componentes del modelo multiplicativo AMMI para los híbridos evaluados en los diferentes ambientes. El análisis AMMI mostró que los dos primeros componentes son significativos (P < 0.01) y que en conjunto explican 62.66 % (CP1 45.09 % y CP2 17.57 %) de la variación total debida a la interacción. Los resultados se plasman en un gráfico "biplot" en dos dimensiones que corresponden a los dos primeros componentes.
El "biplot" de la Figura 1 muestra que los híbridos 2, 7, 15 y 24 fueron los más cercanos al origen, lo que indica su mayor estabilidad; los híbridos 11, 13, 18 y 22 tienen un comportamiento aceptable de rendimiento, pero éste cambia de manera considerable de un ambiente a otro. También se aprecia que los híbridos 10, 11, 12, 19, 23 y 27 son los que más aportaron a la interacción, mientras que los híbridos 14, 19 y 23 ubicados en la porción negativa sobre el eje de las abscisas del "biplot", presentaron baja adaptación a los ambientes evaluados, de acuerdo con el criterio propuesto por Medina et al. (2001).
Con relación a los ambientes se observó que Agua Fría, Puebla, México; San Andrés, El Salvador; Playita, Comayagua, Honduras; Managua, Nicaragua; y Quesada, Jutiapa, Guatemala son localidades que presentaron menor interacción, en contraste con los ambientes de las Monjas, Jalapa, Guatemala; Tlaltizapán; San Gilberto, Chinandenga y Tocorón, Aragua, que fueron los que contribuyeron más a la interacción y por ello discriminan mejor a los genotipos. Yan et al. (2000) señalan que los ambientes que presentan un ángulo menor de 90° clasifican a los genotipos de manera semejante, caso que se presenta en las localidades de Agua Fría; Palmira, Colombia; Tlaltizapán; Cotaxtla; Santa Cruz Porrillo, El Salvador y San Andrés, El Salvador.
La capacidad de reducir el número de ambientes de evaluación sin perder precisión en los resultados es de gran utilidad, debido al consecuente abatimiento de los costos de experimentación, y hace más eficientes a los programas de fitomejoramiento. De acuerdo con los planteamientos de Kempton (1984), las localidades que más discriminaron a los genotipos en la evaluación fueron Las Monjas, Jalapa; Tlaltizapán; San Gilberto, Chinandenga, y Tocorón, Aragua, y las localidades más contrastantes fueron Santa Cruz Porrillo con San Gilberto, Chinandenga y Las Monajas, Jalapa con Tocorón, Aragua.
Método SREG FA
Los dos primeros componentes de SREG FA explican 75.52 % de la variación para el Componente 1 y 10.94 % para el Componente 2, los cuales se representan en el "biplot" de la Figura 2 que exhibe los patrones de respuesta de 29 genotipos tropicales de endospermo blanco evaluados en 15 localidades. Crossa et al. (1990) mencionan que los genotipos y ambientes con coordenadas grandes en la dirección del Componente 1, consideradas en valor absoluto, contribuyen en mayor medida a la interacción G × A, mientras los genotipos y ambientes con valores cercanos a cero para la dirección del Componente 1 tienen poca participación sobre este efecto.
De acuerdo con esta interpretación, se puede apreciar que los ambientes con mayor aporte a la interacción corresponden a Santa Cruz Porrillo; Tlaltizapán y Tocorón, Aragua; mientras que San Gilberto, Chinandenga muestra menor relación con el resto de los ambientes. Por otra parte, los genotipos que se muestran más estables y con buen rendimiento fueron los híbridos 13, 17 y 27. Con relación a las localidades, se observa un grupo de ellas en el cuadrante superior derecho (Managua; Sabana, Escuintla; Yorito, Yoro, Hoduras; Quesada, Jutiapa; Tiucal, Jutiapa, Guatemala; Playita, Comayagua y Cotaxtla) y otro en el cuadrante inferior derecho (Tocorón, Aragua; Palmira y Tlatizapán), mientras que San Gilberto, Chinandenga queda como ambiente único que conforma un grupo en el lado izquierdo de la misma figura, que indica ser un ambiente con condiciones poco favorables para los genotipos. El "biplot" de patrones descriptivos generales de genotipos y localidades ofrece la posibilidad de identificar algunos pares extremos de genotipos y de localidades; por ejemplo, para Agua Fría, Palmira y Tlatizapán el genotipo más sobresaliente fue el 13.
Análisis de conglomerados
El análisis de conglomerados muestra los patrones ambientales de los marcadores para los Componentes 1 y 2 del modelo SREG FA. En el dendrograma de la Figura 3 se percibe el agrupamiento de los genotipos, que con un nivel de corte de 1.2 se aprecian dos grupos. En el Grupo I se observan dos subgrupos, el Subgrupo I-A que sólo alberga a la localidad de Tlatizapán como mayor discriminante de los híbridos, y en el Subgrupo I-B se ubican las localidades de Santa Cruz Porrillo; San Andrés; Palmira y Cotaxtla que presentan patrones diferentes de interacción pero en baja magnitud.
En el Grupo II se ubicaron dos subgrupos, el Subgrupo II-A conformado sólo por la localidad San Gilberto, Chinandenga, la cual en el gráfico de biplot de la Figura 2 se ubica como una localidad que contrasta con el resto. El Subgrupo II-B estuvo integrado por la mayoría de las localidades, entre las cuales se encuentran Tocorón, Aragua; Yorito, Yoro; Tiucal, Jutiapa; Quesada, Jutiapa; Sabana, Escuintla; Managua; Playita, Comayagua; Las Monjas, Jalapa y Agua Fría. La agrupación de estos ambientes refleja patrones similares, definidos probablemente por condiciones ambientales afines entre las localidades.
En la Figura 4 se muestra la distribución de agrupamientos de los 29 genotipos evaluados; en la parte superior del dendrograma se ubican los genotipos que tuvieron puntuaciones extremas, porque pudieron tener los más bajos o altos rendimientos en las localidades evaluadas. También se muestran tres grandes grupos de híbridos, a un nivel de corte de 1.0. El Grupo I se divide en dos subgrupos; en el Subgrupo I-A sólo se encuentra el híbrido 23; en el subgrupo I-B se ubican los híbridos 14 y 19; el híbrido 26 se ubica solo en el Subgrupo II-A, mientras que en el Subgrupo II-B se ubican seis híbridos: 9, 12, 16, 21, 25 y 29; estos subgrupos de híbridos indican que éstos no fueron los mejores en ninguno de los ambientes de evaluación.
El Grupo III se divide en dos subgrupos. El Subgrupo III-A donde se ubica la mayoría de los híbridos evaluados, como 3, 6, 4, 5, 8, 10, 11, 13, 15, 17, 18, 22, 24, 27 y 28; materiales que por su agrupamiento en el dendrograma, su distribución en el grafico "biplot" y de acuerdo al Cuadro 3 donde se reflejan los promedios de los genotipos, se consideran estables y con rendimientos medios. Esta agrupación puede deberse a que los factores ambientales presentan condiciones favorables para este grupo de híbridos ya que pueden responder de manera aceptable en todos los ambientes. En el subgrupo III-B se ubicaron los híbridos 1, 2, 7 y 20, que presentaron rendimientos intermedios, pero que difieren de manera considerables de un ambiente a otro.
CONCLUSIONES
El modelo AMMI fue útil para entender la compleja interacción genotipo por ambiente existente en caracteres cuantitativos como el rendimiento de grano, lo que permite ganar comprensión de la interacción, mejorar el proceso de selección y sumar eficacia experimental al poder formar grupos de ambientes de igual respuesta, así como grupos de ambientes que poco contribuyen a la interacción y a la discriminación de genotipos. La aplicación de SREG FA fue altamente efectiva, porque los ambientes fueron agrupados con mayor precisión.
Estos resultados demuestran la aplicabilidad de este método para definir los mega-ambientes cuando existe poca interacción. Los resultados obtenidos con este método muestran que, desde la perspectiva de un mejorador, es importante tener en cuenta la combinación de ciertos subconjuntos de ambientes en un grupo más amplio (mega-ambientes), ya que esto permite representar mejor a las poblaciones objetivo.
BIBLIOGRAFÍA
Burgueño J., J. Crossa, P. L. Cornelius and R. C. Yang (2008) Using factor analytic models for joining environments and genotypes without crossover genotype x environment interaction. Crop Science 48:1291-1305. [ Links ]
Crossa J. and P. L. Cornelius (1997) Sites regression and shifted multiplicative model clustering of cultivar trial sites under heterogeneity of error variances. Crop Science 37:406-415. [ Links ]
Crossa J. and P. L. Cornelius (2000) Modelos lineales bilineales para el análisis de ensayos de genotipos en ambiente multiples. In: Simposium Interacción Genotipo x Ambiente. Sociedad Mexicana de Fitogenética A.C. 15-20 de octubre 2000. Irapuato, México. pp:61-68. [ Links ]
Crossa J., H. G. Gauch and R. W. Zobel (1990) Additive main effects and multiplicative interaction analysis of two international maize cultivar trials. Crop Science 30:493-500. [ Links ]
Crossa J., J. Burgueño, P. L. Cornelius, G. McLaren and R. Trethowan (2006) Modeling genotype x environment interaction using additive genetic covariances of relatives for predicting breeding values of wheat genotypes. Crop Science 46:1722-1733. [ Links ]
Gauch H. G. Jr. (1988) Model selection and validation for yield trials with interaction. Biometrics 44:705-715. [ Links ]
Gordón-Mendoza R., I. Camargo-Buitrago, J. Franco-Barrera y A. González-Saavedra (2006) Evaluación de la adaptabilidad y estabilidad de 14 híbridos de maíz, Azuero, Panamá. Agronomía Mesoamericana 17:189-199. [ Links ]
Gauch H. G. (2006) Statistical analysis of yield trials by AMMI and GGE. Crop Science 46:1488-1500. [ Links ]
Gauch H. G. and R. W. Zobel (1997) Identifying mega-environments and targeting genotypes. Crop Science 37:311-326. [ Links ]
Gilmour A. G., B. R. Cullis, S. J. Welham and R. Thompson (2002) ASReml Reference Manual. 2nd Ed. Release 1.0. NSW Agriculture Biometrical Bulletin 3. NSW Agriculture. Locked Bag, Orange, NSW, Australia. 186 p. [ Links ]
Glaz B. and M. S. Kang (2008) Location contributions determined via GGE biplot analysis of multienvironment sugarcane genotype-performance trials. Crop Science 48:941-950. [ Links ]
Gollob H. F. (1968) A statistical model which combines features of factor analytic and analysis of variance techniques. Psychometrika 33:73-115. [ Links ]
Kempton R. A. (1984) The use of biplots in interpreting variety by environment interactions. Journal of Agricultural Science 103:123-135. [ Links ]
Martínez G. A. (1988) Diseños Experimentales. Métodos y Elementos de Teoría. Ed. Trillas. México, D.F. 756 p. [ Links ]
Medina M., T. Abadie, D. Vilaró and S. Ceretta (2001) Estudio metodológico de adaptación de cultivares de maíz para silo a las condiciones de Uruguay. Agrociencia (Uruguay) 5:23-31. [ Links ]
Patterson H. D. and E. R. Williams (1976) A new class of resolvable incomplete block designs. Biometrika 63:83-92. [ Links ]
Smith A. B., B. R. Cullis and R. Thompson (2002) Exploring variety-environment data using random effects AMMI models with adjustment for spatial field trends: Part 1: Theory. In: Quantitative Genetic, Genomics, and Plant Breeding. M. S. Kang (ed.). CABI Publishing, Wallingford, UK. pp:323-335. [ Links ]
Sprague G. F. and S. A. Eberhart (1977) Corn breeding. In: Corn and Corn Improvement. G. F. Sprague (ed.). American Society of Agronomy. Madison, Wisconsin. pp:305-362. [ Links ]
Yan W., L. A. Hunt, Q. Sheng and Z. Szlavnics (2000) Cultivar evaluation and mega-environment investigation based on the GGE biplot. Crop Science 40:597-605. [ Links ]
Yan W. and L. A. Hunt (2001) Interpretation of genotype x environment interaction for winter wheat yield in Ontario. Crop Science 41:19-25. [ Links ]














