Introducción
Pyrodinium bahamense Plate es un dinoflagelado tecado, bioluminiscente, perteneciente al orden Gonyaulacales, erigido a partir de organismos colectados por Plate en 1906 en las Bahamas (New Providence Island). Años más tarde, Böhm (1931) encontró ejemplares similares en el Golfo Pérsico, aunque formando cadenas de células comprimidas anteroposteriormente, a los que denominó Pyrodinium bahamense f. compressa Böhm (Pbc). Dos años después en el Mar Rojo, Matzenahuer (1933) describió una especie semejante: Gonyaulax schilleri Matzenahuer, la cual después fue transferida por Schiller (1937) al género Pyrodinum Plate, señalando que P. bahamense f. compressa era su sinónimo. En 1972, en el sureste de Asia en Port Moresby, Papúa Nueva Guinea, ocurrió el primer florecimiento algal nocivo (FAN) de Pbc, produciendo envenenamiento paralítico por mariscos (PSP, por sus siglas en inglés) (MacLean, 1989). Desde ese momento, varios otros episodios ocurrieron en esa región del Pacífico occidental y a partir de 1987 también en el Pacífico oriental, pues de julio a octubre ocurrió un FAN de Pbc en las costas de Guatemala, causando la muerte de 26 personas y la intoxicación de 186 (Rosales-Loessener, 1989a; Cortes-Altamirano et al., 1993). Dado que todos estos eventos tóxicos fueron causados por Pbc, los taxónomos enfatizaron aún más la existencia de las dos variedades; reconociéndose la forma comprimida (compressa) como la tóxica y la f. bahamense como la inocua (Hallegraeff y Maclean, 1989). Steidinger et al. (1980), valorando al menos seis diferencias constantes tanto morfológicas como fisiológicas, estas últimas referente a su toxicidad, consideraron que la variedad compressa era realmente conespecífica con Pyrodinium bahamense, pero le dieron valor a la categoría de variedad. Más tarde el nombre de compressa fue corregido a compressum por Balech (1985). De esta manera, ambas variedades fueron aceptadas por la mayoría de los taxónomos, agregando a las diferencias morfológicas y fisiológicas su distinta distribución geográfica, con excepción de Mertens et al. (2015), quienes desconocen el peso de estos caracteres en el reconocimiento de estas variedades y recomiendan tratarlas como un solo taxón. Así, la var. bahamense ocurre en latitudes tropicales y subtropicales, principalmente en el Atlántico y Mar Caribe (Gómez-Aguirre y Licea, 1998; Usup et al., 2012), aunque Martínez-López et al. (2007), Morquecho (2008), y Gárate-Lizárraga y González-Armas (2011) la reportan en áreas costeras del Pacífico mexicano. Por otro lado, la var. compressum está distribuida prácticamente en todo el Océano Pacífico (tanto en la porción occidental como en la oriental) y en el Océano Índico (Alonso-Rodríguez et al., 2008; Gárate-Lizárraga y González-Armas, 2011; Morquecho, 2019). Ambas variedades han sido reportadas en coocurrencia en la costa occidental de México (Meave del Castillo et al., 2019) y en Costa Rica (Vargas-Montero y Freer, 2003). A estas diferencias se pueden sumar las ambientales: Phlips et al. (2006) señalan que la var. bahamense se desarrolla en sitios tranquilos y someros, con cambios de salinidad importantes, donde la columna de agua es estable y el tiempo de residencia largo, mientras que la var. compressum ocurre en zonas costeras más dinámicas e incluso en el océano abierto, en sitios con tiempos de residencia cortos y temperatura y salinidad estables.
Recientemente, se encontró toxicidad PSP y saxitoxina en una población identificada como var. bahamense (Landsberg et al., 2006; Lewitus et al., 2014), lo que ha llevado a sugerir que P. bahamense es un complejo de especies y que el término “variedades” ya no está justificado (Mertens et al., 2015), a pesar de toda la evidencia adicional anteriormente presentada por otros autores (Steidinger et al., 1980; Hallegraeff y Maclean, 1989), generando así la problemática actual.
En estudios de filogenia y diversidad genética en grupos de fitoplancton marino, que presentan una baja variación morfológica, como el caso de Pyrodinium (Kamikawa et al., 2009; Kuno et al., 2010; Miranda et al., 2012; Cusick et al., 2016; Rhodes et al., 2017; Hernández-Rosas et al., 2018), diferentes marcadores moleculares, como ITS (espaciador transcrito interno), LSU (subunidad grande del ribosoma), SSU (subunidad pequeña del ribosoma), Lcf (gen de la luciferasa) y cox1 (citocromo oxidasa subunidad 1), han sido empleados con éxito en el aporte de evidencia para la delimitación de especies. Adicionalmente, dichos marcadores han permitido comprender la estructura y la conectividad de las poblaciones, con poca o nula diferenciación entre ellas, lo que ha contribuido a un mejor entendimiento de sus relaciones filogenéticas e historia evolutiva (Scholin et al., 1994; Richlen et al., 2008; Litaker et al., 2009; Uwe et al., 2014).
Aunque las poblaciones de Pyrodinium con distribución en los océanos Indo-Pacífico y Atlántico Occidental han sido estudiadas con múltiples marcadores (LSU, SSU, Lcf) y con todos ellos se ha evidenciado, de manera consistente, la estructura genética entre las dos variedades, los autores han mantenido una posición neutral en cuanto al reconocimiento de la independencia genética entre ellas (Leaw et al., 2005; Usup et al., 2012; Mertens et al., 2015; Cusick et al., 2016). Por tanto, nuestro objetivo fue reconocer, desde la evidencia genética, la independencia taxonómica de las dos variedades de Pyrodinium: Pyrodinium bahamense var. compressum y Pyrodinium bahamense var. bahamense.
Materiales y Métodos
Secuencias de P. bahamense var. bahamense y var. compressum de los océanos Atlántico occidental e Indo-Pacífico se obtuvieron de la base de datos (GenBank, 2020) del National Center for Biotechnology Information (NCBI), para los marcadores Lcf, LSU y SSU (Apéndice 1), ya que estos marcadores tenían un mayor número de secuencias disponibles. Las secuencias se alinearon utilizando el programa Bioedit v. 7.0.5 (Hall, 1999). Se analizaron los conjuntos de datos de Lcf, LSU y SSU, por separado. Algunas de estas secuencias se incluyeron, junto con otras secuencias del orden Gonyaulacales (Apéndice 2), en un conjunto de datos combinado de Lcf, LSU y SSU. Para los conjuntos de datos de Lcf, LSU y SSU, se generaron redes de parsimonia estadística utilizando el programa TCS v. 1.21 (Clement et al., 2000). Para el conjunto de datos combinados de Lcf, LSU y SSU, los taxones para los que no estaba disponible una secuencia de algún marcador se trataron como datos faltantes. Se incluyó como grupo externo a Hematodinium perezii Chatton & Poisson; esta especie fue seleccionada por pertenecer a un orden diferente y distante de Gonyaulacales. A partir de este conjunto de datos, se realizaron análisis filogenéticos utilizando máxima verosimilitud (ML) e inferencia bayesiana (IB), dividiendo por separado los datos en posiciones de codones. El modelo evolutivo seleccionado fue Jukes-Cantor (JC) determinado con base a la prueba de razón de máxima verosimilitud implementada por el software TOPALi v. 2 (Milne et al., 2009). El análisis de ML se realizó utilizando el software RAxML v. 8 (Stamatakis, 2006). El soporte para cada rama se obtuvo a partir de 1000 réplicas de bootstrap. Para el análisis de BI se utilizó MrBayes v. 3.2.2 (Ronquist et al., 2012). Se utilizaron cuatro cadenas de la cadena de Markov Monte Carlo, comenzando con un árbol aleatorio y muestreando los datos cada 500 generaciones durante 5 × 106. El 25% de los árboles se descartaron como burn-in.
Resultados
Las redes de parsimonia estadística y análisis filogenético mostraron que existe variación genética y aislamiento reproductivo entre secuencias de P. bahamense var. bahamense y var. compressum (Figs. 1A, B).
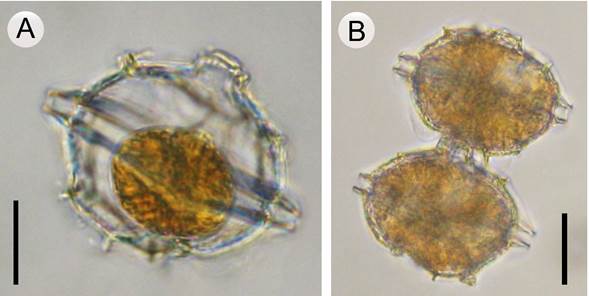
Figura 1: Pyrodinium bahamense Plate (microscopio óptico). A. célula solitaria de Pyrodinium bahamense var. bahamense Plate; B. cadena de dos células de Pyrodinium bahamense var. compressum (Böhm) Steidinger, Tester & Taylor. Un estudio detallado de la morfología de ambas variedades se puede consultar en Meave del Castillo et al. (2019). Barra de escala: A=20 µm; B=30 µm.
Variación genética
Set de datos del marcador Lcf
La comparación de 42 secuencias (365 pares de bases (pb)) reveló 38 sitios polimórficos en muestras de Pyrodinium de los océanos Atlántico occidental e Indo-Pacífico (Apéndice 1). La red de parsimonia estadística (Fig. 2) mostró 13 haplotipos, A1-A13 (Apéndices 1, 2) dispuestos en tres redes separadas (Figs. 2A-C). El haplotipo A1 tuvo el mayor número de muestras (n=14), seguido del haplotipo A9 (n=11). La primera red estuvo formada por los haplotipos A1-A7 (de River Lagoon, Florida), separados de A1 por un solo paso mutacional, el haplotipo A6 separado por tres pasos mutacionales y el haplotipo A7 separado por cuatro pasos mutacionales (Fig. 2A). La segunda red estuvo formada por los haplotipos A8-A12 (de River Lagoon, Florida), todos ellos separados entre sí por un solo paso mutacional (Fig. 2B). La tercera red estaba formada por un solo haplotipo (A13) (de Malasia) con tres secuencias (Fig. 2C).
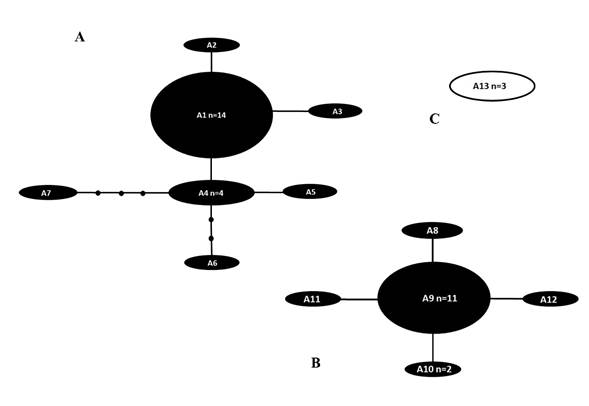
Figura 2: Red de parsimonia estadística para el marcador Lcf de secuencias de Pyrodinium bahamense Plate. Los haplotipos del Océano Atlántico occidental (P. bahamense var. bahamense Plate) se indican en negro, mientras que los haplotipos del Océano Indo-Pacífico (P. bahamense var. compressum (Böhm) Steidinger, Tester & Taylor) se indican en blanco. A, B y C indican cada una de las diferentes redes. Las líneas representan un paso mutacional; los pequeños círculos negros indican la ausencia de haplotipos. n=número de muestras con secuencia idéntica. No se indica el tamaño de la muestra en los haplotipos con una sola muestra.
Set de datos del marcador LSU
La comparación de 15 secuencias (647 pb) reveló siete sitios polimórficos (Cuadro 1). La red de parsimonia estadística (Fig. 3) mostró tres ribotipos interconectados, B1-B3 (Cuadro 1, Apéndice 2). El ribotipo B3 (de Florida y Jamaica) se encontró en nueve secuencias (n=9) y estuvo claramente separado por los ribotipos B2 (de Filipinas) y B3 (de Malasia, Filipinas y Japón) por cinco pasos mutacionales. Los ribotipos B1 y B2, a su vez, se separaron entre sí mediante un único paso mutacional (Fig. 3).
Cuadro 1: Posiciones variables en secuencias de ARN alineadas de la LSU de Pyrodinium bahamense Plate. Ribotipos de P. bahamense var. compressum (Böhm) Steidinger, Tester & Taylor se indican en negritas; de manera contraria, se indica el ribotipo de P. bahamense var. bahamense Plate (ver Fig. 3 para la red de haplotipos).
| Ribotipos/sitios | 2 | 67 | 106 | 108 | 182 | 208 | 575 |
| B1 | A | T | G | T | C | T | G |
| B2 | - | - | - | A | - | A | T |
| B3 | C | C | A | T | T | T | G |
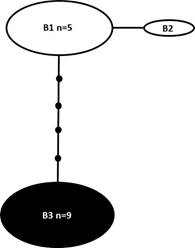
Figura 3: Red de parsimonia estadística para el marcador LSU de secuencias de Pyrodinium bahamense. Los ribotipos del Océano Atlántico occidental (P. bahamense var. bahamense) se indican en negro, mientras que los ribotipos del Océano Indo-Pacífico (P. bahamense var. compressum) se indican en blanco. Las líneas representan un paso mutacional; los pequeños círculos negros indican la falta de ribotipos. n=número de muestras con secuencia idéntica. No se indica el tamaño de la muestra en ribotipos con una sola muestra.
Set de datos del marcador SSU
La comparación de 38 secuencias (593 pb) reveló 288 sitios polimórficos (el cuadro no se muestra debido al gran número de sitios polimórficos). La red de parsimonia estadística mostró 15 ribotipos (Apéndice 2, Fig. 4), C1-C15 dispuestos en dos redes separadas (Figs. 4A, B). El ribotipo C1 tuvo el mayor número de muestras (n=15), seguido del ribotipo C12 (n=7). La primera red estuvo formada por los ribotipos C1-C11 (de Florida), los ribotipos C1-C6 separados de C1 por un solo paso mutacional, el ribotipo C7 separado por dos pasos mutacionales, los ribotipos C8-C10 separados por tres pasos mutacionales y el ribotipo C11 separado por cinco pasos mutacionales. (Fig. 4A). La segunda red la formaron los ribotipos C12-C15 de Florida (Fig. 4B), todos separados de C12 por un paso mutacional.
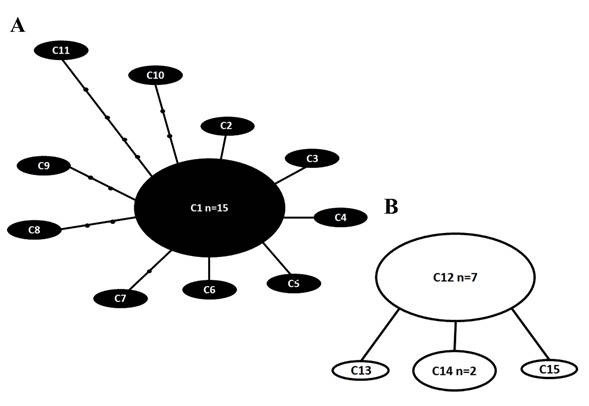
Figure 4: Red de parsimonia estadística para el marcador SSU de secuencias de Pyrodinium bahamense Plate. Los ribotipos del Océano Atlántico occidental (P. bahamense var. bahamense Plate) se indican en negro, mientras que los ribotipos del Océano Indo-Pacífico (P. bahamense var. compressum (Böhm) Steidinger, Tester & Taylor) se indican en blanco. A, B y C indican cada una de las diferentes redes. Las líneas representan un paso mutacional; los pequeños círculos negros indican la falta de ribotipos. n=número de muestras con secuencia idéntica. No se indica el tamaño de la muestra en ribotipos con una sola muestra.
Análisis filogenético
La combinación de Lcf, LSU y SSU constó de 26 secuencias y 1605 pb de longitud. La topología resultante mostró que las secuencias de Pyrodinium bahamense formaron un grupo monofilético, con el máximo soporte filogenético, diferenciado a su vez en dos grupos hermanos (Fig. 5). El grupo del Atlántico occidental estuvo integrado por secuencias de la var. bahamense, mientras que el grupo Indo-Pacífico por secuencias de la var. compressum. Una secuencia de la var. bahamense, de Florida, mostró una relación estrecha con secuencias de la var. compressum de Malasia y se resolvió como su grupo hermano. Estos dos grupos formaron un clado mayor, también con el máximo apoyo, con secuencias de especies del género Alexandrium Halim, como su grupo hermano (Fig. 5).
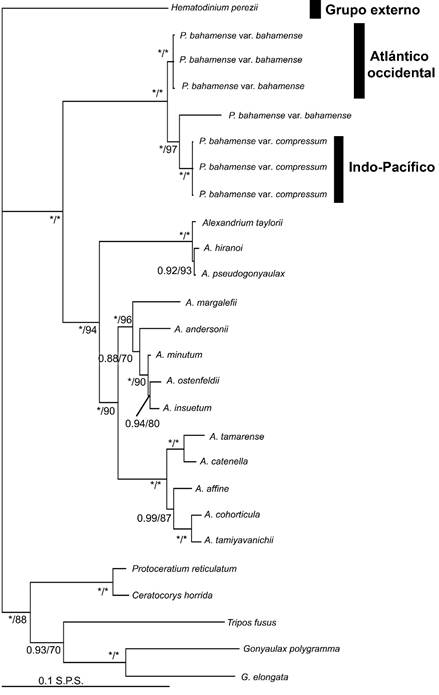
Figura 5: Topología bayesiana basada en datos de secuencias combinadas de los marcadores Lcf, LSU y SSU de Pyrodinium bahamense Plate. IB (izquierda) seguido de los valores de soporte de ML (derecha) en las ramas. Los asteriscos indican soporte total (1.00% IB, 100% ML). El grupo Atlántico occidental indica secuencias de P. bahamense var. bahamense Plate y el grupo Indo-Pacífico indica secuencias de P. bahamense var. compressum (Böhm) Steidinger, Tester & Taylor (ver resultados). S.P.S.=sustituciones por sitio. Los números de acceso de GenBank (2020) para las secuencias utilizadas en este análisis se proporcionan en el Apéndice 3.
Discusión
A pesar de que se han establecido diferencias morfológicas, fisiológicas, ecológicas, genéticas y biogeográficas entre P. bahamense var. bahamense y var. compressum, hoy en día se sugiere que ambas variedades se tratan de la misma especie (Mertens et al., 2015). Balech (1985) fue el primero en hacer esta propuesta con base en un estudio detallado de la teca de ejemplares de la var. bahamense (de Puerto Rico y Jamaica) y de la var. compressum (procedentes de Nueva Guinea y Filipinas) y no encontró diferencias consistentes, aunque sí ciertas tendencias diferenciales entre la morfología de ambas variedades a las que no se les dio peso taxonómico y, aunque propuso que las diferencias de toxicidad encontradas entre ambos taxa podrían deberse a la ocurrencia de una evolución críptica, en la que la diferenciación únicamente era fisiológica, consideró que las diferencias en toxicidad podían deberse a agentes externos, incluyendo las bacterias simbióticas (Balech, 1985; Usup et al., 2012). De esta manera, Balech (1985) señaló que Pyrodinium contiene un solo taxón, sinonimizando las siguientes especies con Pyrodinium bahamense: P. bahamense f. compressum (“compressa”), Gonyaulax schilleri, P. schilleri (Matzenauer) Schiller, P. bahamense var. compressum (“compressa”) (Böhm) Steidinger, Tester & Taylor, así como sus quistes fósiles Polysphaeridium zoharyi (Rossignol) Davey & Williams y Hemicystodinium zoharyi (Rossigniol) Wall. Adicionalmente, la concepción de validar a las variedades, e incluso pensar en que ambos taxa podrían ser especies crípticas, dejó de considerarse cuando Landsberg et al. (2006) y Lewitus et al. (2014) registraron toxicidad tipo PSP y saxitoxinas en una población de P. bahamense var. bahamense de Indian River Lagoon, Florida.
Con respecto a la toxicidad, se han encontrado distintos perfiles de saxitoxinas en diferentes regiones del planeta, e incluso dentro de poblaciones de la misma variedad, como puede apreciarse en el Cuadro 2. Sin embargo, las toxinas no están relacionadas con variaciones genéticas al interior de organismos de la misma especie, sino más bien con una respuesta fisiológica a ciertas condiciones ambientales particulares que generan un tipo de estrés, por lo que existen especies potencialmente tóxicas que no en todos los ambientes expresan toxicidad. Usup et al. (1994) encontraron que el contenido y composición de PSP se afecta por los factores que están relacionados con la tasa de crecimiento de la especie, como es la luz. La mayor concentración de toxinas ocurre en la fase de crecimiento exponencial de la población. En cultivos de Pyrodinium, el contenido de toxina aumentó al disminuir la temperatura (de 8 a 22 °C) y la salinidad (de 24 a 20 g/kg), y disminuyó considerablemente al disminuir la intensidad luminosa de 90 a 15 µE m2 s-1. Dado lo anterior, sugerimos realizar estudios más profundos que permitan explicar cómo puede ser que poblaciones de P. bahamense var. bahamense, que no solían expresar toxicidad en su ambiente, lo hagan de manera espontánea. Dichos estudios proporcionarían evidencia sólida para determinar las condiciones bajo las cuales se expresa la toxicidad en las poblaciones de una u otra variedad, sin que esto represente un criterio decisivo en el reconocimiento de su independencia taxonómica. Al ser individuos de la misma especie, ambas serían potencialmente tóxicas, aunque no siempre lo expresarán, según el ambiente, como el caso de P. bahamense var. bahamense en el Atlántico.
Cuadro 2: Perfil de toxinas de envenenamiento por consumo de mariscos (PSP) de distintas poblaciones de Pyrodinium bahamense var. compressum (Böhm) Steidinger, Tester & Taylor (Pbc) y Pyrodinium bahamense var. bahamense Plate (Pbb). Abreviaturas: dc-STX=decarbamoil saxitoxina, STX=Saxitoxina (grupo carbamoil), NeoSTX=Neosaxitoxina (grupo carbamoil), GTX1, GTX4 Gonyautoxina 1, Gonyautoxina 4 (grupo carbamoil), GTX5, GTX6=Gonyautoxina5, Gonyautoxina 6 (grupo N-sulfodecarbamoil), C1-C4 (grupo N-sulfcarbamoil). Las toxinas marcadas con * son las más potentes. Los símbolos indican: (-) Ausencia de toxina, (+) Bajo porcentaje de toxina >50% y (++) Alto porcentaje de toxina <50%.
| Toxinas PSP/var.-procedencia-referencia | dc-STX | STX* | NeoSTX* | GTX1* | GTX 2 | GTX3 | GTX4* | GTX5 (B1) | GTX6 (B2) | C1-C4 |
| Pbc | ||||||||||
| Malasia Usup et al. (1994) | + | + | ++ | - | - | - | - | ++ | + | - |
| Indopacific populations Hummert et al. (1997), Montojo et al. (2006), Gedaria et al. (2007). | + | + | + | - | - | - | - | + | + | - |
| Guatemala (Rosales-Loessener (1989b) | + | + | - | + | + | + | - | - | - | |
| México (Acapulco) Gárate-Lizárraga et al. (2012) | ++ | ++ | - | - | + | + | + | + | ||
| Pbb | ||||||||||
| Florida Indian River Lagoon (Landsberg et al. (2006) | + | + | - | - | - | - | - | + | - | - |
Todas las secuencias utilizadas en la presente contribución ya han sido publicadas en los trabajos de Mertens et al. (2015) y Cusick et al. (2016); en sus análisis filogenéticos claramente se observa una diferenciación genética entre las variedades bahamense y compressum. Sin embargo, dichos autores desconocen la validez taxonómica de estas variedades, pues discuten que existe solo una ligera variación genética entre ellas.
En los resultados que mostraron nuestras redes de parsimonia estadística, utilizando los tres marcadores (Lcf, LSU y SSU), fue siempre consistente y evidente una estructuración genética completa entre las dos variedades. Las secuencias de P. var. bahamense siempre se agruparon en una red, o incluso en dos redes independientes con el marcador Lcf; mientras que las secuencias de la var. compressum, siempre formaron otra red, igualmente independiente. Asimismo, la correspondencia de los haplotipos y ribotipos resultantes con los tres marcadores fue siempre la misma. Es decir que, en las tres redes estadísticas, las muestras del Océano Indo-Pacífico siempre se colocaron en un grupo diferente al de las del Atlántico occidental; no hubo interconexión. Particularmente, la diferencia de bases entre ambas variedades fue muy superior a lo esperado con SSU, lo cual se suma al hecho de que tradicionalmente los marcadores nucleares suelen ser altamente polimórficos y no permiten realizar hipótesis filogenéticas certeras (Saunders, 2005). Contrariamente el marcador LSU, a pesar de ser nuclear, resultó ser muy poco polimórfico y conservado para este género. No obstante, aunque los dos grupos de variedades estuvieron interconectados en la red, se observó una distancia genética notable entre ellos (cinco pasos mutacionales), lo que sugiere también un aislamiento reproductivo. Para Lcf, la red de parsimonia estadística mostró tres estructuras genéticas representadas en tres redes o clados independientes: dos correspondientes a las poblaciones del Atlántico occidental y una correspondiente con las del Indo-Pacífico. Este resultado igualmente sugiere que, al interior de las poblaciones del Atlántico occidental, particularmente en las de River Lagoon, Florida, podría haber una tercera variedad involucrada dentro del espectro morfológico atribuido a la variedad P. bahamense var. bahamense, aún subestimada.
Los análisis filogenéticos, en concordancia, también mostraron la misma estructura que las redes de parsimonia, donde las secuencias del Indo-Pacífico formaron un grupo monofilético, independiente del clado conformado por las secuencias del Atlántico occidental, lo que implica una diferencia biogeográfica importante entre las variedades. Dado que se realizó un análisis concatenado con los tres marcadores utilizados en nuestro análisis, la filogenia también evidenció una tercera estructura genética al interior de las poblaciones de P. bahamense, hasta ahora no detectada con los marcadores SSU y LSU en estudios anteriores; sino que fue expuesta hasta los análisis filogenéticos realizados con Lcf (Cusick et al., 2016). Esto responde en gran parte al hecho de que los marcadores moleculares utilizados hasta ahora en la construcción de hipótesis filogenéticas, con SSU y LSU, no son específicos para dinoflagelados, sino que son universales para estudios con eucariontes en general (Scholin et al., 1994; Matsuoka et al., 2006). Por esta razón, las distancias genéticas detectadas a nivel interespecífico entre diversos géneros de dinoflagelados es tan corta que, en muchos casos, no resuelve concretamente las relaciones filogenéticas entre especies. Sin embargo, marcadores más específicos y menos conservados o degenerados, como Lcf, permitirán detectar cambios evolutivos más recientes, lo que a su vez resolvería de manera más robusta la filogenia. Particularmente para P. bahamense, el conjunto de análisis que presentamos, concretamente la presencia de esta tercera estructura con Lcf, deja en claro la ausencia de flujo genético entre las dos variedades, por lo que al estar aisladas reproductivamente se sustenta su independencia y validez taxonómica. Incluso la diferencia genética entre ellas, en cuanto al número de sitos polimórficos o variables, mostrada con los marcadores Lcf (38 bp) y SSU (288 bp), sugiere que esta estructura puede corresponder a especies distintas dentro del género Pyrodinium. Esta posibilidad revela la necesidad de realizar un estudio morfológico, molecular y ecológico más amplio, en el que se incluyan además secuencias de poblaciones del Pacífico tropical mexicano y Centro América, y del Golfo de México y el resto del Caribe ya que, hasta ahora, los estudios moleculares involucran solo organismos de Florida, Puerto Rico, Jamaica y del Indo-Pacífico. Si bien no existen caracteres morfológicos que permitan discriminar entre la var. compressum y la var. bahamense, los límites de distribución de las entidades genéticas reconocidas y, en consecuencia, los factores ecológicos asociados a esta distribución, sí las discriminan. Por lo tanto, rechazamos la existencia de un complejo ente ambas variedades. Es importante hacer énfasis en que, si existiera un complejo, este estaría al interior de las poblaciones de P. bahamense var. bahamense, que comparten distribución y condiciones ambientales en el Atlántico. Sin embargo, las poblaciones de P. bahamense var. compressum se encuentran claramente fuera de dicho complejo ya que, aunque filogenéticamente estén emparentadas (porque son variedades de una especie o posiblemente de tres especies de un mismo género), no están interconectadas genéticamente. Consecuentemente, es importante describir a fondo esta tercera estructura genética en un estudio posterior, debido a que de momento no contamos con más información que las tres secuencias moleculares disponibles en Genbank (2020), por lo que no podemos describirla o establecerla como tercera variedad o especie de Pyrodinium.
En conclusión, tanto las redes haplotípicas como los análisis filogenéticos mostraron la ausencia de flujo genético entre las dos variedades de Pyrodinium (var. bahamense y var. compressum), por lo que, al estar aisladas reproductivamente, se sustenta su validez taxonómica, cumpliendo con el concepto biológico de especie (Mayr, 1942). A pesar de que se ha reportado la coocurrencia de ambas, como hemos discutido anteriormente, no existe evidencia de que la variedad tóxica que se reportó en Florida corresponda morfológicamente con var. compressum o var. bahamense, ya que además se ha reportado traslape de caracteres morfológicos diagnósticos entre ambas (Meave del Castillo et al., 2019). Por otro lado, nuestros resultados genéticos proporcionan evidencia sobre la posibilidad de que, al interior de Pyrodinium (género monoespecífico), existan al menos dos especies, en vez de variedades. Dado lo anterior, proponemos la validez e independencia taxonómica de P. bahamense var. compressum y var. bahamense, cuyo sustento no solo es genético, sino ecológico y biogeográfico, aunque no morfológico. Adicionalmente, no solo es necesario realizar estudios más integrales entre todos los enfoques biológicos (ecológico, biogeográfico, taxonómico, evolutivo y fisiológico), que permitan encontrar caracteres más robustos que delimiten a estas variedades u otras especies en conflicto, sino también aceptar la evidencia proporcionada por los genes, aunque esta no empate con la morfología. En la actualidad, el criterio morfológico prevalece, erróneamente, sobre la evidencia molecular en la toma de decisiones taxonómicas, lo que se ve plasmado en un sistema de clasificación artificial e inestable, que hasta ahora no refleja la historia evolutiva ni las relaciones filogenéticas entre las especies de fitoplancton. Tal es el caso del género Alexandrium, que en todos las filogenias no es monofilético (Scholin et al., 1994; Cusick et al., 2016), por lo que la sistemática de todo este grupo debe ser reevaluada. En la toma de decisiones taxonómicas, lo que se ve plasmado en un sistema de clasificación artificial e inestable, que hasta ahora no refleja la historia evolutiva ni las relaciones filogenéticas entre las especies.











 nueva página del texto (beta)
nueva página del texto (beta)



