Introducción
Los métodos estadísticos para el estudio de la variación morfológica de los caracteres taxonómicos han estado disponibles durante muchos años (Jeffers, 1967). Sin embargo, los métodos de trabajo básicos de los sistemáticos han dependido de impresiones “Gestalt” (Jäkel et al., 2016) para las comparaciones cualitativas de las características (Poe y Wiens, 2000; Rieppel y Kearney, 2007). Obviamente no toda la variación es cuantificable, como la presencia/ausencia de estructuras. Pero en el caso de los rasgos mensurables, las descripciones y las comparaciones de la variación deben basarse en mediciones y elementos de juicio cuantitativos si pretendemos que sean conjeturas formales sobre patrones de variación, estados y caracteres taxonómicos (Humphries, 2002; Houle et al., 2011). Tal formalidad es posible con las mediciones de la variación de caracteres morfológicos registradas con un lenguaje numérico y las comparaciones con técnicas de análisis estadísticos (Marcus et al., 1996; Van Valen, 2005). En esta contribución se revisan conceptos y métodos cuantitativos acumulados desde hace décadas usando mediciones lineales (Jeffers, 1967; Reyment et al., 1984; Marcus, 1990) y otros recientemente disponibles usando coordenadas Cartesianas de puntos (Claude, 2008; Zeldtich et al., 2012; Bookstein, 2018) para el análisis de los datos morfométricos. A diferencia de otras revisiones previas sin aplicaciones en sistemática y con un enfoque práctico (Toro-Ibacache et al., 2010; Jaramillo, 2011; Benítez y Püschel, 2014), aquí se enfatizan las bases teóricas de los espacios matemáticos y la lógica geométrica del morfoespacio de un carácter taxonómico. El objetivo de esta revisión es suministrar elementos conceptuales para una comprensión básica de los métodos morfométricos y estadísticos útiles en la sistemática bajo un enfoque filogenético. La tesis central de este artículo es que la descripción de los caracteres morfológicos mensurables del tamaño y la forma requiere el lenguaje numérico que proveen las mediciones lineales y las coordenadas Cartesianas, y que la comparación sólo puede calificarse mediante un procedimiento estadístico.
El desarrollo de la teoría y los métodos morfométricos han proporcionado conjuntos de herramientas para describir y comparar la variación morfológica. Estos métodos morfométricos se están usando ampliamente en aplicaciones de biología comparativa (Monteiro, 2013). Un ejemplo es el modelado de los procesos de tasas de cambio y diversificación morfométrica usando filogenias (Gavrilets, 1999; Revell et al., 2008). Otro ejemplo es el uso de la estructura filogenética para postular procesos explicativos de la variación genética y morfométrica en las especies de cícadas del género Ceratozamia Brongn. (Medina-Villareal et al., 2019). En contraste, la mayoría de las aplicaciones de la morfometría en sistemática con medidas lineales y con coordenadas de puntos se han implementado con el objetivo de agrupar fenéticamente y distinguir grupos taxonómicos (Hodálová et al., 2008; Depypere et al., 2009; Valcárcel y Vargas, 2010). Los análisis morfométricos en los cuales los grupos taxonómicos se obtienen en función de la similitud global equivalen a realizar análisis fenéticos (Rohlf, 1971; James y McCulloch, 1990; Quicke, 1993; Sneath, 1995; Marhold, 2011; Williams et al., 2010). La presente revisión propone que, bajo un enfoque filogenético, los análisis morfométricos se pueden usar para el estudio de la variación de caracteres taxonómicos y los estados, y no sólo para agrupar fenéticamente.
La formación de grupos taxonómicos depende de las hipótesis de los grupos monofiléticos y homologías en una filogenia (Wheeler, 2012). Epistemológicamente los grupos taxonómicos se conceptualizan como grupos monofiléticos (De Luna, 1995; Mishler y De Luna, 1997) y metodológicamente los sistemáticos aplican análisis de agrupación filogenética basados en parsimonia, verosimilitudes o probabilidades Bayesianas posteriores (De Luna et al., 2005). Los métodos y el software para la integración de los datos morfométricos con conceptos filogenéticos, como homología y monofilia, son relativamente recientes (Goloboff et al., 2006; Catalano et al., 2010). Una de las primeras aplicaciones fue el análisis filogenético con parsimonia de las coordenadas de la configuración del ala en cinco especies de chinches de la tribu Rhodniini y cuatro grupos externos (Soto-Vivas et al., 2011), usando un script para TNT (land_search.run, Catalano et al., 2015). El desarrollo del software para los análisis filogenéticos brinda la oportunidad de integrar los datos morfométricos en la investigación taxonómica y filogenética para descubrir homología táxica y homología transformacional (De Pinna, 1991; De Luna y Mishler, 1996; Humphries, 2002; Palci y Lee, 2019). Las configuraciones de puntos se pueden analizar en combinación con otros tipos de caracteres con métodos de parsimonia mediante búsquedas intensivas con una variedad de algoritmos ahora ya incluidos en el software TNT (Goloboff y Catalano, 2016). Ejemplos recientes de análisis filogenéticos que combinan las coordenadas con los datos moleculares en la inferencia de filogenias incluyen los de Botero-Trujillo et al. (2017), Bona et al. (2018) y Solís-Zurita et al. (2019).
Con tantos caracteres moleculares (nucleótidos, transcriptomas) disponibles se podría cuestionar si los caracteres morfológicos son necesarios para la inferencia de filogenias. A este argumento se podrían agregar las dificultades para definir caracteres morfológicos y codificarlos de maneras no ambiguas ni subjetivas (Gift y Stevens, 1997; Hillis y Wiens, 2000). No obstante, argumentos empíricos, metodológicos y filosóficos dejan comprender que los caracteres morfológicos seguirán siendo relevantes. Bajo un enfoque integrativo la morfología no debe considerarse como un complemento opcional a la filogenia molecular, sino como un componente esencial de nuestra comprensión de las especies y su diversificación (Wheeler, 2008). Además, la inclusión de fósiles como terminales en las filogenias para la clasificación y la datación de filogenias señala la necesidad de incorporar datos morfológicos. Los métodos morfométricos pueden reducir la subjetividad en la conceptualización de los caracteres (Humphries, 2002; Freudenstein, 2005; Rieppel y Kearney, 2007) y la identidad de los estados de carácter (Brower y Schawaroch, 1996; Humphries, 2002). Potencialmente, la adquisición y anotación automatizada de fenotipos facilitarían la inclusión de estos datos en los análisis filogenéticos (Giribet, 2015). Conforme se sigan desarrollando los métodos analíticos será más fácil combinar datos morfométricos con datos moleculares para proveer mejores estimaciones de las relaciones filogenéticas de los grupos.
Epistemológicamente se ha argumentado que las filogenias combinadas con todos los tipos de caracteres tendrán mejor soporte, estabilidad y capacidad de explicación del dominio empírico (Nixon y Carpenter, 1996; Kluge, 1997, 2004; Rieppel, 2005; Assis, 2009, 2015). Por ahora las filogenias con datos morfométricos y moleculares combinados se infieren con métodos de parsimonia (Goloboff et al., 2006; Catalano et al., 2010). Aunque han existido modelos probabilísticos para caracteres continuos (Felsenstein, 1973, 1985), los que se han adaptado para usar la morfología en la inferencia de la filogenia (Markov-k y extensiones) sólo aplican a caracteres codificados con estados discretos (Lewis, 2001; Wright y Hillis, 2014; Goloboff et al., 2018; Smith, 2019). El estudio de Ascarrunz et al. (2019) es, a mi conocimiento, el primero en analizar filogenéticamente la forma también con métodos de parsimonia cuadrada mediante scripts y con máxima verosimilitud con el software ContML-PHYLIP (Felsenstein, 1973, 2000), considerando cada coordenada como un carácter por separado. Todavía no hay modelos para poder combinar los datos morfométricos multivariados en la inferencia de la filogenia con verosimilitud o con probabilidades Bayesianas posteriores. Entonces, al lado de las perspectivas científicas y las posibilidades metodológicas, los factores sociológicos influyen considerablemente en la disposición o no de incluir la morfología y los datos morfométricos en la inferencia de la filogenia. Pero ni la convicción personal ni el escepticismo autoritario deberían arbitrar la decisión de usar o no los caracteres morfométricos en la inferencia de la filogenia. Pragmáticamente, una ventaja de la disposición a examinar el valor de los caracteres morfométricos es que puede llevar a desarrollar las habilidades teóricas y analíticas para combinar diferentes tipos de caracteres. Curricularmente, estas capacidades académicas potencialmente serían ventajosas; los taxónomos incrementarían sus oportunidades de competir por financiamiento o para ser contratados en campos afines, y también en su capacidad de interactuar y colaborar fuera de la sistemática.
Los prerrequisitos numéricos para sacar provecho de esta revisión se limitan a un conocimiento básico de geometría y estadística multivariada. Este trabajo no intenta dar una visión completa de las diferentes concepciones teóricas y metodológicas sobre cómo analizar estadísticamente los datos morfométricos. Por ejemplo, aquí no se revisan las aproximaciones teóricas y metodológicas basadas en superposiciones robustas con métricas lineales (Torcida et al., 2014). Tampoco se revisan la teoría y métodos de la morfometría que substituyen las superposiciones por el análisis de matrices de distancias Euclidianas (Euclidean Distance Matrix Analysis, Lele, 1993; Lele y Richtsmeier, 2001). Únicamente se examinan la teoría y los métodos para el estudio de la variación estadística de las formas bajo la perspectiva de la métrica de Procrustes, pues es la más ampliamente usada (Zelditch et al., 2012; Adams et al., 2013). Por lo tanto, en la primera sección se presentan un repaso breve de geometría vectorial, el espacio de Kendall, la distancia Procrustes, proyección de espacios tangenciales y construcción de hipercubos del morfoespacio. Para estimular la imaginación geométrica y facilitar recordar los conceptos (espacio de Kendall, espacios tangentes), se proveen ilustraciones no técnicas de esos conceptos y espacios matemáticos. En la segunda sección se repasan brevemente los conceptos estadísticos útiles para la aplicación de los Análisis de Componentes Principales (PCA) y los Análisis de Variables Canónicas (CVA) en sistemática. En la tercera sección se ubican aplicaciones de la morfometría en el contexto de diversos campos de investigación en la sistemática bajo una visión pluralista. Se identifica un cambio epistemológico necesario para pasar de aplicaciones bajo una sistemática fenética a la integración de los análisis morfométricos como parte de la investigación filogenética. Se dan recomendaciones ejemplificadas sobre el uso de datos morfométricos de mediciones lineales y coordenadas de puntos para la identificación de especies, el análisis de caracteres taxonómicos y el uso de los datos morfométricos en la inferencia de la filogenia y clasificación.
Conceptos de morfometría para sistemática
Aunque la práctica permite la experiencia de operar la amplitud de los procedimientos morfométricos y estadísticos, la pericia con las herramientas y software “point and click” no es suficiente. Como en cualquier otra disciplina científica (Wild y Pfannkuch, 1999), en morfometría hace falta el discernimiento de una estructura teórica con la cual hallarle sentido y sacarle provecho a cada nueva experiencia. Entonces primero se bosqueja aquí la teoría, luego los métodos morfométricos y finalmente las aplicaciones en sistemática. En la práctica, la preparación de las matrices, la exploración de la estructura de la variación y la aplicación de modelos estadísticos es muy fluida cuando es guiada por los manuales en la mayoría de los paquetes de software. Para el análisis de mediciones lineales es muy útil el manual en línea de StatSoft (2013). Para las coordenadas, el manual en línea de MorphoJ es un excelente texto (Klingenberg, 2011). No obstante, la elección de la metodología y el software apropiados se ha convertido en una tarea difícil dada la complejidad inherente de los conceptos y métodos de la morfometría geométrica. Aquí se brinda un panorama general de los aspectos básicos del marco conceptual desarrollado en los últimos años para los métodos en morfometría. Esta visión general de la teoría morfométrica pretende ayudar a los biólogos con diversos niveles de antecedentes estadísticos y sofisticación, en su responsabilidad individual de entender las propiedades y supuestos subyacentes de los métodos morfométricos y estadísticos que usan.
Morfometría
Morfometría es el estudio de la forma y tamaño de objetos empíricos mediante el uso de descriptores cuantitativos (Fig. 1). Una excelente reseña de la historia, el desarrollo de conceptos, métodos y las expectativas para el futuro de la disciplina es el artículo de MacLeod (2017). Aquí sólo se incluye la metodología para el registro numérico de la morfología usando mediciones entre puntos de referencia y coordenadas Cartesianas de los puntos. Un conjunto de estos números constituye un objeto matemático “X” y es la representación matemática de un objeto empírico (Tal, 2013). La colección de objetos matemáticos permite la aplicación de métodos estadísticos para el análisis de los promedios y las varianzas. La métrica para comparar los registros numéricos de mediciones lineales produce espacios Euclidianos que pueden ser directamente analizados con métodos estadísticos regulares de la estadística multivariada (Fig. 1). En contraste, los registros de las coordenadas localizan las muestras (n) en espacios curvos no-Euclidianos (espacio de Kendall, Fig. 1). En este caso, se derivan variables ad hoc (“partial warps”) para los análisis con métodos estadísticos especiales de la morfometría geométrica multivariada (Bookstein, 1992; Zelditch et al., 2012). En esta sección se señalan características deseables de los datos, de las variables y del diseño de muestreo. Posteriormente se resumen los teoremas geométricos sobre los espacios matemáticos definidos por las mediciones y por las coordenadas. Finalmente se repasan los ajustes para la selección de ejes latentes para la construcción de un hipercubo de análisis del morfoespacio (Fig. 1). Estos conceptos geométricos básicos facilitan entender los conceptos formales del tamaño y de la forma, los métodos para seleccionar ejes latentes y los métodos estadísticos para poder analizar la variación morfológica.
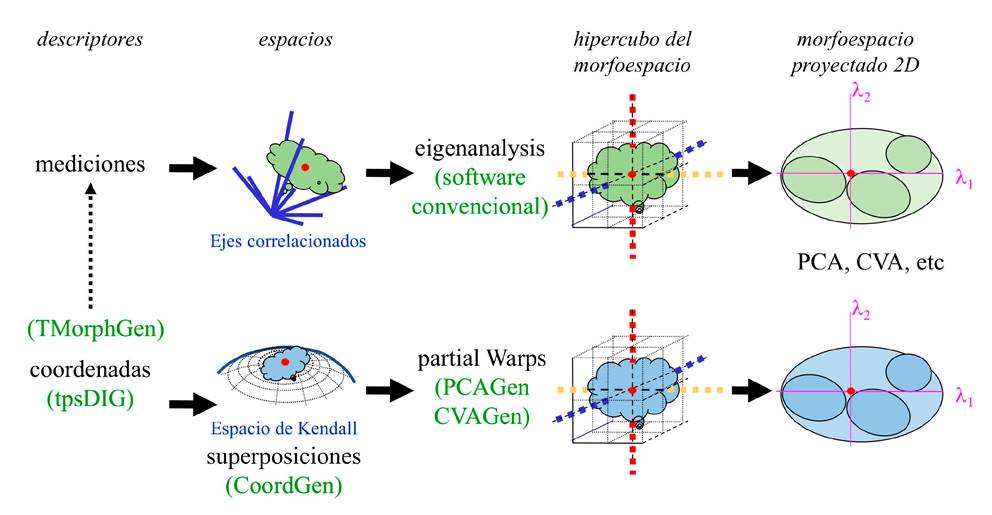
Figura 1: Morfometría. Desde las mediciones y coordenadas Cartesianas de puntos a espacios geométricos, ajustes para la formación del hipercubo del morfoespacio y la proyección 2D del espacio multivariado ortogonal. El diagrama muestra también la secuencia de operaciones con los programas típicos en cada fase por las dos rutas hasta la ejecución de análisis estadísticos.
Tamaño
En el contexto matemático y geométrico, el tamaño es una cantidad escalar. El tamaño de un objeto físico es una cantidad que mide la magnitud en alguna escala. Una cantidad escalar sólo tiene “magnitud“, no hay “dirección”, como en el caso de una cantidad vectorial. El tamaño es una cantidad escalar que puede ser referida a la unidad de medida de un sistema físico y también a cualquier objeto matemático. Cuando se refiere a un objeto físico en 2D o 3D, el tamaño se puede expresar como la medida de longitud, área, volumen, masa, o combinaciones de estas cantidades escalares. El tamaño también puede medirse como un escalar multivariado. Por ejemplo, la combinación de varias mediciones de longitudes y anchos de un objeto miden el tamaño de manera multivariada. Considerando los seis “pentagonoides” de la Figura 2 (modificados de la Fig. 2.31. p 49, Banchoff y Wermer, 1991), ¿cuál es el objeto más grande? El Cuadro 1 informa los valores de seis mediciones observadas en cada “pentagonoide”. La colección con los valores más altos es el “pentagonoide b”, los valores más pequeños son los del “pentagonoide a” y el “d”. Esos valores corresponden con la ordenación intuitiva de los diferentes tamaños.
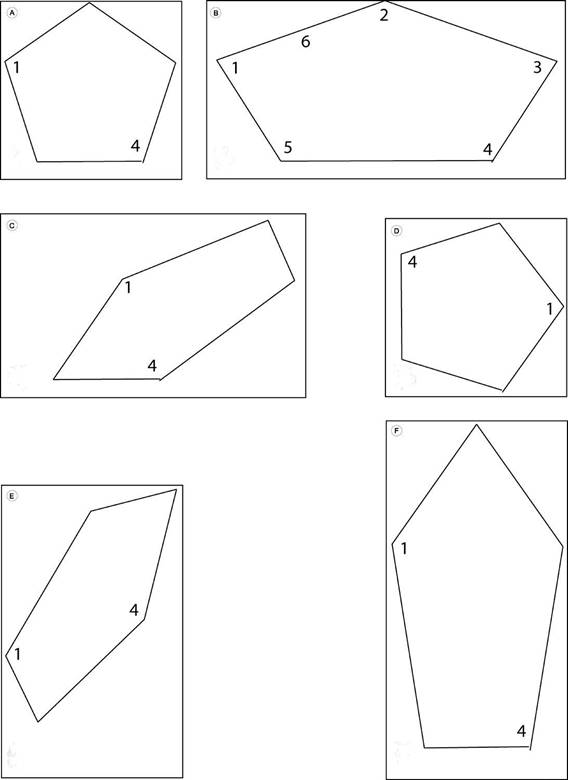
Figura 2: “Pentagonoides”. Seis objetos geométricos con puntos numerados en los vértices. Los puntos 1 al 5 se usaron para registrar las coordenadas Cartesianas, con el origen localizado en la esquina inferior izquierda del rectángulo contenedor de cada “pentagonoide”. El punto 6 se usó además para calcular la distancia entre los puntos 6 y 4 y las cinco mediciones de cada lado.
Cuadro 1: Mediciones lineales de los seis “pentagonoides” en la Figura 2. Se miden las longitudes “m1-6” de cada lado empezando desde el punto etiquetado con un “1”. La sexta longitud medida m6 es entre el punto “6” y el punto “4”. En cada “pentagonoide” el punto “6” lo ubicamos a la mitad del lado definido por los puntos “1” “y 2”. Los seis valores medidos en centímetros en ese orden son mostrados en la siguiente matriz (n=6, v=6).
| m1 | m2 | m3 | m4 | m5 | m6 | |
| a | 18 | 18 | 18 | 18 | 18 | 27 |
| b | 30 | 30 | 20 | 35 | 20 | 37 |
| c | 25 | 11 | 28 | 18 | 20 | 22 |
| d | 18 | 18 | 18 | 18 | 18 | 27 |
| e | 29 | 10 | 23 | 25 | 12 | 17 |
| f | 25 | 25 | 34 | 18 | 34 | 45 |
Cuando el objeto empírico se registra con las coordenadas Cartesianas (2D, 3D) de varios puntos se produce un objeto geométrico “X”. Un triángulo, un pentágono o un polígono irregular es el objeto geométrico derivado del registro de las coordenadas de tres, cinco o varios puntos. En este caso, el tamaño de un objeto geométrico es una cantidad que mide la magnitud sin escala, es decir, un escalar matemático sin unidades de medida físicas. En los objetos geométricos, una manera de medir esta magnitud escalar es estimar las distancias de cada punto al centro del polígono. Geométricamente esto equivale primero, a calcular la distancia cuadrada entre cada punto y el centro, luego el tamaño geométrico se obtiene al extraer la raíz cuadrada de la suma de esas distancias. Esta magnitud escalar de una configuración de puntos se expresa sólo con un número real sin unidades de medida y se conoce como Tamaño Centroide (CS, “Centroid Size”, Bookstein, 1992; Zelditch et al., 2012).
El ejemplo con los seis “pentagonoides” (Fig. 2) transmite la idea intuitiva y el concepto matemático de tamaño. Las coordenadas de registro superpuestas usando CoordGen (Sheets, 2014a) generan la matriz de nuevas coordenadas de la forma (definida en la siguiente sección) y los valores de CS para cada figura (Cuadro 2). Los valores de la variable Tamaño Centroide (CS) van ubicados antes del símbolo “%”, en el formato de CoordGen. Según el valor máximo (625.47), el “pentagonoide b” es el más grande. Nótese también que los “pentagonoides a” y “d” tienen prácticamente el mismo valor de CS (a= 394.89 y d=394.417). Estas estimaciones de valores del CS son congruentes con las estimaciones de los valores de tamaño a partir de la colección de mediciones. Por consiguiente, la colección de mediciones lineales mide fundamentalmente variación en tamaño, mientras que las coordenadas superpuestas también miden variación en tamaño, pero en la variable CS separada de, y no correlacionada con, las variables de la forma.
Cuadro 2: Coordenadas Procrustes de los seis “pentagonoides” en la Fig. 2. Las coordenadas iniciales de registro se sometieron a una superposición Procrustes usando CoordGen. La primera columna contiene la identidad de los “pentagonoides “a-d, según la Figura 2; “Xn, Yn” son los pares de coordenadas rectangulares de cada punto 1-5; CS=centroid size; archivo=nombre de la fotografía digital; e=etiqueta del objeto matemático en la matriz.
| p | X1 | Y1 | X2 | Y2 | X3 | Y3 | X4 | Y4 | X5 | Y5 | CS | archivo | e | |
| A | -0.424423 | 0.135222 | -0.00152474 | 0.444174 | 0.428978 | 0.137765 | 0.266914 | -0.36111 | -0.269944 | -0.356051 | 394.89 | % | pentagonoideA.jpg | 0 |
| B | -0.540261 | 0.0603161 | -0.00967607 | 0.283481 | 0.536079 | 0.112025 | 0.338318 | -0.212483 | -0.32446 | -0.243338 | 625.476 | % | pentagonoideB.jpg | 1 |
| C | -0.262228 | -0.000649298 | 0.181629 | 0.485097 | 0.368288 | 0.295787 | 0.0532519 | -0.299723 | -0.340942 | -0.480511 | 486.563 | % | pentagonoideC.jpg | 2 |
| D | -0.427852 | 0.135429 | 0.000888755 | 0.446924 | 0.427657 | 0.135413 | 0.264258 | -0.360665 | -0.264952 | -0.357102 | 394.417 | % | pentagonoideD.jpg | 3 |
| E | -0.413761 | -0.0600367 | 0.146796 | 0.327243 | 0.51607 | 0.267203 | 0.163814 | -0.170507 | -0.412919 | -0.363903 | 481.34 | % | pentagonoideE.jpg | 4 |
| F | -0.261422 | 0.185373 | 0.0254773 | 0.566392 | 0.276484 | 0.161401 | 0.146046 | -0.46831 | -0.186585 | -0.444855 | 624.029 | % | pentagonoideF.jpg | 5 |
Forma
El concepto matemático de forma depende de los conjuntos de valores de las coordenadas Cartesianas registrados en una configuración de puntos en varios objetos empíricos. Para el objetivo de la presente revisión se denominan puntos tanto a las marcas (landmarks) como a las semi-marcas (semi-landmarks). La colocación de los puntos considerados marcas está condicionada por lugares anatómicos de referencia, mientras que la ubicación de los puntos considerados semi-marcas se determina mediante algún algoritmo para registrar una curva. Se denomina “configuración” (configuration, Small, 1996; Dryden y Mardia, 1998) al conjunto de puntos “p” registrados para un objeto, sean marcas, semi-marcas, o combinaciones de ambos tipos. Consideremos un mapa de “p” puntos en un plano “A” con las coordenadas (x1, y1, x2, y2, ….., xn, yn) y otro mapa en otro plano B con el mismo número de puntos y con un sistema de coordenadas (u, v). La pregunta es si estos dos mapas o configuraciones de puntos tienen la misma forma o no. El ejemplo con los seis “pentagonoides” (Fig. 2) ilustra la noción intuitiva y el concepto matemático de forma. Si la esquina inferior izquierda es el origen del sistema rectangular (x=0, y=0), los valores de las coordenadas Cartesianas 2D de un mapa de los cinco puntos en cada “pentagonoide” serán muy diferentes. Intuitivamente percibimos que el “pentagonoide a” y el “pentagonoide d” tienen la misma forma, pero no es difícil imaginar que los cinco puntos obtendrán diferentes valores de coordenadas por diferente rotación de los dos mapas de puntos. En general, cuando registramos coordenadas de varios puntos “p” en muchos objetos, cada mapa podría diferir en tamaños, rotaciones y translaciones.
Matemáticamente, la forma es descrita por las propiedades geométricas entre los puntos (marcas y semi-marcas) de cada configuración, luego de haber sido superpuestos los mapas. Las diferencias debido a las rotaciones de los objetos físicos y las diferencias del registro de coordenadas por el desplazamiento del punto de referencia (translación) deben ser eliminadas antes de hacer la comparación de los objetos. Cuando sólo se remueve el efecto de translación, el conjunto de puntos se denomina “configuración centrada” (Helmertized/centered, Small, 1996; Dryden y Mardia, 1998). El conjunto de puntos contiene información combinada de “tamaño y forma” (size and shape, Small, 1996; Dryden y Mardia, 1998) cuando se remueven los efectos de translación y rotación. Alternativamente, las coordenadas contienen información de “pre-forma” (pre-shape, Small, 1996; Dryden y Mardia, 1998) cuando se remueve los efectos de translación y del tamaño, pero no el de rotación. Finalmente, se denomina “forma” al conjunto de las coordenadas cuando se han removido los efectos de translación, rotación y tamaño (shape, Small, 1996; Dryden y Mardia, 1998). En la presente revisión se prefiere este sistema de nomenclatura en español, en acuerdo con traducciones establecidas en revisiones previas (Toro-Ibacache et al., 2010; Benítez y Püschel, 2014) en lugar de la traducción shape=conformación sugerida por Jaramillo (2011).
En la práctica, los puntos se registran con una referencia local de un sistema de coordenadas rectangulares a cada figura. Por ejemplo, la esquina inferior izquierda de cada foto digital por convención tendrá las coordenadas x=0, y=0. Las coordenadas de registro de los “pentagonoides” se colectaron en cinco puntos con tpsDig (Rohlf, 2017) y se sometieron a una superposición usando CoordGen (Sheets, 2014a). Los valores en el Cuadro 2 son las nuevas coordenadas en un sistema Cartesiano común a todas las configuraciones y entonces describen forma. Las dos colecciones de los valores pareados “x, y” en la primera y la cuarta hilera prácticamente son los mismos, lo cual revela que el “pentagonoide a” y el “pentagonoide d” tienen la misma forma. Este ejemplo ilustra que matemáticamente la forma se define como las propiedades de las coordenadas Cartesianas de puntos en un mapa que permanecen después de remover los efectos de tamaño, ubicación y orientación de los objetos mediante algún tipo de superposición (Zelditch et al., 2012).
Superposición
Los métodos de superposición separan el efecto del tamaño y eliminan las diferencias por translación y rotación (Zelditch et al., 2012). Las coordenadas iniciales registradas respecto a la esquina izquierda de cada fotografía se re-expresan en coordenadas de superposición comunes para todos los mapas. Estos valores de la forma describen información geométrica de “ubicación” y “magnitud” que es “invariante” (en el sentido matemático, Lele y McCulloch, 2002) bajo translaciones, rotaciones o re-escalamientos isotrópicos (expansión o contracción uniforme) del mapa de puntos de un objeto. Es decir, estas tres operaciones numéricas pueden cambiar los valores de las coordenadas, pero no alteran la figura, pues las relaciones geométricas entre los puntos de las configuraciones superpuestas se conservan.
Los diversos métodos de superposición ajustan la dispersión de perturbación en cada punto minimizando las sumas de las distancias cuadradas o las distancias lineales entre los mapas de coordenadas de diferentes figuras. Después de la superposición, dos objetos con forma distinta tendrán valores diferentes en al menos un punto de la configuración. La Figura 3A muestra el caso más sencillo con la comparación de dos triángulos desde la perspectiva de la “Superposición Bookstein”. Bajo este modelo, el usuario elige dos puntos de referencia fijos (varianza=cero, error=cero) con coordenadas p1= “0,0” y p2= “1,0” para interpretar la variación relativa en los demás puntos libres (Zelditch et al., 2012). Por definición, la diferencia entre la forma de los dos triángulos es el desplazamiento de un punto. En contraste, la comparación entre los dos primeros “pentagonoides” de la Figura 2 ilustra otro aspecto de los efectos de la superposición basada en mínimos cuadrados. Intuitivamente percibimos que los valores en las coordenadas en “y” permanecen igual y las diferencias entre los dos “pentagonoides” sólo son un estiramiento uniforme en el eje de las “x”s (Fig. 3B). Evidentemente las distancias entre los puntos 1-3 y 5-4 son mayores en el “pentagonoide b” que en el “pentagonoide a”. No obstante, usando la “Superposición Procrustes Parcial”, el punto de referencia fijo es el centro de cada figura (c=0,0) y se infieren desplazamientos en todas las direcciones “x, y” de los cinco puntos (Fig. 3C). Un ejemplo más complicado es la comparación de la forma del cáliz en varias especies de hierbas del género Salvia L. (Lamiaceae). Las coordenadas de varios puntos en los contornos (semi-marcas, Fig. 3D) se ajustaron con tres tipos de superposición en CoordGen (Sheets, 2014a): “Bookstein”, “Sliding Base Registration” y “Procrustes Parcial”. Las diferencias entre formas, dadas por la dispersión alrededor de cada punto, se perciben de manera distinta según la superposición empleada (Fig. 3E-G). El efecto de la superposición en los patrones de dispersión de puntos es todavía más notable en las configuraciones de mentones de coleópteros de la familia Passalidae (Fig. 3H-J).
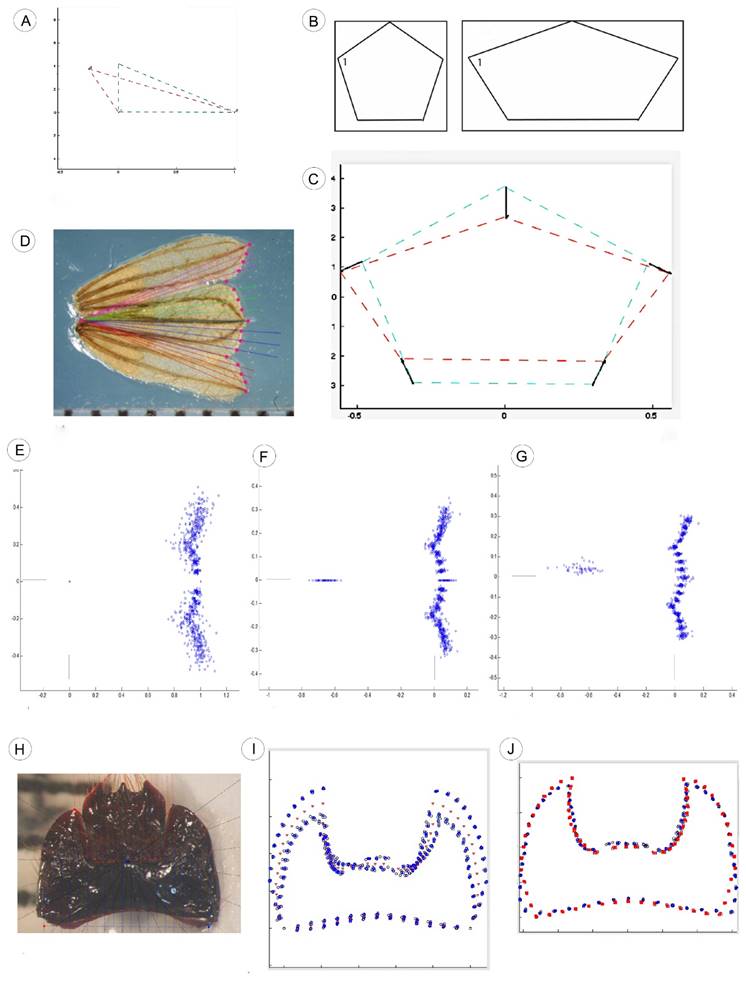
Figura 3: Superposiciones. A. El caso más sencillo con la comparación de dos triángulos desde la perspectiva de la superposición Bookstein. La diferencia entre la forma de los dos triángulos es el desplazamiento de un punto; B. Comparación entre dos “pentagonoides”; C. La diferencia en forma se infiere con desplazamientos en los cinco puntos, usando la superposición Procrustes; D. Contornos del cáliz de especies de Salvia L. (Lamiaceae) registrado con semi-marcas; E Superposición Bookstein; F. Superposición “sliding base registration”; G. Superposición Procrustes; H. Contornos del mentón de coleópteros de la familia Passalidae registrados con semi-marcas; I. Desplazamientos mayores en los puntos superiores usando la superposición Bookstein; J. Desplazamientos distribuidos en todos los puntos cuando se usa la superposición Procrustes. Fotografía y datos en D-G, cortesía de Itzi Fragoso. Fotografía y datos en H-J, cortesía de Larry Jiménez.
Estos ejemplos alertan que, desde la perspectiva biológica, el cambio inferido en la forma se modifica con la aplicación de modelos de superposición y que los desplazamientos son siempre relativos entre los puntos de una configuración y entre configuraciones. Esto se debe a que, desde una perspectiva estadística paramétrica particular, la variación en las coordenadas Cartesianas se analiza especificando un modelo de perturbación Gaussiana, el cual presupone dispersión (varianza y error) en todas direcciones en cada punto de la configuración, exista o no empíricamente (Goodall, 1991; Richtsmeier et al., 2005; von Cramon-Taubadel et al., 2007). Consecuentemente, una limitación importante de los métodos de morfometría geométrica es que es imposible localizar qué partes de la forma permanecen fijas y cuáles han cambiado biológicamente (Polly, 2018). Matemáticamente se modelan “residuales” en todos los puntos, pues los métodos de superposición distribuyen diferentes componentes de la deformación, varianzas y errores en proporciones diferentes punto a punto, incluso en direcciones donde no esperaríamos desplazamientos, como el caso de los dos “pentagonoides” (Fig. 3B). Ésta es una consideración importante, pues a partir de los componentes de la deformación en todas las direcciones en todos los puntos entre todos los objetos se calculan los “partial warps” como nuevas variables para los análisis estadísticos.
Los resultados de los análisis de varianza en la forma dependen sensiblemente del método particular usado para superponer configuraciones y no hay razones biológicas aparentes para elegir entre métodos (Strauss, 2010a). En términos estadísticos, el método menos recomendado es la “Superposición Bookstein”, pues los dos puntos fijos inducen correlaciones en los puntos libres, dependiendo de su distancia a la línea base. Bookstein (1996b) mismo, explícitamente advirtió de las limitaciones de estas coordenadas para PCA. Usualmente el método más recomendado para análisis estadísticos es la “Superposición Procrustes parcial”, pues los distintos errores (von Cramon-Taubadel et al., 2007) y la varianza (Rohlf, 2003; Webster y Sheets, 2010) se minimizan y distribuyen en todos los puntos. También se ha sugerido que cuando se percibe intuitivamente que la variación se concentra en regiones específicas de la configuración de puntos, el ajuste por la “superposición de medianas repetidas” (RFTRA en CoordGen) sería el más adecuado (Torcida y Pérez, 2012). En general, para los análisis estadísticos, “un análisis exploratorio de los resultados proporcionados por ambos métodos (Procrustes y medianas repetidas) suele ser de gran utilidad” (Torcida y Pérez, 2012). Para los análisis filogenéticos, las mejores alternativas son preparar las coordenadas inicialmente mediante la superposición Procrustes y/o la superposición de medianas repetidas (Palci y Lee, 2019).
Objetos
Una de las primeras preocupaciones en un proyecto de morfometría es la amplitud del muestreo en términos del número mínimo de especímenes o muestras. Esto equivale a diseñar la estructura de una matriz de datos que permita los tipos de análisis que se pretenden realizar. Por ejemplo, unos cuántos especímenes (n=5) son suficientes para un análisis exploratorio como el de componentes principales (PCA). Esto es particularmente un límite en el caso de grupos con representantes fósiles. Por ejemplo, para la variación en un grupo de cinco géneros de erizos de mar (equinoideos), se dispuso de unas cuantas muestras y sólo se realizaron los análisis exploratorios con PCAGen (Sheets, 2014c) a partir de la variación en configuraciones con semi-marcas (Martínez-Melo et al., 2017). Pero para un análisis inferencial (ANOVA, MANOVA, CVA) se requiere un mínimo de muestras por cada grupo o nivel de comparación. El diseño de muestreo puede visualizarse en dos direcciones: el número de grupos o niveles de comparación (g) y el número de variables (v). Los requerimientos mínimos de muestreo se establecen en relación con la amplitud del proyecto (número de grupos a comparar), los tipos de datos a colectar (mediciones, coordenadas) y los métodos estadísticos de análisis (univariados, bivariados, multivariados) necesarios para responder las preguntas biológicas investigadas. El Cuadro 3 presenta una organización recomendada de las operaciones que puede ayudar a seccionar, ordenar y planear las fases de un proyecto morfométrico; este arreglo de fases también puede ayudar a organizar las secciones en la escritura de los métodos y resultados para publicación.
Cuadro 3: Organización recomendada para las operaciones en un protocolo de investigaciones morfométricas. TPS: Thin Plate Spline.
| 0 |
Especificación del tipo de pregunta de
investigación |
| ¿Cuántas
formas? -vs.- ¿Son diferentes? -vs.- ¿Cómo cambian? ¿Cuáles son los grupos a priori o las variables factoriales? (especies, fases ontogenéticas, grupos geográficos, grupos funcionales) |
|
| 1 |
Diseño de muestreo Especímenes
(n) por cada grupo o factor Fotografía digital (tamaño vs. resolución, captura vs. despliegue) Ubicación y densidad de marcas y semi-marcas (plantillas, curvas) |
| 2 |
Descripción de la
variación Registro de puntos para mediciones lineales y coordenadas Cartesianas Ajuste de las configuraciones (Superposición por Análisis de Procrustes) |
| 3 |
Cuantificación de la deformación
relativa (distancias Procrustes) Extracción de los vectores de deformación (Warps) Ordenación de la varianza generalizada (Análisis de Componentes Principales) |
| 4 |
Análisis estadísticos de
la variación (modelos e hipótesis estadísticos) Regresión, Correlación, Anovas, Manovas, etc. Gradillas de deformación relativa (TPS local) Evaluación de la robustez y soporte de hipótesis estadísticas (bootstraps, jackknifes) |
| 5 | Interpretaciones (modelos e hipótesis
biológicos) Caracteres y estados, identificación de grupos, etc |
Las descripciones y estimaciones de los dos parámetros estadísticos (promedio, desviación estándar) se basan en la aplicación del modelo Normal (univariado y multivariado). Entonces se deben disponer de un número de datos (n) suficientes para que las frecuencias observadas de los valores de una variable se aproximen a la frecuencia esperada por el modelo. Una muestra muy pequeña (n=5) no permite una estimación estable de los límites del rango de los valores extremos, de la frecuencia de los valores intermedios posibles, ni del promedio y ni de la desviación estándar. La expectativa teórica es que las estimaciones del promedio y la desviación estándar se estabilizan a partir de unas 12 o 15 observaciones. Por lo tanto, en la situación más simple considerando sólo la distribución esperada por el modelo Normal de la variación de un sólo grupo (T1), se puede recomendar un mínimo de unos 12-15 especímenes (Campbell, 1974; Giere, 1979; Byrkit, 1987).
Sin embargo, en el caso de dos o más taxones (T) como unidades de muestreo a comparar, la variación se estima entre y dentro de cada grupo (T1, T2, T3, Tn). Explícitamente no hay un número mínimo, pero considerando otros factores además de Normalidad, como el nivel de significancia y el poder de la prueba estadística, la comparación entre promedios o varianzas puede requerir idealmente al menos unas 20 observaciones por cada grupo (Campbell, 1974; Giere, 1979; Byrkit, 1987). Por ejemplo, si el nivel de comparación es entre dos especies (T1 y T2), mínimamente se esperaría contar con una muestra de 20 especímenes de cada una (nT1=20, nT2=20). Este tamaño de muestras permitiría calcular un promedio para T1 y otro para T2, sea la comparación en una o muchas variables. El número de especímenes por cada grupo puede ser desigual, pero el límite crítico es el grupo que tiene menos muestras. En algunos casos, se han logrado los análisis de varianza del CVA aún con el mínimo de 12 especímenes en uno de los grupos (De Luna y Gómez-Velasco, 2008; Ramírez-Sánchez et al., 2016). Cuando sólo hay uno o pocos ejemplares por grupo, lo único que puede hacerse son análisis de componentes principales. Otros análisis requieren todavía más muestras por grupo o nivel (n>30), porque se comparan los patrones de variabilidad (variación de la varianza), por ejemplo, en estudios de asimetría (Medina et al., 2018) y de disparidad (Chollet-Villalpando y De Luna, 2020).
Variables
Las variables (v) son aspectos medidos de los objetos físicos o geométricos. Por ejemplo, el tamaño y la forma de los “pentagonoides”, de las hojas o de mandíbulas. Las observaciones para describir la variación morfológica pueden registrarse en valores discretos o continuos. El dominio examinado aquí es exclusivamente el de los datos continuos de las mediciones lineales y de las coordenadas Cartesianas. La variación de las medidas lineales puede ser analizada en las escalas originales o con la distribución de los valores re-expresados respecto de alguna variable latente (Cuadro 4). Ejemplos de estos ejes latentes pueden ser el de una regresión o una co-regresión. No obstante, para dos variables mutuamente interdependientes, donde no hay variable independiente como en las mediciones lineales, lo recomendable es usar los valores respecto al “eje mayor” o los del “eje mayor reducido” (Smith, 2009). En el caso multivariado, los valores respecto a todos los “componentes principales” (Cuadro 4) pueden ser tomados como datos para los análisis estadísticos (Van Valen, 2005).
Cuadro 4: Valores en la escala de los componentes principales PC1-6 como nuevos ejes estimados a partir de la varianza en las seis mediciones m1-6 de los seis “pentagonoides” A-F.
| pentagonoides | PC 1 | PC 2 | PC 3 | PC 4 | PC 5 | PC6 |
| A | -4.8085 | -1.9195 | -8.2493 | -0.059731 | 0 | 0 |
| B | 11.429 | 16.553 | 0.69409 | 0.28444 | 0 | 0 |
| C | -8.1897 | -7.0227 | 5.4893 | 0.81969 | 0 | 0 |
| D | -4.8085 | -1.9195 | -8.2493 | -0.059731 | 0 | 0 |
| E | -16.184 | 3.5293 | 7.0986 | -0.6479 | 0 | 0 |
| F | 22.562 | -9.2209 | 3.2167 | -0.33677 | 0 | 0 |
Las variables para análisis morfométricos “v” son simples en el sentido de que no se usan variables compuestas, como las proporciones de dos variables “v=x1/x2”. El uso de proporciones, por ejemplo, la división de largo sobre ancho es adecuado para propósitos descriptivos, pero no para los análisis estadísticos y morfométricos (Simpson et al., 1960; Barraclough y Blackith, 1962; Atchley y Anderson, 1978; Phillips, 1983; Strauss y Bond, 1990). Por lo tanto, las proporciones no se deben agregar al conjunto de las variables simples, a no ser que se hagan ajustes y análisis especiales (Douma y Weedon, 2019). Tampoco se deben sustituir dos variables simples x1 y x2 por las proporciones (Jeffers, 1967). Una manera adecuada de mezclar dos variables (largo, ancho) en una variable simple conmensurable es producir algebraicamente la “combinación lineal”, es decir, la suma vectorial de las dos variables “v=x1+x2”. Por ejemplo, los análisis del “eje mayor”, o del “eje mayor reducido” o los “análisis de componentes principales” seleccionan una combinación lineal estimada mediante diferentes criterios. Estas técnicas combinan las dos variables de interés en una nueva variable simple, con propiedades correctas para los análisis estadísticos exploratorios e inferenciales.
Bajo un enfoque filogenético la variación de las mediciones o las coordenadas se analiza para cada carácter taxonómico por separado. Dependiendo de la complejidad de cada carácter, algunos podrán ser descritos numéricamente con una o dos variables (largo y ancho), pero otros deberán ser descritos por un conjunto de varias mediciones entre puntos, o por las coordenadas Cartesianas de varios puntos. Los conjuntos de mediciones lineales para describir un sólo carácter pueden llegar a una decena de variables. Pero, los conjuntos de variables pueden ser muy grandes cuando un carácter se describe con coordenadas 2D; cada punto en un plano define dos variables “x,y”. Aun las formas sencillas pueden requerir unos 20 puntos o más para ser muestreadas, es decir, se generan 40 variables o más. En el estudio de la variación del cráneo de especies de murciélagos del género Myotis Kaup se analizaron filogenéticamente cinco caracteres por separado. La forma sencilla de la sección de la hilera de dientes de la mandíbula se registró con 14 puntos, pero la forma del contorno de la región parieto-occipital del cráneo requirió 32 puntos (Ospina-Garcés y De Luna, 2017). Las formas con contornos más complejos pueden requerir un diseño de muestreo con una mayor densidad de puntos. Por ejemplo, la variación poblacional percibida a priori en el contorno de las hojas de Jatropha curcas L. (Euphorbiaceae) va desde ligera hasta profundamente lobulada (Fig. 4). Para optimizar la superposición y revelar cambios en el contorno se registraron dos marcas y 48 semi-marcas de cada lado, usando dos curvas en tpsDig (Rohlf, 2017). Empíricamente hablando, 50 puntos de cada lado de la hoja son demasiados. Las 50×2×2=200 variables en una hoja generan un problema metodológico identificado en la literatura estadística como “hiperdimensionalidad” (HDLSS, High Dimension, Low Sample Size, Gündüz y Fokoué, 2015). El artículo de Jung y Marron (2009) es una buena introducción general. Una explicación detallada de los problemas potenciales en las aplicaciones morfométricas se puede encontrar en la publicación de Bookstein (2017).

Figura 4: Densidad de puntos para registrar la forma. Se usaron dos contornos para comparar la forma de la hoja en poblaciones de Jatropha curcas L. A. los puntos 1 y 50 son marcas en la base y el ápice de la hoja. Cada contorno se registró con estas dos marcas y otros 48 puntos (semi-marcas) en el lado izquierdo y derecho; B. la superposición Procrustes da una idea de la localización de las diferencias en los lóbulos de las hojas más o menos pronunciados en seis poblaciones indicadas con colores. Fotografía y datos para la gráfica, cortesía de Leonardo Daniel Arévalo Monterrubio.
Los procedimientos empíricos para el caso de demasiadas variables como el de este ejemplo son relativamente simples. Una opción es disminuir el número de puntos en la superposición y alineamiento de las semi-marcas. Por ejemplo, usando SemiLand (Sheets, 2014b), el protocolo de alineamiento de las semi-marcas puede definir varios “helper points” alternados para eliminarlos. Los análisis estadísticos se ejecutan con sólo unos 15 a 25 puntos en cada lado de la hoja. Obviamente después de algunos experimentos uno puede encontrar el límite menor de puntos cuando todavía se registran las diferencias en el contorno con un análisis multivariado de varianza (CVA). Otra opción es reducir el número de variables después de la superposición durante los análisis estadísticos. Los PCA son precisamente la herramienta para reducir dimensiones. Usando la opción “PCA reduction” en CVAGen (Sheets, 2014d), los análisis multivariados de varianza (MANOVA) para la comparación de promedios se ejecutan con los valores de sólo unos pocos componentes. Posiblemente se puedan usar menos de 10 componentes, cuando la varianza acumulada es mayor a 95% de la varianza total. El registro de muchos puntos mejora la superposición y las variables se pueden disminuir después de la superposición y alineamiento de semi-marcas para evitar problemas en la comparación estadística de varianzas entre grupos.
Datos
Todos los métodos morfométricos se basan en la ubicación de puntos de referencia “p” sobre una estructura biológica para registrar información numérica. Los métodos de la “morfometría tradicional” (sensuMarcus, 1990) se basan en datos de medidas lineares “m” entre los puntos “p”. Las coordenadas Cartesianas 2D o 3D de los mismos puntos son la base para los métodos de la “morfometría geométrica” (sensuRohlf y Marcus, 1993). Los dos tipos de puntos de referencia más usados son las marcas (landmarks) y semi-marcas (semi-landmarks). Las marcas son puntos, los cuales localizan posiciones explícitas sobre una morfología. La variación alrededor de una marca, ajustada después de la superposición, informa sobre cambios en la forma. Consideremos los puntos 1-5 en los “pentagonoides” de la Figura 2. Los cinco puntos pueden ser reconocidos en todas las muestras por referencia a su ubicación relativa o similitud topológica. Las semi-marcas son conjuntos de puntos que registran la información matemática de un contorno. No toda la variación alrededor de una semi-marca es informativa. Los métodos de alineamiento de semi-marcas minimizan la variación tangencial a la curva (Perez et al., 2006; Gunz y Mitteroecker, 2013). Sólo la variación ortogonal en cada punto de la curva informa sobre cambios en el contorno.
La densidad de semi-marcas en un contorno puede ser variable dependiendo de la complejidad de la curva. Por ejemplo, pocos puntos describen bien cada uno de los cuatro contornos en la forma del cáliz en varias especies de Salvia (Fig. 3D). En cambio, un contorno complejo tal vez requiera registrarse con muchos puntos, como el caso de los dos contornos en las hojas de Jatropha L. (Fig. 4). La correspondencia de los conjuntos de puntos (marcas y semi-marcas) entre mapas comparables se ha denominado “homología”, pero es puramente geométrica y no debe confundirse con los varios tipos de homología biológica (Donoghue, 1992). En el contexto de la morfometría, “homología” explícitamente significa que la colección de puntos genera un “mapa homólogo” como conjunto de descriptores numéricos comparables matemáticamente con otro conjunto de puntos (Bookstein et al., 1985; Kim y Kim, 2001). En el contexto biológico, los datos son el mapa numérico de comparación que describe un carácter taxonómico.
La unidad estructural o anatómica que cuenta como unidad de comparación (homología biológica) es una decisión a priori dependiente de teoría y conocimiento del dominio empírico del objeto de estudio (Ruse, 1988; Rieppel y Kearney, 2007). La definición y selección de cuál estructura morfológica medir viene antes de cómo medirla. La estrategia inversa, primero colectar muchas mediciones o coordenadas, subdividir el mapa numérico en sectores y luego conceptualizar el carácter (Griffiths, 1999), no es epistemológicamente robusta para conjeturas de homología biológica en general (Donoghue, 1992; Rieppel y Kearney, 2007), ni para hipótesis de homología filogenética en particular (de Pinna, 1991; De Luna y Mishler, 1996; Palci y Lee, 2019). En la aplicación en sistemática, no es recomendable decidir la conceptualización de qué constituye un carácter taxonómico dependiendo de los análisis de correlación entre subconjuntos de puntos.
Operativamente, los módulos o caracteres son la base para comparar entidades en estudios sobre procesos del desarrollo, eventos históricos o procesos ecológicos (Fristrup, 1992; DeSalle, 2006). Si todo un cráneo se considera biológicamente homólogo con otro cráneo, entonces se diseñará un conjunto de puntos para comparar morfométricamente la variación entre los mapas numéricos de los cráneos. Pero si la unidad de comparación es un módulo o un pequeño sector anatómico del cráneo, entonces la configuración de puntos se registrará y analizará con el propósito de medir la variación en ese rasgo morfológico, por separado de los otros rasgos anatómicos del cráneo. Por ejemplo, el cráneo se dividió a priori en cinco caracteres en el estudio filogenético de la variación morfométrica en especies de Myotis (Ospina-Garcés y De Luna, 2017). Con el fin de ahorrar tiempo y otras cuestiones prácticas se registraron las coordenadas de todos los puntos en todo el cráneo. Antes de la superposición, las coordenadas se subdividieron en varias matrices, según el conjunto de los puntos que a priori definen un carácter. Del mismo modo, por conveniencia se registraron las coordenadas de todos los puntos (marcas y semi-marcas) en el cuerpo de los ácaros del género Arrenurus Dugès. Pero se extrajeron matrices de subconjuntos de puntos para las superposiciones separadas y el análisis de la variación en cinco caracteres de formas definidos a priori (Ramírez-Sánchez et al., 2016).
Matrices
Las matrices morfométricas contienen la colección de objetos matemáticos “X” usados como representaciones numéricas de una colección de objetos empíricos. Los mapas numéricos de los objetos empíricos convencionalmente se arreglan en las hileras. Las celdas contienen los valores de los descriptores numéricos de la morfología (n hileras) expresados en función de un conjunto de variables. Los valores observados en cada objeto pueden ser mediciones lineales (Cuadro 1) o las coordenadas de los puntos sobre el objeto (Cuadro 5). Si los valores morfométricos son mediciones (largos y anchos), una hilera (n1) de números es un vector. En este caso, la colección de hileras (n especímenes) es una colección de vectores que geométricamente configura una nube de objetos o puntos (N) en un espacio Euclidiano (Fig. 5). Por otro lado, si los valores son coordenadas Cartesianas de puntos morfológicos (marcas o semi-marcas), cada par contiguo de números es un vector y entonces un espécimen (una hilera) es una colección de vectores (un tensor). En este caso, la matriz con las coordenadas de todos los objetos (n hileras) es una colección de tensores los cuales configuran una nube de puntos (N) en un espacio curvo no Euclidiano (Fig. 6).
Cuadro 5: Coordenadas Cartesianas 2D en el formato tps para los seis “pentagonoides” de la Figura 2. n=objetos, p=puntos, k=dimensiones del objeto, v=variables. LM=punto, IMAGE=archivo digital, ID=etiqueta de objetos matemáticos.
| Matriz de coordenadas Cartesianas 2D para seis pentagonoides, en el formato TPS (n=6, p=5, k=2, v=10) |
| LM=5 74.00000 325.00000 241.00000 447.00000 411.00000 326.00000 347.00000 129.00000 135.00000 131.00000 IMAGE=pentagonoideA.jpg ID=0 LM=5 97.00000 328.00000 435.00000 452.00000 771.00000 329.00000 638.00000 132.00000 223.00000 132.00000 IMAGE=pentagonoideB.jpg ID=1 LM=5 318.00000 343.00000 614.00000 465.00000 657.00000 343.00000 395.00000 146.00000 184.00000 148.00000 IMAGE=pentagonoideC.jpg ID=2 LM=5 396.00000 279.00000 273.00000 110.00000 75.00000 175.00000 76.00000 381.00000 275.00000 444.00000 IMAGE=pentagonoideD.jpg ID=3 LM=5 82.00000 352.00000 246.00000 636.00000 419.00000 686.00000 356.00000 423.00000 145.00000 220.00000 IMAGE=pentagonoideE.jpg ID=4 LM=5 120.00000 564.00000 289.00000 809.00000 456.00000 563.00000 391.00000 167.00000 183.00000 173.00000 IMAGE=pentagonoideF.jpg ID=5 |
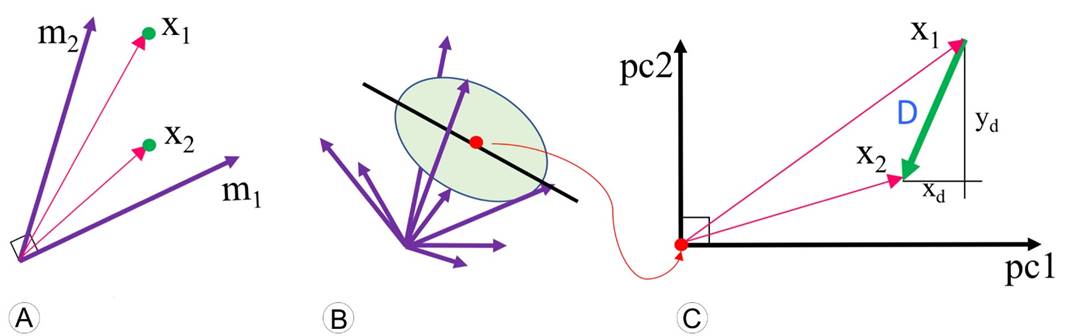
Figura 5: Distancias Euclidianas. La matriz de mediciones lineales es una colección de vectores que geométricamente configura una nube de objetos o puntos (n) en un espacio Euclidiano. A. dos objetos empíricos se representan numéricamente por dos objetos matemáticos “x1” y “x2” con valores en dos variables “m1” y “m2”. Las dos variables están correlacionadas, por lo tanto, su representación vectorial es con los dos ejes oblicuos; B. espacio multidimensional configurado por todas las mediciones (mn) y todos los objetos (xn). Los ejes son oblicuos debido a que están correlacionados; C. dos ejes ortogonales (pc1, pc2) centrados en el promedio de la nube de datos. Ahora es posible usar los ejes ortogonales como catetos para calcular la distancia D entre los dos objetos “x1” y “x2” como una hipotenusa.
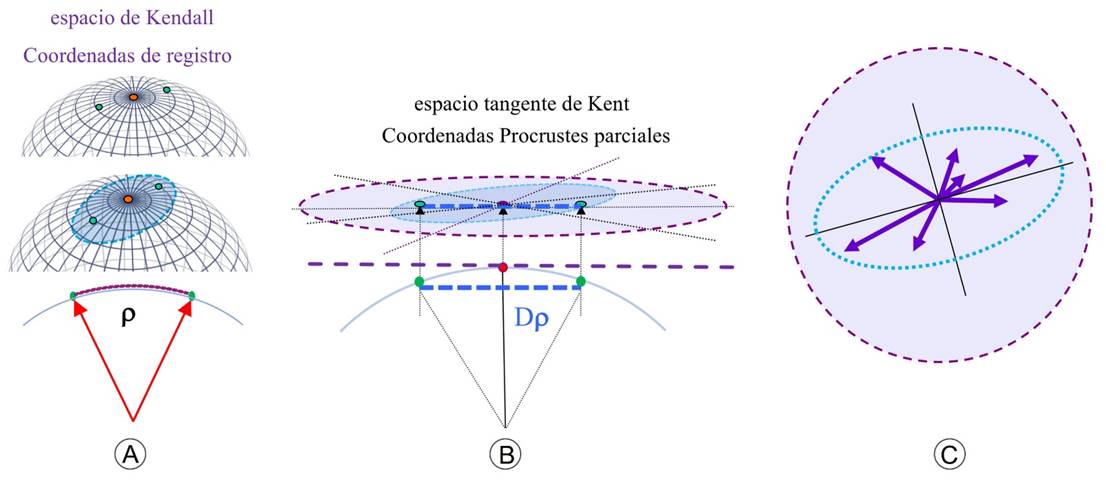
Figura 6: Distancias Procrustes. La matriz de coordenadas Cartesianas es una colección de formas que configura una nube de puntos (n) en un espacio curvo no Euclidiano. A. las coordenadas de registro de un objeto empírico se representan matemáticamente como un punto en la superficie curva del espacio de Kendall. La colección de objetos configura una nube de puntos sobre un sector de la hiperesfera. En este espacio curvo, la geodésica es la distancia Procrustes (() entre dos puntos; B. proyección de los objetos desde el espacio curvo hacia un espacio tangente en el punto promedio de todas las formas. En el espacio tangente de Kent, las distancias Procrustes parciales (D() se calculan como el equivalente de la cuerda entre pares de puntos; C. Los “partial warps” se calculan como nuevas variables de deformación de cada figura en relación con la forma promedio.
Las variables convencionalmente se arreglan en las columnas de la matriz de datos. Las variables por definición son los “ejes base” de un espacio matemático (Healy, 2000). Por ejemplo, cada longitud y ancho medido es una variable o eje (Cuadro 1). En este caso, el cambio de orden de las columnas no cambia las características cuantitativas de la matriz ni las propiedades geométricas de la nube de datos en el espacio multidimensional. Pero el orden de las variables es muy informativo cuando cada espécimen (n) es descrito por una colección de pares (x, y) o tríos (x, y, z) de coordenadas Cartesianas de cada punto. El orden de los pares o tríos de números registra las relaciones geométricas entre puntos y definen los ejes base de un espacio geométrico y su dimensión. En el formato TPS (Thin Plate Spline) (Rohlf, 2015) estos pares o tríos de números se escriben en varias hileras sucesivas, una hilera de dos o tres números por punto registrado. Un bloque con las coordenadas de un objeto empírico termina con un código de identificación ID=x para cada objeto matemático (Cuadro 5).
Espacios geométricos
Una matriz de datos morfométricos define un espacio geométrico vectorial (Healy, 2000). Los conceptos de vectores y geometría de espacios útiles en la teoría estadística pueden ser estudiados en los tratamientos detallados de análisis vectorial (Hausner, 1965; Banchoff y Wermer, 1991; Wickens, 1995; Lattin et al., 2003). El concepto geométrico básico en morfometría es la visión de un espécimen u objeto “n” como un vector o punto geométrico con cierta ubicación, dirección y magnitud en un espacio matemático (Mitteroecker y Huttegger, 2009). En ese espacio multidimensional, la ubicación de un objeto representado por un punto x depende de una colección de valores definidos por los “ejes base” de las mediciones lineales o de las coordenadas Cartesianas. En el contexto biológico, un objeto empírico puede ser una hoja, una mandíbula, un cráneo, la aleta de un pez, etc. Un objeto biológico se representa como un vector x cuando se registran largos y anchos (mediciones “m”, Fig. 5). La representación matemática resulta de concatenar cada medición lineal (m) y la representación vectorial sería la siguiente colección de valores: x = (m 1 , m 2 , m 3 , …, m n ). Cuando se registran las coordenadas Cartesianas de varios puntos un objeto biológico se representa como un tensor x (Fig. 6). La representación matemática resulta de concatenar cada par de coordenadas “x y” de cada punto P, si son coordenadas Cartesianas en un plano (k=2). La representación vectorial para un objeto sería la siguiente colección de coordenadas: x = (x 1 y 1 , x 2 y 2 , x 3 y 3 , …, x n y n ) T. La ubicación de cada objeto en el espacio matemático de comparación depende de todos los valores para cada variable. Por eso se deben registrar todas las mediciones (largos y anchos) o todos los valores de las coordenadas de los puntos en cada objeto o espécimen (n). Si los valores son incompletos para alguna variable, el espécimen se elimina de la matriz. Los datos faltantes todavía son un problema en los análisis estadísticos y morfométricos (Strauss y Atanassov, 2006).
La matriz de datos de las mediciones entre marcas define la geometría de un espacio Euclidiano (Fig. 5) con tantas variables como el número de mediciones “m” registradas. El conjunto de ejes base “m” se intersectan en el origen y divergen con relaciones angulares diferentes dependiendo del grado de dependencia lineal. Por lo tanto, los ejes base “m” característicamente son oblicuos (Hausner, 1965; Wickens, 1995; Healy, 2000), es decir, los ángulos entre los ejes no son a 90˚. La matriz de correlaciones provee un diagnóstico de las relaciones angulares entre todos los pares de ejes base (Wickens, 1995; Kendall, 2004). El ángulo entre dos ejes indica el grado en el que las dos variables varían juntas. Los valores de la correlación cercanos a 1 corresponden a un ángulo muy pequeño entre un par de ejes, lo cual sugiere dependencia lineal. Los valores cercanos a 0 indican un ángulo cercano a 90˚, lo cual revela independencia. Los ejes son ortogonales cuando el ángulo entre ellos es exactamente 90˚ (Hausner, 1965; Wickens, 1995; Healy, 2000). Además, los ejes no son de extensión homogénea por el efecto de distintas escalas de medición. La transformación a valores de logaritmos puede remediar estas diferencias de escalas. La dispersión de la nube de objetos tampoco es homogénea en la dirección de cada variable, es decir, la nube de datos no configura una esfera, sino un elipsoide multidimensional. La heterogeneidad de las varianzas en la matriz de varianza-covarianza es un indicador de esta dispersión. Dadas estas características geométricas, el espacio de registro con ejes base “m” oblicuos debe ser ajustado para encontrar nuevos ejes base ortogonales para la estimación de distancias entre objetos como la hipotenusa de un triángulo rectángulo. Por ejemplo, los análisis de componentes principales seleccionan un nuevo sistema de ejes base ortogonales (Fig. 5).
La matriz de coordenadas de la forma en varios objetos (n) define la geometría de un espacio curvo no Euclidiano (Fig. 6A), el cual se denomina espacio de Kendall (Kendall, 1984, 1989). Este espacio geométrico es un manifold Riemanniano (Mitteroecker y Huttegger, 2009). Un manifold es un espacio curvo no Euclidiano, el cual localmente puede ser mapeado con mosaicos Euclidianos, por ejemplo, un pequeño plano cuadrado tangente. La superficie de una esfera es el manifold más sencillo; también la superficie de una dona es un manifold, la cual geométricamente es un “toro” (Ramírez-Galarza y Seade-Kuri, 2002; Martínez-Naveira, 2007). El formalismo matemático de la morfometría geométrica se ha desarrollado gradualmente desde hace unos cuarenta años, especialmente a partir de las contribuciones teóricas y aplicaciones metodológicas simultáneas de Kendall (1977, 1981, 1984) y Bookstein (1978, 1982, 1984). La teoría y herramientas matemáticas consisten en teoremas sobre la geometría del manifold Riemanniano de la forma (Small, 1996; Kendall et al., 1999). Las características geométricas del espacio de Kendall pueden imaginarse a partir del caso más sencillo de la colección de coordenadas Cartesianas 2D “x, y” (k=2) de tres marcas (p=3). Si varios objetos empíricos (n) en dos dimensiones (k=2) son registrados con tres puntos (p=3), el espacio de Kendall es un sector de una esfera (Fig. 6A). Un objeto (n=1) registrado por una constelación de coordenadas, matemáticamente se representa como un punto en la superficie curva del espacio de Kendall (Bookstein, 1996a).
Cuando los objetos planos (k=2) se registran con configuraciones de más de tres puntos, como es lo usual, el espacio de Kendall se vuelve más complejo. El registro de objetos 3D con coordenadas “x, y, z” (k=3) genera espacios matemáticos “substancialmente más complicados” (Goodall, 1991; Rohlf, 1996). En el caso de muchos objetos (n) registrados por coordenadas 2D de varios “p” puntos (marcas o semi-marcas), el espacio de Kendall es una superficie curva de la clase de objetos geométricos denominados “toros”. Se usa la notación “n-toro” para definir la dimensión del espacio matemático. Por ejemplo, la superficie matemática de una dona es un “3-toro” simple generado por la rotación de un círculo en un espacio tridimensional. El espacio de Kendall es un “(p-2)-toro” construido por el producto Cartesiano de esferas en un espacio hiperdimensional. Las propiedades matemáticas de este espacio son “sorprendentemente complejas” (Rohlf, 1996). Desde la perspectiva práctica, la descripción anterior brinda el contexto para visualizar la nube de “n” objetos registrados con configuraciones de marcas como un elipsoide de “n” puntos sobre la superficie curva de un sector en una hiperesfera (Fig. 6A). También nos permite comprender la particularidad de los análisis estadísticos especiales requeridos para los datos de coordenadas en la morfometría geométrica (Bookstein, 1996b).
Distancias morfométricas
Los objetos biológicos o muestras son “n” puntos posicionados en espacios definidos por las mediciones lineales o por las coordenadas. Por lo tanto, la diferencia entre un par de objetos x1 y x2 siempre es una distancia morfométrica entre dos puntos (D=x1-x2). Se han propuesto varias medidas de distancias, por ejemplo, la distancia Euclidiana o la distancia de Manhattan y seleccionar cual es la mejor o correcta lleva cierto grado de arbitrariedad o conveniencia. En esta revisión, sólo se considera la distancia Euclidiana, pues es la más comúnmente usada en los análisis estadísticos multivariados (Manly, 1986). Si cada objeto morfométrico lo representamos con una flecha, la comparación de dos objetos equivale a la medida del desplazamiento de las puntas de las flechas x1 y x2. Esto define la distancia Euclidiana D=x1-x2 como un “vector del desplazamiento” con cierta magnitud y dirección, el cual a su vez puede visualizarse como otra flecha. La punta de esta flecha estaría colocada en el punto x2, lo cual informaría el sentido de la dirección del movimiento entre x1 y x2 (Fig. 5C). La comparación entre dos objetos morfométricos se cuantifica con un vector de desplazamiento. Esta distancia Euclidiana es la métrica de comparación entre objetos cuando las variables morfométricas son mediciones entre puntos (Manly, 1986). El ajuste del espacio geométrico de ejes oblicuos “m” consiste en centralizar, normalizar y seleccionar ejes latentes ortogonales para construir un hipercubo multidimensional (Coxeter, 1973). En este espacio ortogonal se procede al cálculo de la distancia D entre un par de objetos como la hipotenusa de algún triángulo rectángulo implícito. Un espacio ortogonal muy útil es el definido por los componentes principales (PCA). Los nuevos ejes ortogonales funcionan como catetos en ese triángulo para la estimación de la diferencia mediante el teorema de Pitágoras (Fig. 5C). El Cuadro 6 muestra los valores de las distancias Euclidianas entre seis “pentagonoides” cuando los datos son mediciones lineales. Estas distancias pareadas entre los seis objetos se estimaron con el programa InfoStat (Di Rienzo et al., 2017).
Cuadro 6: Distancias morfométricas entre los seis “pentagonoides” A-F de la Figura 2. Las distancias Euclidianas (abajo de la diagonal) fueron calculadas a partir de los valores de seis mediciones m1-6. Las distancias Procrustes (arriba de la diagonal) se calcularon con CoordGen a partir de las coordenadas superpuestas de cinco puntos. Los valores en negritas destacan el mínimo y el máximo.
| a | b | c | d | e | f | |
| a | 0.00 | 0. 3133 | 0.4220 | 0.0081 | 0.4063 | 0.3279 |
| b | 26.17 | 0.00 | 0.6010 | 0.3154 | 0.3560 | 0.6328 |
| c | 15.07 | 31.05 | 0.00 | 0.4219 | 0.3396 | 0.3786 |
| d | 0.00 | 26.17 | 15.07 | 0.00 | 0.4084 | 0.3256 |
| e | 19.87 | 31.21 | 13.42 | 19.87 | 0.00 | 0.6057 |
| f | 30.56 | 28.20 | 30.94 | 30.56 | 40.98 | 0.00 |
Cuando los datos son las coordenadas Cartesianas y cuando se superponen los mapas de puntos con métodos de Procustes, la diferencia entre dos objetos es una geodésica (Kendall, 1984) y se mide con las distancias Procrustes “(“ (Fig. 6A). Esta distancia curva entre puntos en el espacio de Kendall es la manera convencional de cuantificar la diferencia entre formas en los análisis de morfometría geométrica. Dos objetos con forma idéntica ocuparán el mismo punto en este espacio, después de la superposición habiendo eliminado las diferencias debido a localización, rotación y tamaño de los objetos. Los valores de las coordenadas superpuestas son la base para calcular las distancias Procrustes. El Cuadro 6 presenta los valores de las distancias Procrustes entre los seis “pentagonoides” de la Figura 2. Aun teniendo la misma forma, la distancia Procrustes entre los “pentagonoides a” y “d” es en el orden de 0.0081, lo cual podemos interpretar como el error de registro. En el Cuadro 6 la máxima distancia es entre los “pentagonoides b” y “f”, lo cual es congruente con la percepción intuitiva de la máxima diferencia entre las figuras.
Geométricamente, la cuerda es la línea recta entre los dos puntos de una geodésica. La línea tangente en el punto promedio entre los dos puntos es paralela a la cuerda y puede ser vista como una proyección de la cuerda (Fig. 6B). Entonces la distancia curva entre los dos puntos de la geodésica también se estima usando la distancia lineal de la proyección tangencial de la cuerda. Las distancias Procrustes parciales “D(“ son una aproximación lineal de la geodésica del espacio curvo de Kendall (Fig. 6B). Generalizando para todas las formas, las diferencias pareadas se estiman con las distancias Procrustes parciales “D(“ en un espacio multidimensional tangente a la hiperesfera (espacio de Kent) en el punto que corresponde al promedio de todas las formas (Kent y Mardia, 2001; Kume et al, 2007). Los métodos de análisis de la forma proyectan toda la nube de puntos a un espacio tangente definido por nuevos ejes. Las coordenadas de registro son re-expresadas como coordenadas Procrustes parciales (Fig. 6B).
Variables de la deformación relativa
La varianza remanente en las coordenadas ajustadas por superposición se descompone (“eigenanalisis”) para seleccionar variables latentes ortogonales (“warps”). Estos nuevos ejes de deformación son los parámetros de un modelo que describe la ubicación, la magnitud y dirección de los desplazamientos relativos en cada punto de cada configuración comparada con la forma promedio (Goodall, 1991; Rohlf, 1999). Cada eje es un vector univariado y la varianza de la deformación es independiente de los otros vectores. En conjunto, estas son las variables convencionalmente usadas para los análisis estadísticos de la variación de la forma. Las variables de la deformación punto a punto se pueden separar en dos tipos: las que describen transformación uniforme (“affine component”) y las que describen transformación local o no uniforme (“non-affine component”). Los “ partial warps ” son ejes o componentes de la deformación relativa no uniforme (Rohlf, 1996). El espacio de estas variables de la ubicación, dimensión y dirección de la deformación define un hipercubo multidimensional Euclidiano (Fig. 1). Los valores en estas variables basadas en deformación geométrica (“partial warp scores”) pueden ser usados como datos para analizar la varianza con métodos de ordenamiento para la distribución de los objetos en una variedad de espacios latentes (PCA, CVA). No obstante, debe considerarse que tales variables de deformación relativa (“partial warps”) representan ejes abstractos, no biológicos y no estables, pues siempre son dependientes de una colección particular de muestras, de un método de superposición y de una forma usada como referencia para calcular la varianza. Las diferentes combinaciones de muestras, tipos de superposiciones y selección de referencias resultan en diferentes matrices de varianza-covarianza en los “partial warp scores” (Richtsmeier et al., 2005; Webster y Sheets, 2010). A su vez, los componentes principales también pueden ser inestables, pues se calculan a partir de la covarianza ordenada de mayor a menor en los “partial warps” (Bookstein, 1992; Rohlf, 1996, 2015; Zeldithch et al., 2012).
Visualización de la deformación relativa
La diferencia en forma entre dos objetos se visualiza con los valores diferentes del desplazamiento relativo entre configuraciones en cada punto. La deformación puede ser visualizada con un mapa que muestra los vectores de desplazamiento en cada punto mediante cuatro tipos de funciones de interpolación (Márquez et al, 2012): interpolación espacial penalizada (interpolating splines), interpolaciones con errores suaves (smoothing splines), interpolación espacial Gaussiana (kriging) y elementos finitos rígidos (finite elements). Una buena descripción y comparación de las propiedades de estos métodos alternativos de interpolación es el artículo de Márquez et al. (2012). Entre las alternativas del primer tipo de modelos espaciales penalizados, Bookstein (1992) adoptó las funciones de interpolación de placa delgada ( Thin Plate Spline, TPS). Según este modelo, las deformaciones que tienen un alcance a más puntos en un mapa tienden a verse favorecidas debido a su menor contribución a la energía de flexión total (bending energy) que las deformaciones entre puntos cercanos (Márquez et al., 2012). El modelo de interpolación espacial para el mapa de deformación TPS no es parte necesaria en los cálculos estadísticos ni para la cuantificación de la variación de la forma, pues tales estimaciones se basan en la distancia Procrustes y los valores de los “partial warps”.
En esta revisión sólo se examina la función TPS pues es el modelo más ampliamente usado, pero debe enfatizarse que no es la única solución al problema de cómo visualizar la deformación relativa. Así como se aplica el modelo de regresión para dibujar la línea continua con el mejor ajuste de los datos bivariados, del mismo modo se aplica la función TPS para visualizar el plano de deformación que mejor ajusta las diferencias multivariadas entre todas las formas (Rohlf y Marcus, 1993). Las deformaciones relativas en el plano de la función TPS se dibujan comparando cada objeto respecto a una configuración de referencia, usualmente el promedio (Fig. 6C). La función TPS es un modelo de interpolación conveniente que permite estimar los parámetros del desplazamiento gradual entre punto y punto de la configuración de la forma (Bookstein, 1989, 1992). Como ejemplo, consideremos la variación en la forma de las hojas (n=691) en una colección de diez especies de arbustos del género Hoffmania Sw. (Rubiaceae, datos cortesía de G. Castillo-Campos). Se registraron las coordenadas de dos contornos con 25 puntos de cada lado usando MakeFan (Sheets, 2005) y tpsDIG (Rohlf, 2017). Las coordenadas se ajustaron con una superposición Procrustes en CoordGen y se alinearon con Semiland (Fig. 7A). El análisis de varianza con un CVA muestra que al menos cuatro especies son diferentes en la forma de las hojas, dos en cada extremo de los dos primeros ejes discriminantes (Fig. 7B). En lugar de dibujar el mapa TPS de la deformación global entre todas las especies explicada por el primer eje, lo recomendable es graficar la gradilla de diferencias locales entre dos formas usando CVAGen, pues la pregunta taxonómica de interés era comparar esas dos especies en particular (Fig. 7C).
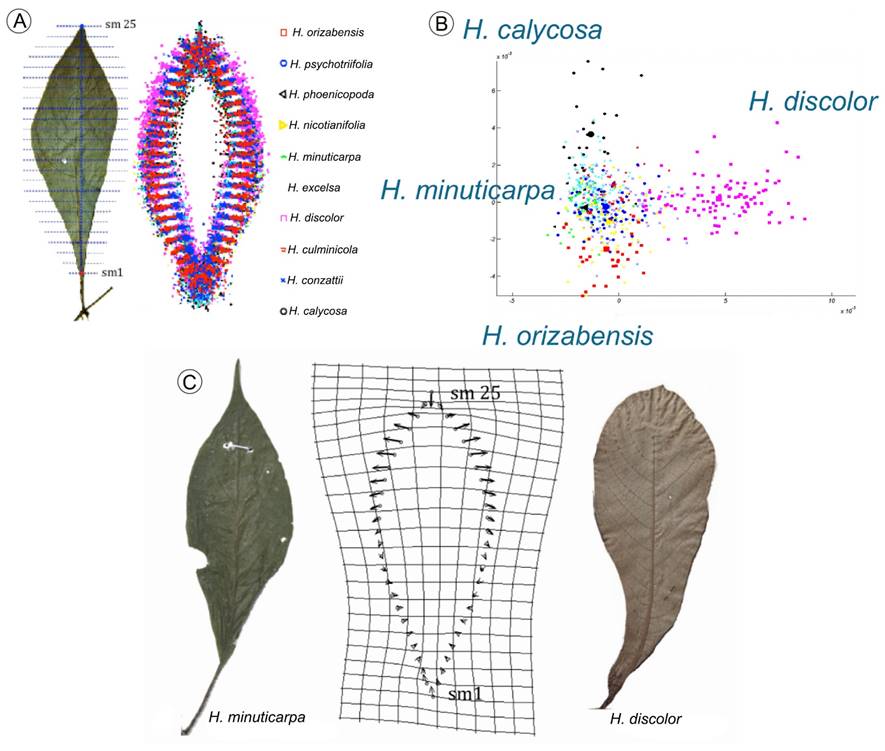
Figura 7: Interpolación de la deformación. A. los dos contornos de la hoja se registraron colocando 25 puntos con un peine usando MakeFan y tpsDIG. Se ilustra la superposición y alineamiento de las muestras de diez especies del género Hoffmania Sw. (Rubiaceae); B. variación entre especies en la forma de la hoja en el plano de los dos primeros ejes discriminantes calculados con CVAGen; C. comparación de la forma entre dos especies visualizada con la gradilla de deformación relativa de la función TPS, estimada en el morfoespacio del CVA. Las diferencias se ubican en el acumen y en el ángulo de la base de la hoja. La dimensión de los vectores de deformación y la amplitud de las cuadriculas de deformación es mayor en el acumen que en la base. Fotografías y datos, cortesía de G. Castillo-Campos.
Este ejemplo ilustra que las cuadrículas de deformación en el plano de la función TPS son un mapa visual de las correspondencias bidireccionales entre las coordenadas (x1, y1, x2, y2, ….., xn, yn) de los “p” puntos en un plano “A” y otro mapa en otro plano “B” con el mismo número de puntos y con un sistema comparable de coordenadas (u, v). Si los dos planos A y B son diferentes (D = A-B), la función TPS es el modelo de un plano D que estima la dimensión de los vectores de desplazamiento punto a punto D = ((x1, y1) - (u1, v1), …., (xn, yn ) - ( un, vn)). La expansión o contracción de cuadriculas y la dirección de los vectores de deformación permiten inferir cambios de forma en zonas particulares del mapa de la configuración de puntos. No obstante, cuando los cambios son muy complejos, ningún análisis de coordenadas de puntos revela las deformaciones en las zonas vacías entre los puntos. Este es un problema de baja densidad de datos, ya sea por causa de muy pocos puntos, o demasiado separados para describir los cambios en forma (Sheets et al., 2002).
Morfoespacios
Existe un conjunto de muchos ejes latentes en un espacio ajustado, tanto en el espacio geométrico configurado por ejes centrados y normalizados derivados de las mediciones lineales, como en el espacio definido por los “partial warps” calculados desde los datos de las coordenadas superpuestas. En ambos espacios geométricos, el centroide de la nube de datos (promedio global) ubica el origen de todos los ejes (Fig. 1). Si la dispersión de la nube de datos fuera homogénea en todas las direcciones desde el centro, la nube configuraría una esfera multidimensional y todos los ejes latentes en todas las direcciones posibles tendrían la misma extensión. Un hipercubo en cualquier orientación serviría para los análisis estadísticos. Cualquier par ortogonal de ejes latentes en cualquier dirección serviría como catetos para calcular la distancia entre dos objetos (n=1, n=2) como una hipotenusa (Fig. 5C). En un elipsoide multidimensional, los distintos análisis estadísticos escogen ciertos ejes para la comparación de la variación entre objetos o grupos de objetos, sea que el morfoespacio ortogonal es el de las mediciones o el morfoespacio definido por “partial warps”. La lógica geométrica de esta selección de ejes en los dos espacios multidimensionales es semejante y el resultado es un hipercubo particular en cierta orientación (Fig. 1). Por esa misma geometría, los análisis reciben el mismo nombre, PCA, CVA o Regresión, sea que se usen para mediciones lineales o que se usan para las coordenadas. Sin embargo, como el álgebra no es idéntica, en lugar de PCA, Rohlf (1996) prefiere llamar “Relative Warp Analysis” al ordenamiento de la varianza generalizada. Los “relative warps” son los “partial warps” rotados rígidamente en la dirección de la máxima deformación generalizada.
Métodos estadísticos para el análisis de la variación morfológica
La parte analítica central en la morfometría la constituyen los métodos estadísticos de la variación del tamaño, la forma y también su co-variación. Un obstáculo para el uso de descriptores cuantitativos de variación en la sistemática podría ser la complejidad relativa de las matemáticas y las estadísticas involucradas. No es el propósito profundizar en las matemáticas de las técnicas, pues éstas se desarrollan en textos especializados (Kachigan, 1991; Wickens, 1995; Carroll y Green, 1997). Más bien, se sugiere el uso de los diferentes análisis estadísticos disponibles dependiendo del tipo de comparación de la variación de la morfología entre grupos de especímenes para tres propósitos básicos en la investigación sistemática: identificar especies, evaluar la utilidad de los caracteres taxonómicos y usarlos para inferir filogenias y grupos monofiléticos. En los apartados que siguen se proporcionan las bases conceptuales de los modelos para análisis estadísticos en una estrategia práctica basada en el uso de mediciones lineales y las coordenadas Cartesianas de puntos de referencia. La ruta de procedimientos en la exploración y análisis de la variación de un carácter multivariado se ilustra en el esquema de la Fig. 8. El uso de mediciones lineales y de las coordenadas no han de verse como alternativas en competencia, sino complementarias epistemológicamente. Las dos herramientas deberían ser usadas en todos los estudios morfométricos ya que ambas herramientas tienen limitaciones. Uno debe recordar que la varianza en las coordenadas es dependiente de un sistema de registro, un modelo de superposición y la referencia. La varianza en las mediciones es una combinación de la forma y del tamaño, aunque sin depender del método de registro. Esa diferencia en la capacidad de medir distintos aspectos de la variación morfológica permite usar ambas herramientas bajo una epistemología de análisis confirmatorios (Rodgers, 2010). Los análisis morfométricos confirmatorios pueden robustecer o modificar las conclusiones derivadas usando sólo una herramienta (O´Higgins, 2010).
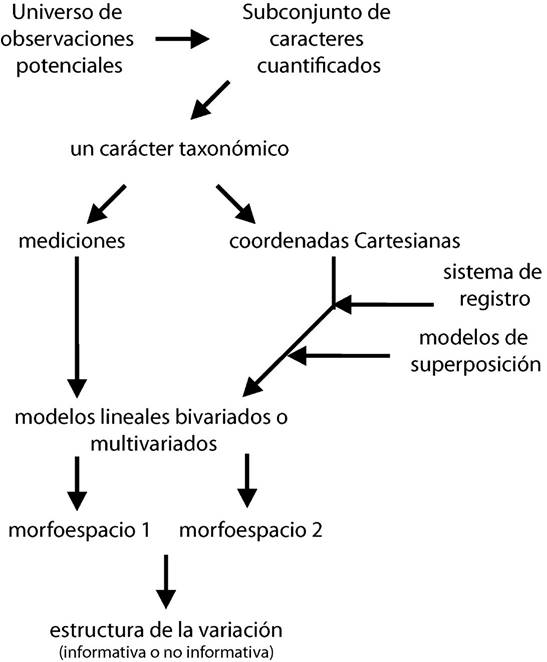
Figura 8: Ruta de operaciones para la exploración y el análisis de la variación de un carácter taxonómico cuantitativo. Por un lado, el conjunto de las mediciones que describen un carácter se analiza mediante la aplicación de algún modelo multivariado como el CVA. Por otro lado, la estructura de la variación en la forma registrada con las coordenadas de un conjunto de puntos no sólo depende de la aplicación de algún modelo multivariado, sino también del uso de algún modelo de superposición.
Especificación de Modelos
Los análisis estadísticos permiten comparar las formas biológicas y calcular distancias morfométricas en algún espacio matemático (Fig. 9). La detección cuantitativa de la diferencia morfológica se basa en el despliegue de distancias Euclidianas entre muestras en un espacio de vectores ortogonalizado (bivariado o multivariado). El algoritmo central en el cálculo de las distancias entre objetos es el teorema de Pitágoras, con la suma polinomial de un número finito de catetos, tantos como variables definan el espacio matemático. La ejecución de un análisis estadístico es de hecho la “especificación” (en el sentido estadístico, Spanos, 2006; Dennis et al., 2019) de algún modelo lineal al conjunto de datos en cuestión, sean mediciones o coordenadas. Tal aplicación consiste en la estimación de parámetros de tendencia central y de la dispersión (promedios y varianzas y los errores de estimación). Estas estimaciones se calculan mediante la suma de mínimos cuadrados en una dirección implícita en el modelo seleccionado. Distintos modelos, PCA o CVA, estiman esas distancias en direcciones diferentes (Fig. 9). Las inferencias estadísticas derivadas de un análisis pueden consistir en la resolución de que los promedios de muestras son “significativamente” diferentes entre sí. En el caso de los análisis multivariados de varianza (MANOVA), una hipótesis estadística puede ser la de la existencia de diferencia “significativa” entre dos o más grupos de datos (nA, nB, nC, etc.).

Figura 9: Especificación de modelos estadísticos. Cada modelo selecciona un eje latente en una dirección diferente, entre muchos posibles en un elipsoide multidimensional. Para comparación se incluye el modelo de regresión, el cual calcula la función lineal que minimiza la suma de cuadrados en la dirección de la varianza de la variable “y”. Los ejes en el PCA, en cambio, se seleccionan como combinaciones lineales que minimizan la suma de cuadrados en la dirección de cada punto respecto al eje. En el caso del CVA, los ejes van en la dirección en la que se maximiza la proporción F y la distancia proyectada entre los promedios de cada grupo es máxima.
Normalmente la dispersión de la nube de datos no es homogénea en todas las direcciones desde el centroide, por lo que la nube configura un elipsoide multidimensional (Fig. 9). Los distintos análisis estadísticos (PCA, CVA) emplean diferentes criterios al calcular la suma de mínimos cuadrados para seleccionar heurísticamente una dirección óptima de los ejes latentes ortogonales. La distinción entre los métodos multivariados en sistemática se facilita cuando uno decide el procedimiento apropiado con la clave presentada por Atchley y Bryant (1975). La primera opción en la clave pregunta si los objetos están o no agrupados. Los Análisis de Componentes Principales (PCA) seleccionan el primer eje PC1 en la dirección de la máxima dispersión, sin considerar la estructura de grupos. La “varianza generalizada” (sensuVan Valen, 2005) es calculada entre todos los especímenes respecto de un sólo promedio global. En cambio, los análisis de variables canónicas (CVA) seleccionan el primer eje en la dirección de la máxima diferencia entre al menos tres promedios de tres grupos definidos a priori. Los componentes y las variables canónicas discriminantes son seleccionados en orientaciones diferentes respecto del elipsoide multidimensional. Los dos tipos de ejes pasan por el centroide de la misma nube de datos, pero su dirección y extensión puede ser diferente, dada la distribución espacial de los especímenes y la ubicación de los promedios de los grupos (Fig. 9).
Uso de los Análisis de Componentes Principales (PCA)
Los análisis de ordenamiento de la varianza generalizada (PCA) se usan para reducir dimensiones y explorar la estructura total de la nube de datos. La nube puede imaginarse como un elipsoide en “v” dimensiones. El objetivo del análisis es tomar las “v” variables X1, X2, X3, …, Xv y encontrar combinaciones lineares de estas que producen ejes PC1, PC2, PC3, …, PCv, los cuales no están correlacionados entre sí (Manly, 1986; Wickens, 1995; Lattin et al., 2003). Estos ejes están ordenados según la cantidad de varianza en cada dimensión. El eje mayor del elipsoide es el componente uno (PC1), dirigido en el sentido de la máxima varianza generalizada. La selección sucesiva de ejes PCi es en función de la suma de cuadrados de las distancias pareadas entre cada punto y la línea que pasa por el promedio. Cada componente PCi es una combinación lineal ortogonal (rotación rígida) transformada a partir de la matriz de covarianza (o correlaciones). Por lo tanto, existen tantos componentes como variables de registro. Pero si la varianza es muy heterogénea, entonces será posible re-expresar la mayoría de la dispersión en los datos en unos pocos componentes. Para disminuir dimensiones en los análisis estadísticos subsecuentes, por ejemplo, CVA, eliminar los últimos componentes siempre será la alternativa apropiada en vez de eliminar algunas de las variables originales (Wickens, 1995; Lattin et al., 2003).
La gráfica de los dos primeros componentes (PC1 vs. PC2) es muy útil pues visualiza una “sombra” en 2D del elipsoide multidimensional dado por todas las variables. Por lo tanto, se simplifica interpretar las posiciones relativas de todos los objetos en dos o muy pocas dimensiones. Pero para la inspección visual de la estructura de los datos se debe recordar que las distancias entre puntos observadas en la gráfica 2D no son lo mismo que las distancias en el espacio multidimensional. Las diferencias entre puntos se pueden visualizar con un fenograma de similitud (p. ej., UPGMA) calculado con las distancias Euclidianas si los datos son las mediciones o con las distancias Procrustes para las coordenadas de las formas. Otra advertencia al interpretar la disposición de los puntos en la gráfica 2D es que los cuadrantes positivos y negativos son un artificio de la rotación particular empleada para el despliegue de la nube de datos. Análisis repetidos con el mismo software de la misma matriz pueden intercambiar el signo (+, -) de los valores y producir una diferente ubicación de los cuadrantes en los mismos ejes. También debe cuidarse que los dos o tres ejes en la gráfica estén dibujados con la misma extensión y escala (Jolliffe, 2005; Rohlf, 2015). La interpretación cuantitativa depende de los porcentajes de varianza sumados por cada componente. Además, en morfometría geométrica, cada componente principal permite visualizar las ubicaciones del cambio parcial de la forma entre todos los objetos usualmente en un mapa de deformación del modelo TPS. En cambio, en el CVA, cada plano de deformación relativa de la función TPS para cada eje discriminante permite ver la diferencia en forma entre grupos de objetos. Esta estimación y visualización de la ubicación y magnitud de la deformación en el espacio del CVA es de particular interés para el taxónomo (Fig. 7).
Dado que los detalles del cálculo para la transformación ortogonal de las variables en los componentes principales no involucran la varianza de subconjuntos de datos, este método de ordenación no agrupa ni califica grupos a priori (Bartlett, 1965; Manly, 1986). No obstante, una práctica común, pero errónea, es usar el PCA para inferir la existencia de grupos taxonómicos. Por ejemplo, De Souza et al. (2010) ejecutaron un Análisis de Coordenadas Principales (PCO, un equivalente del PCA) para analizar las mediciones de 38 caracteres vegetativos, de frutos y de flores. Interpretaron los resultados de este análisis de ordenamiento expresando: “PCO applied to this data set does not recover groups, with axes 1 and 2 explaining only 15% of the variation”. Además, concluyeron: “Analysis failed to identify a single morphological character or combination of characters that allows the separation of A. molaris and A. leiocarpa or the recognition of any well-supported species within Apuleia”. Este es simplemente un ejemplo de muchos donde se usa el PCA para justificar la separación o no de los grupos. Aun cuando los grupos se vean separados o no, los métodos de ordenamiento, como el PCA o el PCO, no pueden justificar tales interpretaciones. Si los grupos no existen a priori, la alternativa es usar muchos caracteres, estimar distancias y usar algoritmos de agrupación fenética como el UPGMA o aplicar los métodos de agrupación filogenéticos con parsimonia, verosimilitud o probabilidades Bayesianas. Pero si los grupos ya existen a priori, entonces la mejor alternativa son los análisis de varianza y el CVA para las inferencias de distinción entre los grupos.
Uso de los Análisis de Variables Canónicas (CVA)
Los análisis de Variables Canónicas de la varianza particionada por grupos (CVA) se usan para explorar la separación de grupos definidos a priori . Cuando son dos grupos los análisis se conocen como “análisis discriminantes” y son “funciones discriminantes múltiples” o “análisis de variables canónicas” cuando son más de dos grupos (Lattin et al., 2003). El objetivo del análisis es tomar “v” variables X1, X2, X3, …, Xv en “g” grupos predefinidos y encontrar funciones (combinaciones lineares) de estas que producen ejes CV1, CV2, CV3, …, CVg-1 los cuales maximicen la distancia entre los promedios de los grupos (Manly, 1986; Kachigan, 1991; Carroll y Green, 1997; Lattin et al., 2003). La primera función CV1 se selecciona en la dirección que maximiza la “proporción F” de la varianza entre grupos dividida sobre la varianza dentro de los grupos. El grupo más diferente es separado en la dirección del CV1. Cada función CV siguiente maximiza la distancia entre los grupos restantes en una dirección diferente de los ejes CV previos. El par de promedios más cercanos define la dirección del último eje. Entonces para “g” grupos existen un máximo de “g-1” ejes. Las diferencias entre los promedios evaluadas con la “proporción F” pueden ser o no significativas en cada uno de los ejes. Por ejemplo, el último eje muy probablemente no sea significativo cuando el valor de la proporción F es bajo (F<2.0) y la distancia entre los dos promedios es muy pequeña. Por eso sólo cierto número de ejes se califican como “ejes significativos”. En el CVA la orientación de cada eje sucesivo rota a diferentes ángulos. En una rotación rígida, todos los ejes rotan con el mismo ángulo, cualquiera que sea el valor, por lo cual la posición relativa de cada punto en la nube de datos se conserva. En cambio, en el CVA dado que dependen de la posición de los promedios, la rotación de los ejes discriminantes no es rígida. El ángulo de rotación de un eje es independiente del ángulo de la rotación de los otros ejes, por lo que no necesariamente son ortogonales y la dispersión relativa de la nube de puntos se distorsiona. Por esta razón las varianzas explicadas por cada eje discriminante no se suman. En este espacio no ortogonal, la separación entre promedios se calcula con la distancia de Mahalanobis. La fórmula para esta hipotenusa pondera la desviación de ortogonalidad usando las covarianzas entre todos los ejes del CVA considerados como catetos (Manly, 1986; Fielding, 2007).
En la aplicación en sistemática, el análisis discriminante y el CVA permiten detectar cuáles son las mediciones lineales que más contribuyen a la distinción entre grupos, dado que los ejes son en la dirección donde existe mayor separación entre promedios de los grupos (Campbell y Atchley, 1981; Reyment et al., 1984). Un buen ejemplo de la aplicación de los PCA y CVA es el estudio de la variación en tres especies de arbustos del género Cestrum L. (Solanaceae, Del Castillo-Batista et al., 2017). No obstante, en la literatura taxonómica se razona erróneamente que el PCA identifica esos caracteres importantes. Cuando los datos son coordenadas, se razona de manera similar cuando se interpretan los resultados del PCA de la forma. Por ejemplo, los datos de coordenadas de la forma de las hojas se usaron para análisis de “relative warps” (PCA) en especies de helechos del género Ophioglossum L. (Magrini y Scoppola, 2010). Los autores usaron erróneamente las deformaciones del PCA para ubicar diferencias diagnósticas entre especies: “The graphical outputs of the PCA of outlines show the differences between the three species, confirming shape and base of the leaf as the main diagnostic characters”. Lo cierto es que sólo el CVA y el MANOVA asociado calculan la ubicación y magnitud del cambio de forma entre grupos (Webster y Sheets, 2010; Zelditch et al., 2012). Aun suponiendo que las diferencias en forma se estimaran igual, sólo el CVA podría identificar si esas diferencias en forma son significativas o no entre especies.
Igualmente, los PCA a partir de las mediciones informan sobre la dimensión de la varianza generalizada pero no informan sobre la estructura de la varianza entre y dentro de grupos. Las variables con mayor varianza entre especímenes “v1” pueden ser diferentes de las variables “v2” que tienen una varianza mayor entre grupos de especímenes. En el estudio citado de la variación en tres especies de Cestrum, el PCA identificó el largo de la porción libre del filamento como el carácter más variable entre todos los especímenes. Pero el CVA identificó la porción adnada del filamento como el carácter de mayor importancia en la discriminación de especies (Del Castillo-Batista et al., 2017).
Del mismo modo, en el estudio de dos especies de musgos del género Braunia Bruch & Schimper, el PCA indicó que los caracteres con el mayor peso absoluto en el PC1 fueron el ancho de urna de la capsula y la longitud de la lámina de hoja. Pero el CVA detectó que la extensión del margen de la hoja revoluta y el largo de las células superiores de la hoja contribuyeron más a la distinción entre especies (De Luna y Gómez-Velasco, 2008). Estos ejemplos ilustran que los PCA a partir de las mediciones no distinguen entre una variable unimodal “v1” con mucha varianza generalizada y una variable multimodal “v2” igualmente amplia, pero con mucha varianza entre grupos y poca varianza dentro de los grupos. Ambos tipos de variables “v1” y “v2” podrían ser las que más contribuyen a la dirección del PC1, por lo que la identificación de los caracteres taxonómicamente importantes sería potencialmente errónea. En cambio, los CVA sí se basan en las variables tipo “v2”, pues esas son las que contribuyen a la diferencia entre grupos. Cuando se hacen los dos análisis, PCA y CVA, es posible que se descubra que los caracteres que tienen más varianza generalizada no sean los mismos que maximizan las diferencias entre grupos, especialmente cuando los grupos son marginalmente diferentes.
Software
Los análisis estadísticos de las mediciones se realizan mediante programas de cómputo como InfoStat (Di Rienzo et al, 2017), PAST (Hammer et al., 2001), paquetes en R y otros disponibles comercialmente como Systat (Systat Software, 2019) y Statistica (StatSoft, Inc. 2013). La lógica, presuposiciones y soluciones a las desviaciones de Normalidad de las técnicas estadísticas basadas en la suma de mínimos cuadrados son semejantes, sea que los datos de origen son las mediciones de largos y anchos o si los datos son las coordenadas Cartesianas. Sin embargo, como hemos visto, los ajustes y preparativos de las variables y los procedimientos algebraicos en detalle son diferentes, por lo que se han desarrollado herramientas de software exclusivos para los conjuntos de datos de coordenadas. En el espacio curvo de Kendall la dirección de la máxima varianza podría estimarse con “análisis de geodésicas principales” (Fletcher et al., 2004; Huckemann y Ziezold, 2006). Pero en morfometría geométrica es convencional que la nube de datos se proyecte a un espacio Euclidiano tangente a la hiperesfera, para ahí generar variables de la deformación (“partial warps”). La base de estos métodos estadísticos especiales de la morfometría geométrica es el uso de las distancias Procrustes en substitución de la suma de cuadrados tradicionales en la estimación de los modelos lineares (Bookstein, 1996b). Por lo tanto, debe evitarse llevar las matrices de datos de coordenadas Cartesianas a programas diseñados para métodos estadísticos regulares, como Systat (Systat Software, 2019), Statistica (StatSoft, Inc. 2013) y otros.
El programa más usado y recomendable para el registro de las coordenadas de marcas y semi-marcas es sin duda tpsDig (Rohlf, 2015). Las operaciones básicas, como la superposición, los métodos estadísticos y la visualización de las gradillas de deformación TPS están disponibles en la mayoría de los paquetes. Algunos programas agregan diferentes tipos de superposición y alineamiento de semi-marcas (Cuadro 7). No obstante, sin relación con los límites o la diversidad de opciones, algunos programas son más conocidos o populares que otros. Una encuesta reciente con posibilidad de respuestas múltiples (De Luna, 2016) reveló que el software más usado en la comunidad académica latinoamericana es MorphoJ (29% de los usuarios, Klingenberg, 2011). Al parecer la razón es que MorphoJ es el más amigable y cómodo de usar (Benítez, H., com. pers.). Otros programas comunes son la serie TPS (21%, Rohlf, 2015), Geomorph en R (13%, Adams y Otárola-Castillo, 2013) y con 12% de los participantes usando IMP (Sheets, 2014a-d) y PAST (Hammer et al., 2001). Los programas menos usados son Morpho en R (Schlager, 2017), Morphologika (ahora EVAN, O´Higgins y Jones, 2006), Morpheus (Slice, 2013), Shapes en R (Dryden, 2018) y Morpho-tools (Koutecký, 2015). Un programa reciente que será necesario conocer más es XYOM (Dujardin y Dujardin, 2019). La aplicación se accede en línea (https://xyom.io) y permite registrar puntos y realizar análisis basados en marcas y semi-marcas. El desarrollo futuro es posiblemente para los programas en R, los cuales son los más flexibles y potencialmente diversos en las rutinas de cálculo y despliegues de gráficos aplicables (Ospina-Garcés, S., 2019, com. pers.). Por ahora, sería un error usar MorphoJ si los análisis incluyen semi-marcas; este software sólo contiene métodos para el análisis de conjuntos de marcas. En el caso de combinaciones de marcas y semi-marcas (o sólo semi-marcas), los programas apropiados son los de la serie TPS, los de IMP y las funciones en Geomorph con R (Cuadro 7).
Cuadro 7: Operaciones disponibles en algunos programas para morfometría geométrica. Se incluyen sólo los más usados, en orden decreciente de popularidad o preferencia de uso, según una encuesta reciente (De Luna, 2016). Tipos de superposición, BR: Bookstein; SBR: “sliding base registration”; P: Procrustes; RFTRA: medianas repetidas o “resistant‐fit theta‐rho analysis”. A: alineamiento de semi-marcas. Análisis, PCA: análisis de componentes principales (=TPSRelw en la serie TPS); DA: análisis de discriminantes (=TwoGroup en IMP); CVA: análisis de variables canónicas. Sistema operativo, W: Windows; M: MacOS; L: Linux; R: programas en lenguaje R. S: si existe la opción. -: no existe la opción. P: es posible ejecutar el programa nativo de Windows con la ayuda de software emulador, como WINE para Windows en Mac, o para Windows en Linux.
| Software | Superposición | A | Análisis | Sistema operativo | |||||||
| BR | SBR | P | RFTRA | SM | PCA | DA | CVA | W | M | L | |
| MorphoJ | - | - | s | - | - | s | s | s | JAVA | ||
| Serie TPS | - | - | s | - | s | s | - | - | s | p | p |
| Geomorph | - | - | s | - | s | s | s | s | R | ||
| IMP | s | s | s | s | s | s | s | s | s | s | p |
| PAST 3.0 | - | - | s | - | - | s | s | s | s | s | p |
Aplicaciones de la morfometría en el contexto de la sistemática pluralista
Vista como una disciplina científica epistemológicamente pluralista (Miller et al., 2008; Suri, 2013), la sistemática es el estudio comparativo de la diversidad de organismos fósiles y vivientes, por ejemplo, especies, e incluye su descubrimiento, descripción, distribución y reconstrucción de las relaciones filogenéticas que comparten como linajes. A partir de esta información, se producen clasificaciones científicas que son el fundamento para organizar el resto del conocimiento biológico de las especies y para inferir el cambio de sus propiedades, funciones e interacciones con otras especies y con el mundo abiótico (Claridge, 1995). La sistemática es pluralista en el sentido de que el estudio científico para la clasificación de la diversidad biológica se ha orientado en tres áreas diferentes: 1) Exploración para descubrir y documentar la diversidad de organismos, 2) Entender su historia para clasificar la diversidad de organismos, y 3) Difundir y usar ese conocimiento. Cada actividad con propósitos igualmente valiosos permite la síntesis complementaria del conocimiento sobre la biodiversidad (Fig. 10). Consecuentemente, las distintas orientaciones epistemológicas han requerido de diferentes habilidades, recursos tecnológicos, educación y metodologías numéricas de análisis.
Figura 10: Sistemática pluralista. Las tres orientaciones epistemológicas principales en la actividad sistemática. En cada dirección, la dependencia es biunívoca. Por lo tanto, bajo esta visión no cabe argumentar que hay una taxonomía “alfa” anterior a las otras dos áreas. La morfometría tiene aplicación evidente en la identificación de taxones, en el análisis de caracteres taxonómicos y en la inferencia de la filogenia.
Identificación
Bajo esta visión pluralista de la sistemática, una posibilidad del uso de datos morfométricos es en el problema de la identificación de taxones previamente conocidos. En la primera área de la sistemática el objetivo es presentar estimaciones del número de especies en regiones particulares. Mediante los inventarios y las guías de identificación, los biólogos aprendemos a observar las características morfológicas que distinguen a una especie de otra y a determinar el nombre científico apropiado de un organismo particular. Indudablemente la morfometría ha jugado un papel importante en la identificación de especies y en la detección de grupos de organismos diferentes aún sin nombre. Aplicaciones de la morfometría en sistemática tales como los análisis de ordenación (CVA) y los análisis de varianza (MANOVA) son muy informativos para la identificación de taxones conocidos a priori usando la variación de caracteres cuantitativos (Humphries, 2002; Strauss, 2010b). Lo común es intentar la identificación fenética de grupos taxonómicos analizando todos los datos morfométricos combinados. Por ejemplo, Martínez-Domínguez et al. (2017) incluyeron 17 mediciones lineales de varios tipos de caracteres en un solo análisis de discriminantes para evaluar “morphometric diagnosability” de seis especies de cícadas del género Ceratozamia. Lo recomendable desde la perspectiva filogenética es detectar la diagnosis de taxones haciendo los análisis estadísticos por particiones sólo con las variables que describen un carácter a la vez.
Un ejemplo es la aplicación de la morfometría de mediciones lineales en la identificación de dos especies de musgos del género Braunia, presumiblemente sinónimas. Para examinar los patrones de variación de caracteres taxonómicos, además de un CVA global que incluye todas las 21 variables, también ejecutamos un “CVA particionado” para cada conjunto de variables que describen hojas (siete mediciones), células de hoja (ocho mediciones) o cápsulas (seis mediciones) por separado. Se evaluaron las diferencias entre ocho grupos geográficos en los tres “CVA particionados”. Las correlaciones con las variables canónicas revelaron que la extensión de la base revoluta de las hojas y los anchos y largos de las células foliares son las que contribuyen más a la identificación de las dos especies (De Luna y Gómez-Velasco, 2008). En otro ejemplo, se consideró la morfometría geométrica de las alas en la distinción de dos especies de abejas del género Euglossa Latreille. Un análisis discriminante detectó que las intersecciones de las venas del ala anterior y la forma de la célula radial identifican las dos especies (Quezada-Euan et al., 2015).
En la primera área de exploración, otra oportunidad es evidentemente el uso de los métodos morfométricos en el descubrimiento de nuevas especies (Strauss, 2010b). Con la aplicación de los análisis multivariados, la identificación de un morfo sin nombre puede postularse como un grupo potencialmente monofilético al nivel de especie. Por ejemplo, el análisis de la variación de las hojas complementó la distinción de una nueva especie de Salvia (Fragoso-Martínez et al., 2015). La varianza de las coordenadas de la forma de las hojas se examinó con un Análisis de Variables Canónicas comparando cuatro grupos a priori. Los resultados indicaron que la forma de la hoja de la nueva especie es diferente de otras tres morfológicamente muy similares. La distinción de la nueva especie de Salvia incluye además varios caracteres de la forma de crecimiento, número de flores por verticilastro, la forma de los lóbulos posteriores del cáliz, la posición de los tricomas en el cáliz y la forma de las hojas. En aplicaciones morfométricas como ésta, la evaluación de la nueva especie como grupo monofilético potencial puede ser abordada en la siguiente ronda de estudios filogenéticos.
Homología filogenética
En la segunda área de la sistemática pluralista (Fig. 10), la clasificación requiere entender la historia de las especies y cómo se relacionan filogenéticamente entre sí. Estas deducciones derivan del examen minucioso de la variación de varios tipos de caracteres (morfología, secuencias del ADN, etc.) en muchos organismos y los análisis numéricos para la agrupación filogenética (parsimonia, verosimilitud, probabilidades posteriores). El resultado de estos análisis es la formación de grupos monofiléticos a varios niveles. Clasificar entonces es construir grupos monofiléticos con la combinación de todos los tipos de caracteres disponibles. Los datos morfométricos se pueden utilizar para el análisis de la variación de caracteres taxonómicos en la formación de hipótesis de homología primaria y para la inferencia de la filogenia. El concepto de homología filogenética es central en estos dos tipos de análisis.
Epistemológicamente homología filogenética es la relación o correspondencia histórica que existe entre dos o más rasgos de diferentes organismos (de Pinna, 1991; De Luna y Mishler, 1996). Esta relación histórica entre características moleculares o morfológicas de dos o más organismos puede ser de dos tipos: homología táxica y homología transformacional (Fig. 11). Homología táxica es la relación histórica entre “estados similares” observados entre organismos distintos. El mismo estado entre diferentes organismos sugiere la herencia sin modificación a partir de un ancestro común que tenía ese rasgo. La similitud molecular o morfométrica que existe entre dos o más organismos, pero que no es resultado de ancestría común, es “homoplasia”. Homología transformacional es la relación histórica entre “estados diferentes”, cada uno observado en distintos organismos. Los diferentes estados entre organismos sugieren una correspondencia debido a modificaciones históricas de un estado a partir del otro. La homología filogenética no es una relación directamente observable, sino que se detecta como resultado de un método de análisis inferencial mediante la congruencia entre caracteres. El cladograma más parsimonioso permite que las inferencias primarias de homología sean refutadas por su incongruencia con otros caracteres. Sólo así, los estados similares en cualquier tipo de carácter ya sean nucleótidos o morfométricos (Fig. 11), pueden declararse como homoplasia (de Pinna, 1991; De Luna y Mishler, 1996; Palci y Lee, 2019).
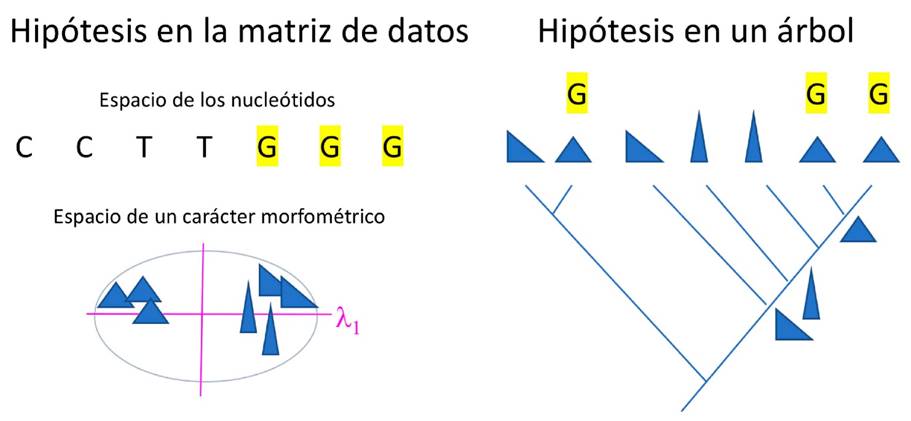
Figura 11: Homología táxica y transformacional. Distribución de estados en siete terminales. Tres terminales comparten una guanina (G) y también tienen un carácter morfométrico (definido por mediciones o coordenadas) muy similar (triángulos equiláteros). En ambos espacios, por ser el mismo estado se postula “homología táxica”. En la filogenia combinada de muchos caracteres, esta similitud en los nucleótidos resulta ser homoplasia para las tres terminales. Como se explica en el texto, parte de la similitud en un carácter morfométrico continuo (triángulo equilátero) se explicaría como homoplasia. La variación del carácter morfométrico con al menos tres estados diferentes puede postularse como homología transformacional (triángulo rectángulo -> isósceles -> equilátero).
Morfometría filogenética
La integración de los análisis morfométricos y análisis filogenéticos requiere un cambio epistemológico para pasar de la morfometría fenética a una morfometría filogenética. Bajo un enfoque de similitud global, todos los caracteres morfométricos se combinan en una matriz y se procede en una o dos direcciones para valorar la similitud total. Primero, para agrupar los especímenes se calcula algún tipo de distancia pareada y con esas distancias se ejecuta algún algoritmo de agrupación como el UPGMA (Crisci y López-Armengol, 1983; Fielding, 2007). Segundo, para ordenar y visualizar la dispersión de los especímenes en el espacio de todos los caracteres se usa comúnmente el Análisis de Componentes Principales y/o el Análisis de Variables Canónicas (Marcus, 1990; Kachigan, 1991; Carroll y Green, 1997). Los grupos de similitud en el fenograma generado y la visualización de nubes de puntos separados con métodos de ordenación (CVA), se interpretan como grupos taxonómicos. Además, dado que los ejes del CVA identifican la máxima separación de los grupos y los caracteres correlacionados con estos ejes son los que más contribuyen a la distinción, se interpreta que esos son los caracteres de mayor importancia taxonómica. Un ejemplo reciente de esta doble estrategia fenética es el estudio de la variación de la similitud total en especímenes del arbusto Gymnopodium floribundum Rolfe (Ancona et al., 2019). Se combinaron datos de mediciones (largos y anchos) y una configuración de coordenadas “x, y” para la forma de la hoja. Se aplicaron los dos tipos de análisis de agrupación y ordenación (UPGMA, CVA). Los resultados revelaron la existencia de tres grupos de especímenes, los cuales se interpretaron taxonómicamente como subespecies. El análisis morfométrico de la similitud total con todos los caracteres combinados así no es la única manera de usar estos datos en sistemática.
Bajo un enfoque filogenético, los análisis morfométricos proceden examinando la variación particionada, es decir, un carácter a la vez. Eventualmente todos los caracteres se combinan en una matriz filogenética y se procede a postular los grupos taxonómicos en la filogenia inferida. El concepto de “carácter taxonómico” como hipótesis de homología filogenética es el fundamento epistemológico para este proceder en el estudio morfométrico de la variación particionada de caracteres y la similitud de estados entre organismos. Esto implica substituir las preguntas fenéticas sobre la formación de grupos taxonómicos con similitud morfométrica total con preguntas filogenéticas sobre la variación cuantitativa de un carácter y la existencia de estados. Con las mediciones o coordenadas de un carácter C1 en una colección de T taxones la pregunta filogenética es ¿cuántos estados se pueden agrupar y distinguir? La selección de un carácter como taxonómico implica una hipótesis de homología transformacional entre al menos dos estados. Entonces la expectativa teórica es que un carácter taxonómico mostrará más varianza entre taxones que dentro de los taxones, es decir, la distribución de mediciones será aproximadamente bimodal o multimodal (Fig. 12). Pero antes, la primera pregunta es exploratoria: ¿qué conjunto de mediciones o coordenadas describe la variación de un carácter taxonómico? La respuesta requiere una estimación cuantitativa de la comparabilidad y la inspección de la correspondencia topológica (Hawkins et al., 1997; Rieppel y Kearney, 2002, 2007).
Figura 12: Espacio geométrico de un carácter taxonómico y expectativas de los patrones de la variación cuantitativa. En los cuatro casos (A, B, C, D), el espacio puede considerarse bivariado, por ejemplo, como cuando medimos el largo y ancho de un carácter. A. variación homogénea, probablemente no informativa; B. variación amplia pero continua; C. variación amplia multimodal, potencialmente informativa; D. la misma distribución que en (C), pero la variación ahora se expresa con los valores en un nuevo eje resultado de la combinación lineal de las dos variables originales. La variación expresada en este eje es muy probablemente informativa. T1, T2, T3, son grupos de especímenes de taxones diferentes.
Cuando se analiza la variación de un carácter taxonómico se deben explorar varios espacios matemáticos alternativos. Consecuentemente, además de analizar estadísticamente las mediciones colectadas, se construyen nuevas variables y espacios de análisis, particularmente, cuando los valores de los largos y anchos de un carácter están correlacionados. Ese es el caso de la variación de las valvas de especies de las diatomeas del género Chaetoceros Ehrenberg (Fig. 13A). Para ilustrar este ejercicio se dispuso de datos (Ake, J., com. pers.) de los largos (eje pervalvar) y anchos (eje apical) de las valvas en 20 especímenes en varias especies de Chaetoceros. Un análisis estadístico nos revela una distribución multimodal, pero con mucho sobrelape entre las distribuciones de cada especie, para las dos mediciones. Por ejemplo, los promedios y desviaciones estándar de los valores en micras del eje pervalvar no son muy diferentes (Fig. 13B). La conclusión rápida es que esa variación no es informativa, pues la distinción de estados no es evidente. Sin embargo, falta explorar si el problema puede ser que esas dos variables no son buenas descriptoras del espacio matemático del carácter taxonómico. Para explorar otro espacio matemático, las dos variables se sometieron a un análisis de variables canónicas (CVA) usando Statistica (Fig. 13C). El despliegue de los valores en la nueva variable el eje canónico 1 (CV1, Fig. 13D) muestra un espacio donde es evidente la existencia de al menos dos estados de carácter entre las especies. El análisis de varianza seguido de una comparación múltiple de promedios sobre los valores del CV1 para cada especie efectivamente muestra que hay dos grupos de promedios. Este ejemplo ilustra que el análisis de la variación sobre los valores de una combinación lineal de las dos variables puede revelar que en ese espacio rotado sí hay variación informativa, claramente multimodal. El carácter taxonómico no se describe ni con el largo ni con el ancho por separado, sino una combinación lineal de ambas variables. Los análisis morfométricos pueden resultar en la redefinición de los descriptores numéricos y un cambio del espacio matemático de los caracteres.
Figura 13: Exploración de espacios matemáticos alternativos en la definición de un carácter taxonómico. Variación de las valvas en un conjunto de especies de diatomeas del género Chaetoceros Ehrenberg. A. largo (eje pervalvar) y ancho; B. variación casi homogénea en el largo de las valvas; C. gráfica de dos ejes en un análisis de variables canónicas; D. distribución claramente bimodal de los valores en el primer eje CV1. Fotografía y datos, cortesía de José Aké.
Morfometría y la identidad de estados de caracteres
Después de explorar las variables para describir la variación de un carácter, los métodos estadísticos se aplican filogenéticamente con el objetivo de cuantificar niveles de similitud para discernir la existencia de estados entre organismos. La similitud en la variación de un carácter taxonómico puede ser un concepto vago en la sistemática porque a veces se refiere a la similitud posicional (correspondencia topográfica) y a veces al parecido en forma o tamaño (Rieppel y Kearney, 2002). Aquí la primera opción se relaciona con la conceptualización del carácter como entidad discreta, relevante y comparable (Farris et al., 1970; Freudenstein, 2005; Sereno, 2007; “theory-laden objects” sensuWheeler, 2012). Operativamente esto involucra seleccionar o delimitar cual estructura se medirá o registrará con coordenadas de puntos. La segunda aplicación se circunscribe al concepto de parecido o identidad de estados (“character state identity”, sensuBrower y Schawaroch, 1996; Humphries, 2002). Decidir sobre la identidad de los estados taxonómicos entre organismos es una estimación estadística, la cual requiere considerar la estructura de correlaciones y varianzas en algún espacio matemático univariado, bivariado o multivariado. La evaluación cuantitativa de la prueba de similitud para hipótesis de homología primaria (“similarity test”, sensuPatterson, 1982) permite dar una respuesta formal de si es o no el mismo estado taxonómico en la variación multimodal de un carácter.
Los conceptos de probabilidad y los procedimientos estadísticos se pueden usar para evaluar si los taxones T comparados en el análisis son “iguales” o “diferentes” para los estados en la variación de un carácter (Jardine, 1969; Rae, 1998). Las mediciones de un carácter C1 para cada taxón se resumen en los parámetros estimados de la distribución y tendencia central (en un sentido estadístico). Las decisiones sobre qué distribuciones unimodales o multimodales (y las tendencias centrales asociadas) son diferentes o no, deben evaluarse con los niveles de variación dentro de y entre taxones incluidos en el análisis (Rae, 1998). La identidad de estados se estima entonces con análisis de varianza univariados o multivariados de las mediciones o de las coordenadas. Con un objetivo filogenético, los métodos multivariados (CVA, PCA, UPGMA) se usan para explorar si en dos o más taxones Tn, un carácter taxonómico C1, descrito por dos variables o más Vn, es potencialmente informativo con al menos dos estados. El árbol de similitud total derivado del UPGMA puede interpretarse como la hipótesis primaria del orden de estados o la serie de transformación de estados del carácter (“character state tree” sensuFreudenstein, 2005).
Un ejemplo reciente de la construcción del árbol de los estados es el caso de la variación en el sulcus acusticus del otolito sagittae en varias especies de la familia de peces Gerreideae. La variación de los contornos de este pequeño canal se describió con semi-marcas. El árbol de las distancias Procrustes con un UPGMA se interpretó como “the character state tree of the variation in the sulcus” (Chollet-Villalpando et al., 2019). Los árboles de los estados de un carácter morfométrico también se pueden estimar con métodos de verosimilitud usando el programa ContML-PHYLIP v. 3.6 (Felsenstein, 1973, 2000). Por ejemplo, la variación de los frutos en especies del género Lithocarpus Blume descrita con coeficientes de Fourier y tres mediciones se agrupó con máxima verosimilitud y el árbol se interpretó como “the most likely transformation series of shape change” (Cannon y Manos, 2001). En otro ejemplo, la variación del cuerpo de peces del género Diapterus Ranzani, descrita con los “partial warps scores” derivados de las coordenadas de 16 puntos, se analizó con parsimonia (usando TNT) y con máxima verosimilitud (ContML) para construir el árbol de los estados de la forma (Vergara-Solana et al., 2014). Otro ejemplo ilustra el árbol de la transformación de estados en la variación de la forma de la cauda entre especies de ácaros del género Arrenurus, descrita con coordenadas Cartesianas construido con parsimonia en TNT (Ramírez-Sánchez et al., 2016, Fig. 14).
Figura 14: Uso de métodos morfométricos para la justificación empírica de las hipótesis de identidad de estados taxonómicos como homología primaria. Variación de las brácteas florales (n=470) en 12 especies de bromelias del grupo de Tillandsia bulbosa Hook. A. inflorescencia; B. bráctea floral con dos contornos registrados con semi-marcas; C. tres grupos en un fenograma (UPGMA con Distancias Mahalanobis); D. variación en la forma de las brácteas. Las fotografías están arregladas según el ordenamiento en los dos primeros ejes de un PCA; E. despliegue de tres nubes de puntos en el plano de las dos primeras variables canónicas (CVA). La similitud en diez especies (en rojo) sugiere que comparten el mismo estado. Queda por ver en un análisis filogenético si esa similitud es homología táxica u homoplasia. Fotografías y datos, cortesía de Tania Chew.
El siguiente ejercicio ejemplifica la interpretación del despliegue de tres nubes discretas de puntos en el plano de las dos primeras variables canónicas para revelar la identidad de al menos tres estados distintos en la variación morfométrica de un carácter taxonómico. La interpretación de estos tres grupos en el fenograma también permite postular una hipótesis primaria de la serie de transformación de los tres estados, independientemente del número de taxones comparados. Para este ejercicio se analizaron los datos de la variación de las brácteas florales (Fig. 14A) en 470 especímenes de 12 especies de bromelias del grupo de Tillandsia bulbosa Hook. (Chew et al., 2010). Se registraron tres marcas (una en el ápice y dos en la base) y dos contornos con 45 semi-marcas (Fig. 14B) usando MakeFan (Sheets, 2005) y tpsDig (Rohlf, 2017). Se procedió a la superposición Procrustes y alineamiento de las semi-marcas con CoordGen y SemiLand (Sheets, 2014a, b). Para el análisis de agrupamiento se usaron las distancias de Mahalanobis estimadas en el espacio de todas las variables canónicas con CVAGen (Sheets, 2014d). Se usó el algoritmo de agrupamiento UPGMA en NTSys (Rohlf, 2000). La variación de la forma de las brácteas florales en las 12 especies es muy amplia (Fig. 14D). Las distancias de Mahalanobis indican que diez especies, aunque variables, son muy similares entre sí. El fenograma muestra claramente las diez especies en un sólo grupo (Fig. 14C). El despliegue de las dos primeras variables canónicas muestra los resultados del análisis multivariado de varianza (MANOVA) revelando tres formas en la variación de las brácteas florales (Fig. 14E).
Las preguntas sobre la variación de los caracteres y estados se examinan con los análisis estadísticos y morfométricos particionados, es decir, sólo con la o las variables usadas para cuantificar un carácter. En la práctica no es difícil seleccionar el conjunto de mediciones o puntos para describir un carácter. Para el estudio de la variación de especies de ácaros acuáticos del género Arrenurus (Acari, Hichnidia, Arrenuridae) se analizaron ocho caracteres multivariados (Ramírez-Sánchez et al., 2016). Estos caracteres se describieron cuantitativamente con tres conjuntos de mediciones y cinco configuraciones de coordenadas “x, y”. Para cada conjunto de variables por separado, se usaron las técnicas y análisis morfométricos (CVA, UPGMA) para la cuantificación de la identidad de estados entre 13 especies. Los resultados revelaron que algunos caracteres son muy variables y tienen muchos estados diferentes, mientras que otros caracteres son menos variables y tienen pocos estados. Este estudio ejemplifica el uso de métodos morfométricos para la justificación empírica en las hipótesis de identidad de estados como homología primaria. Dado que la similitud morfométrica no es homología filogenética, tal juicio cuantitativo de la existencia de estados de carácter sigue siendo hipótesis de homología primaria, que debe analizarse para congruencia. Los análisis filogenéticos y la congruencia entre caracteres permiten distinguir si un sistema de estados de carácter morfométrico son homología u homoplasia (De Pinna, 1991; De Luna y Mishler, 1996; Palci y Lee, 2019).
Datos morfométricos en la inferencia de la filogenia
El uso de los datos morfométricos para la inferencia de la filogenia ha provocado reacciones en dos direcciones epistemológicas opuestas. Por un lado, investigadores opinan “a priori” que absolutamente TODOS los caracteres morfométricos (mediciones o coordenadas) no contribuyen a la filogenia y por lo tanto no se deben incluir (Bookstein, 1994; Rohlf, 1998, 2002; Monteiro, 2000). De acuerdo con esta visión, los datos morfométricos se pueden mapear “a posteriori” sobre una filogenia preestablecida (Bookstein, 1994; Rohlf, 1998, 2002; Monteiro, 2000; Felsenstein, 2002; Klingenberg y Gidaszewski, 2010; Adams et al., 2011, 2013). Por otro lado, con la idea de examinar si los datos morfométricos son homología u homoplasia, se han desarrollado métodos de parsimonia para el análisis de la congruencia entre varios tipos de caracteres (sinapomorfía). Bajo este enfoque, los caracteres morfométricos siempre se incluyen combinados con otros caracteres para la inferencia de homología y grupos monofiléticos en una filogenia (Naylor, 1996; MacLeod, 2002; Goloboff et al., 2006; Gonzalez-José et al., 2008; Catalano et al., 2010; Clouse et al 2011; Palci y Lee, 2019). Bajo el enfoque de los métodos probabilísticos, también ya se han explorado análisis filogenéticos con parsimonia cuadrada y con máxima verosimilitud, aunque con modelos muy elementales (Ascarrunz et al., 2019).
Históricamente, las discusiones y los argumentos sobre los datos morfométricos en la inferencia de la filogenia se han centrado en, 1) el uso de estados morfométricos discretos (Monteiro, 2000; Adams et al., 2011) o los datos continuos (Catalano et al., 2010), 2) el uso de las variables de la deformación relativa (partial warps, Naylor, 1996; Adams et al., 2011) o las coordenadas (Catalano et al., 2010) directamente, 3) el uso de los métodos alternativos de parsimonia (Goloboff et al., 2006) o de parsimonia cuadrada (Rohlf, 2002) o verosimilitud (Felsenstein, 2002) para la inferencia de la filogenia y 4) sobre si los datos morfométricos contienen o no señal filogenética (Klingenberg y Gidaszewski, 2010). Dado que hay problemas con la discretización de estados morfométricos, metodológicamente es preferible usar los datos continuos. También se ha recomendado el uso de las coordenadas, tanto con métodos de parsimonia como con máxima verosimilitud, pues en ambos casos no hay necesidad de extraer los partial warps como estimadores de deformación relativa para la inferencia de la filogenia (Catalano et al., 2010; Felsenstein, 2010). El punto controversial sigue siendo si los datos morfométricos aportan señal filogenética o no.
Esta revisión promueve la posición epistemológica de que todos los tipos de datos deben ser escudriñados para análisis filogenético potencial (Thiele, 1993; Stevens, 2000; Humphries, 2002). Además, la expectativa teórica en la reconstrucción filogenética es que la combinación de todos los tipos de datos disponibles (Nixon y Carpenter, 1996; Kluge, 1997, 2004; Rieppel, 2005; Assis, 2015), incluyendo los morfométricos codificados o continuos, puede proveer las mejores hipótesis sobre las relaciones filogenéticas. Empíricamente, la inclusión de los caracteres de las formas, al igual que el impacto de otros caracteres, tiene un efecto mínimo en la reconstrucción de las relaciones filogenéticas, especialmente cuando se combinan con muchos datos moleculares (Ospina-Garcés y De Luna, 2017; Solís-Zurita et al., 2019). Si se incluyen en la inferencia de la filogenia, algunos caracteres cuantitativos de mediciones o coordenadas sobrevivirán como homología y algunos se replantearán como homoplasia. Como con cualquier tipo de datos, la calificación de homología u homoplasia en los datos morfométricos debe depender de usar índices de consistencia para evaluar filogenéticamente su utilidad. Los índices de consistencia en varios análisis han revelado niveles semejantes de homología y homoplasia en los datos morfológicos, conductuales y moleculares en la inferencia de la filogenia (Sanderson y Donoghue, 1989; De Queiroz y Wimberger, 1993; Philippe et al., 1996; Sanderson y Hufford, 1996; Engstrom et al., 2004). Conforme se examinen más casos de estudio, es posible que la variación en el nivel de homoplasia en los datos morfométricos sea semejante a la variación en el nivel de homoplasia que se ha documentado en todos los tipos de datos.
Para la inferencia de la filogenia la variación de un carácter morfométrico puede analizarse con códigos discretos o con los valores continuos. La decisión cuantitativa sobre la identidad de estados es independiente de la necesidad o no de aplicar un sistema de codificación de estados discretos (Hawkins, 2000; Stevens, 2000; Jenner, 2002), puesto que es factible usar los valores continuos al incluir un carácter morfométrico en la matriz filogenética, tanto para análisis con parsimonia (Goloboff et al., 2006; Catalano et al., 2010), como para análisis con parsimonia cuadrada y máxima verosimilitud (Ascarrunz et al., 2019). En un estudio comparativo del desempeño de los caracteres morfométricos con parsimonia se encontró que tanto la matriz de caracteres codificados como la de los caracteres cuantitativos resultan en árboles similares (Thiele, 1993). Por ejemplo, antes del desarrollo de métodos para el análisis de datos continuos, la variación de caracteres morfométricos en especies de orquídeas del género Bletia Ruiz & Pav. se codificó en estados discretos para analizarlos filogenéticamente (Sosa y De Luna, 1998). Un re-análisis con parsimonia de los mismos caracteres, pero ahora con los datos continuos (promedios y rangos) produce una filogenia congruente con la filogenia basada en los caracteres codificados discretamente (Fig. 15). Estas dos filogenias a su vez son congruentes con el subclado de Bletia reflexa Lindl. en otra filogenia posterior basada en caracteres moleculares, aunque con los morfométricos todavía codificados (Sosa, 2007). Este ejemplo ilustra que la prueba de similitud (sensuPatterson, 1982) para homología primaria es diferente del procedimiento para re-expresar la variación continua en códigos discretos en una matriz filogenética. La frecuencia observada en la distribución de las mediciones en los especímenes describe numéricamente los estados (Jardine, 1969; Thiele, 1993), aunque no obliga a codificarlos como estados discretos (Farris, 2007). Los promedios y las varianzas pueden ingresarse en la matriz y analizarse filogenéticamente como datos continuos (Goloboff et al., 2006). Otros ejemplos ya han demostrado la utilidad filogenética de los caracteres descritos con mediciones de largos y anchos analizados con códigos discretos (Guerrero-Enríquez et al., 2003) y también directamente con los valores continuos de los promedios y desviaciones estándar con métodos de parsimonia (De Bivort et al., 2010; Parins-Fukuchi, 2018; Solís-Zurita et al., 2019).
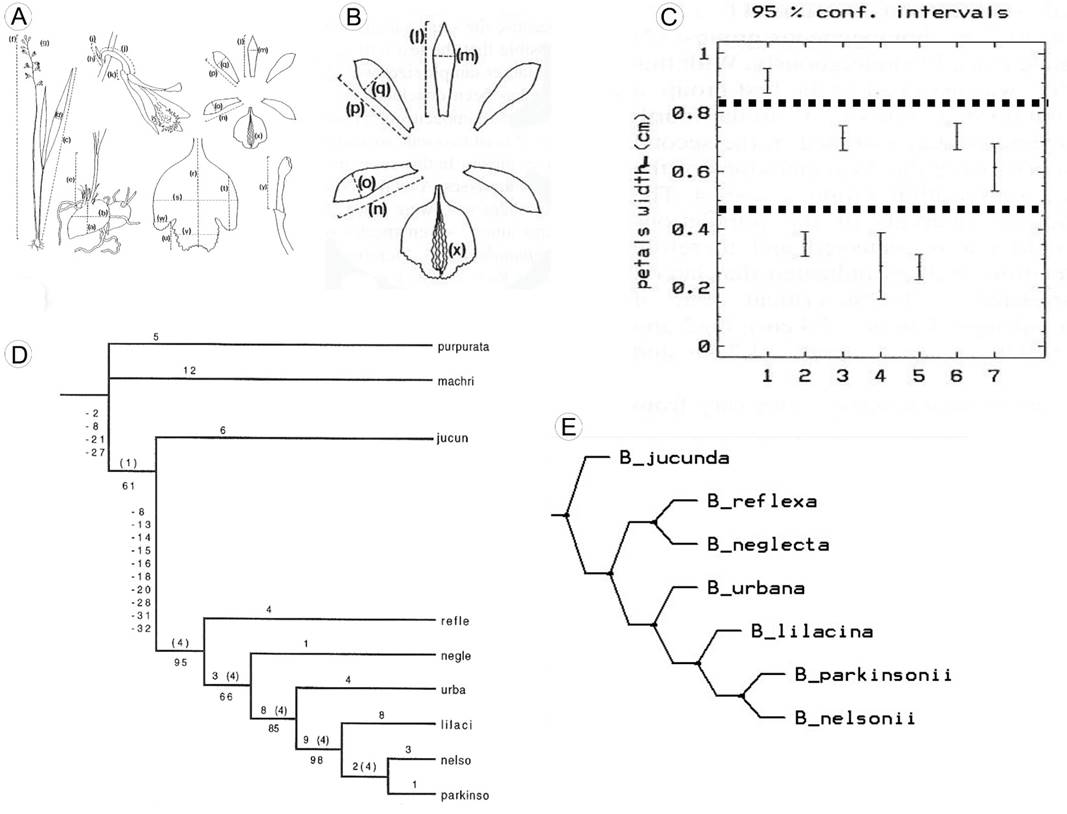
Figura 15: Uso opcional de códigos discretos o continuos para la inferencia de la filogenia. Variación de caracteres morfométricos en especies de orquídeas del género Bletia Ruiz & Pav. A. mediciones lineales de varios caracteres colectados en siete especies (1-7); se dispuso de entre 10 y 24 especímenes de cada especie; B. detalle de las mediciones de la flor; C. tres estados del ancho de los pétalos [[1] [3,6,7,] [2,4,5]] con códigos discretos 0, 1, 2, respectivamente; D. filogenia usando PAUP a partir de caracteres morfológicos discretos combinados con todos los morfométricos codificados en estados discretos (Sosa y De Luna, 1998); E. filogenia con los mismos caracteres morfométricos, usando TNT (Goloboff y Catalano, 2016) para analizar los promedios y rangos como datos continuos sin codificar.
La ruta de procedimientos en la integración de los datos morfométricos para los análisis filogenéticos con métodos de parsimonia se ilustra en el esquema de la Figura 16. Una de las maneras de incluir datos morfométricos en un análisis filogenético es con el software TNT (Goloboff y Catalano, 2016). La preparación de los caracteres morfométricos, por ahora, depende de disponer de este programa que permite combinar datos continuos y otros tipos de datos. Los conjuntos de mediciones o las coordenadas de puntos para describir un carácter se ingresan definidos como bloques para el formato de las matrices en este software. El Cuadro 8 ejemplifica con las coordenadas superpuestas de los “pentagonoides” el formato y sintaxis de comandos básicos requeridos para poder analizar filogenéticamente el cambio en las formas en TNT. Cada forma definida por una configuración de puntos (marcas y/o semimarcas) es un bloque que inicia con un identificador, las terminales, los valores de las coordenadas y el comando de fin del bloque. La tarea de convertir las coordenadas superpuestas desde los formatos comunes en los programas morfométricos (IMP, tpsDig) al formato de TNT puede facilitarse con el programa LMs4TNT ( Sandría y De Luna, 2016).

Figura 16: Protocolo para el análisis filogenético de datos morfométricos. Los conjuntos de diferentes caracteres morfológicos discretos, conteos, mediciones, las coordenadas superpuestas y los nucleótidos de marcadores moleculares se ingresan definidos como bloques distintos en el formato de las matrices [X] para el programa de análisis filogenético TNT (Goloboff y Catalano, 2016).
Cuadro 8: Coordenadas superpuestas 2D en el formato TNT para los seis “pentagonoides” A-F de la Figura 2. Estas son las mismas coordenadas del Cuadro 1 convertidas al formato TNT con el programa LMs4TNT (Sandría y De Luna, 2016). Este programa lee las coordenadas superpuestas y el tamaño (CS) para escribir dos bloques de datos. Los dos primeros números después de dos líneas de comandos de TNT son el número de caracteres (nc=2) y el número de terminales (nt=6). El primer bloque (carácter 1=CS) empieza con el comando “& [Cont]” usado para los datos continuos de mediciones. El bloque para el segundo carácter empieza con “& [landmark 2d]” para las coordenadas de cinco puntos de la forma de los “pentagonoides”. Los valores del par de coordenadas de cada punto ahora llevan una coma en medio.
| nstates
cont; xread 2 6 & [Cont] pentagonoideA 394.89 pentagonoideB 625.476 pentagonoideC 486.563 pentagonoideD 394.417 pentagonoideE 481.34 pentagonoideF 624.029 & [ landmark 2d ] pentagonoideA -0.424423,0.135222 -0.00152474,0.444174 0.428978,0.137765 0.266914,-0.36111 -0.269944,-0.356051 pentagonoideB -0.540261,0.0603161 -0.00967607,0.283481 0.536079,0.112025 0.338318,-0.212483 -0.32446,-0.243338 pentagonoideC -0.262228,-0.000649298 0.181629,0.485097 0.368288,0.295787 0.0532519,-0.299723 -0.340942,-0.480511 pentagonoideD -0.427852,0.135429 0.000888755,0.446924 0.427657,0.135413 0.264258,-0.360665 -0.264952,-0.357102 pentagonoideE -0.413761,-0.0600367 0.146796,0.327243 0.51607,0.267203 0.163814,-0.170507 -0.412919,-0.363903 pentagonoideF -0.261422,0.185373 0.0254773,0.566392 0.276484,0.161401 0.146046,-0.46831 -0.186585,-0.444855 ; proc/; |
La teoría y métodos sobre el flujo de operaciones a partir de una matriz con caracteres combinados está fuera del alcance de esta revisión, pues son materia de los métodos de análisis filogenéticos con parsimonia que se ejecutan para la inferencia de filogenias y para la optimización de hipótesis de cambio en los caracteres morfométricos (Goloboff et al., 2006; Catalano et al., 2010; Catalano y Goloboff, 2012). En términos generales, cuando se analizan varias formas, las coordenadas promedio de cada terminal (especie) se estandarizan para igualar el efecto del diferente número de puntos en cada configuración. Estos procedimientos de estandarización se implementan según lo sugerido por Catalano et al. (2015). Independientemente del número de puntos, cada configuración (marcas y semi-marcas) se pondera de tal manera que la contribución de cada forma a la longitud total del árbol es similar a la de un solo carácter taxonómico. En este procedimiento para la búsqueda del árbol más corto, TNT optimiza simultáneamente la longitud total del árbol y la superposición de las configuraciones.
La superposición filogenética no es una superposición con mínimos cuadrados para estimar una hipotenusa ni tampoco es referida al promedio de todas las formas, como la superposición Procrustes, lo cual es usual en la morfometría geométrica. En la superposición con parsimonia, los desplazamientos de puntos se minimizan con distancias Euclidianas a través de los catetos (distancia Manhattan) y superponiendo jerárquicamente las configuraciones de ancestros y descendientes considerando la topología de un árbol. La optimización de la superposición filogenética es entre las tres configuraciones (a, b, c) conectadas a un nodo interno (d), minimizando las medianas geométricas de Fermat de las distancias en las coordenadas de cada punto (Catalano et al., 2010). El procedimiento estima una configuración ancestral para el nodo interno “d” aproximando posiciones óptimas para cada punto (marcas o semi-marcas). La posición ancestral de un punto se optimiza para los valores alternativos de las posiciones observadas o estimadas de ese punto en las configuraciones vecinas (a, b, c). Se explora sólo un conjunto limitado de estados continuos posibles muestreados en subdivisiones (p. ej., unas 4, 6, o 10) de una cuadrícula. Las aproximaciones de las configuraciones en cada nodo interno se optimizan con el algoritmo de Sankoff usando una cuadrícula local más pequeña en cada paso para afinar las posibles posiciones de cada punto (“grid nesting”, Goloboff y Catalano, 2011). Además, con la ayuda de scripts, la superposición filogenética en TNT se puede hacer optimizada dinámicamente para todos los nodos internos en cada examen sucesivo de la longitud de cada árbol con todos los caracteres combinados, hasta encontrar la solución óptima para la topología del árbol y para la superposición (Catalano y Goloboff, 2012).
Un ejemplo reciente que ilustra la ruta de procedimientos es el estudio filogenético con parsimonia de las especies de lagartijas del grupo Sceloporus variabilis Wiegmann (Squamata: Phrynosomatidae). En este análisis se combinaron tres marcadores moleculares, datos morfológicos discretos y datos morfométricos sin codificar. En la matriz se incluyeron los promedios y los rangos de 17 mediciones lineales. También se incluyeron en el análisis tres configuraciones de coordenadas “x, y” para describir la forma de escamas cefálicas (Solís-Zurita et al., 2019). En el caso de los estados discretos, los estados similares pueden declararse homología u homoplasia. Pero cuando los datos son continuos, sólo es posible decir si parte de toda la similitud de un estado morfométrico en un nodo es homología o si cierta proporción de la similitud es homoplasia. En la filogenia del grupo Sceloporus variabilis, la identidad de estados morfométricos entre nueve especies de lagartijas en algunos casos (forma de las escamas cefálicas, longitud de la pata delantera) resulta ser mayormente por homología y en otros (longitud del cuerpo) la mayor proporción de la similitud es por homoplasia. Este estudio ejemplifica la interpretación a posteriori de homología y homoplasia en los datos morfométricos continuos.
La idea “a priori” de que los caracteres morfométricos (mediciones o coordenadas) no contribuyen a la filogenia no se justifica ni epistemológica ni empíricamente. Aunque la disposición dominante es la de no incluir los datos morfométricos en la inferencia de la filogenia, la discusión está migrando al campo analítico, pues se ha reconocido que la decisión de si son útiles o no sólo es posible después de analizarlos combinados para inferir una filogenia. Se sabe que Felsenstein ha estado experimentando con métodos de máxima verosimilitud para el análisis de las coordenadas con superposiciones que consideran la topología de un árbol (Felsenstein, 2010). Sólo es cuestión de tiempo para que nuevos métodos se implementen en software para enriquecer con más casos empíricos la siguiente ronda de discusiones sobre el análisis de los datos morfométricos con enfoques filogenéticos bajo parsimonia, parsimonia cuadrada y máxima verosimilitud.
Recomendaciones
1. Se requiere un entendimiento básico de la estructura teórica, tanto de la morfometría como de los métodos de análisis estadísticos para una buena aplicación y para aprovechar cada nueva experiencia de investigación.
2. Los diferentes métodos de superposición de coordenadas Cartesianas distribuyen los componentes de la deformación y varianzas punto a punto en ubicaciones y magnitudes diferentes. Conviene entonces explorar la inestabilidad de resultados bajo diferentes modelos de superposición para construir el mapa de la deformación relativa con la función TPS.
3. En el caso de las mediciones lineales, se debe evitar el uso de variables compuestas en la forma de proporciones. Para los análisis estadísticos y morfométricos se pueden usar las dos variables para construir una combinación lineal. En el caso multivariado es mejor usar todas las variables sencillas disponibles. En cualquier caso, los análisis estadísticos son particionados, usando sólo las variables que describen numéricamente un carácter taxonómico a la vez.
4. En el caso de las coordenadas de puntos, es mejor usar relativamente muchos puntos para optimizar las superposiciones y tener mejor poder de interpolación de las deformaciones entre puntos con la función TPS. En el análisis filogenético de las coordenadas con parsimonia, ni las superposiciones ni la visualización con estas interpolaciones tienen efecto alguno en la filogenia inferida. En los análisis estadísticos, si se requiere se puede disminuir el número de variables para los análisis de varianza en un CVA, mediante el uso de los primeros componentes principales. Entre más heterogénea sea la varianza, será posible analizar la mayoría de la dispersión en los datos (> 95% de la varianza) con menos componentes.
5. Las mediciones lineales y las coordenadas Cartesianas de puntos no son alternativas en competencia. Epistemológicamente los dos tipos de análisis son confirmatorios. Ambos descriptores numéricos tienen limitaciones y ventajas. Conviene usar las dos herramientas en todos los estudios morfométricos, ya que los análisis confirmatorios pueden robustecer o modificar las conclusiones derivadas usando sólo una herramienta. Así como en los análisis estadísticos, las mediciones y las coordenadas se analizan por separado; también conviene explorar su utilidad y su independencia. Si las mediciones y las coordenadas de la misma estructura o morfología no son independientes, entonces ambos conjuntos no se deben incluir combinados en los análisis filogenéticos.
6. Los análisis de ordenación PCA y CVA no son métodos de agrupación estadísticos ni taxonómicos. Si los grupos existen a priori, ambos métodos de ordenación permiten explorar la ubicación y distinción relativa entre los promedios de esos grupos. Si los grupos taxonómicos no existen a priori, estos deben construirse mediante métodos filogenéticos de agrupación. La identificación de los caracteres (mediciones lineales o forma) que más contribuyen a la distinción taxonómica se debe explorar en los análisis de discriminantes o el CVA, pero no en los análisis de componentes principales.
7. Bajo un enfoque filogenético, los análisis morfométricos se usan para el estudio de la variación de caracteres taxonómicos y los estados. Los métodos estadísticos permiten el objetivo de cuantificar niveles de similitud o identidad de los estados de carácter.
8. Los caracteres morfométricos de las mediciones y coordenadas se incluyen en la inferencia de la filogenia usando los valores continuos. Se recomienda combinarlos con otros tipos de caracteres para efectivamente calificar mediante la prueba de congruencia en los análisis filogenéticos cuales mediciones y formas pueden postularse a posteriori como homoplasia y cuales sobreviven como hipótesis de homología táxica y homología transformacional.











 nueva página del texto (beta)
nueva página del texto (beta)



