Introducción
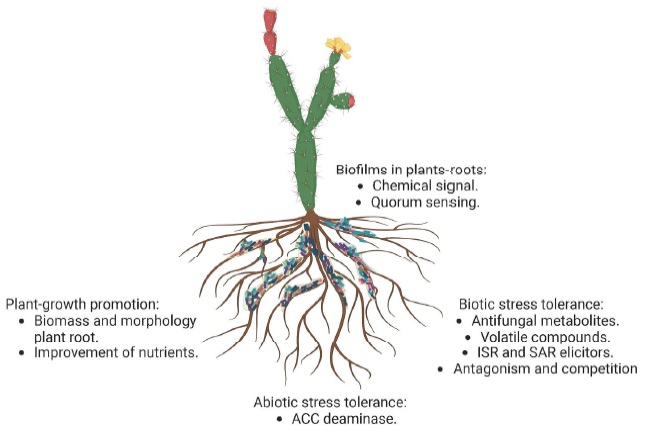
Las plantas son muy diversas y habitan la mayor parte de los ecosistemas terrestres, donde juegan un papel fundamental como productoras en la cadena trófica (Delaux y Schornack, 2021). Las plantas albergan muchos microorganismos en una relación altamente regulada y específica conocida como: microbioma vegetal, el cual es diverso y dinámico. El microbioma está compuesto por organismos procarióticos y eucarióticos, incluidas bacterias, arqueas, virus, hongos y protozoos (Lemanceau, Blouin, Muller y Moënne-Loccoz, 2017; Abdelfattah, Wisniewski, Schena y Tack, 2021). Las plantas tienen varios mecanismos para modular su microbioma, incluidas las modificaciones estructurales, la exudación de metabolitos secundarios y la acción coordinada de diferentes respuestas de defensa (sistema inmunológico de la planta). Además, las composiciones del microbioma difieren entre los compartimentos espaciales de las plantas y su entorno, así pues, se definen como: endosfera para las comunidades que se encuentran dentro de los tejidos de las plantas, y filosfera, rizoplano o rizósfera para las comunidades microbianas adheridas al exterior de los diferentes órganos o que habitan en el suelo cercano a la raíz (Santoyo, 2022). Asimismo, el medio ambiente, principalmente el suelo, es ampliamente considerado el origen y fuente principal de los microorganismos que componen el microbioma de las plantas (Berendsen, Pieterse y Bakker, 2012; Gopal y Gupta, 2016), cuyo ensamblaje es un proceso de varios pasos que está determinado por: la dispersión, las interacciones de las especies, el medio ambiente y el huésped (Mhlongo, Piater, Madala, Labuschagne y Dubery, 2018; Trivedi, Leach, Tringe, Sa y Singh, 2020). Las interacciones entre los microorganismos de la rizósfera son decisivas para la salud de la planta huésped, donde pueden coexistir patógenos del suelo y microorganismos benéficos (Yu, Pieterse, Bakker y Berendsen, 2019). Así pues, los miembros que forman el microbioma de la planta confieren ventajas a la planta huésped, tales como: la promoción del crecimiento, la absorción de nutrientes, la tolerancia al estrés y la resistencia a los patógenos, siendo este último un campo de estudio de interés y poco investigado (Figura 1). Recientemente, se ha observado que diversos microorganismos que habitan en la rizósfera y bacterias endófitas sintetizan una amplia gama de metabolitos secundarios que tienen actividad antimicrobiana, siendo de interés en biotecnología para su aplicación en la industria farmacéutica, entre otras más. Ante esto, se ha observado la necesidad de comprender el complejo proceso de formación del microbioma vegetal, donde la comunicación bacteriana y la formación de biopelículas son puntos clave.
Figura 1: El microbioma vegetal confiere ventajas al huésped, como la promoción del crecimiento vegetal, la tolerancia al estrés abiótico y la tolerancia al estrés biótico a través de diferentes mecanismos, donde uno de los más importantes es la formación de biopelículas en las raíces y la producción de compuestos antimicrobianos.
Los microorganismos que componen el microbioma de las plantas usan la quimiotaxis para detectar y responder a las señales derivadas de las plantas presentes en los exudados de las mismas para iniciar, posteriormente, la colonización. Una vez que se percibe una señal, los microorganismos se mueven hacia la planta, principalmente, a través del uso de estructuras celulares llamadas: flagelos. Posteriormente, los microorganismos se adhieren a la superficie de la raíz y forman estructuras conocidas como biopelículas (Trivedi et al., 2020). Sin embargo, el sistema inmunitario de la planta tiene un papel principal en el ensamblaje del microbioma, los miembros de la microbiota pueden suprimir las respuestas inmunitarias de la raíz, lo que probablemente promueva la colonización. Además, el reconocimiento de microorganismos comensales y la exclusión de patógenos es un punto clave en este proceso (Leach, Triplett, Argueso y Trivedi, 2017). La primera línea son los receptores de reconocimiento de patrones (PRR, por sus siglas en inglés) ubicados en la membrana plasmática que reconocen diferentes MAMP (Patrones Moleculares Asociados a Microorganismos, MAMP por sus siglas en inglés). Este reconocimiento activa el primer mecanismo de respuesta conocido como inmunidad activada por MAMP (MTI, por sus siglas en inglés). Sin embargo, un alto número de microorganismos comensales, así como patógenos, pueden evadir este sistema produciendo moléculas llamadas: efectoras. Entonces, la planta responde utilizando receptores pertenecientes a la familia de proteínas NLR (Repetición rica en leucina de unión a nucleótidos o Nucleotide-binding Leucine-rich Repeat) y funcionan como sensores intracelulares que reconocen la presencia de proteínas efectoras específicas. Esto desencadena una segunda respuesta conocida como: inmunidad activada por efectores (ETI, por sus siglas en inglés) (Garrido-Oter et al., 2018; Ayliffe y Sørensen, 2019; Teixeira, Colaianni, Fitzpatrick y Dangl, 2019). En este punto, es la interacción entre los microorganismos y la respuesta inmunitaria de la planta la que da forma al ensamblaje del microbioma (Yu et al., 2019).
Desarrollo del Tema
Estudios recientes indican que, el microbioma vegetal es dinámico y responde a la presencia de patógenos y plagas, lo que respalda la hipótesis de que las plantas seleccionan activamente comensales protectores para combatir enfermedades en determinadas circunstancias (Teixeira et al., 2019). Sin embargo, el reconocimiento por parte de la planta a los microorganismos comensales de los patógenos para formar el microbioma indica un área de mayor interés entre los investigadores.
La rizósfera y el suelo
Las plantas sirven como ricas fuentes de nutrientes para diferentes microorganismos que viven dentro y alrededor de sus tejidos, o en el suelo (Pausch y Kuzyakov, 2018). Los microorganismos que forman el microbioma de las plantas pueden habitar en diferentes ambientes vegetales: las raíces (rizósfera; desde la superficie de la raíz hasta el suelo inmediatamente adyacente a la raíz), hoja (filósfera), semilla (espermósfera) e interna (endósfera) (Levy, Conway, Dangl y Woyke, 2018). Las plantas liberan compuestos orgánicos en el suelo, tales como: carbohidratos, aminoácidos (exudados), metabolitos secundarios, entre otros, que contribuyen a la acumulación de carbono derivado de las plantas a la biomasa microbiana circundante, lo que permite la acumulación de bacterias del suelo y en la rizósfera (Pausch y Kuzyakov, 2018). Así mismo, la producción de exudados por parte de la raíz puede variar según la especie, teniendo un papel importante en el establecimiento de la comunidad microbiana que habita la rizósfera (Garcia-Lemos et al., 2019). Estos compuestos pueden modular activamente los rasgos fisiológicos bacterianos, como la formación de biopelículas y la biosíntesis de metabolitos secundarios (Brescia et al., 2020).
El suelo contiene la diversidad microbiana más alta reportada en la tierra. Estos microorganismos son atraídos a distintas superficies de las plantas, como por ejemplo las raíces, por el microambiente rico en nutrientes. Lo anterior sirve como un recurso para las raíces para el reclutamiento de comunidades microbianas específicas para la rizósfera. Por otra parte, se necesita una comunicación química mediada por una diversa gama de moléculas de señalización para que estas comunidades microbianas en la rizósfera formen redes complejas. Estas interacciones se establecen, regulan o inhiben mediante la producción y percepción de compuestos físicos y químicos (Leach et al., 2017). Uno de los mecanismos por los que se produce esta comunicación es a través del denominado Quorum Sensing (QS). El QS en bacterias puede describirse como: un mecanismo de comunicación intra o interespecífico, en el que intervienen mediadores químicos (llamados autoinductores) capaces de inducir respuestas dependientes de la densidad de la población (Dos Santos Lima et al., 2019). El QS en bacterias constan de dos componentes principales: una proteína receptora y un autoinductor. La unión del autoinductor a su receptor activa el gen diana, que luego realiza la función correspondiente en bacterias (Yi, Dong, Grenier, Wang y Wang, 2021). Muchas bacterias Gram negativas utilizan autoinductores como las N-acil-homoserina lactonas (AHL) para controlar la expresión génica (Dos Santos Lima et al., 2019). El QS en estas bacterias suele ser un sistema de dos componentes integrado por una sintasa (LuxI), responsable de la producción de las moléculas autoinductoras, y un receptor sensor (LuxR), responsable de la detección de las moléculas en el ambiente (Barriuso y Martínez, 2018). Las bacterias Gram-positivas utilizan regularmente un pequeño péptido como molécula señal. Por lo general, este sistema consta de un conjunto de genes codificados en un casete (Barriuso y Martínez, 2018). El QS juega un papel importante tanto en los microorganismos patógenos como en casi todos los microorganismos ya que controla diferentes procesos, como la formación de biopelículas, los factores de virulencia y la adhesión, entre otros (Yi et al., 2021).
Biopelículas microbianas en la rizósfera
La rizósfera es un microhábitat crucial para los procesos de producción agrícola. Sin embargo, la composición y las funciones de las biopelículas bacterianas en la rizósfera son poco conocidas (Bogino, Abod, Nievas y Giordano, 2013). Las raíces de las plantas proporcionan uno de los entornos más ricos debido a los exudados producidos para reclutar el microbioma que habita en la rizósfera (Al-Ali et al., 2018). Los exudados de las raíces sirven como un importante factor responsable de desencadenar la colonización de las raíces y la formación de biopelículas. Se estima que las plantas secretan alrededor del 20% de sus fotosintatos como exudados de raíces (Bogino et al., 2013; Rudrappa, Biedrzycki y Bais, 2008; Poole, 2017). De forma general, la idea más aceptada de cómo ocurre la colonización de las raíces es: 1) las plantas producen exudados en las raíces para reclutar microorganismos, 2) diferentes especies de bacterias del suelo con la capacidad de percibir los exudados vía quimiotaxis se mueven hacia el lugar para colonizar la raíz, 3) esos microorganismos usan el carbón y fuentes de nitrógeno proporcionadas por los exudados de las raíces, 4) comienza la respuesta de la planta, 5) comienzan las interacciones bacteria-planta, y 6) las bacterias se adhieren y forman una biopelícula en la superficie de la raíz (Figura 2), (Al-Ali et al., 2018).
Figura 2: Formación de biopelículas en las raíces de las plantas. 1) Las plantas producen exudados de raíces para reclutar a los microorganismos del suelo, 2-3) diferentes especies de bacterias con la capacidad de percibir exudados a través de la quimiotaxis se mueven hacia el lugar para colonizar, 4) las bacterias llegan a la raíz de la planta y la planta responde (interacción bacteria-planta), 5) las bacterias se adhieren a la superficie de la raíz, 6) división celular y formación de microcolonias, 7) formación de biopelículas maduras, 8) formación de biopelículas multiespecies y 9) dispersión de bacterias para colonizar nuevas raíces.
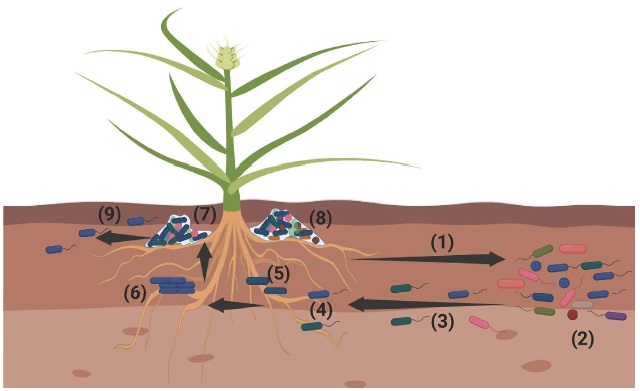
Figure 2: Biofilm formation in plant roots. 1) Plants produce root exudates to recruit soil microorganisms, 2-3) different species of bacteria with the ability to perceive exudates via chemotaxis move toward the place to colonize, 4) bacteria arrive to plant root and plant response (bacteria-plant interaction), 5) bacteria adhere at the root surface, 6) cell division and formation of microcolonies, 7) formation of mature biofilms, 8) multi-species biofilms formation, and 9) dispersion of bacteria to colonize new roots.
La formación de las biopelículas es un proceso importante que representa la base de la colonización de las raíces y las comunidades de agregados en la superficie de las partículas del suelo por parte de las rizobacterias (Tan et al., 2013; Al-Ali et al., 2018; Koua, N’golo, Alloue-Boraud, Konan, y Dje, 2020). Las bacterias y en general los microorganismos, normalmente viven en comunidades multiespecies con patrones de organización espacial denominadas biopelículas, donde ocurren diferentes interacciones (Liu et al., 2016). Una ventaja importante del estilo de vida del biofilm para las bacterias del suelo es la protección contra el estrés abiótico (Bogino et al., 2013). Las biopelículas son agregados de microorganismos incrustados en una matriz extracelular y adheridos a superficies vivas o inertes, donde exhiben un fenotipo diferente al de su contraparte planctónica. Por ejemplo, más del 50% de los metabolitos detectados fueron producidos diferencialmente por cuatro cepas de Pseudomonas en condiciones de biopelícula con respecto al planctónico (Rieusset et al., 2020). Las biopelículas formadas en las raíces de las plantas juegan un papel importante en el mantenimiento de un ambiente óptimo en la rizósfera para apoyar el crecimiento de las plantas (Ganin et al., 2019; Rieusset et al., 2020).
Las biopelículas microbianas están formadas por células incrustadas en una matriz extracelular producida por ellas mismas, que puede estar compuesta por ADN, exopolisacáridos, proteínas y otros compuestos, que generalmente se conocen como sustancias poliméricas extracelulares (EPS) (Dominiak, Nielsen y Nielsen, 2011). Por ejemplo, al igual que en las biopelículas producidas por patógenos como Pseudomonas aeruginosa, el papel esencial del ADN extracelular es mantener la masa y la estructura en la matriz extracelular de las biopelículas formadas en las raíces de las plantas (Wang, Yu, Dorosky, Pierson III y Pierson, 2016). Asimismo, la producción de exopolisacáridos es necesaria para la formación de biopelículas en las raíces y de manera general. Por ejemplo, se ha visto que mutantes defectuosos para la producción de exopolisacáridos en Bacillus velezensis no pudieron formar biopelículas en raíces de tomate (Al-Ali et al., 2018). Por otra parte, la expresión génica de las sustancias poliméricas extracelulares (EPS por sus siglas en inglés) y la formación de biopelículas dependen de las condiciones ambientales. En B. subtilis, la fosforilación del regulador de respuesta Spo0A es fundamental para la formación de biopelículas. Spo0A-P gobierna la vía reguladora para la expresión del gen EPS al controlar la actividad del regulador maestro SinR (Ganin et al., 2019). En otro ejemplo, las rizobacterias producen una serie de exopolisacáridos específicos de la cepa que ayuda en la adherencia y durante el inicio de la simbiosis, aumentando la nodulación y la formación de biopelículas. Las rizobacterias también producen celulosa bacteriana. La celulosa está involucrada en las etapas secundarias de unión a las raíces de las plantas y maduración de la biopelícula (Augimeri, Varley y Strap, 2015).
Azospirillum brasilense, una bacteria que forma parte del rizobioma de diferentes plantas, forma biopelículas en las raíces de su planta huésped. Esta biopelícula tiene una matriz extracelular compuesta de flagelina, exopolisacáridos, proteína de membrana externa (OmaA) y ADN extracelular (eDNA) (Ramírez-Mata, Pacheco, Moreno, Xiqui y Baca, 2018; Viruega-Góngora, Acatitla, Reyes, Baca y Ramírez, 2020). El primer paso en la formación de la biopelícula de A. brasilense está dado por la adhesión a la raíz por parte del flagelo polar y la proteína de membrana externa (OmaA). Posteriormente, se da el inicio de la producción de exopolisacáridos y eDNA, que formarán la matriz extracelular cuando la biopelícula esté madura. Finalmente, después de 7 días, la biopelícula comienza a declinar, lo que marca el comienzo de las últimas etapas de formación de la biopelícula que conduce al desprendimiento y la dispersión (Viruega-Góngora et al., 2020).
En otras especies, como las del género Bacillus, la matriz extracelular de la biopelícula se compone, principalmente, de un esqueleto básico de fibras amiloides que consisten en una proteína llamada: TasA, un exopolisacárido y proteína BslA (Zhou et al., 2016). Así mismo, la localización correcta de la proteína TasA depende de las actividades de las proteínas SipW e YqxM (Xu et al., 2014). TasA y el exopolisacárido γ-poliglutamato (γ-PGA), juegan un papel complejo en la formación de biopelículas en diferentes cepas de B. subtilis. Asimismo, la producción de lipopéptidos (LP), como la surfactina y las fengycinas, son necesarias para la formación de biopelículas en diferentes especies de Bacillus (Zhou et al., 2016).
La producción de moléculas antimicrobianas en biopelículas de la rizósfera
Las interacciones entre bacterias y raíces son fundamentales para el ecosistema terrestre (Poole, 2017). En la naturaleza, las plantas viven en un ambiente rico en microbios y deben interactuar con microbios patógenos, comensales y beneficiosos. Un mecanismo mediante el cual las plantas aprovechan las funciones beneficiosas proporcionadas por los microbios y, al mismo tiempo, combaten los patógenos microbianos, es a través de la producción de moléculas antimicrobianas como los antibióticos, producidos por la planta u otro microorganismo que habita en la rizósfera para eliminar competidores estrechamente relacionados (Cheng, Zhang y He, 2019).
Las especies del género Pseudomonas pueden producir varios productos con actividad antimicrobiana que pueden proteger a las plantas contra patógenos cuando forman biopelículas, ejemplos: 2.4-diacetilfloroglucinol (DAPG), fenazinas, pirrolnitrina o alquilresorcinol (Rieusset et al., 2020). Pseudomonas chlororaphis puede producir fenazinas. Este compuesto es capaz de inhibir el crecimiento del patógeno Gaeumannomyces graminis var. tritici en trigo. Las fenazinas comprenden un gran grupo de compuestos heterocíclicos que contienen nitrógeno que son sintetizados solo por bacterias que habitan en la rizósfera, principalmente por especies pertenecientes a los géneros Pseudomonas y Streptomyces. Asimismo, la producción de fenazina promueven la liberación de ADN extracelular, lo que se correlaciona con la producción de una matriz más estructurada en la biopelícula (Wang et al., 2016).
Otra molécula antimicrobiana producida por bacterias del género Pseudomonas son las tailocinas. Las tailocinas son bacteriocinas de alto peso molecular producidas por una variedad de bacterias, que se asemejan a las colas de los bacteriófagos. Las fibras de las tailocinas tipo R interactúan con receptores de superficie específicos (lipopolisacáridos) de competidores bacterianos susceptibles e insertan un núcleo similar a una aguja a través de la pared celular. Esto despolariza el potencial de membrana y detiene la síntesis de macromoléculas, lo que lleva a la muerte celular (Dorosky, Yu, Pierson y Pierson III, 2017). Dorosky et al. (2017) muestran que P. chlororaphis produce dos tipos de R-tailocinas que solo inhibían el crecimiento de otras especies de Pseudomonas. Además, también señalan que la producción de estas dos moléculas permitió una ventaja en biopelículas adheridas a la superficie contra P. putida. Así mismo, se observó que la actividad de la tailocina no influye en la capacidad de P. chlororaphis para colonizar la rizósfera en suelos con poblaciones microbianas nativas. En otro estudio, Schoenborn, Clapper, Eckshtain y Shank (2021) encontraron que P. fluorescens Pf-5 secreta ciertos metabolitos que son capaces de reducir, significativamente, la adhesión y la formación de biopelículas del patógeno Listeria monocytogenes en raíces de Arabidopsis thaliana.
Por otro lado, las bacterias del género Bacillus, encontradas normalmente en suelos y en la rizósfera, son capaces de producir diferentes compuestos que tienen actividad antimicrobiana y que además están involucrados en la formación de la biopelícula, como las familias de las surfactinas, iturinas y fengicinas. Por ejemplo, las fengicinas y las iturinas tienen actividad antifúngica e inhiben el crecimiento de varios patógenos de plantas. Bacillus atrophaeus produce fengicinas, iturinas y surfactinas con actividades antifúngicas al formar biopelículas. Se conoce que puede proteger a los cultivos de: tomate, lechuga y remolacha azucarera contra la infección por Rhizoctonia solani (Aleti et al., 2016). Así mismo, Zhou et al. (2016) encontraron que el gen gltB en B. subtilis cumple un papel clave en la regulación de la producción de lipopolisacáridos, particularmente, en el bacilomicina L y fengicina, que cumple un papel clave en el control del tizón de la vaina del arroz y en la formación de biopelículas en la planta huésped. Además, la producción de surfactinas es necesaria para el desarrollo de biopelículas y la colonización de raíces en cultivos de melón y lechuga por B. subtilis y B. atrophaeus (Zeriouh, de Vicente, Pérez y Romero, 2014; Aleti et al., 2016). Por otra parte, las surfactinas tienen efectos sinérgicos sobre la actividad biológica de las iturinas y son necesarias para la colonización de la rizósfera y la formación de biopelículas por B. velezensis (Aleti et al., 2016).
Chen et al. (2013) descubrieron en una evaluación de 60 aislamientos recolectados en varios lugares de China, que la producción de matriz extracelular es fundamental en B. subtilis para la colonización bacteriana de las superficies de las raíces de las plantas. Asimismo, la protección por parte de estas cepas a su planta huésped requería de varios genes involucrados en la formación de biopelículas, principalmente en la formación de la matriz extracelular (Chen et al., 2013). Recientemente, Chen et al. (2021) secuenciaron el genoma de B. subtilis BS-Z15 debido a su capacidad inhibitoria frente al hongo Verticillium dahliae. Los autores encontraron que este aislado es capaz de sintetizar, principalmente, dos compuestos con actividad antifúngica: la micosubtilina y bacilibactina. Asimismo, los autores reportan varios genes que codifican productos relacionados con la producción de biopelículas, lo que puede estar relacionado con la capacidad de colonización de la cepa en la rizósferas (Chen et al., 2021).
Finalmente, en el género Bacillus, se ha encontrado que el regulador de respuesta DegU regula: la competencia genética, la motilidad de enjambre, la formación de biopelículas, la arquitectura compleja de colonias y la producción de proteasas. Además, su sobreexpresión produce una arquitectura de colonia compleja, formación de biopelículas, actividades de colonización, producción de los antibióticos bacilomicina D y dificidina, y eficacia del control biológico de la enfermedad del marchitamiento por Fusarium (Xu et al., 2014).
Los microorganismos del género Lysobacter, habitantes comunes de la rizósfera vegetal, tienen la acción de inhibir el crecimiento de microorganismos patógenos de plantas cuando forman biopelículas en las raíces de la planta huésped. Dentro de los patógenos a los cuales inhibe su crecimiento se encuentran: Ralstonia solanacearum, Rhizoctonia solani y Streptomyces spp. a través de la producción de metabolitos secundarios pertenecientes a la familia de las macrolactamas de tetramato policíclico, ejemplos: xantobacinos A, B y C, y dihidromaltofilina, éste último también llamado Factor Antifúngico Estable al Calor (HSAF) (Brescia et al., 2020). Además, ciertos compuestos producidos por bacterias del género Lysobacter poseen acción contra ciertos tipos de carcinoma humano (alteramida A, lisobacteramida A y B (Xu et al., 2014) y contra bacterias patógenas humanas (lipodepsipéptidos WAP-8294A), (Brescia et al., 2020).
Aunque los exudados de las raíces pueden atraer una amplia variedad de bacterias, la colonización de las plantas por bacterias endófitas es un proceso complejo que se puede dividir en cinco etapas distintas: a) reconocimiento de los exudados de las raíces y la motilidad hacia la planta por parte de las bacterias, b) adhesión a la superficie de las raíces , c) formación de biopelículas, d) penetración en la superficie de la raíz, y e) colonización de las partes internas de una planta (Pinski, Betekhtin, Hupert-Kocurek, Mur y Hasterok, 2019). Cada vez hay más pruebas de que las bacterias endofíticas son una fuente potencial para el descubrimiento de nuevos compuestos antimicrobianos; varios metabolitos producidos por estas bacterias actúan como agentes antimicrobianos contra patógenos humanos y vegetales (Christina, Christapher y Bhore, 2013). La diversidad de bacterias endófitas puede variar con el tipo de especie vegetal hospedante. Se ha observado, generalmente, que en aislados predomina el phylum Proteobacteria y a nivel de las clases α-, β-, y γ-Proteobacteria. Esta última es la más diversa. Con menor frecuencia podemos encontrar a las clases Acidobacteria, Planctomycetes y Verrucomicrobia. Los géneros bacterianos más comúnmente aislados son: Bacillus, Burkholderia, Microbacterium, Micrococcus, Pantoea, Pseudomonas y Stenotrophomonas, donde Bacillus y Pseudomonas son los géneros predominantes (Afzal, Shinwari, Sikandar y Shahzad, 2019). Los Actinomycetes endófitos (bacterias Gram-positivas), donde encontramos a los géneros: Actinomyces, Actinoplanes, Amycolatopsis, Micromonospora, Saccharopolyspora y Streptomyces, son reconocidos como productores de metabolitos bioactivos con actividad antimicrobiana (Ek-Ramos et al., 2019). Las cepas de Streptomyces aisladas de varias plantas han producido kakadumicinas y xiamicinas que tienen fuertes actividades antimicrobianas (Christina et al., 2013; Jiang et al., 2018).
En los últimos años, se ha visto que bacterias endófitas pueden ser agentes de biocontrol en la agricultura. Por ejemplo: Pseudomonas brassicacearum aislado como un endófito de las raíces de Artemisia sp. y Bacillus velezensis PCSE10 aislados de Piper colubrinum, demostraron controlar al patógeno Phytophthora capsici, causante de la pudrición por tizón en varios cultivos de importancia comercial (Chung et al., 2008; Kollakkodan, Anith y Nysanth, 2021).
Conclusiones
El papel del microbioma bacteriano vegetal en la salud del huésped y su capacidad de producir compuestos bioactivos para contrarrestar patógenos vegetales y humanos genera la necesidad de comprender cómo se forma este microbioma. Conocer cuales factores activan la síntesis de estos metabolitos secundarios y cómo se relacionan con los complejos mecanismos de la planta hospedante, especialmente el localizado en la rizósfera. Debido a que el suelo es la mayor fuente de esta diversidad microbiana, sumado a la capacidad de las bacterias para comunicarse, formar biopelículas y contrarrestar los mecanismos de defensa de las plantas, es necesario un estudio integrador y multidisciplinario para contrarrestar el vacío que aún existe en este campo.











 nueva página del texto (beta)
nueva página del texto (beta)