Introducción
La necrosis pancreática infecciosa (NPI) es una de las enfermedades infecciosas más importantes en la industria del salmón, debido a que causa alta mortalidad en crías de primera alimentación; a nivel mundial fue identificada en el año 1960. Wolf, Dunbar y Snieszko (1981) Sin embargo en México se identificó hasta el año 2000 en granjas de truchas arcoíris (Oncorhynchus mykiss) que importaban huevos oculados de Estados Unidos de América (Ortega et al., 2002; Ortega, Vega y Enriquez, 2007).
El agente etiológico de esta enfermedad es el virus de la necrosis pancreática infecciosa (VNPI), un miembro de la familia Birnaviridae, género Aquabirnavirus, de aproximadamente un diámetro de 60 nm, tiene un genoma de ARN de doble hebra organizado en dos segmentos. El segmento A mide aproximadamente 3097 Pb y contiene dos marcos abiertos de lectura (ORF´s del inglés Open Reading Frames) Duncan, Mason, Nagy, Leong y Dobos, (1991), Barrera-Mejía, Martínez, Ortega y Ulloa, (2011). El ORF menor codifica una proteína de 17 kDa rica en argininas, no estructural (NS) denominada VP5; Magyar y Dobos (1994) y el ORF mayor codifica una poliproteína NH2 pVP2-NS (VP4)-VP3-COOH, la cual en su proceso de maduración es fragmentada por la proteasa VP4 dando origen a las proteínas VP2, VP3 y VP4.
El genoma del segmento B mide aproximadamente 2783 Pb y codifica una ARN polimerasa dependiente de ARN llamada VP1 (RdRp del inglés RNA-dependent RNA polymerase) Dobos (1995), Duncan y Dobos (1986), pero también puede dar origen a una proteína de 94 kDa llamada VPg, que está unida a través de enlaces covalentes al extremo 5´ de los segmentos A and B. (Calvert, Nagy, Soler y Dobos, 1991).
La cápside del VNPI está formada externamente por la proteína VP2, mientras que el interior por la proteína VP3 de 31 kDa, y junto con la VP1 forman el núcleo de ribonucloproteinas (Petit, Lejal, Huet y Delmas, 2000).
En el gen que codifica para la VP2 está la región del genoma del virus donde se han identificado la mayoría de polimorfismos de nucleótidos. Bain, Gregory y Raynard (2007), Hvarstein, Kalland, Christie y Endresen (1990), Santi, Vakharia y Evensen (2004) En una región hipervariable entre los residuos 243 y 335 de la proteína VP2 se encuentran los epítopos responsables para el reconocimiento antigénico, esta región de la proteína es importante porque determina la especificidad de la neutralización por unión de anticuerpos, por lo tanto, esta región es importante para inducir inmunidad y protección a los animales. (Blake et al., 1995).
También ha sido propuesto que la virulencia del VNPI podría estar relacionada al segmento A en la proteína VP2, Sano, Okamoto, Fukuda, Saneyoshi y Sano (1992) por lo tanto, las cepas virulentas poseen Treonina en la posición 217 y Alanina en la posición 221, Treonina o Alanina en la posición 247 y Tirosina o Histidina en la posición 500, mientras que las cepas de mediana virulencia tienen Prolina 217 y Alanina en 221, y las de baja virulencia tienen Treonina 221 independientemente del residuo en la posición 217. Blake et al. (1995), Bruslind y Reno (2000), Santi et al. (2004), posteriormente Song, Santi, Evensen y Vakharia (2005) observaron que después de cuatro pasajes del VNPI en células CHSE-214, la VP2 presenta un cambio de aminoácido en el residuo 221 de Alanina por Treonina, pero no cuando se utilizan células RTG-2, la cual está asociada con atenuación del virus debido a su adaptación.
Debido a la existencia de diversos aislados del VNPI distribuidos a nivel mundial, diferentes clasificaciones de este aislado han sido propuestas, la primera de estas se realizó utilizando los serotipos del virus, en esta clasificación Hill y Way (1995) proponen dos serogrupos, el serogrupo A con nueve serotipos A1 (West Buxton), A2 (Spajarup), A3 (Abild), A4 (Hecht), A5 (Tellina), A6 (Canadá 1), A7 (Canadá 2), A8 (Canadá 3) and A9 (Jasper), el serotipo con mayor frecuencia en los Estados Unidos de América es el A1, en Canadá los serotipos A6, A7, A8 y A9, y en Europa el serotipo A2 y A3.
El serogrupo B tiene un solo serotipo B1 (TV-1). Con relación a la clasificación genómica de los Aquabirnavirus varios autores han contribuido a la clasificación de los aislados del VNPI. Bain et al. (2007), Blake et al. (1995), Cutrín et al. (2004), Hvarstein et al. (1990), Lee, Blake, Singer y Nicholson (1996) y Nishizawa, Kinoshita y Yoshimizu (2005), sin embargo esta clasificación por genogrupo no siempre coincide con los serotipos propuestos arriba. Hill y Way (1995) Blake et al. (1995), propusieron seis genogrupos, en el genogrupo I fueron incluidos los aislados de Estados Unidos de América (A1) y dos aislados de Canadá (A9), En el genogrupo II se incluyen los aislados de Asia y Europa (A3), En el genogrupo III se consideran dos aislados de Canadá (C1 and ASV) (A6) y el aislado de Europa Te (A5), El genogrupo IV incluye los aislados C2 (A7) and C3 (A8) de Canadá, El genogrupo V contiene cinco aislados de Europa y uno de Asia ( A2) y el genogrupo VI contiene el aislado He (A4). En adición a este genogrupo VI, Nishizawa et al. (2005) propone la inclusión de un genogrupo VII, que contienen los aislados encontrados en Japón y Corea.
En México el primer aislado de VNPI fue obtenido en el año 2000 Actualmente, solo secuencias parciales del genoma de este aislado han sido reportado Ortega et al. (2002). En este estudio fue secuenciado el genoma completo de este primer aislado identificado en nuestro país; utilizando esta secuencia genómica, se realizó una caracterización molecular de este prototipo del VNPI aislado mexicano, y se determinó sus relaciones genéticas con otros aislados a nivel mundial y, por lo tanto, su origen genético.
Material y Métodos
Cultivo viral
Células CHSE-214 (Chinook Salmon Embryo) fueron utilizadas para el aislamiento y propagación del VNPI. El crecimiento de las células se realizó a 20 °C en medio MEM (Earle’s Minimum Essential) suplementado con 10% de Suero fetal bovino, 400 UI mL-1 penicilina y 400 mg mL-1 de estreptomicina. El sobrenadante fue obtenido de las placas y posteriormente centrifugado a 3500 rpm durante 10 min a 10 °C, el sobrenadante de esta centrifugación conteniendo el virus fue depositado en crioviales con 10% de suero fetal bovino a -85 °C hasta su uso.
Extracción del ARN viral y RT-PCR
La extracción del ARN viral fue realizada utilizando 250 mL del sobrenadante del medio de cultivo que contenía al virus, el procedimiento fue realizado utilizando la técnica de Trizol, Siguiendo las instrucciones del fabricante; una vez obtenido el ARN fue eluido en 10 mL agua DEPC (diethylpyrocarbonate 1%), e inmediatamente fue realizado el procedimiento de RT-PCR para amplificación de las regiones genómicas correspondientes, esta técnica fue realizada utilizando el paquete Access Quick RT-PCR System, como se describe a continuación: fueron adicionados 25 mL de la reacción buffer 2X, 1 mL (10 mM) de los iniciadores forward y reverse, 17 mL de agua DEPC, 1 mL de la enzima de (5 U) y 5 mL (5 ng) de ARN.
Secuenciación del genoma del VNPI (prototipo aislado mexicano)
La amplificación del genoma completo del primer aislado mexicano reportado por Ortega et al. (2002) se realizó utilizando siete pares de iniciadores, seleccionados para amplificar fragmentos sobrelapados, los nombres y las características de amplificación de estos iniciadores están indicados en la Cuadro 1.
Table 1: Primers for the amplification of the entire genome of the Mexican NPV.
| Nombre | Secuencia | Posición | Tamaño |
| bp | |||
| A-A5´NCa A-ApaR*a | 5´-GGAAAGAGAGTTTCAACG- 3´ 5´-CCATCGCACCAAGGAGGTAT- 3 | 1-18 711-731 | 731 |
| A-ApaF*a A-Sala | 5´-GGTCAACAATCAACTAGTGACC- 3´ 5´-GCATCACACCTGATTCAG 3´ | 571-593 2129-2146 | 1576 |
| A-NS-Fb A-3´NC-Rb | 5´-GCATACCCGGAACTTCTTGA- 3´ 5´-ACTGATCCCCTGGCCTTAAC- 3´ | 1818-1837 3057-3076 | 1259 |
| B-B5´NCa B-HindR*a | 5´-GGAAACAGTGGGTCAACG-3´ 5´-GAAGGTGAGTTGCTTCAGAAGTG-3´ | 1-18 477-499 | 500 |
| B-HindF*a B-PstRa | 5´-GGAAGACGGCAAGCTTAAGGACAC-3´ 5´-GTGTTGTCCTGCAGTATGTAGATG-3´ | 352-375 1267-1290 | 938 |
| B-PstF*a B-BstR*a | 5´-AGAGACAGCCTGGACAA-3´ 5´-GAGTTTGGTCCTCTGGTCTAG-3´ | 1217-1233 2285-2305 | 1072 |
| B-SmaDF*a B-Sma3´NCa | 5´-AGAAAACCCGGAGCCGAGATTG-3´ 5´-GGGGTCCCTGGCGGAACCGGATGT-3´ | 1898-1919 2761-2784 | 886 |
| Pr A2c | 5´-GACAGGATCATCTTGGCATAGT-3´ | 1309-1330 | 760 |
*Iniciadores modificados, los nucleótidos modificados fueron remarcados con negritas. aIniciadores previamente reportados por Yao y Vakharia (1998); bIniciadores diseñados para amplificar un producto de PCR de 1259 Pb del 3´ RNC de la poliproteína; cIniciadores previamente reportados por Blake et al. (2001).
*Modified initiators, modified nucleotides were highlighted in bold. aInitiators previously reported by Yao and Vakharia (1998); bInitiators designed to amplify a 1259 Pb PCR product of the 3' RNC of the polyprotein; cPrimers previously reported by Blake et al. (2001).
Clonación y secuenciación de los fragmentos amplificados. Los amplicones de las RT-PCR fueron purificados de los geles de agarosa con el paquete (QiaQuick Purification Kit) y fueron clonados en un vector comercial p-GEM T, posteriormente se obtuvo un purificado de 50 ng del plásmido recombinante y este fue secuenciado utilizando el Sistema Big Dye Terminator kit v.3.1 Trade Cycle Sequencing. El análisis de las secuencias fue realizado utilizando el secuenciador Genetic Analyzer 3 100. Las secuencias obtenidas fueron editadas y alineadas con el programa Mega X, Kumar, Stecher, Li, Knyaz y Tamura (2018). Para obtener la edición del genoma completo del VNPI los fragmentos secuenciados fueron concatenados y comparados a un total de 24 secuencias de genomas completos de otros aislados a nivel mundial, las secuencias de estos aislados fueron obtenidos del banco de genes del NCBI (GenBank) (Cuadro 2).
Table 2: Sequences used in the phylogenetic tree with both segments of the VNPI genome.
| Nombre | Origen Geográfico | Número de acceso al GenBank | |
| Segmento A | Segmento B | ||
| West Buxton | Estados Unidos | AF078668 | AF078669 |
| JasperD | Canadá | M18049 | M58756 |
| 20G1d | Canadá | AY780924 | AY780931 |
| 19G7e | Canadá | AY780923 | AY780930 |
| 17F2 | Canadá | AY780920 | AY780927 |
| 19G3e | Canadá | AY780922 | AY780929 |
| 5B1d | Canadá | AY780918 | AY780925 |
| 19F3b | Canadá | AY780921 | AY780928 |
| 6B1a | Canadá | AY780919 | AY780926 |
| México | México | JX174178 | EU665685 |
| AM-98 | Japón | AY283780 | AY129664 |
| 2310 | España | AJ489225 | AJ489241 |
| DRT | Corea | D26526 | D26527 |
| H1 | Japón | D61386 | AY129665 |
| AY-98 | Japón | AY283785 | AY123970 |
| 2284 | España | AJ489223 | AJ489239 |
| 3175 | Francia | AJ622822 | AJ622823 |
| NVI 016 | Noruega | AY379742 | AY379743 |
| 88R | España | AJ489229 | AJ489245 |
| Sp116 | Noruega | AY354520 | AY354523 |
| Sp103 | Noruega | AY354519 | AY354522 |
| NVI 013 | Noruega | AY379738 | AY379739 |
| Sp122 | Noruega | AY354521 | AY354524 |
| NVI-015 | Noruega | AY379740 | AY379741 |
Análisis filogenéticos. Los árboles filogenéticos fueron construidos con el método Neighbour-joining. Saitou y Nei (1987), y fueron utilizadas 1000 repeticiones de bootstrap para la confiabilidad. Una red filogenética fue construida utilizando el algoritmo (median joining) con el software Network 4.6 (Bandelt, Forster y Röhl, 1999).
Virulencia de los aislados mexicanos utilizando identificación molecular. La identificación de los residuos 217 and 221 fue realizada obteniendo la secuencia de aminoácidos de la proteína VP2, la clasificación fue realizada como fue reportada previamente en diferentes bibliografías. (Blake et al., 1995; Bruslind y Reno, 2000; Santi et al., 2004).
Resultados y Discusión
Análisis genómico del aislado mexicano
Los resultados derivados del análisis aportan evidencia de que el genoma completo del primer aislado mexicano del VNPI tiene la siguiente composición de nucleótidos: timina 16%, citocina 29.2%, adenina 29.6% y guanina 25.2%. El segmento A del primer aislado mexicano del VNPI mide 3076 Pb y está organizado de la siguiente manera: un fragmento de máximo 67 nucleótidos localizados en la región no codificante 5’, posteriormente se encuentra el ORF menor formado por 68 a 511 nucleótidos, que codifica a la proteína VP5 de 148 aminoácidos. El ORF mayor esta sobre puesto con el ORF menor, y por lo tanto comienza en el nucleótido 120 y termina en el nucleótido 3035, el ORF mayor está organizado por tres genes: el gen que codifica la proteína preVP2, la cual comienza en el nucleótido 120 al 1592, el gen para la proteína VP4 que comienza en el nucleótido 1629 y termina en el 2294 y el gen para la VP3 comienza en el nucleótido 2328 al 3035; y finalmente está la región no codificante 3’ del nucleótido 3036 al 3076. Los tres genes de este ORF codifican una sola poliproteína de 972 aminoácidos, la cual es fragmentada después de su traducción. Los sitios de rompimiento para separar pVP2 de VP4 en el aislado mexicano fueron identificados entre los residuos 508-509 y los sitios para separar a VP3 de VP4 fueron encontrados entre los residuos 734-735. En la poliproteína fueron identificados seis potenciales sitios de N-glicosilación, cuatro en la VP2 y dos en la VP4 en los siguientes nucleótidos: 73-75, 118-120, 234-236, 254-256, 584-586 y 698-700.
El segmento B tiene un ORF de 2535 nucleótidos, comenzando en el 100 y terminando en el 2635 codifica una proteína de 845 aminoácidos. En la secuencia de aminoácidos fueron encontrados seis sitios de N-glicosilación, 7 sitio de fosforilación y un sitio tirosina fosforilación y los siguientes motivos GLPYIGKT, DLEKGE, SGNAFTFLNN, LKN y R, en los residuos 248-255, 402-407, 471-480, 521-523 y 569 respectivamente.
Caracterización genética del aislado mexicano
El genoma completo del aislado mexicano fue comparado con genomas completos de otros 23 aislados del VNPI obtenidos de diferentes países. Utilizando estas 24 secuencias fue construido un árbol filogenético. La topología del árbol se dividio en cinco genogrupos. Solo cuatro de estos genogrupos fueron organizados de acuerdo con la clasificación de Blake et al. (1995) y Nishizawa et al. (2005). El genogrupo I tiene dos principales ramas, una contiene los aislados 19G7e, 17F2, 19G3e, 5B1d, 19F3e y West Buxton, y la otra los aislados mexicanos y los aislados AM-98, 2310, DRT y Jasper. En el genogrupo III se encuentran los aislados 2284, 578 y 6B1a, El genogrupo V tiene a los aislados 3175, NVI 016, 88R, SP116, SP 103, AMP 013, AMP 122 y Sp 015, El genogrupo VII los asilados H-1 and AY-98. Los aislados 20G1d fueron agrupados independientemente, y no fueron caracterizados en algún genogrupo descrito actualmente (Figura 1).
Figura 1: Árbol filogenético de genomas completos de IPNV de diferentes aislados en el mundo. Para la construcción del árbol filogenético se utilizaron 22 secuencias de aislamientos previamente reportadas más la secuencia del aislamiento mexicano prototipo, utilizando el método Neighbour-Joining. La confianza en la construcción del árbol se evaluó utilizando 1000 repeticiones de arranque. La barra de escala de distancia filogenética indica el cambio estimado por nucleótido.
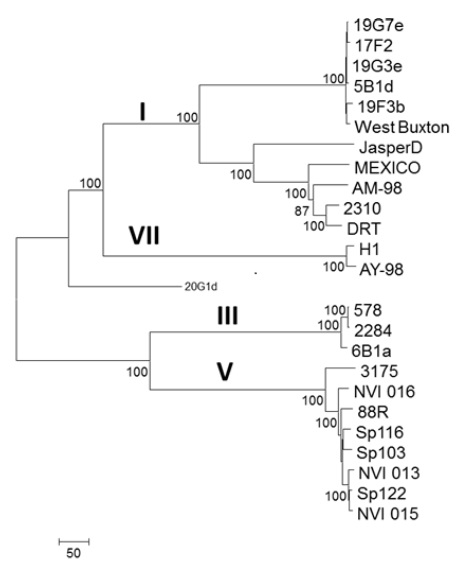
Figure 1: Phylogenetic tree of IPNV complete genomes of different isolates in the world. 22 sequences of isolates previously reported plus the sequence of prototype Mexican isolate were used for constructing the phylogenetic tree, utilizing the method Neighbour-Joining. Confidence on tree construction was assessed using 1000 bootstrap replicates. The phylogenetic distance scale bar indicate estimated change per nucleotide.
En la red filogenética de los 25 aislados, se pudo observar que el aislado 20G1d se agrupo entre los aislados del genogrupo I y genogrupo III, el resto de los aislados fueron organizados de la misma forma como se organizaron en el árbol filogenético (Figura 2).
Figura 2: Red filogenética con los nucleótidos haplotipo 624-1264 del segmento A del genoma del IPNV. La red se fue construyendo con cualquiera de los aislamientos de los siete genogrupos informados previamente. Los aislamientos mexicanos se identificaron con la etiqueta de genotipos mexicanos. El tamaño del círculo es proporcional al número de muestras.
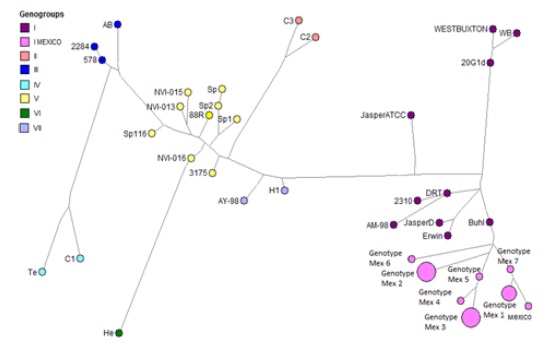
Figure 2: Phylogenetic network with nucleotides haplotype 624-1264 of segment A of IPNV genome. The network was constructing with any isolates of the seven genogroups previously reported. Mexican Isolates were identified with label genotypes Mexican. The size of circle is proportional to number of samples.
Alternativamente, dos árboles filogenéticos fueron construidos, uno con el ORF mayor del segmento A con 44 secuencias completas disponibles en el GenBank y otro con el ORF del segmento B con 27 secuencias completas también disponibles en el GenBank. Aunque ambos árboles mantienen la distribución de los aislados bajo la misma topología, En el árbol del segmento A del ORF mayor aparecen los aislados de los VII genogrupos previamente reportados, Pero en los árboles del segmento B solo se observan cuatro genogrupos, debido a que las secuencias para esta región se han reportado escasamente en el GenBank. Interesantemente, el aislado 20G1d en el árbol filogenético del segmento A fue localizado en el genogrupo I, pero si este mismo aislado se analiza con el segmento B se encuentra separado entre el genogrupo I y el III y forma una rama diferente dentro del árbol filogenético (Datos no presentados).
Comparando las secuencias del ORF mayor del segmento A de los diferentes aislados, los aislados genéticamente más cercanos al asilado mexicano son Buhl con una identidad de nucleótidos del 98.8% y una identidad de aminoácidos del 99.6%, y el más alejado fue He con una identidad de 74.4% nucleótidos y 80.3% en aminoácidos.
Mientras que en el análisis del ORF del segmento B, encontramos que los asilados más cercanos al aislado mexicano fueron el 2310 con una identidad del 98.2% y 98.8% en nucleótidos y aminoácidos, respectivamente, y el más distante fueron el 578 y el 2284, ambos con identidades de 81% y 90% en nucleótidos y aminoácidos, respectivamente.
Identificación de los aminoácidos asociados con la virulencia
En el análisis de los motivos de aminoácidos relacionados a la virulencia que han sido descritos en otros aislados del VNPI, nosotros encontramos que el aislado mexicano tiene el residuo 217 correspondiente a una alanina, en el 221 a una treonina, y en el 247 a un ácido glutámico.
El genoma del VNPI consiste en dos segmentos de ARN de doble hebra, denominados A y B, ambos codifican un total de cinco proteínas VP5, VP2, VP4, VP3 and VP1. La secuencia del segmento A del aislado mexicano tiene 3076 nucleótidos, otros aislados han reportado que su segmento A puede llegar a tener hasta 3098 nucleótidos; La región 3’ no codificante tiene 21 nucleótidos y fue conservada en 15 aislados. Santi et al. (2004), Yao y Vakharia (1998). El ORF menor de este segmento tuvo la misma longitud en todos los aislados disponibles en el GenBank (584 nt), mientras que el ORF mayor vario en 468 nt, dependiendo con el aislado que se comparó.
Los resultados obtenidos demuestran que la poliproteína del aislado mexicano tuvo la mayor identidad con la cepa Buhl y difiriendo solo en cuatro aminoácidos, específicamente tres en VP2 (72, 287, 405) y uno en VP3 (834), en el caso de las secuencias para VP5 estas aún no han sido reportadas para Buhl, por lo tanto, nuestros resultados presentan que la proteína VP5 del aislado mexicano tuvo una alta identidad con la cepa JasperD, la cual pertenece al serotipo A1. (Duncan et al., 1991).
Utilizando las secuencias de los genes VP2, VP3 y VP4 en el aislado mexicano se determinó que este pertenece al serotipo A1 de acuerdo con su identidad con la secuencia de la cepa Buhl. Esto posteriormente fue confirmado en este estudio por sueroneutralización (datos no presentados).
Las secuencias del segmento B obtenidas en este estudio fueron consistentes con las obtenidas previamente por Barrera-Mejía et al. (2011). El segmento es de 2782 nucleótidos con un ORF que codifica una proteína de 845 aminoácidos, similar a West Buxton y JasperD, serotipos reportados por Yao y Vakharia (1998). En la comparación del ORF que codifica para VP1, nosotros encontramos que el aislado mexicano genéticamente está estrechamente relacionado con el aislado 2310 y los más alejados fueron los aislados 578 y 2284, sin embargo, un análisis con He y Buhl no pudo ser realizado, debido a que las secuencias de este segmento no están disponibles en el GenBank para estas cepas.
Con respecto al segmento A, en la proteína VP2 están los residuos 217, 221, 247 y 500 relacionados a la virulencia del VNPI. Blake et al. (1995), Bruslind y Reno (2000) y Santi et al. (2004) en esta posición los aislados virulentos contienen treonina, alanina, treonina/alanina, tirosina/histidina respectivamente; aislados moderadamente virulentos tienen prolina y alanina en los residuos 217 y 221 y los aislados de baja virulencia o avirulentos contienen treonina en los residuos 221, estos residuos fueron analizados en el aislado mexicano para determinar su virulencia y este tuvo alanina en el residuo 217, treonina en el 221, acido glutámico en el 247 y tirosina en el 500; Por lo tanto, el prototipo del aislado mexicano fue clasificado como avirulento.
En el árbol y la red filogenética construidos con las 24 secuencias de los genomas completos de VNPI obtenidas del GenBank, se demostró que el aislado mexicano pertenece al genogrupo I como fue previamente reportado por Ortega et al. (2002), Barrera-Mejía, Simón, Ulloa, Salgado y Soriano (2010), Barrera-Mejía et al. (2011). En la misma rama del árbol filogenético fueron agrupados los aislados AM-98, 2310 and DRT con los cuales el aislado mexicano presentó los valores más altos de identidad genética 97%, 98% and 98% respectivamente, estos aislados habían sido reportados como serotipos americanos aunque los tres fueron aislados en diferentes países (Japón, España y Corea) pero es sabido que estos países importaban huevos-oculados de truchas de los Estados Unidos de América (Cutrín et al., 2004; Chung, Lee, Lee, Lee, y Kim, 1994).
Nuestros datos también confirman que utilizando árboles filogenéticos con genomas completos puede ayudar a identificar aislados del VNPI que han presentado recombinación de segmentos de genes como es el caso del aislado 20G1d que tiene el segmento A americano y el segmento B europeo, así como también el aislado 6B1a. Romero-Brey, Bandín, Cutrín, Vakharia, y Dopazo (2009). La secuenciación del genoma completo en este estudio nos permitió obtener información importante sobre el VNPI aislado en México, por ejemplo, su caracterización genómica y sus relaciones genéticas con otros aislados a nivel mundial.
Conclusiones
Aunque, existe una gran cantidad de reportes de secuencias nucleotídicas, para estudiar la diversidad genética del VNPI a nivel mundial, esta información es poco informativa, debido a que la mayoría de estos trabajos se han realizado con secuencias parciales de los genomas de los aislados virales en cuestión, por otro lado, hay muy pocos reportes de este tipo de estudios donde se utilice la secuencia de genomas completos del VNPI. Este es el primer reporte de la secuencia completa del genoma del VNPI en México.
La información de la secuencia del genoma completo obtenida en este trabajo es muy importante, porque permite entender de una manera más precisa las relaciones genéticas entre los diferentes aislados distribuidos a nivel mundial.
Declaración de Ética
Este estudio experimental, fue realizado de acuerdo a la guía de experimentación en animales de investigación de la norma oficial mexicana, NOM-062-ZOO-1999.
Consentimiento para Publicación
Los autores han leído el contenido del artículo y están de acuerdo con el mismo, por lo tanto otorgan el consentimiento a la editorial de la revista TERRA Latinoamericana para que este sea publicado.
Conflicto de Intereses
Los autores declaran que no existe un potencial conflicto de interés respecto a esta investigación, para la publicación de este artículo.
Fondos
El financiamiento de este estudio experimental fue otorgado por el Consejo de Ciencia y tecnología (CONACYT) número de proyecto 61119.
Contribución de los Autores
Todos los autores contribuyeron a la concepción y diseño del estudio. Preparación del artículo, la colección de datos y análisis de los mismos fue realizado por: S.M.C. el Análisis filogenético fue realizado por L.G.B.G. revisión del artículo y conceptos importantes del VNPI fueron realizados por C.O.S. Todos los autores comentaron las versiones previas del manuscrito y aprobaron el manuscrito final.











 nueva página del texto (beta)
nueva página del texto (beta)



