Introducción
El término infección de transmisión sexual (ITS) hace referencia a una infección que se transmite por la sangre, el semen, los fluidos vaginales u otros líquidos corporales durante el sexo oral, anal o genital con una pareja infectada1. El término enfermedad de transmisión sexual (ETS) hace referencia a una enfermedad que se ha desarrollado a partir de una ITS. Algunos ejemplos son el VIH, la sífilis, el papiloma, la gonorrea, la clamidiasis o la hepatitis, entre otras1.
La implementación de métodos moleculares para la detección de microorganismos dio una nueva perspectiva en el diagnóstico mediante pruebas basadas en la técnica de reacción en cadena de la polimerasa (PCR). La Organización Mundial de la Salud ha puesto énfasis en desarrollar métodos de diagnóstico rápidos, sensibles y específicos, para aplicar un tratamiento rápido y eficaz, por lo que se ha considerado la PCR como una técnica confiable para el análisis de enfermedades2.
El uso de métodos moleculares como la PCR se considera de bajo costo en comparación con otras técnicas convencionales utilizadas para la detección de estos patógenos y cuyos resultados han demostrado no ser tan eficaces3,4. Hoy en día, los centros de salud, hospitales e institutos del Sistema Nacional de Salud basan su diagnóstico de ITS en la microbiología convencional apoyado por equipos automatizados como el Sistema Vitek2, que son relativamente costosos y requieren hasta dos o tres días de incubación para obtener resultados5. Las pruebas de detección microbiológica convencional para bacterias se consideran los métodos de referencia, pero tienen una utilidad limitada en casos subclínicos o crónicos debido a su menor sensibilidad e ineficacia en microorganismos no cultivables6. Los ensayos serológicos como el ensayo inmunoabsorbente ligado a enzimas (ELISA), la prueba de anticuerpos fluorescentes indirectos (IFA) y los ensayos de inmunotransferencia son sensibles, pero adolecen del problema del costo incurrido, la reactividad cruzada y la incapacidad de distinguir entre infección presente o pasada7. Sin embargo, las técnicas de diagnóstico molecular, como la PCR convencional y los ensayos de PCR en tiempo real, ofrecen pruebas de diagnóstico sensibles y específicas para la detección de patógenos3,8.
De las variantes de la PCR la más utilizada para la detección e identificación específica de microorganismos es la PCR múltiplex, en donde se amplifican dos o más segmentos de ADN blanco utilizando iniciadores diseñados que mantienen una alta sensibilidad y especificidad en la detección génica de los patógenos9. Sin embargo, en México no se utiliza esta técnica molecular para la detección simultánea de patógenos en una sola PCR, solo existen reportes de trabajos con PCR simple y PCR anidada para la detección individual de los patógenos. Recientemente, los ensayos convencionales de PCR se han utilizado ampliamente para la detección de microorganismos denominados atípicos, como Chlamydia trachomatis, Mycoplasma spp. y Ureaplasma spp., entre otros10,11. Así mismo, estas pruebas se han implementado en el diagnóstico de patógenos que pueden ser identificados por pruebas microbiológicas convencionales como Neisseria, Haemophilus ducreyi, etc. Además, se ha demeritado el rol de Gardnerella vaginalis en afecciones como la vaginosis bacteriana a pesar de haber estudios que la relacionan directamente en la implantación de infecciones por Neisseria gonorrhoeae y C. trachomatis12. Actualmente, las compañías biotecnológicas ofrecen diferentes alternativas para el diagnóstico tanto de manera individual como en paneles de enfermedades; estas nuevas metodologías nos permiten identificar desde 6 hasta 11 agentes etiológicos en el caso de marcas como Seegene e Hybrispot 12 respectivamente, en donde se incluyen tanto bacterias, como virus y protozoos. Sin embargo, la implementación de las nuevas tecnologías de multidiagnóstico por biología molecular que se encuentran en el mercado son excesivamente costosas y en ocasiones insostenibles por el número de pruebas solicitadas.
El desarrollo de PCR múltiplex por su rapidez, sensibilidad y especificidad se transforma en una herramienta para el uso en epidemiológico, de control y prevención de enfermedades3. Es por ello que numerosos grupos de trabajo han desarrollado sus propias pruebas diagnósticas para cubrir las necesidades de cada hospital o centro de salud para una rápida identificación y diferenciación de los patógenos y así ofrecer un tratamiento inmediato. Ante este panorama, el propósito de este estudio es el de desarrollar pruebas de PCR múltiplex para el diagnóstico de microorganismos atípicos. Con base en lo anterior, se propone la elaboración de pruebas de PCR de tipo simple o múltiple para satisfacer las necesidades del instituto. La finalidad es ofrecer una prueba diagnóstica alcanzable pero que mantenga la sensibilidad y especificidad que tienen los métodos moleculares.
Materiales y métodos
Extracción de ADN genómico
La extracción del ADN total de las muestras endocervicales se realizó utilizando el reactivo de purificación TripureTM de acuerdo con las especificaciones del fabricante (Boehringer Mannheim, Ingelheim, Alemania). Así mismo, el ADN plasmídico se aisló con el sistema QIAprep Miniprep Kit (QiagenTM, Hilden, Alemania).
Identificación individual de Chlamydia trachomatis, Mycoplasma spp., Ureaplasma spp. y Gardnerella vaginalis por PCR
La identificación de los patógenos asociados a las ETS (Tabla 1) se realizó mediante la prueba de la PCR convencional dirigida a diferentes genes utilizando los cebadores sintetizados por IDT® (Coralville, IA, EE.UU.). Cabe destacar que para el caso de C. trachomatis se consideraron dos dianas génicas, el gen ompA y el orf6 plasmídico, debido al bajo número de bacterias y para aumentar la sensibilidad de la prueba de PCR para este microorganismo. Por otra parte, para micoplasmas y G. vaginalis se recurrió a iniciadores que han sido reportados como altamente sensibles y específicos. La mezcla de reacción para las pruebas de PCR consistió en TaqPCR Master Mix Kit (QiagenTM), 10 pM de cada iniciador y 5 μl del ADN. Para cada ensayo de PCR se utilizó como control positivo el ADN de cepas cada uno de los microorganismos estudiados y como control negativo el ADN de células HeLa. La amplificación se realizó en un termociclador PTC-100 (MJ Research Inc.TM, Hercules, CA, EE.UU.) con una desnaturalización inicial a 95 °C durante 5 minutos, seguido de 30 ciclos (95 °C/45 s, 59 °C/45 s y 72 °C/1 min) y extensión final a 72 °C durante 10 minutos. Los productos de la PCR se analizaron mediante electroforesis en gel de agarosa al 2%.
Tabla 1 Generalidades de los oligonucleótidos seleccionados para la PCR
| Microorganismo | Iniciadores | Sitio | Referencia |
|---|---|---|---|
| Chlamydia trachomatis | CT3 CT4 |
Cromosoma Gen ompA |
Jian-Hong et al., 201013 |
| Chlamydia trachomatis | CDS6 CDS6 |
Plásmido orf6 |
Escobedo-Guerra et al. (datos no publicados) |
| Mycoplasma spp. Ureaplasma spp. | GPO1 MGSO |
Cromosoma Gen rRNA16s |
van Kuppeveld et al., 199214 |
| Gardnerella vaginalis | GV16sF GV16sR |
Cromosoma Gen rRNA16s |
Pillay et al., 202015 |
PCR: reacción en cadena de la polimerasa.
Identificación múltiplex de Chlamydia trachomatis, Mycoplasma spp., Ureaplasma spp. y Gardnerella vaginalis por PCR
De acuerdo con las características de los iniciadores para cada uno de los microorganismos estudiados (Tabla 2) se estableció un protocolo para el ensayo del sistema de PCR múltiple. De manera inicial se realizaron pruebas dúplex y posteriormente tríplex para la identificación de los diversos patógenos contemplados (Tabla 3). La mezcla de reacción para las pruebas de PCR consistió en TaqPCR Master Mix Kit (QiagenTM), 10 pM de cada iniciador y 5 μl del ADN. Para cada uno de los ensayos se utilizó como control positivo el ADN de cada una de las cepas de los microorganismos estudiados y como control negativo el ADN de células HeLa. La amplificación se realizó en un termociclador PTC-100 (MJ Research Inc.TM, Hercules, CA, EE.UU.) con una desnaturalización inicial a 95 °C durante 5 minutos, seguido de 30 ciclos (95 °C/45 s, 59 °C/45 s y 72 °C/1 min) y extensión final a 72 °C durante 10 minutos. Los productos de la PCR se analizaron mediante electroforesis en gel de agarosa al 2%.
Tabla 2 Características de los oligonucleótidos seleccionados para la PCR múltiplex
| Microorganismo | Tm °C | Amplicón pb | Secuencia 5´-3´ |
|---|---|---|---|
| Chlamydia trachomatis ompA | 55 | 460 | ACTTTGTTTTCGACCGTGTTTG GATTGAGCGTATTGGAAAGAAGC |
| Chlamydia trachomatis orf6 | 58 | 303 | GCAAAATAAAAGAAAAGTGAGGGACG TCAGCCTTGGAAAACATGTCT |
| Mycoplasma spp. Ureaplasma spp. | 59 | 715 | ACTCCTACGGGAGGCAGCAGTA TGCACCATCTGTCACTCTGTTAACCTC |
| Gardnerella vaginalis | 54 | 1300 | TTCGATTCTGGCTCAGG CCATCCCAAAAGGGTTAGGC |
PCR: reacción en cadena de la polimerasa.
Tabla 3 Esquema de trabajo para el análisis del desempeño de la PCR múltiple
| Dúplex | Tríplex | Cuádruplex |
|---|---|---|
|
C. trachomatis ompA C. trachomatis orf6 C. trachomatis ompA Mycoplasma/Ureaplasma C. trachomatis ompA G. vaginalis Mycoplasma/Ureaplasma C. trachomatis orf6 Mycoplasma/Ureaplasma G. vaginalis |
C. trachomatis ompA C. t rachomatis orf6 Mycoplasma/Ureaplasma |
C. trachomatis ompA C. trachomatis orf6 Mycoplasma/Ureaplasma G. vaginalis |
PCR: reacción en cadena de la polimerasa.
Validación de resultados
Los resultados obtenidos de las pruebas de PCR simple y múltiple fueron validados mediante el análisis en paralelo de algunas muestras con el equipo HybriSpot 12 (Master Diagnostica, Vitro Group, Sevilla, España) y por secuenciación de los productos de amplificación.
El ADN obtenido de las muestras fue procesado en el equipo HybriSpot 12 de acuerdo con las indicaciones del fabricante. El equipo HybriSpot 12 es un sistema automatizado de PCR e hibridación que permite la identificación múltiple de patógenos asociados a ITS como C. trachomatis, Mycoplasma hominis, Mycoplasma genitalium, Haemophilus ducreyi, Ureaplasma spp., N. gonorrhoeae, Treponema pallidum, herpes virus I, herpes virus II y Trichomonas vaginalis.
Así mismo, los productos de PCR de cada uno de los microorganismos identificados se purificaron con el sistema NucleoSpin (Macherey-Nagel, Düren, Alemania) y se enviaron al Instituto de Biología (Universidad Nacional Autónoma de México) para su secuenciación mediante el secuenciador ABI PRISM (Applied Biosystems, Waltham, MA, EE.UU.). Las secuencias obtenidas se analizaron con el software de Chromas (Technelysum Pty Ltd., South Brisbane QLD, Australia), identificado por BLASTn (NCBI, MD, EE.UU.)16,17 y alineado con el software ClustalW18 y Multalin (versión 5.4.1, alineación de secuencia múltiple con agrupamiento jerárquico, Francia)19.
Resultados
PCR simple
Se obtuvieron los productos esperados de la amplificación de cada uno de los genes de los microorganismos analizados y que correspondieron a los resultados reportados (Tabla 1). Los productos de amplificación fueron secuenciados y el análisis in silico demostró su homología y alineación a las secuencias de C. trachomatis (gen ompA cromosómico, orf6 plasmídico), Mycoplasma spp. (gen rRNA16s), Ureaplasma spp. (gen rRNA16s) y G. vaginalis (gen rRNA16s) reportadas en el GenBank (National Library of Medicine, National Center of Biotechnology Information). Datos no publicados.
PCR múltiple
De acuerdo con el esquema propuesto se realizaron diferentes pruebas para la realización de la PCR múltiple (Tabla 3). Durante el desarrollo de las pruebas se observó que la PCR dúplex para C. trachomatis con los iniciadores para el gen ompA y el orf6 plasmídico fueron satisfactorias. También se obtuvieron resultados para el dúplex de C. trachomatis (orf6) con G. vaginalis y C. trachomatis (gen ompA) con Mycoplasma/Ureaplasma (Fig. 1). Así mismo, se obtuvieron resultados limitados en la PCR dúplex tanto de C. trachomatis (orf6) con Mycoplasma/Ureaplasma y de Mycoplasma/Ureaplasma con G. vaginalis.
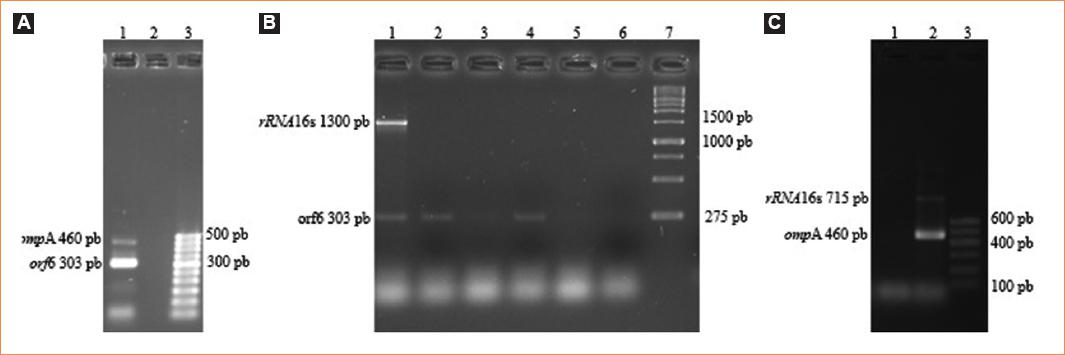
Figura 1 Desarrollo electroforético de las PCR dúplex para Chlamydia trachomatis. A: se observa la amplificación del orf6 y del gen ompA con amplicones de 303 y 460 pb respectivamente; en el pozo 1, amplificación de 100 ng de ADN de C. trachomatis; en el 2, control negativo, y en el 3, marcador de tamaño molecular. B: se muestra el desarrollo electroforético del gen orf6 de C. trachomatis y del gen rDNA16s de G. vaginalis con amplicones de 303 y 1,300 pb respectivamente. En el pozo 1, amplificación de 100 ng de ADN de C. trachomatis y G. vaginalis; del 2 al 5, muestras clínicas; en el 6, control negativo, y en el 7, marcador de tamaño molecular. C: se muestra el desarrollo electroforético del gen ompA de C. trachomatis y del gen rRNA16s de Mycoplasma/Ureaplasma con amplicones de 460 y 715 pb respectivamente. En el pozo 1, amplificación de 100 ng de ADN de C. trachomatis y Mycoplasma hominis; en el 2, control negativo, y en el 3, marcador de tamaño molecular.PCR: reacción en cadena de la polimerasa.
Posteriormente, con base en los resultados anteriores se realizó la PCR tríplex para C. trachomatis (gen ompA y orf6 plasmídico) y Mycoplasma/Ureaplasma (gen rRNA16s) obteniéndose amplificación de los tres sitios génicos analizados (Fig. 2). Así mismo, se prosiguió a implementar una PCR cuádruplex, en la cual se utilizaron los oligonucleótidos para la detección de C. trachomatis, Mycoplasma/Ureaplasma y G. vaginalis con resultados satisfactorios (Fig. 3). Cabe señalar que en estas dos fases de la investigación además de incluir los controles positivos para cada uno de los microorganismos estudiados se utilizaron muestras clínicas para determinar la sensibilidad de la PCR múltiplex.
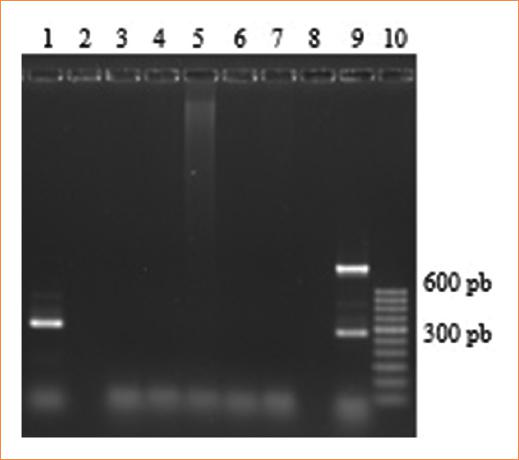
Figura 2 Desarrollo electroforético de la PCR tríplex para el gen ompA y orf6 de C. trachomatis y el gen rRNA16s de Mycoplasma/Ureaplasma. Del pozo 1 al 7, muestras clínicas, de las cuales el pozo 1 presenta amplificación con productos de 303 y 460 pb correspondientes al orf6 y gen ompA de C. trachomatis; en el 8, control negativo; en el 9, control positivo con amplicones de C. trachomatis (orf6 y del gen ompA) y de Mycoplasma/Ureaplasma (gen rRNA16s), y en el 10, marcador de tamaño molecular.PCR: reacción en cadena de la polimerasa.
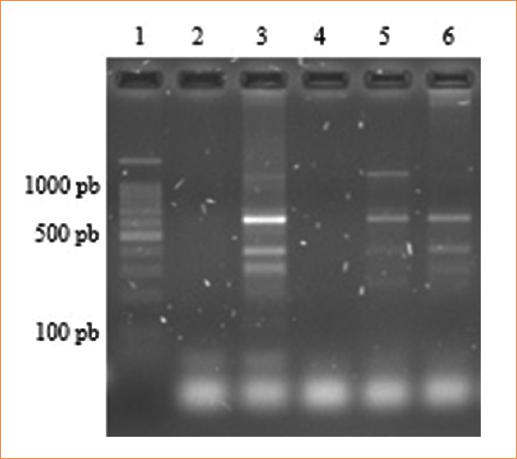
Figura 3 Desarrollo electroforético de la PCR cuádruplex para el gen ompA y orf6 de C. trachomatis, el gen rRNA16s de Mycoplasma/Ureaplasma y el gen rRNA16s de G. vaginalis. El pozo 1, marcador de tamaño molecular; en el 2, control negativo; en el 3, control positivo donde se observan los productos de amplificación de los cuatro sitios génicos estudiados (orf6 y ompA de C. trachomatis, rRNA16s de Mycoplasma/Ureaplasma y rRNA16s de G. vaginalis); en el 5, muestra clínica positiva a C. trachomatis, Mycoplasma/Ureaplasma y G. vaginalis, y en el 6, muestra clínica positiva a C. trachomatis y a Mycoplasma/Ureaplasma.PCR: reacción en cadena de la polimerasa.
Discusión
El objetivo más importante del diagnóstico clínico es el de obtener resultados veraces y rápidos que repercutan de manera directa en la calidad de vida del paciente4,6. Desafortunadamente, el sistema de salud no posee las herramientas adecuadas para cubrir estas necesidades, además de que el diagnóstico de microrganismos atípicos como C. trachomatis y micoplasmas no se realiza de manera sistemática y en el caso de G. vaginalis no se considera como patógeno primario10,12.
En cuanto a C. trachomatis y micoplasmas, los métodos utilizados en los laboratorios requieren de personal calificado e infraestructura especializada. Afortunadamente, la inclusión de las técnicas de biología molecular en el diagnóstico de estos microorganismos ha permitido su identificación oportuna y un tratamiento adecuado5,6. Sin embargo, estos métodos moleculares representan un gran costo para los servicios de salud como para ser considerados como ensayos de rutina para los pacientes que asisten o ingresan a la clínicas u hospitales.
En esta investigación se analizaron las posibilidades de implementar pruebas de PCR dúplex, tríplex y posiblemente cuádruplex con las dificultades técnicas que se tienen que sortear. El establecimiento de pruebas múltiplex conlleva el estudio de los todos los factores que intervienen en la PCR, principalmente la interacción de los oligonucleótidos para cada uno de los sitios génicos estudiados20. Las variables que se tienen que investigar son la selección de la temperatura adecuada de hibridación del iniciador y que esta sea específica, además de observar si no hay dimerización o formación de estructuras que impiden su correcta alineación e identificación con la cadena de ADN21. Así mismo, se tienen que establecer las concentraciones adecuadas de cada uno de los componentes de la reacción de PCR para que no haya elementos limitantes que impidan su correcto funcionamiento22.
Durante el desarrollo de este trabajo se obtuvieron resultados satisfactorios no solo en pruebas dúplex sino en la tríplex y en la cuádruplex. Lo anterior representa un logro que posibilita que nuestras pruebas se utilicen para el diagnóstico, ya que los resultados fueron verificados en paralelo con el sistema automatizado HybriSpot 12 (Vitro). Este sistema de diagnóstico utiliza la tecnología DNA-Flow que mediante una PCR múltiple y la posterior hibridación en membrana con sondas de ADN específicas permiten la identificación de hasta 11 microorganismos en su panel de ETS. Los resultados obtenidos en nuestras pruebas caseras fueron similares a los del HybriSpot 12 (Vitro) a excepción de G. vaginalis, que no se incluye en el panel de ETS de Master Diagnostica®.
Además, se corroboró que los productos obtenidos en cada una de las pruebas de PCR tanto simple como múltiples pertenecen a los microorganismos estudiados. Lo anterior se obtuvo mediante el análisis in silico de las secuencias de los amplicones obtenidos; estos datos fueron sometidos a métodos bioinformáticos, permitiéndonos establecer la correcta identidad de los genes y el microorganismo al que pertenecen.
En concreto, se logró obtener una prueba de PCR tríplex que permite la identificación de los microorganismos denominados atípicos como C. trachomatis y micoplasmas. Esta prueba permitirá al clínico establecer un correcto diagnóstico y dar un tratamiento oportuno para impedir que se desarrollen las secuelas de una infección crónica por estas bacterias. Se conoce que la infección por C. trachomatis en mujeres puede derivar en endometriosis, oclusión tubo-ovárica, enfermedad pélvica inflamatoria e infertilidad23. En recién nacidos de madres positivas se puede presentar neumonía atípica y en hombre epididimitis y proctitis24. En el caso de los micoplasmas se ha observado el desarrollo de inflamación pélvica, adherencias epiteliales y probable infertilidad en mujeres, además que promueven la ruptura prematura de membranas durante el embarazo11. Así mismo, en el hombre los micoplasmas se han asociado a problemas de fertilidad por su capacidad de adherencia a los espermatozoides25.
Finalmente, se consiguió implementar una prueba de PCR cuádruple que además de identificar a C. trachomatis y micoplasmas nos permite reconocer a G. vaginalis. Este logro es de suma importancia, ya que aunque G. vaginalis no se considera como patógeno primario en las ETS se encuentra asociado al desarrollo de la vaginosis bacteriana y a la implantación de bacterias como N. gonorrhoeae y C. trachomatis26. En consecuencia, estos resultados pueden ofrecer una alternativa para el diagnóstico de aquellos microorganismos considerados atípicos o secundarios en México, pero que representan un gran impacto en la salud según estudios de países del primer mundo.
Conclusiones
Como se indicó, se logró obtener un panel de pruebas de PCR múltiple para el diagnóstico de C. trachomatis, Mycoplasma spp., Ureaplasma spp. y G. vaginalis que pueden ser utilizados como estudios sistemáticos con la ventaja de ser de bajo costo sin perder su especificidad y sensibilidad.











 nueva página del texto (beta)
nueva página del texto (beta)


