Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista mexicana de micología
versión impresa ISSN 0187-3180
Rev. Mex. Mic vol.36 Xalapa dic. 2012
Contribuciones
Caracterización morfológica y molecular de una cepa silvestre mexicana perteneciente al género Ganoderma
Morphologic and molecular characterization of a Mexican wild strain belonging to Ganoderma genus
Jorge Suárez-Medellín1,2, Mauricio Luna-Rodríguez1, Guillermo Mendoza1, Alejandro Salinas1, Mirna Leonor Suárez Quiroz2, Ángel Trigos1*
1 Laboratorio de Alta Tecnología de Xalapa, Universidad Veracruzana. Calle Médicos No. 5, Col. Unidad del Bosque. C.P. 91010, Xalapa, Veracruz, México.
2 Unidad de Investigación y Desarrollo en Alimentos, Instituto Tecnológico de Veracruz. Av. Miguel A. de Quevedo # 2779 C Formando Hogar, 91680 Veracruz, Veracruz, México.
*Autor para correspondencia:
Ángel Trigos atrigos@uv.mx
Recibido 8 de Mayo 2012;
Aceptado 5 de Diciembre 2012.
Abstract
Fungi belonging to the Ganoderma genus have been widely studied because of their phytopatogenic and medicinal potential. However, due to its morphological variability, the genus has been considered in a status of "taxonomic chaos". In this study, a specimen of the Ganoderma genus originary of the State of Veracruz (México) was isolated and described from a morphological and molecular viewpoint, and also was identified according to its phylogenetic affinity.
Keywords: PCR, ITS, taxonomic chaos.
Resumen
Los hongos pertenecientes al género Ganoderma han sido ampliamente estudiados por su potencial fitopatógeno y medicinal. No obstante, su variabilidad morfológica ha ocasionado que en la actualidad se considere que el género se encuentra en un estado de "caos taxonómico". El presente trabajo reporta la caracterización morfológica y molecular de un ejemplar del género Ganoderma aislado en el estado de Veracruz (México), el cual fue identificado de acuerdo a su afinidad filogenética, combinando el enfoque molecular con el morfológico tradicional.
Palabras clave: PCR, ITS, caos taxonómico.
Introducción
Los hongos del género Ganoderma (P. Karst), han sido estudiados desde distintos puntos de vista, dependiendo de los intereses de cada grupo de investigación: como fuente de productos nutricéuticos y/o medicinales (Wasser et al., 2000; Mau et al., 2002; Sliva et al., 2003; Wachtel-Galor et al., 2004; Han et al., 2005; Paterson, 2006; Trigos y Suárez- Medellín, 2011), como fitopatógenos en cultivos de palma aceitera, coco, hule, té, café, cacao y árboles forestales (Zakaria et al., 2005; Paterson, 2007; Karthikeyan et al., 2009), como causa de asma debido a la dispersión aérea de sus esporas (Craig y Levetin, 2000), como degradadores selectivos de lignocelulosa con posibles aplicaciones en la fabricación de pulpa de papel y en la biodegradación de contaminantes como policloruro de bifenilo y otros fenoles clorados (Hong y Jung, 2004; Wang y Ng, 2006), e incluso, como complemento alimenticio para pollos de granja (Ogbe et al., 2008). Sin embargo, siendo un género particularmente variable desde el punto de vista morfológico, su identificación taxonómica presenta serias dificultades (Buchanan, 2001).
Este género perteneciente a los hongos poliporales, es uno de los más amplios al contar con más de 250 especies reportadas en todo el mundo (Moncalvo et al., 1994; Buchanan, 2001). De acuerdo con Ryvarden (2000), actualmente el estatus de dicho género podría describirse como de "caos taxonómico". La metodología empleada en la taxonomía tradicional suele tomar en cuenta características tales como: especificidad del hospedero, distribución geográfica y rasgos morfológicos macroscópicos del cuerpo fructífero, incluyendo el color del contexto, la forma del margen del píleo y la presencia o ausencia de estípite (Seo y Kirk, 2000; Pérez et al., 2005; Hood, 2006).
Otras características comúnmente consideradas como útiles en la identificación del género, son las siguientes: estructura microscópica de la corteza del píleo; forma, textura y tamaño de las basidiosporas; presencia o ausencia de clamidosporas en cultivos in vitro y reacción amiloide o dextrinoide en presencia del reactivo de Melzer (Seo y Kirk, 2000; Ryvarden, 2000).
Siguiendo un consenso ampliamente aceptado, el género Ganoderma, ha sido divido en dos subgéneros de acuerdo con la textura de la cubierta superior de su basidiocarpo: subgénero Ganoderma para los ejemplares laqueados y subgénero Elfvingia para los no laqueados (Buchanan, 2001). Más allá del límite impuesto por dichos subgéneros, la clasificación taxonómica basada en características morfológicas deja de ser confiable, debido a la amplia variabilidad existente entre los miembros del género. Se ha reportado que características tradicionalmente consideradas como diagnósticas, tales como la forma del basidiocarpo, el tamaño y forma de las basidiosporas y el color del contexto, dependen en gran medida de las condiciones ambientales en las que creció el hongo, por lo que para identificar a nivel de especie es necesario recurrir a técnicas derivadas de la biología molecular (Steyaert, 1972; Steyaert, 1975; Chen, 1993; Buchanan, 2001).
Entre los enfoques empleados para complementar la identificación taxonómica de los hongos del género Ganoderma cabe mencionar el uso de métodos basados en la amplificación de secuencias de DNA mediante la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés), especialmente aquellas relacionadas con el análisis de la región espaciadora interna transcrita (ITS, por sus siglas en inglés) (Hseu et al., 1996; Pérez et al., 2005; Karthikeyan et al., 2009). Adicionalmente a los enfoques relacionados con el uso de marcadores moleculares, también se han ensayado otras estrategias de identificación en este género, entre las que destacan: quimiotaxonomía basada en el análisis del patrón de producción de metabolitos secundarios mediante HPLC (Shi et al., 2008) y ensayos inmunológicos tipo ELISA (Karthikeyan et al., 2009).
Por lo anterior, el objetivo del presente trabajo consistió en realizar la caracterización morfológica y molecular de una muestra de ejemplares veracruzanos del hongo Ganoderma perteneciente al subgénero Ganoderma, aplicando el enfoque molecular junto con el morfológico tradicional para lograr una correcta identificación.
Materiales y métodos
Aislamiento y caracterización morfológica
La cepa fue aislada a partir de cuerpos fructíferos silvestres colectados en el Campus para la Cultura y las Artes de la Universidad Veracruzana zona Xalapa (N 19° 30' 33'' O 96a 54' 58'', 1368 msnm), a los cuales se les realizó una identificación preliminar basada en su morfología utilizando las claves dicotómicas de Guzmán (1980) y Ryvarden (2000). Las preparaciones microscópicas se montaron con lactofenol claro y azul de lactofenol. Para observar las reacciones amiloides o dextrinoides de las hifas esqueléticas se empleó reactivo de Melzer.
El aislamiento de la cepa se llevó a cabo a partir de cuerpos fructíferos frescos, los cuales fueron lavados a chorro de agua, desinfectados en hipoclorito de sodio al 2% y colocados en cámara húmeda a 25 °C ± 2, y posteriormente trasladados a cajas Petri con medio PDA. Las características micromorfológicas del micelio fueron comparadas con las reportadas por Moncalvo et al. (1995).
Se depositó un resguardo de la cepa por quintuplicado en el cepario interno de LATEX con la clave LAT-054. Esta fue mantenida inoculando cinco tubos inclinados de medio PDA con la cepa en cuestión incubando a 25 °C ± 2. Cuando el micelio estaba completamente desarrollado, se agregó agua estéril, se selló con parafilm y se almacenó a 4 °C, a partir de la cual se realizaron posteriormente resiembras cada cuatro meses (Smith y Onions, 1994).
Caracterización molecular de la cepa
La extracción de ADN se realizó a partir de micelio fresco, siguiendo el protocolo reportado por Cheng y Jiang (2006) de la siguiente manera: una muestra de 50 mg de micelio fresco se homogeneizó en mortero congelado con 200 µL de Buffer TE (10 mM Tris/HCl, 1 mM EDTA, pH 8.0) y 100 µL de Tris saturado de fenol (pH 8.0), hasta quedar totalmente molido. La mezcla se centrifugó a 1000xg durante 5 min (Eppendorf Mini Spin Plus). Posteriormente, 160 µL de la fase acuosa fueron transferidos a un tubo limpio de 1.5 mL y se añadieron 40 µL de buffer TE. Se mezcló con 100 µL de cloroformo y se centrifugó a 1000xg durante 5 min. Esta operación se repitió de tres a cuatro veces, hasta que la interfase de color blanco dejó de aparecer. A continuación, 160 µL de la fase acuosa fueron transferidos a un tubo limpio y se añadieron 40 µL de buffer TE y 5 µL de RNAsa (a una concentración de 10 mg/mL), se dejó incubar a 37 °C durante 10 min para degradar el ARN presente. Una vez concluido este paso se añadieron 100 µL de cloroformo al tubo, se mezcló en vortex y se centrifugó a 1000 xg durante 5 min. Finalmente, 150 µl de la fase acuosa fueron transferidos a un tubo limpio de 1.5 ml y se almacenó a -20° C hasta su uso.
Para la amplificación de las regiones ITS1 e ITS2 incluyendo el gen 5.8S del ADNr, se emplearon los iniciadores BMB-CR (5'-GTACACACCGCCCTCG-3') y LR0 (5'-GCTTAAGTTCAGCGGGT-3') (Gottlieb et al, 2000). La reacción fue llevada a cabo en un volumen final de 25 µL de solución compuesta por 25 pM de cada primer, una mezcla de 0.25 mM de cada uno de los dNTPs, 2.5 mM de MgCl2 1 X de buffer Taq, 1.5 unidades de Taq DNA polimerasa (Promega) y 50 ng de DNA. La amplificación se llevó a cabo en un termociclador Mastercycler (Eppendorf AG), empleando los siguientes parámetros: 30 ciclos de 94 °C para la desnaturalización durante 1 min, 50 °C para el anillamiento durante 45 s, y 72 °C para la extensión durante 1 min, finalmente un ciclo de extensión a 72 °C por 7 min para asegurar la polimerización completa de cualquier remanente de los productos de PCR (Vilgalys y Hester, 1990). Los productos de PCR fueron analizados usando un gel de agarosa al 1.8%, revelados con bromuro de etidio y purificados mediante el kit comercial Wizard SV Gel and PCR Clean-Up System (Promega). La secuenciación de los productos de amplificación se realizó en un secuenciador automático Genetic Analyzer 3500. La secuencia obtenida fue comparada con todas las secuencias de Ganoderma en la base de datos de DNA (GenBank and EMBL) usando el Basic Local Alignment Search Tool (BLAST). Posteriormente, se realizó una búsqueda de secuencias de especies más representativas, según el análisis BLAST. La secuencia seleccionada fue alineada con CLUSTALW y ajustada manualmente y se llevó a cabo un análisis filogenético empleando el algoritmo de máxima parsimonia (MP), con 500 réplicas (Bootstrap) con el software Mega 4.1 (Mendoza et al., 2011).
Resultados y discusión
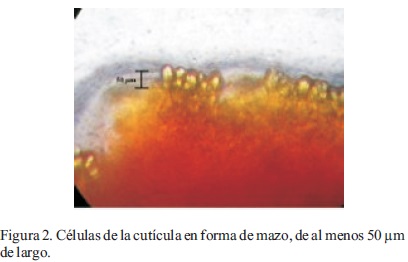
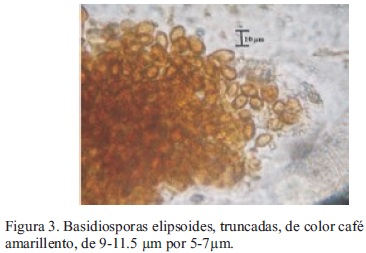
Las características morfológicas macroscópicas de los especímenes coinciden con las reportadas por Guzmán (1980) como Ganoderma sessile Murr.; sin embargo, de acuerdo con Bandala et al. (1993) y Ryvarden (2000), dicho nombre es un sinónimo de G. resinaceum Boud. Por otra parte, las características morfológicas tanto macroscópicas como microscópicas de los cuerpos fructíferos coinciden con la descripción de Ryvarden (2000) para la especie G. resinaceum, la cual se resume en: basidiocarpos perennes, pileados, sin estípite. De consistencia leñosa o semejante al corcho. Superficie superior plana, surcada, glabra, con una costra distintiva, al principio rojiza y brillante, y con la edad de color cada vez más café rojizo a bayo o grisáceo, debido a una capa resinosa excretada que se pone amarillenta y se derrite al contacto con la flama de un encendedor. Superficie de los poros de color blanco cremoso al principio y posteriormente ocrácea a gris pálido con tintes cafés. Tres a cuatro poros angulares o circulares por mm. Contexto café grisáceo pálido. Sistema hifal dimítico; hifas generativas hialinas, de pared delgada, con uniones de 2-5 µm de diámetro; hifas esqueléticas (Figura 1) abundantes, de pared gruesa y color café amarillento, de 3-6 µm de diámetro. En contacto con el reactivo de Melzer, las hifas esqueléticas presentan una leve reacción amiloide. Células de la cutícula (Figura 2) en forma de mazo, de al menos 50 µm de largo. Basidiosporas (Figura 3) elipsoides, truncadas, de color café amarillento, de 9-11.5 µm por 5-7 µm (Ryvarden, 2000).
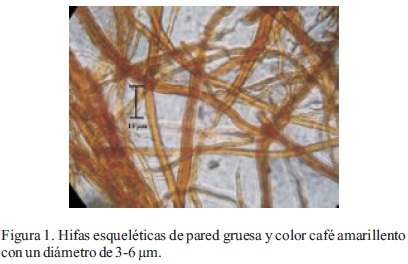
La cepa fue exitosamente cultivada en medio PDA y a partir de 14 días, el micelio comenzó a presentar numerosas clamidosporas ovoides y hialinas con doble pared celular. De acuerdo con Moncalvo et al. (1995), la presencia de un gran número de clamidosporas ovoides es común tanto a G. oerstedtii Murrill, como a G. resinaceum, está última especie, es nuevamente concordante para la identidad del hongo en estudio.
Con respecto a la caracterización molecular, fue posible extraer un promedio de 160 ng/µL de ADN con un factor de calidad de 1.9. Los productos de la PCR fueron de ≈ 730 pb, tamaño similar a lo reportado por Gottlieb et al. (2000). La secuencia de nucleótidos generada fue de 476 bases.
La comparación de dicha secuencia con las reportadas en GenBank, mediante la herramienta BLAST, arrojan una similitud de 99% con un porcentaje de cobertura del 90% y un valor E de 0.0 entre nuestra cepa de estudio y las cepas GU726925.1 y HM053467.1, ambas originarias de la India e identificadas como G. lucidum.
El subsecuente análisis filogenético, utilizando el algoritmo de máxima parsimonia y las secuencias reportadas en GenBank para cepas pertenecientes a 6 especies relacionadas dentro del género Ganoderma, una cepa local identificada como G. oerstedii (Mendoza et al., 2011) y Fomes fomentarius como clado externo (Figura 4), muestra que la cepa es conespecífica con la cepa GU726925.1 (identificada como Ganoderma lucidum), y no con las cepas identificadas como G. resinaceum (AM906065 y AY884177), como cabría esperar por su morfología, ni con la cepa local de G. oerstedii, reportada por Mendoza et al. (2011).
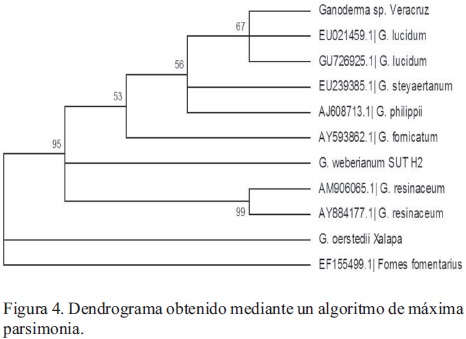
Dicha afinidad entre G. lucidum y G. resinaceum, no es completamente inesperada. Se sabe que G. resinaceum, junto con G. pfeifferi Bres., G. carnosum Pat., G. valesiacum Boud., y G. lucidum sensu stricto, pertenecen a un complejo de especies que han sido tradicionalmente identificadas como G. lucidum en sensu lato, sin necesariamente ser conespecíficas (Moncalvo et al., 1995). Los orígenes de dicho conflicto en la identificación pueden rastrearse hasta 1881, año en que Karsten estableció el género, conteniendo como única especie a G. lucidum, previamente identificado como Boletus lucidus por Curtis a partir de un ejemplar europeo. Desde entonces hasta la fecha, poco más de dos siglos después, el número de nuevas especies reportadas ha ido en aumento, en gran medida debido a la abundancia de sinonimias y errores de identificación. El grado de confusión taxonómica dentro del género es tal, que a pesar de que gran parte de los estudios químicos y clínicos acerca de Ganoderma lucidum son realizados en Asia empleando cepas autóctonas, estudios basados en métodos de clasificación molecular sugieren que la especie G. lucidum sensu stricto, se encuentra limitada al continente europeo (Moncalvo et al., 1995; Buchanan, 2001). De hecho, en las claves de identificación de Ryvarden (2000) para miembros laqueados del género Ganoderma presentes en regiones neotropicales, ni siquiera aparece una descripción de G. lucidum sensu stricto, debido a su supuesta restricción a las regiones templadas de Europa. Este mismo autor, reporta que los basidiocarpos de G. resinaceum son muy variables en forma y tamaño, por lo que han sido comúnmente descritos bajo diversos nombres, y en muchas ocasiones confundidos con G. lucidum, especie que a pesar de contar con múltiples especímenes depositados bajo este nombre en muchos herbarios americanos, de acuerdo con el autor antes citado, podría no estar presente en las regiones tropicales de América (Ryvarden, 2000).
Por otro lado, Hong y Jung (2004), basándose en estudios filogenéticos de las secuencias mitocondriales de la subunidad pequeña de rADN (SSU DNAr, por sus siglas en inglés), también encontraron que cepas procedentes de Norte América y Taiwán identificadas como G. lucidum y G. resinaceum eran conespecíficas entre sí.
Moncalvo et al. (1995) por su parte, reportan la presencia de un grupo monofilético de cepas asiáticas (procedentes de Taiwán, Filipinas y - significativamente - la India), todas ellas identificadas como G. lucidum, las cuales sin embargo demostraron no pertenecer a G. lucidum sensu stricto (Moncalvo et al., 1995; Buchanan, 2001). No obstante, otros autores, como Bhosle et al. (2010) sí reportan la presencia tanto G. lucidum como de G. resinaceumen la India, aunque haciendo referencia solamente a sus características morfológicas.
Tomando en consideración los reportes de los autores antes mencionados, parece probable que la cepa asiática GU726925.1 (conespecífica con la nuestra) haya sido identificada como G. lucidum sensu lato, no sensu stricto. Esto explicaría porque los basidiocarpos de nuestra cepa, aún a pesar de su afinidad filogenética con la anterior, presenten características macro y micromorfológicas más afines con las descritas para G. resinaceum (en el supuesto, obviamente, de que la cepa asiática GU726925.1 esté correctamente identificada, lo cual al no tener acceso a sus materiales biológicos no nos consta). Ahora bien, considerando el análisis molecular, y en virtud de que la cepa en estudio demostró no ser conespecífica con otras secuencias de cepas identificadas como G. resinaceum depositadas en Genbank, cabría la posibilidad de que la cepa en estudio corresponda a una especie aún no descrita, por lo que se deberá aplicar estudios complementarios que ayuden a la correcta identificación de ésta y evitar caer en las irregularidades taxonómicas del género. Sin embargo, este estudio deja en claro, como ha sucedido con muchas otras especies, las oportunidades que ofrecen las técnicas moleculares para ser usadas como herramientas en trabajos taxonómicos.
Por otro lado, dado que la mayor parte de los estudios químicos y farmacológicos (uno de los principales intereses prácticos responsables de la importancia de las investigaciones acerca del género) se han llevado a cabo en ejemplares asiáticos identificados como G. lucidum sensu lato y no sensu stricto, quizás valdría la pena reconsiderar la utilidad de identificar a los ejemplares europeos como G. lucidum sensu stricto, especialmente cuando la viabilidad de su producción de metabolitos secundarios sea el interés principal de la investigación.
Agradecimientos
Al Dr. Roberto Zenteno del Instituto de Salud Pública de la Universidad Veracruzana por su apoyo para la secuenciación del ADN. Este trabajo fue financiado en parte por la Beca No 14358, concedida por CONACyT a Jorge Manuel Suárez-Medellín para la realización de su doctorado.
Literatura citada
Bandala V., G. Guzmán, L. Montoya, 1993. Los hongos del grupo de los poliporaceos conocidos en México. Reporte Científico No. Especial 13:1-55. [ Links ]
Bhosle S., K. Ranadive, G. Bapat, S. Garad, G. Deshpande, J. Vaidya, 2010. Taxonomy and diversity of Ganoderma from the Western parts of Maharashtra (India). Mycosphere 1(3): 249-262. [ Links ]
Buchanan, P. K., 2001. A taxonomic overview of the genus Ganoderma with special reference to species of medicinal and neutriceutical importance. Proc. Int. Symposium Ganoderma Sci., Auckland, Nueva Zelandia. [ Links ]
Chen, C. S., 1993. Methods for inducing various morphological fruiting body of Ganoderma tsugae Murr. Transactions of the Mycological Society of Republic of China 8: 9-16. [ Links ]
Cheng H. R., N. Jiang, 2006. Extremely rapid extraction of DNA from bacteria and yeasts. Biotechnology Letters 28:55-59. [ Links ]
Craig, R. L., E. Levetin, 2000. Multi-year study of Ganoderma aerobiology. Aerobiologia 16: 75-81. [ Links ]
Gottlieb A. M., E. Ferrer, J. E. Wright, 2000. rDNA analyses as an aid to the taxonomy of the species of Ganoderma. Mycological Research 104: 1033-1045. [ Links ]
Guzmán, G., 1980. Identificación de los hongos comestibles, venenosos, alucinantes y destructores de la madera. Limusa, México D. F. [ Links ]
Han, J. R., C. H., J. M. Yuan, 2005. Solid-state fermentation of cornmeal with the basidiomycete Ganoderma lucidum for degrading starch and upgrading nutritional value. Journal of Applied Microbiology 99: 910-915. [ Links ]
Hong, S. G., H. S. Jung, 2004. Phylogenetic analysis of Ganoderma based on nearly complete mitochondrial small-subunit ribosomal DNA sequences. Mycologia 96(4): 742-755. [ Links ]
Hood, I. A., 2006. The mycology of the basidiomycetes. In: Potter, K., A. Rimbawanto, C. Beadle (Eds.). Heart rot and root rot in tropical Acacia plantations. Canberra, ACIAR Proceedings No. 124, pp. 34-59 [ Links ]
Hseu, R. S., H. H. Wang, H. F. Wang, J. M. Moncalvo, 1996. Differentiation and grouping of isolates of the Ganoderma lucidum complex by random amplified polymorphic DNA-PCR compared with groiuping on the basis of internal transcribed spacer sequences. Applied and Environmental Microbiology 62(4): 1354-1363. [ Links ]
Karthikeyan, M., K. Radhika, R. Bhaskaran, S. Mathiyazhagan, R. Velazhahan, 2009. Rapid detection of Ganoderma lucidum and assessment of inhibition effect of various control measures by immunoassay and PCR. African Journal of Biotechnology 8(10): 2202-2208. [ Links ]
Mau J.L., H.C. Lin, C.C. Chen, 2002. Antioxidant properties of several medicinal mushrooms. Journal of Agricultural and Food Chemistry 50: 6072-6077. [ Links ]
Mendoza, G., G. Guzmán, F. Ramírez-Guillén, M. Luna, A. Trigos, 2011. Ganoderma oerstedii (Fr.) Murrill (Higher Basidiomycetes), a tree parasite species in Mexico: Taxonomic description, rDNA study and review of its medical applications. International Journal for Medicinal Mushrooms 6: 545-552. [ Links ]
Moncalvo, J. M., H. F. Wang, H. H. Wang, R. S. Hseu, 1994. Molecular studies in the Ganoderma lucidum complex. In. 94'Int.Symp. Ganoderma Res. Lin, Z. B. (Ed.). Beijing Medical University Press, Beijing. [ Links ]
Moncalvo, J. M., H. F. Wang, R. S. Hseu, 1995. Gene philogeny of the Ganoderma lucidum complex based on ribosomal DNA sequences: Comparison with traditional taxonomic characters. Mycological Research 99:1489-1499. [ Links ]
Ogbe, A.O., L.O. Mgbojikwe, A.A. Owoade, S.E. Atawodi, P.A. Abdu, 2008. The effect of a wild mushroom (Ganoderma lucidum) supplementation of feed of the immune response of pullet chickens to infectious bursal disease vaccine. Electronic Journal of Environmental, Agricultural and Food Chemistry 7(4): 2844-2855. [ Links ]
Paterson, R. R. M., 2006. Ganoderma - a therapeutic fungal biofactory. Phytochemistry 67:1985-2001. [ Links ]
Paterson, R. R. M., 2007. Ganoderma disease of oil palm - A white rot perspective necessary for integrated control. Crop Protection 26:1369-1376. [ Links ]
Pérez, L. I., A. Santerre, A. R. Villalobos, A. Galván, M. G. Torres-Torres, A. Rodríguez, L. Guzmán-Dávalos, 2005. Extracción de DNA y amplificación de secuencias del ITS del DNAr de Ganoderma (Fungi, Basidiomycetes) para su uso en el análisis filogenético. Avances en la Investigación Científica en el CUCBA 450-457. [ Links ]
Ryvarden, L., 2000. Studies in neotropical polypores 2: A preliminary key to neotropical species of Ganoderma with a laccate pileus. Mycologia 92(1): 180-191. [ Links ]
Seo, G. S., P. M. Kirk, 2000. Ganodermataceae nomenclature and classification. In: Ganoderma diseases of perennial crops. J. Flood, P. D. Bridge y M. Holderness (Eds.) CABI Publishing, Nueva York. Pp 3-22. [ Links ]
Shi, X. M., J. S. Zhang, Q. J. Tang, Y. Yang, R. X. Hao, Y. J.Pan, 2008. Fingerprint analysis of Lingzhi (Ganoderma) strains by high-performance liquid chromatography coupled with chemometric methods. World Journal of Microbiology and Biotechnology 24:2443-2450. [ Links ]
Sliva, D., M. Sedlak, V. Slivova, T. Valachovicova, F.P. Lloyd, N.W.Y. Ho, 2003. Biologic activity of spores and dried powder from Ganoderma lucidum for the inhibition of highly invasive human breast and prostate cancer cells. Journal of Alternative and Complementary Medicine 9(4):491-497. [ Links ]
Smith, D., A. H. S. Onions, 1994. The preservation and maintenance of living fungi. Second edition. CAB International, Wallingford. [ Links ]
Steyaert, R. L., 1972. Species of Ganoderma and related genera mainly of the Bogor and Leiden herbaria. Persoonia 7:55-118. [ Links ]
Steyaert, R. L., 1975. The concept and circunscription of Ganoderma tornatum. Transactions of the British Mycological Society 65: 451-467. [ Links ]
Trigos, A., J. Suárez-Medellín, 2011. Biologically active metabolites of the genus Ganoderma: Three decades of myco-chemistry research. Revista Mexicana de Micología 34: 63-82. [ Links ]
Vilgalys, R., M. Hester, 1990. Rapid genetic identification and mapping of enzymatically amplified ribosomal DNA from several Cryptococcus species. Journal of Bacteriology 172(8): 4238-4246. [ Links ]
Wachtel-Galor, S., B. Tomlinson, I.F.F Benzie, 2004.Ganoderma lucidum ('Lingzhi'), a Chinese medicinal mushroom: biomarker responses in a controlled human supplementation study. British Journal of Nutrition 91(2): 263-269. [ Links ]
Wang, H.X., T.B. Ng, 2006. Alaccase from the medicinal mushroom Ganoderma lucidum. Applied Microbiology and Biotechnology 72: 508-513. [ Links ]
Wasser S.P., E. Nevo, D. Sokolov, S. Reshetnikov, M. Timor-Tismenetsky, 2000. Dietary supplements from medicinal mushrooms: Diversity of types and variety of regulations. International Journal of Medicinal Mushrooms 2: 1-19. [ Links ]
Zakaria, L., H. Kulaveraasingham, T.S. Guan, F. Abdullah, O.H. Wan, 2005. Random amplified polymorphic DNA (RAPD) and random amplified microsatellite (RAMS) of Ganoderma from infected oil palm and coconut stumps in Malaysia. Asia Pacific Journal of Molecular Biology and Biotechnology 13(1): 23-34. [ Links ]














