Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Universidad y ciencia
versão impressa ISSN 0186-2979
Universidad y ciencia vol.27 no.1 Villahermosa Abr. 2011
Artículos
Discontinuidad geográfica y variabilidad genética en Crassostrea rhizophorae guilding del sureste de México
Geographic discontinuity and genetic variability in Crassostrea rhizophorae guilding in southeastern Mexico
F Rodríguez Romero1 * , J Tello Cetina2
1 Unidad Académica de Geología marina y Ambiental Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México. Avenida Universidad 3000, México D. F. México. Apartado Postal 70-305. Código Postal 04510. (FRR). * Correo electrónico: faustrod@cmarl.unam.mx
2 IT Mérida. (JTC)
Artículo recibido: 01 de octubre de 2008,
Aceptado: 07 de abril de 2011
RESUMEN
Se estudió el impacto de la fragmentación del hábitat sobre la variabilidad genética en dos poblaciones de Crassostrea rhizophorae Guilding 1828 del sistema estuarino de la laguna de Términos, Campeche, México, mediante la expresión de isoenzimas de 10 sistemas enzimáticos y proteína total. Se utilizó el programa TFPGA 1.3 para analizar las frecuencias génicas de las poblaciones en estudio. Muestras de músculo de 50 organismos de cada población se utilizaron para caracterizar la expresión genotípica. Los parámetros determinados fueron estadística descriptiva, estadística F, distancia genética, equilibrio Hardy-Weinberg, UPGMA y el número de migrantes como indicador del flujo de genes. Los valores de polimorfismo proteico fueron 36.35% (p95) y 90.90% (p99). Los datos de heterocigosis se ubicaron entre 0.2491 para Idh1 hasta 0.01 en Pt1, el valor de heterocigocidad media fue He = 0.1044. Los promedios de Fis = 0.1574 y Fit = 0.1727 indicaron deficiencia de heterocigotos. La media de Fst = 0.0181 indicó que las diferencias genéticas correspondieron a variación interpoblacional con endogamia moderada. El número de migrantes obtenido por la ecuación de Slatkin resultó de 13.5621 por generación, indicativo de cierto grado de variabilidad entre las poblaciones, es consistente con valores de distancia genética de Nei. Se concluye que las dos poblaciones reflejan sensibilidad a los procesos adaptativos interpoblacionales producidos por la discontinuidad del medio geográfico sin detrimento de su variabilidad genética y no reflejan fragilidad que las exponga a proceso de formación de metapoblaciones.
Palabras clave: Flujo de genes, Golfo de México, isoenzimas, ostiones, metapoblaciones.
ABSTRACT
A study was carried out on the impact of habitat fragmentation on the genetic variability of two populations of Crassostrea rhizophorae Guilding 1828 in the estuarine system of Terminos lagoon, Campeche, Mexico, through the expression of isoenzymes of 10 enzyme systems and total protein. The software TFPGA 1.3 was used to analyse the genetic frequencies of the populations under study. Muscle samples of 50 organisms of each population were used to characterise the genotypic expression. The parameters determined were descriptive statistics, F statistic, genetic distance, Hardy-Weinberg equilibrium, UPGMA and the number of migrants as an indicator of gene flow. The protein polymorphism values were 36.35% (p95) and 90.90% (p99). The heterozygosis data were recorded between 0.2491 for Idh1 and 0.01 in Pt1, and the mean heterozygosity value was He = 0.1044. The averages of Fis = 0.1574 and Fit = 0.1727 indicated a heterozygote deficiency. The mean Fst = 0.0181 indicated that the genetic differences corresponded to an inter-population variation with a moderate endogamy. The number of migrants obtained by the Slatkin equation was 13.5621 per generation, indicated a certain degree of variability between populations, and is consistent with values of Nei genetic distance. It is concluded that both populations reflect a sensibility to the inter-population adaptive processes produced by the discontinuity of the geographical environment, with no affectation to their genetic variability, and do not reflect a fragility that subjects them to a process of formation of meta-populations.
Key words: Gene flow, Gulf of Mexico, isoenzymes, oysters, meta-populations.
INTRODUCCIÓN
En México, la familia Ostreidae se encuentra representada por los géneros Ostrea y Crassostrea con 16 especies (Rodríguez-Romero et al. 1988). Los ostiones con valor comercial pertenecen al género Crassostrea representados por una especie introducida: Crassostrea gigas (Thunberg 1795) y tres especies autóctonas: Crassostrea corteziensis (Herlein 1955) en el Pacífico Mexicano y Crassostrea virginica (Gmelin 1791) y Crassostrea rhizophorae (Guilding 1828) en la costa Atlántica. C. rhizophorae se localiza en la laguna de Términos y regiones adyacentes adherida a raíces de Rhizophora mangle en donde el ambiente acuático está influenciado por las aguas marinas. Biogeográficamente, reemplaza a C. virginica que se encuentra en la costa Atlántica del Continente Americano desde el Golfo de San Lorenzo, Canadá hasta la costa de Campeche (Ahmed 1975), donde las poblaciones sureñas de esta especie presentan una transición gradual con poblaciones de C. rhizophorae en cuanto al límite sur biogeográfico de C. virginica. A partir de la península de Yucatán hasta América del Sur, C. rhizophorae es un recurso de importancia económica. Se cree que la sobrevivencia de las poblaciones de estos ostiones en la zona de transición se puede poner en peligro por un manejo inadecuado debido a la falta de información sobre abundancia y distribución de larvas, de la estructura genética de sus poblaciones y el flujo génico entre ellas que oriente su explotación racional.
Los estudios de García-Cubas (1981) señalaron que Crassostrea rhizophorae se encuentra restringida a la zona con influencia marina de Isla del Carmen, Campeche. Esta franja de distribución en Isla del Carmen, se encuentra entre dos subsistemas ecológicos de la Sonda de Campeche cuya definición ambiental presenta entre sí diferencias significativas (Yañez-Arancibia & Sanchez-Gil 1986) y está interrumpida por la presencia de la boca de Puerto Real de aproximadamente 4 km de apertura en el lado norte, en la cual se produce un flujo neto predominante de aguas marinas del Golfo de México al interior de la laguna y configura una estratificación de salinidades (Vázquez-Botello 1978). Se ha inferido que el aislamiento por distancia (Rose et al. 2006) y la fragmentación del hábitat pueden provocar flujo génico limitado en poblaciones panmícticas y conformar metapoblaciones con riesgo de extinciones locales (Endler 1973; Lande & Barrowcloud 1987; Harrison & Hastings 1996; Lande 1999; Booy et al. 2000).
El recurso ostrícola que representa Crassostrea rhizophorae en esta región, no se encuentra sometido a un manejo adecuado de explotación ni de conservación debido a que los pescadores ribereños prefieren encauzar sus esfuerzos a la captura de C. virginica que forma bancos ostrícolas abundantes, sumergidos en las zonas de aguas salobres fáciles de capturar, mientras que C rhizophorae es obtenida mediante corte con machete de las raíces de mangle a las cuales se encuentra adherida. La determinación de la estructura genética poblacional y del comportamiento del flujo génico intra e intepoblacional representan sólidos criterios de utilidad para la preservación de especies, ya que son indicadores de salud poblacional, de riqueza genética y de heterogeneidad de las poblaciones sobre regiones geográficas (Bates & Innes 1995).
Información sobre la citogenética de estas especies ha sido producida mediante estudios cromosómicos y citotaxonómicos (Longwell et al. 1967; Menzel 1968; Ahmed 1973; 1975; Rodríguez-Romero et al. 1978; 1979a; 1979b; 1979c; Ladrón De Guevara et al. 1996; Zhang et al. 1999; Xu et al. 2001; Wang et al. 2001; 2004; Leitao et al. 1999; 2007; Leitao & Chaves 2008; Bouilly et al. 2008); de genética de poblaciones mediante el análisis de isoenzimas (Buroker et al. 1979; Buroker 1983; Hedgecock & Okazaki 1984; De la Rosa-Vélez & Rodríguez-Romero 1988; Perez-Enriquez et al. 2008; Enriquez-Espinoza & Grijalva-Chon 2010) de variabilidad genética molecular (Reeb & Avise 1990; Hrincevich et al. 1995; Gaffney 1996; Hare & Avise 1998) y de hibridación interespecífica (Rodríguez-Romero & Gasca-Montes de Oca 1995; 1998; Cruz et al. 2007; Leitao et al. 2007).
El objetivo del presente trabajo fue determinar si la discontinuidad de la costa producida por la presencia de la boca de Puerto Real y la dominancia del flujo de aguas oceánicas hacia el interior de la laguna de Términos, limita el libre flujo génico interpoblacional de Crassostrea rhizophorae que modifique la variabilidad genética entre dos poblaciones.
MATERIALES Y MÉTODOS
Obtención de los organismos de este estudio
La obtención de organismos se realizó en dos localidades en la costa del Estado de Campeche: estero del Pargo, ubicado entre las coordenadas 91° 44' 31" y 91° 46' 54" LO y 18° 38' 22" y 18° 40' 27" LN. El estero se encuentra comunicado con la laguna de Términos mediante una boca angosta de aproximadamente 25 m de ancho, es de forma alargada y se proyecta dentro de la Isla del Carmen en dirección noreste, con una longitud aproximada de 5 300 m y una profundidad promedio de 2 m. Los aportes de agua dulce a este estero ocurren únicamente durante la época de lluvias de julio a septiembre (Ayala-Pérez et al. 2003) El estero de Sabancuy se localiza a 910 11' 09" y 910 37' 32" LO y 180 78' 54" y 910 37' 32" LN, a una distancia aproximada de 90 km del estero del Pargo, su desembocadura a la laguna de Términos se encuentra adyacente a la boca de Puerto Real y presenta una boca artificial que permite el acceso de agua marina en las inmediaciones del puente de acceso a la población de Sabancuy, lo cual influye en la condición salobre de este cuerpo de agua. 50 ejemplares de C. rhizophorae de tallas entre 6 y 8 cm de cada sitio fueron recolectados a mano, desprendidos de las raíces de mangle.
Técnicas de laboratorio y procesamiento de muestras
Los organismos fueron trasladados al Laboratorio de Biología de la Estación El Carmen del Instituto de Ciencias del Mar y Limnología de la UNAM, se colocaron en acuarios con agua de la zona de recolección. Muestras de la porción semitransparente del músculo aductor fueron extraídas e inmediatamente se congelaron a -20 °C, homogeneizadas en un volumen igual de buffer de extracción consistente de Tris HCl 12.1 g, 336 mg de EDTA, 20 mg de NAD+, ajustado a un pH de 7 (Shaklee & Keenan 1986). Geles de almidón hidrolizado (Sigma Chemical Co.) al 12% fueron preparados con el buffer correspondiente. Se usaron tres sistemas de amortiguadores para el análisis de los diferentes sistemas enzimáticos para ser utilizados en electroforesis horizontal. Se siguieron los procedimientos recomendados por Brewer (1970), Schaal & Anderson (1974), y Shaw & Prasad (1970) para el revelado histoquímico y la determinación fenotí-pica de las muestras. Siempre que fue posible, se siguió la metodología utilizada por Tello-Cetina et al (2005). Loci y alelos revelados fueron designados por medio del sistema de nomenclatura utilizado por Shaklee & Keenan (1986). Múltiples loci de una enzima en particular fueron identificados, según su movilidad anodal relativa. Se asignó al alelo más común como 100. Un locus se consideró polimórfico si el alelo más frecuente tuvo una probabilidad menor que 95 %. (Towsend & Shing 1984) y el nivel de heterocigosis se determinó con relación a la ley del Equilibrio de Hardy-Weinberg con las pruebas de Haldane y χ2 (Miller 2000), mediante el programa TFPGA versión 1.3 (Tools for population genetic analyses), para efectuar el análisis de datos genéticos de aloenzimas de poblaciones. Los parámetros que este programa permitió determinar fueron: estadística descriptiva, estadísticos F, distancia genética, pruebas de robustez para Hardy-Weinberg y UPGMA. Se utilizaron únicamente loci polimórficos para determinar el nivel de polimorfismo.
RESULTADOS
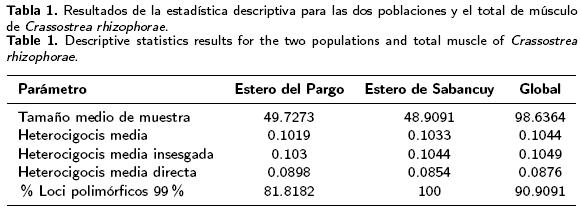
En la tabla 1, se presenta el promedio de heterocigosis y polimorfismo. El valor de la heterocigosis media poblacional fue 0.1044. El del polimorfismo en estero del Pargo, fue 45.45% y 54.54%; en el estero de Sabancuy, con valor global del 36.3636% en todas las poblaciones, los cuales indicaron la existencia de diversidad intrapoblacional con poca heterogeneidad entre ambas poblaciones de ostión.
La Tabla 2, presenta los valores globales obtenidos con las pruebas de Haldane y Chi Cuadrada para las dos poblaciones analizadas. Los loci Cat1, G6pdh, Idh1 y Mdh1 presentaron significancia para la prueba de %2, lo que significa que se apartan de la condición de equilibrio, sin embargo, estos mismos loci aparecieron como no significativos en los resultados de la prueba de Haldane. Se observa que de los 11 loci analizados que presentaron alguna variación, solo 4 de ellos (36.3%), se apartaron de la condición de equilibrio en términos de la prueba de X2 (P < 0.05).
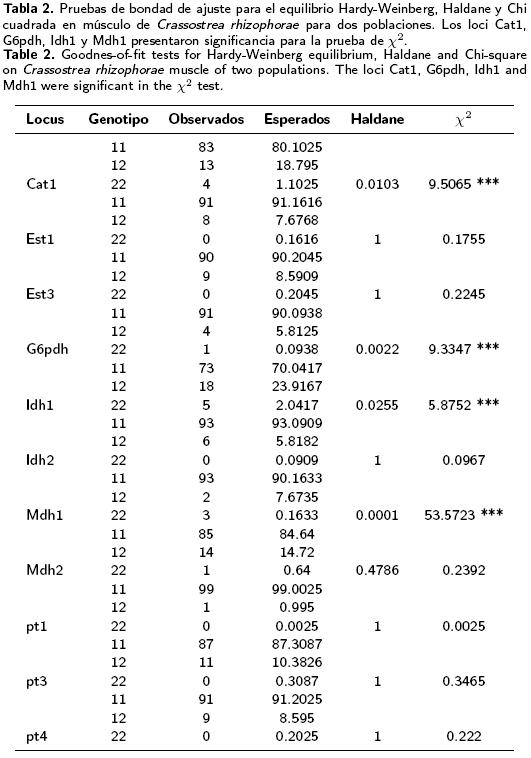
Del análisis de remuestreo por los métodos de Monte Carlo y la Cadena de Markov (Miller 2000) para cada sitio, se observó que los loci no presentaron significancia a diferencia de la prueba de X2. Los mayores valores de Fis se presentaron en Mdh1 con 0,7440 y del índice de fijación Fst, en Idh1 con 0.0869. Los totales en los loci indicaron para F = 0.1727 y Fis = 0.1574 deficiencia de heterocigotos, mientras que el de Fst = 0.0181 determinó que menos del 2% de las diferencias genéticas observadas corresponden a variación interpoblacional (Tabla 3).

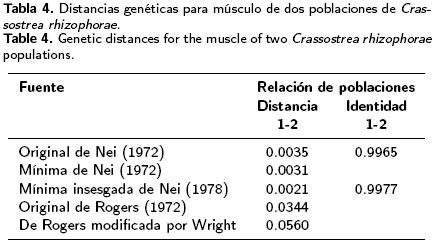
En la tabla 4, se presentan los valores resumidos de distancia genética de las poblaciones de ostión comparadas. Se observa que los obtenidos por el modelo de la distancia de Rogers modificada por Wright ( 0.0560) fueron los más altos y los de mínima insesgada de Nei (1978) = 0.0021 los de menor valor. Los de identidad original de Nei (1972) = 0.9965 y mínima insesgada de Nei (1978) = 0.9977 indicaron fuerte identidad genética.
DISCUSIÓN
Las dos poblaciones de Crassostrea rhizophorae que fueron analizadas se encuentran restringidas a la zona de manglar con influencia marina, los ostiones están adheridos a las raíces y tallos de mangle. Estas dos poblaciones están separadas en subsistemas ecológicos distintos de la Sonda de Campeche cuya definición ambiental presenta entre sí diferencias significativas (Yañez-Arancibia & Sanchez-Gil 1986). Asimismo, esta franja de distribución en la Isla del Carmen está interrumpida por la presencia de la boca de Puerto Real en el lado norte, por la cual se registra el ingreso de aguas oceánicas mediante una corriente que ingresa por el canal de la boca con un flujo neto de aguas marinas al interior de la laguna de Términos con salida por la boca del Carmen, configurando una estratificación de salinidad (Vázquez-Botello 1978). Al respecto, Bravo-Núñez & Yáñez-Arancibia (1979) han determinado que dicha corriente funciona como una barrera física para las comunidades bióticas que se encuentran a ambos extremos, siendo principalmente la causa de diferencias a nivel estructural de las comunidades nectónicas que se encuentran en la zona de la boca de Puerto Real. El sistema de corrientes que se forman por influencia de la entrada de aguas oceánicas debido a las corrientes superficiales provocadas por el viento (Martínez- López & Parés- Sierra 1998) al interior de la laguna de Términos, promueve la formación de franjas de salinidad o isohalinas que se localizan en función de los patrones de corrientes internas, del perfil de la costa y de la presencia e influencia de las aguas limnéticas que aportan los diferentes ríos que desembocan en esta laguna, entre ellos, Candelaria, Palizada y Chumpán. Este flujo de aguas de origen distinto, funciona como una barrera que limita en todas las épocas del año la dispersión homogénea de las especies, y en particular a poblaciones de Crassostrea virginica que se ubica en la zona de aguas de menor salinidad entre 5 y 20 ppm (Rogers & García-Cubas 1981; George-Zamora et al.. 2003), preferentemente en la zona de influencia de las desembocaduras de ríos y del sistema fluvio-lagunar Atasta-Pom (García-Cubas 1981) en el lado continental. Mientras que C. rhizophorae se ubica en una franja angosta en la región de influencia oceánica por encima de las 27 ppm de salinidad (Ley 1979), en la costa interior de Isla del Carmen y en el interior del estero de Sabancuy, con muy poca influencia de agua dulce en regiones de poca profundidad en donde lo somero del fondo y la evaporación contribuyen a la elevación de la salinidad de las aguas que se encuentran en la parte más distante de la desembocadura. En el estero de Sabancuy, las poblaciones reciben el impacto del estrato de aguas de alta salinidad que se forma por la presencia de aguas marinas que penetran por la boca original y por la artificial abierta por los pescadores en la región distal de la boca de Puerto Real. Por lo anterior, es necesario investigar sobre el conocimiento del flujo genético que se produce entre las poblaciones de Crassostrea rhizophorae que se encuentran a ambos lados de la discontinuidad de la costa y la posibilidad de que, al igual que como sucede en comunidades de peces de esta región, (Bravo-Núñez & Yañez-Arancibia 1979) presenten diferenciaciones significativas para la conservación de la continuidad y variabilidad genética (Reeb & Avise 1990) como ha sido indicado en C. virginica en la costa norte del Golfo de México, en la cual, Buroker (1983) mediante el análisis de isoenzimas, ha descubierto una interrupción del flujo genético en la región de la laguna Madre de Texas y un efecto de selección en dos loci asociados a las condiciones ambientales. Para el caso de las poblaciones mexicanas de C. virginica, De la Rosa-Velez & Rodríguez-Romero(1988) utilizando la misma metodología de análisis de isoenzimas, encontraron una variación clinal en donde la mayor similitud genética se encontró entre las poblaciones del sureste de México que ha sido interpretada como una tendencia a la evolución independiente de las poblaciones de ostiones a ambos lados de la laguna Madre de Tamaulipas. Estos resultados son congruentes con la caracterización de la circulación baroclínica en el Golfo de México definida por Vidal-Lorandi et al. (2003a) y la influencia de los vórtices (Vidal-Lorandi et al. 2003b).
Según Ahmed (1975), Crassostrea rhizophorae se encuentra en el límite de su dispersión biogeográfica hacia el norte del Continente Americano, lo que justifica que se realicen estudios sobre la evaluación de la riqueza alélica de estas poblaciones que supuestamente ya no avanzan hacia el norte y el impacto que puede ocasionar la discontinuidad del hábitat.
Se realizó este estudio mediante el análisis enzimático, con el fin de poder comparar con mayor objetividad los resultados aquí obtenidos con los ya publicados con esta misma metodología en el género Crassostrea, con la certeza de que este tipo de análisis es vigente en la determinación de la estructura genética poblacional (Mamuris et al. 1998; 1999; Ridgway 2005) y para el estudio de la variabilidad genética y diferenciación poblacional (Backeljau et al. 1994; Boisselier-Dubayle & Gofas 1999; Reed & Frankham 2001; Ridgway 2005) y porque aunque los métodos de varianza molecular del ADN ofrecen mayor precisión para detectar las diferencias entre los individuos y poblaciones, el número limitado de marcadores que han sido identificados para Crassostrea rhizophorae, no necesariamente ofrecen mayor precisión que la que aportan los estudios de aloenzimas, los que permiten además comparar la información previamente producida con estos procedimientos en ostiones (Bossart & Prowell 1998; Reed & Frankham 2001). Los valores de polimorfismo de 36.35 % y de heterocigocidad media de 0.1044 son relativamente bajos si se comparan con el promedio encontrado para diferentes invertebrados marinos (Ayala & Kiger 1984), pero si se encuentran dentro de los valores determinados para especies de moluscos (Zouros & Foltz 1984; Saavedra et al. 1993), debido a que la deficiencia de heterocigotos es una característica común encontrada en estos organismos (Mamuris et al. 1998; Boisselier-Dubayle & Gofas 1999). Los niveles bajos de variabilidad pueden ser atribuidos a diversas causas, como el esfuerzo de explotación de las poblaciones en el área, al tamaño de muestra utilizado (Heist et al. 1995) o la eficiencia del flujo génico intrapoblacional e interpoblacional, entre otros. En los ostiones del género Crassostrea se ha determinado heterocigocidad entre 6 y 22% (Buroker et al. 1979). En C. rhizophorae, los valores de Fst determinados indican que no se puede considerar al efecto Wahlund como causa de este fenómeno, aunque se mantiene la posibilidad de que existan movimientos interpoblacionales como migrantes (0.0181). La determinación de identidad y distancia genética son de utilidad cuando los valores de heterocigosis resultaron bajos (Bierley et al. 1996). Los valores de distancia genética bajos, son comunes para especies con vida planctónica de gran dispersión (Toro & González 2009; Ward et al. 1992), principalmente debido a que las larvas y huevos pelágicos son conducidos por movimientos pasivos de las corrientes oceánicas durante semanas, transportándolas a grandes distancias y favoreciendo la presencia de poblaciones genéticamente homogéneas (Bohonak 1999; Colin et al. 2006). En ambas poblaciones de ostiones, la distancia e identidad genética cuantificadas, se encontraron dentro de los valores que caracterizan a las poblaciones bien mezcladas de organismos con vida planctónica de dispersión profusa (Burton 1983; Hedgecock 1986).
Las medidas de distancia y el análisis UPGMA fueron similares al agrupar las poblaciones, estos resultados son congruentes con el patrón de corrientes que privan en el área del Golfo de México y que propicia la estructura y variación genética observada en estas poblaciones. No obstante, se debe tener en cuenta que otras variaciones estocásticas como los vientos de superficie, remolinos localizados y diferencias significativas en la salinidad, podrían afectar la dispersión de larvas y modificar el reclutamiento de éstas en localidades como lo señalado por Buroker (1983) y Bucklin et al. (1989), quienes han concluido que tales eventos pueden mantener el estado de discreción de las poblaciones en forma espacial o temporal, lo que finalmente evita la homogeneización del plancton durante el transporte. Al respecto, en C. virginica de bahía de Chesapeake, las diferencias interpoblacionales encontradas podrían indicar un patrón sutil de aislamiento por distancia (Colin et al. 2006).
Se concluye que las dos poblaciones de Crassostrea rhizophorae estudiadas reflejan sensibilidad a los procesos adaptativos interpoblacionales que se producen por la discontinuidad genética del medio geográfico pero aún sin detrimento de sus niveles de variabilidad genética y en consecuencia, no reflejan en el presente fragilidad genética poblacional que las exponga a proceso de formación de metapoblaciones.
Finalmente, los resultados reflejan la estructura genética poblacional de ambas poblaciones estudiadas en el presente. La continuidad de estas investigaciones relacionadas con aspectos ecológicos y oceanográficos son de interés debido a que permiten una visión más detallada de los factores que podrían afectar la genética de las poblaciones de Crassostrea rhizophorae del Sureste de México ante el impacto de los factores geográficos y de la dinámica ambiental de este medio acuático.
AGRADECIMIENTOS
A Arturo Toledano Granados de la estación Puerto Morelos del Instituto de Ciencias del Mar y Limnología de la UNAM por su valiosa ayuda en el trabajo de campo, a Ernesto Guerrero Padilla por la revisión crítica del manuscrito, a David Xibillé Friedman por su ayuda en la traducción del resumen y a los tres revisores anónimos por sus valiosas sugerencias.
LITERATURA CITADA
Ahmed M (1973) Cytogenetics of Oysters. Cytology 38(2): 337-346. [ Links ]
Ahmed M (1975) Speciation in living oysters. Advances in Marine Biology 13: 357-397. [ Links ]
Ayala JF, Kiger JA (1984) Modern Genetics. The Benjamin/Cummings Publishing Company, Inc California, USA. 923 pp. [ Links ]
Ayala-Pérez LA, Ramos-Miranda J, Flores-Hernández D (2003) La comunidad de peces de la Laguna de Términos: estructura actual comparada. Rev. Biol. Trop. 51(3-4): 783-79. [ Links ]
Backeljau T, Bouchet P, Gofas S, de Bruyn L (1994) Genetic variation, systematic and distribution of venerid clam Chamelea galline. J Mar Biol Ass UK 74: 211-223. [ Links ]
Bates JA, Innes DJ (1995) Genetic variation among populations of Mytilus spp. in eastern Newfoundland. Marine Biology 124: 417-424. [ Links ]
Bierley AS, Alicock AL, Torpe JP (1996) Biochemical genetic evidence supporting the taxonomic separation of Loligo edulis and Loligo chinensis (Cephalopoda:Teuthoidea) from the genus Loligo. Marine Biology 127: 97-104. [ Links ]
Bohonak AJ (1999) Dispersal, gene flow, and population structure. The Quarterly Review of Biology 74(1): 21-45. [ Links ]
Boisselier-Dubayle MC, Gofas S (1999) Genetic relationships between marine and marginal-marine populations of Cerithium species from the Mediterranean Sea. Marine Biology 135(4): 671-682. [ Links ]
Bossart JL, Prowell DP (1998) Genetic estimates of population structure and gene flow: limitations, lessons and new directions. TREE 13(5): 201-207. [ Links ]
Booy G, Hendriks RJJ, Smulders MJM, Van Groenendal D, Vosman B (2000) Gene diversity and the survival of populations. Plant Biology 2: 379-395. [ Links ]
Bravo-Núñez E, Yáñez-Arancibia A (1979) Ecología en la Boca de Puerto Real, laguna de Términos I. Descripción del área y análisis estructural de las comunidades de peces. Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 6(1): 69-118. [ Links ]
Brewer GY (1970) An introduction to isozyme techniques. Academic Press, N.Y. USA. 186 pp. [ Links ]
Bouilly K, Chaves R, Leitao A, Benabdelmouna A, Guedes-Pinto H (2008) The chromosomal organization of simple sequence repeats in the Pacific oyster (Crassostrea gigas): (GGAT)(4), (GT)(7) and (TA)(10) chromosome pattern. Journal of Genetics 87(2): 119-125. [ Links ]
Bucklin A, Rienecker MM, Mooers CNK (1989) Genetic traces of zooplankton transport in coastal filaments of Northern California. Journal of Geophysical Research 94: 8277-8288. [ Links ]
Buroker NE, Hershberger WK, Chew KK (1979) Population genetics of the family Ostreidae. II. Interspecific studies of the genera Crassostrea and Saccostrea. Marine Biology 54: 171-184. [ Links ]
Buroker NE (1983) Population genetics of the American oyster Crassostrea virginica along the Atlantic coast and the Gulf of Mexico. Marine Biology 75: 99-112. [ Links ]
Burton RS (1983) Protein polymorphisms and genetic differentiation of marine invertebrate populations. Marine Biology Letters 4: 193-206. [ Links ]
Colin GR, Kennedy TP, Matthew PH (2006) Isolation by distance in the Eastern Oyster, Crassostrea virginica in Chesapeake Bay. Journal of Heredity 97(2):158-170. [ Links ]
Cruz P, Yañez-Jácome A, Ibarra AM, Rangel-Becerril J (2007) Isolation and Characterization of microsatellite loci in the Pacific pleasure oyster, Crassostrea corteziensis, and their cross-species amplification in four other oyster species. Molecular Ecology Notes 7: 448-450. [ Links ]
De la Rosa-Vélez J, Rodríguez- Romero F (1988) Applicability of genetic variability measurements to the fishery of the American oyster Crassostrea virginica Gmelin in the Gulf of Mexico. Ciencias Marinas 14(4): 43-56. [ Links ]
Endler JA (1973) Gene flow and population differentiation. Science 179(4070): 243-250. [ Links ]
Enriquez-Espinoza TL, Grijalva-Chon JM (2010) Variabilidad genética de Crassostrea gigas y Crassostrea corteziensis de un laboratorio de producción del noroeste de México. Ciencias Marinas 36(4): 333-344. [ Links ]
Gaffney P (1996) Nuclear DNA markers for Crassostrea species identification. Journal of Shellfish Research 15(2): 510-511. [ Links ]
García-Cubas A (1981) Moluscos de un sistema lagunar tropical en el sur del Golfo de México (laguna de Términos, Campeche). Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México, Publicación Especial Número 5. 182 pp [ Links ]
George-Zamora A, Sevilla-Hernández ML, Aldana-Aranda D (2003) Ciclo gonádico del ostión americano Crassostrea virginica (Lamellibranchia: Ostreidae) en Mecoacán, Tabasco, México. Rev. Biol. Trop. 51 Supl. 4: 109-117. [ Links ]
Hare MP, Avise JC (1998) Population structure in the American oyster as Inferred by nuclear gene genealogies. Molecular Biology and Evolution 15(2): 119-128. [ Links ]
Harrison S, Hastings A (1996) Genetic consequences of metapopulation structure. Trends in Ecology and Evolution 11: 180-183. [ Links ]
Heist EJ, Graves JE, Musick JA (1995) Population genetics of the Sanbar shark (Carcharhinus plumbeus) in the Gulf of Mexico and Mid Atlantic Bight. Copeia 1995(3): 555-562. [ Links ]
Hedgecock D (1986) ¿Is gene flow from pelagic larval dispersal important in the adaptation and evolution of marine invertebrates? Bulletin of Marine Science 39: 550-564. [ Links ]
Hedgecock D, Okazaki NB (1984) Genetic diversity within and between populations of American oysters (Crassostrea). Journal International of Malacology 25(2): 535-549. [ Links ]
Hrincevich AW, Foltz DW, Ya-Ping Hu (1995) Discrimination between two closely related species of oysters, Crassostrea virginica and C. rhizophorae, using nucleotide sequence variation of the mitochondrial cytochrome oxidase I gene. Journal of Shellfish Research 14 (1): 269. [ Links ]
Ladrón De Guevara B, Winkler F, Rodríguez-Romero F, Palma-Rojas C (1996). Comparative Karyology of four American oyster species. The Veliger 39(3): 260-266. [ Links ]
Lande R, Barrowclough GF (1987) Effective population size, genetic variation, and their use in population management. En: Soulé ME (ed) Viable Populations for Conservation. New York: Cambridge University Press 87-123. [ Links ]
Lande R (1999) Extinction risks from anthropogenic, ecological, and genetic factors. En: Landweber LA and Dobson AP (eds) Genetics and Extinction of Species. Princeton NJ Princeton University Press 1-22. [ Links ]
Leitao A, Thiriot-Quievreux C, Boudry P, Malhiero I (1999) A "G" chromosome banding study of three cupped oysters species: Crassostrea gigas, Crassostrea angulata and Crassostrea virginica (Mollusca: Bivalvia). Genetics Selection Evolution 31: 519-527. [ Links ]
Leitao A,Chaves R, Santos S, Guedes-Pinto H, Boudry P (2007) Interspecific hybridization in oyster: Res-triction enzyme digestion chromosome banding confirms Crassostrea angulata x Crassostrea gigas F1 hybrids. Journal of Experimental Marine Biology and Ecology 343(2): 253-260. [ Links ]
Leitao A, Chaves R (2008) Banding for Chromosomal Identification in Bivalves: A 20-year History. Dynamic Biochemistry, Process Biotechnology and Molecular Biology 2 (special Issue 1), 44-49. Global Science Books. [ Links ]
Ley LF (1979) Algunos factores ecológico-abióticos en estero Pargo, Campeche. Tesis profesional, Facultad de Ciencias Universidad Nacional Autónoma de México. 51 pp [ Links ]
Longwell AC, Stiles SS, Smith DG (1967) Chromosome complement of the American oyster Crassostrea virginica as seen in meiotic and cleaving eggs. Canadian Journal of Genetics and Cytology 9: 845-856. [ Links ]
Mamuris Z, Apostolidis AP, Triantaphyllidis C (1998) Genetic protein variation in red mullet (Mullus barbatus) and striped red mullet (M. surmuletus) populations from the Mediterranean Sea. Marine Biology 130: 353-360. [ Links ]
Mamuris Z, Stamatis C, Triantaphyllidis C (1999) Intraspecific genetic variation of striped red mullet (Mullus surmuletus L.) in the Mediterranean Sea assested by allozyme and random amplified polymorphic DNA (RAPD) analysis. Heredity 83(1): 30-38. [ Links ]
Martinez-López B, Parés-Sierra A (1998) Circulación del Golfo de México inducida por mareas, viento y la corriente de Yucatán. Ciencias Marinas 24(1): 65-93. [ Links ]
Menzel RW (1968) Cytotaxonomy of species of clams (Mercenaria) and Oysters (Crassostrea).Proceedings of the Symposium on Mollusca, Marine Biological Association of India. Part 1, 75-84. [ Links ]
Miller M (2000) TFPGA, a Windows program for the analyses of allozyme and molecular population genetic data. Version 1.3. Dept. of Biological Sciences Northern Arizona University. [ Links ]
Nei M (1972) Genetic Distance between populations. The American Naturalist 106: 283-292. [ Links ]
Nei M (1978) Estimation of average heterocigosity and genetic distances from small number of individuals. Genetics 80: 583-590. [ Links ]
Perez-Enriquez R, Avila S, Ibarra AM (2008) Genética poblacional del ostión de placer Crassostrea corteziensis en el Golfo de California. Ciencias Marinas 34(4): 479-490. [ Links ]
Reed DH, Frankham R (2001) How closely correlated are molecular and quantitative measures of genetic variation? A meta-analysis. Evolution 55(6): 1095-1103. [ Links ]
Reeb CA, Avise JC (1990) A genetic discontinuity in a continuously distributed species: mitochondrial DNA in the American oyster Crassostrea virginica. Genetics 124: 397-406. [ Links ]
Ridgway T (2005) Allozyme electrophoresis still represents a powerful technique in management of coral reefs. Biodiversity and Conservation 14: 135-149. [ Links ]
Rodríguez- Romero F, Uribe M, Laguarda A (1978) Cytogenetic study of an oyster population of Crassostrea virginica. Venus, the Japanese Journal of Malacology 37: 83-86. [ Links ]
Rodríguez- Romero F, Laguarda A, Diupotex M (1979a) The karyotype of Crassostrea rhizophorae (Guilding, 1828). Venus, the Japanese Journal of Malacology 38: 135-140. [ Links ]
Rodríguez- Romero F, Uribe M, Laguarda A (1979b) The karyotype of Crassostrea corteziensis Hertlein 1951 (Mollusca: Ostreidae). Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 6(1): 15-18. [ Links ]
Rodríguez-Romero F, Laguarda A, Uribe M, Rojas ML (1979c) Distribution of "G" Bands in the karyotype of Crassostrea virginica. Venus, the Japanese Journal of Malacology 38(3): 180-184. [ Links ]
Rodríguez-Romero F, García-Saez C, Laguarda A (1988) Electrophoretic patterns variation in two oyster populations of Crassostrea corteziensis from the Mexican coast. Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 15(1): 177-184. [ Links ]
Rodríguez-Romero F, Gasca-Montes de Oca M (1995) Chromosome complements of the experimental interspecific hybrid of the oysters Crassostrea virginica (Gmelin) vs Crassostrea corteziensis (Hertlein). Marine Research 4(2): 127-133. [ Links ]
Rodríguez-Romero F, Gasca-Montes de Oca M (1998) Chromosomes of the experimental hybrid of Crassostrea virginica Gmelin 1791 and Crassostrea rhizophorae Guilding 1828 (Pseudolamellibranchiata: Ostreidae). Ciencias Marinas 24(1): 55-63. [ Links ]
Rogers P, García-Cubas A (1981) Evolución gonádica a nivel histológico del ostión Crassostrea virginica (Gmelin, 1791) del sistema fluvio lagunar Atasta-Pom, Laguna de Términos, Campeche, México. Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 1(8): 1-20. [ Links ]
Rose CG, Paynter KT, Hare MP (2006) Isolation by Distance in the Eastern Oyster, Crassosotrea virginica, in Chesapeake Bay. Journal of Heredity 97(2): 158-170. [ Links ]
Saavedra C, Zapata C, Guerra A, Alvarez G (1993) Allozyme variation in european populations of the oyster Ostrea edulis. Marine Biology 115: 85-95. [ Links ]
Shaklee JB, Keenan, CP (1986) A practical laboratory guide to the techniques and methodology of electrophoresis and its application to fish fillet identification. CSIRO Marine Research Laboratories Australia Report 177. [ Links ]
Schaal BA, Anderson WW (1974) An outline of techniques for starch gel electrophoresis of enzymes from the American oyster Crassostrea virginica G. Technical Report Series. Georgia Marine Science Number 74-3. [ Links ]
Shaw RCh, Prasad R (1970) Starch gel electrophoresis of enzymes. A compilation of recipes. Biochemical Genetics 4: 297-320. [ Links ]
Tello-Cetina JA, Rodríguez-Gil LA, Rodríguez-Romero F (2005) Genética poblacional del caracol rosado Strombus gigas en la Península de Yucatán: Implicaciones para su manejo y pesquería. Ciencias Marinas 31(2): 379-386. [ Links ]
Toro JE, González CP (2009) La estructura genética de la ostra chilena (Ostrea chilensis Philippi, 1845) en poblaciones naturales del sur de Chile, basada en análisis con marcadores RAPDs. Revista de Biología Marina y Oceanografía 44(2): 467-476. [ Links ]
Towsend DR, Shing RS (1984) Genetic variation for a monomer and dimmer equilibria of Esterase 5 in Drosophila pseudoobscura, D. persimilis and D. miranda. Canadian Journal of Genetics and Cytology 28: 374-381. [ Links ]
Vázquez-Botello A (1978) Variación de los parámetros hidrológicos en las épocas de sequía y lluvias (mayo y noviembre de 1974) en la Laguna de Términos, Campeche, México. Anales del Instituto de Ciencias del Mar y Limnología, Universidad Nacional Autónoma de México 5(1): 1-306. [ Links ]
Vidal-Lorandi VM, Vidal-Lorandi F, Zambrano-Salgado L, Portilla-Casillas J (2003a) Avances sobre la circulación de las masas de agua en el Golfo de México. En: Soto LA (ed) Agustín Ayala-Castañares, Universitario impulsor de la investigación científica. Universidad Nacional Autónoma de México. 139-154. [ Links ]
Vidal-Lorandi F, Vidal Lorandi VM, Zambrano-Salgado L, Portilla-Casillas J (2003b) Simulaciones numéricas de la interacción y evolución de vórtices en el Golfo de México. En: Soto LA (ed) Agustín Ayala-Castañares, Universitario impulsor de la investigación científica. Universidad Nacional Autónoma de México. 155-160. [ Links ]
Wang Y, Wu Z, Guo X (2001) A centromeric satellite sequence in the Pacific oyster (Crassostrea gigas Thunberg) identified by fluorescence in situ hybridization. Marine Biotechnology 3(5): 486-492. [ Links ]
Wang Y, Wu Z, Guo X (2004) Differences in the rDNA-bearing chromosome divide the Asian-Pacific and the Atlantic species of Crassostrea (Bivalvia,Mollusca). Biological Bulletin 206(1): 46-54. [ Links ]
Ward RD, Skibinski DOF, Woodwark M (1992) Protein heterozygosity, protein structure, and taxonomic differentiation. Evolutionary Biology 26: 73-159 [ Links ]
Xu Z, Guo X, Gaffney PM, Pierce JC (2001) Chromosomal location of the major ribosomal RNA genes in Crassostrea virginica and Crassostrea gigas. The Veliger 44(1): 79-83. [ Links ]
Yáñez-Arancibia A, Sanchez-Gil P (1986) Los peces demersales de la plataforma continental del sur del golfo de México. 1. Caracterización ambiental, ecología y evaluación de las especies, poblaciones y comunidades. Anales del Instituto de Ciencias del Mar y Limnología, Publicación Especial Número 9. 230 pp. [ Links ]
Zhang Q, Yu G, Cooper RK, Tiersch TR (1999) High-resolution analysis of karyotypes prepared from different tissues of eastern Crassostrea virginica. Journal of Shellfish Research 18(1): 115-120. [ Links ]
Zouros E, Foltz DW (1984) Possible explanation of heterozygote deficiency in bivalve mollusks. Journal International of Malacology 25(2): 583-591. [ Links ]














