Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Universidad y ciencia
versión impresa ISSN 0186-2979
Universidad y ciencia vol.25 no.2 Villahermosa ago. 2009
Artículos
Aislamiento, identificación y caracterización de bacterias ácido lácticas para la elaboración de queso crema tropical
Isolation, identification and characterisation of lactic acid bacteria for the manufacture of tropical cream cheese
B Ramos–Izquierdo1, A Bucio–Galindo*1, C Bautista–Muñoz1, E Aranda–lbáñez1, F Izquierdo–Reyes1
1 Colegio de Postgraduados, Campus Tabasco. Periférico Carlos A. Molina km. 3.5 Ap. 24. Ciudad de Cárdenas, estado de Tabasco, 86500. *Correo electrónico: adbucio@colpos.mx
Artículo recibido: 10 de diciembre de 2006
Aceptado: 30 de julio de 2009
RESUMEN
Se aislaron, identificaron y caracterizaron 20 cepas de bacterias ácido lácticas (BAL) de un "queso crema tropical" tradicionalmente fabricado con leche bronca. Las 20 cepas de BAL utilizadas crecieron en leche descremada y cada una de ellas fue evaluada mediante su poder acidificante (pH) en cuatro tiempos de incubación (6, 24, 48 y 72 h) y tres temperaturas de fermentación (30, 37 y 42 °C). En base al pH más bajo, se seleccionaron seis cepas para fabricar un "queso tipo crema tropical" con leche pasteurizada. Las cepas fueron caracterizadas mediante pruebas bioquímicas (fermentación de carbohidratos y producción de amonio a partir de arginina) y de secuenciación del 16S rDNA. Se identificaron cinco especies de Lactobacillus fermentum y una especie de Lactobacillus pentosus. Entre las temperaturas de fermentación y el tiempo de incubación se estimaron diferencias significativas (p < 0.05) en cada una de las cepas. En las temperaturas de 37 °C y 42 °C se produjeron los valores de pH más bajos a las 72 horas de incubación. Además, el ácido láctico, ácido acético, ácido propiónico y ácido butírico en leche fermentada se evaluaron con cada cepa y no hubo diferencias significativas en los niveles de producción de dichos metabolitos (p > 0.05). Asimismo, se realizaron pruebas que incluyeron cuentas totales de flora no láctica, cuenta de BAL, coliformes totales, Escherichia coli, hongos y levaduras en el queso crema elaborado con leche pasteurizada y como referencia se uso al queso crema artesanal. Las poblaciones más bajas de esos microorganismos se encontraron en el queso fabricado con leche pasterizada (p < 0.05). El número de bacterias lácticas no resultó diferente entre ambos tipos de queso (p > 0.05).
Palabras clave: Bacterias ácido lácticas, 16S rDNA, queso, cultivos iniciadores.
ABSTRACT
The purpose of this study was the isolation, identification and characterisation of 20 strains of lactic acid bacteria (LAB) from a "tropical cream cheese" traditionally prepared with non–pasteurised milk. The 20 strains of LAB were grown in de–creamed milk, and each one was evaluated with respect to its acidifying capacity (pH) at four incubation times (6, 24, 48 and 72 h) and three incubation temperatures (30, 37 and 42 °C). Based on the lowest pH, 6 strains were selected to prepare a "tropical type cream cheese" with pasteurised milk. The strains were characterised by means of biochemical tests (fermentation of carbohydrates and ammonia production from arginine) and 16S rDNA sequencing methods. Five species of L. fermentum and one species of L. pentosus were identified. Each strain presented significant differences (p < 0.05) with respect to fermentation temperatures and incubation times. The lowest pH values were recorded at temperatures of 37 °C and 42 °C after 72 h of incubation. Lactic acid, acetic acid, propionic acid and butyric acid were evaluated in the fermented milk for each strain, and there were no significant differences in the production levels of these metabolites (p > 0.05). Total counts were recorded for non–lactic flora, LAB, total coliforms, Escherichia coli, yeast and moulds in the cream cheese made with pasteurised milk. The artisanal cream cheese was used as a reference. The lowest counts of microorganisms were found in the cheese prepared with pasteurized milk (p < 0.05). The number of lactic bacteria was similar in both types of cheese (p > 0.05).
Key words: Lactic acid bacteria, 16S rDNA, cheese, starter cultures.
INTRODUCCIÓN
El queso crema tropical es un queso de gran aceptación en el sureste de México, principalmente elaborado en los estados de Chiapas y Tabasco. Aunque se desconoce su origen histórico, su manufactura de elaboración a nivel artesanal, es muy similar en ambos estados (Villegas 1989). El queso crema tropical, se elabora de forma tradicional, mediante la acidificación de la leche bronca con suero ácido del día anterior. Posteriormente, un ml de cuajo de ternera se agrega por cada 10 L de leche para obtener la cuajada, la cual se deja reposar por un periodo de 24 h. Después, la cuajada se deja escurrir por un día, se sala, se moldea y se prensa durante cuatro horas y finalmente se empaca en papel encerado (Villegas 1989).
Un problema con ese proceso es que la Norma Oficial Mexicana 121–SSA1–1994 exige que la leche sea pasteurizada (Anónimo 1994). La pasteurización de la leche permite obtener productos lácteos con mejores características higiénicas, pues con este proceso se destruyen la mayor parte de los microorganismos patógenos que podrían estar presentes en la leche bronca. Sin embargo, para que se pueda fabricar el queso con leche pasterizada, es necesario agregar microorganismos acidificantes a la leche, para cubrir con esta parte del proceso. Los microorganismos a utilizar deben ser parte de la biota responsable de impartir características típicas de los quesos que se requiere fabricar.
Los consumidores aprecian los quesos artesanos por sus singulares características de sabor y aroma, que es generalmente atribuida a la actividad metabólica de la microbiota autóctona presente en la leche cruda (Beuvier et al. 1997). La utilización de cultivos lácticos comerciales ayuda a llevar a cabo el proceso de acidificación, pero generalmente se asocian con otras propiedades organolépticas, pues esos microorganismos han sido seleccionados de quesos de otras regiones. Además, el uso de cultivos lácticos industriales en substitución de la microbiota autóctona, podría llevar a la perdida de las cepas autóctonas en el largo plazo (González et al. 2003).
Generalmente, la selección de cepas lácticas autóctonas ha ayudado a obtener productos lácteos con características similares a los productos elaborados con leche cruda (Centeno et al. 1996; Méndez et al.1998). Existe un gran interés en el estudio de los cultivos lácticos autóctonos para emplearlos en la producción industrial de quesos tradicionales que conserven sus propiedades típicas y sean simultáneamente más seguros desde el punto de vista higiénico (Centeno et al. 1996; Méndez et al. 1998). Los objetivos de este trabajo fueron aislar, identificar y caracterizar a un cultivo láctico con bacterias autóctonas, las cuales fueron inoculadas en leche pasteurizada para obtener un producto con el perfil de queso deseado.
MATERIALES Y MÉTODOS
Aislamiento de BAL. Las cepas de BAL fueron aisladas a partir de muestras de queso crema tropical artesanal, el cual fue elaborado por empresas regionales localizadas en el municipio de Huimanguillo, Tabasco, México. Las muestras de 10 g de queso se cortaron asépticamente y se homogenizaron en 90 ml de PBS [NaCl (8.0 g L–1), KCl (0.2 g L–1), Na2HPO4(1.15 g L–1), K2HPO4 (0.2 g L–1), agua destilada estéril a 1 L, pH = 7.3 ± 0.2 a 25 °Cj.
Las diluciones fueron sembradas por duplicado en medio de cultivo Man, Rogosa, Sharpe (MRS) con la técnica de vaciado en placas, incubadas a 30 °C durante 48 h. Las bacterias fueron aisladas mediante resiembras sucesivas por el método de estría cruzada y se formó una colección de 20 cepas de BAL. La pureza e identificación de las BAL fue evaluada de acuerdo a Walstra et al.(2001). Aunque en el medio MRS crecieron varios géneros de bacterias lácticas, solamente se seleccionaron aquellas identificadas como Lactobacillus para estudios posteriores.
Identificación bioquímica de las BAL de acuerdo al tipo de fermentación producida. Las seis cepas seleccionadas fueron transferidas a medio de cultivo líquido MRS con campana de Durham, se incubaron a 30 °C por 48 h, para evaluar la formación de burbujas, que fueron considerados como positivos a la formación de CO2. El sistema API 50 CH (BioMerieux) fue usado para la identificación bioquímica. Además, la prueba de hidrólisis de arginina, en MRS modificado (sin extracto de carne y con arginina, 0.3% w/v; citrato de sodio, 0.2%; y glucosa, 0.05%) se realizó para detectar la producción de amonio. Después de cinco días de incubación se agregaron cinco gotas del reactivo de Nessler para detectar la presencia de amonio (Sánchez et al. 2000).
Identificación molecular de las BAL mediante amplificación y secuenciación del gen 16S DNAr. El DNA genómico de las seis cepas presuntivamente caracterizadas como Lactobacillus spp por pruebas morfológicas y bioquímicas fue aislado a partir de 1.5 ml de un cultivo de toda la noche en caldo MRS, mediante lisis mecánica con perlas de vidrio y siguiendo en método descrito por Ausebel et al. (1994). El DNA extraído fue usado como molde en las subsecuentes reacciones de amplificación por Polymerase Chain Reaction (PCR) con un volumen final de 25 μl usando un termociclador de DNA (TECHNE Inc. Burlington, NJ, U.S.A.).
Los iniciadores 16S SENSE (5'–TGCCTAATACATGCAAGTCG–3') y 16S ANTISENSE (5'–ACCGCTACACATGGAGTTC–3') fueron diseñados de regiones conservadas de genes 16S rDNA de especies del género Lactobacillus, a partir de un alineamiento múltiple realizado con el software CLUSTAL X versión 1.8 (Thompson et al. 1997). El gen 16S rDNA se ha usado sistemáticamente para caracterizar a estas y muchas otras bacterias (Boldo et al. 2003).
Las secuencias fueron obtenidas de la base de datos de la National Center for Biotechnology Information (Anónimo 2009). La edición de los alineamientos se realizó con el programa GeneDoc versión 2.3 (Nicholas & Nicholas 1997). El alineamiento gráfico mostró las regiones con un alto grado de identidad, mismos que fueron empleados para el diseño de los iniciadores. Los iniciadores fueron analizados con el software Oligo versión 4.1, para ajustes y cálculo de la Tm y Ta (Temperatura de fusión y alineamiento, respectivamente). Los iniciadores fueron sintetizados por Invitrogen. La mezcla de PCR contenía 10 ng de DNA molde, regulador de la Taq DNA Polimerasa (1X) (Invitrogen, Brazil), MgCl2 (2 mM), cada uno de los desoxinucleótidos trifosfatos (200 μm) (Invitrogen), los iniciadores (16S SENSE y 16S ANTISENSE) (0.4 μm), Taq DNA Polimerasa (1U) (Invitrogen). El DNA fue amplificado con 35 ciclos usando las condiciones de amplificación: desnaturalización a 94 °C, 1 min; alineamiento a 54 °C, 1 min; polimerización a 72 °C, 1 min. La desnaturalización inicial fue ejecutada a 94 °C por 5 min y un paso de extensión final a 72 °C por 10 min. Los productos de PCR (10 ml de cada mezcla de reacción) fueron analizados en un gel de agarosa al 1 % (p/v) (Invitrogen) con regulador TAE 1X (Trisacetato 40 mM, EDTA 1 mM, pH 8.0) y teñido con Bromuro de etidio. Un marcador de talla molecular de DNA de 100 pb fue usado como estándar. Los geles fueron corridos durante dos horas.
La purificación de los productos de PCR a partir de agarosa se llevó a cabo con el kit QIAquick Gel Extraction (Qiagen Inc., Valencia, California) aproximadamente a 100 V y el DNA fue visualizado con un transiluminador UV.
Los fragmentos de DNA del gen 16S de las cepas fueron enviados a secuenciar a la Unidad de Diagnóstico y Análisis Molecular del Instituto Nacional de Salud Pública (INSP) en Cuernavaca, Morelos.
Huella genética empleando el análisis RAPD–PCR. El RAPD–PCR (Polimorfismo del DNA Amplificado al Azar) fue realizado a cada una de las cepas de BAL seleccionadas, empleando 10 ng de DNA genómico y cada uno de los iniciadores OPA–18 (5'–GGACTGCAGA–3') Y OPE–04 (5'–GTGACATGCC–3'), siguiendo los métodos previamente descritos (Lockhart et al. 1997; Boldo et al. 2003).
Relaciones de similitud entre las secuencias de genes 16S rDNA de Lactobacillus descritos y las cepas BAL aisladas. Las secuencias de nucleotides que codifican los genes del 16S DNAr de bacterias BAL (3L, 5L, 6L, 14L, 19L y 20L) fueron depositadas en la base de datos de la NBCI (http://www.ncbi.nlm.nih.gov); posteriormente se compararon con las secuencias disponibles en el GenBank Database: http://www.ncbi.nlm.nih.gov/entrez/query. Cinco alineamientos múltiples de secuencias de nucleótidos de genes 16S rDNA se realizaron para 47 especies del género Lactobacillus y Pediococcus acidolactici. Se construyó un árbol de similitud con MEGA (Molecular Evolutionary Genetics Analysis) versión 2.1 (Kumar et al. 2001), usando el método de agrupamiento de neighbor–joining y el índice P (Poisson). La evaluación estadística incluyó 1 000 aleatorizaciones tipo bootstrap.
Evaluación de la capacidad de disminución de pH de las cepas BAL aisladas. Cada una de las cepas (500 micro litros; 108 UFC ml–1) fue inoculada a tubos con 50 ml de leche semidescremada (1.6 g l–1 de grasa) ultra pasteurizada, se incubaron a 30, 37 y 42 °C. El pH fue medido a las 0, 6, 12, 24, 48 y 72 h de incubación. En cada uno de los tratamientos se hicieron cinco repeticiones.
Análisis de los productos de la fermentación. La leche fermentada (72 h de incubación) con cada una de las cepas fue centrifugada a 13,000 r.p.m. por 10 minutos, el sobrenadante fue colectado, pasteurizado a 85 °C por 5 min y usado para el análisis de las siguientes variables: ácido láctico, ácido acético, propiónico y butírico. Los precursores y metabolitos del ácido láctico fue determinada por el método de Taylor citado por Madrid et al. (1999), mientras que el resto fueron cuantificados por cromatografía de gases (Erwin et al. 1961). En cada uno de los tratamientos se hicieron cinco repeticiones.
Análisis fisicoquímico del queso crema tropical. Para la determinación del ácido láctico, la acidez total fue medida usando como medio de titulación volumétrica NaOH 0.1 N y el indicador de fenolftaleína. La acidez total se reportó en % de ácido láctico (g de materia seca)–1.
Para la determinación del pH, 10 g de muestra se diluyeron en 90 ml de agua y se tomó lectura utilizando potenciómetro digital Hanna.
La densidad (g cm–3) de las muestras fue calculada mediante cubos de 3x3x3 cm de lado con una regla con escala vernier y su masa fue medida en una balanza granataria con una aproximación de ± 0.1 g. En cada uno de los tratamientos se hicieron cinco repeticiones.
Elaboración del queso crema tropical utilizando las BAL seleccionadas. El queso crema tropical manufacturado con las cepas de BAL seleccionadas fue elaborado en el laboratorio del Colegio de Post–graduados Campus Tabasco. La leche se estandarizó a 3 % de grasa, se pasteurizó a 63 °C por 30 min, se bajó la temperatura a 37 °C, se inoculó con 1 % de leche semidescremada acidificada con los cultivos lácticos seleccionados (108 UFC ml–1).
Por cada 10 L de leche, se agregó 1 ml de cuajo (fuerza 1:10 000). La leche se dejó en reposo por aproximadamente 12–18 h (pH 4.9–5.3), se cortó la cuajada y se dejó escurrir por 12 h a 4 °C. A la cuajada se agregó NaCl en una proporción de 4 % de leche, se prensó durante 4 h y se empacó en papel de polietileno, y se refrigeró a 4 °C. La manufactura de los quesos se repitió cinco veces.
Materia seca. Las muestras de 10 g de queso se secaron a 102 °C por 4 h, se enfriaron en un desecador y se pesaron.
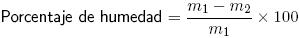
m1= peso de la muestra húmeda;
m2= Peso de la muestra seca.
En cada uno de los tratamientos se hicieron cinco repeticiones.
Análisis microbiológico. La presencia de coliformes y Escherichia coli se determinó en la leche bronca, leche pasteurizada, queso artesanal y queso elaborado con leche pasterizada.
Para las pruebas microbiológicas de coliformes y Escherichia coli, se utilizó el medio de cultivo RAPID Coliform HiCrome Broth (Sigma Aldrich).
El substrato cromogénico permite detectar la presencia de la enzima β–galactosidasa en coliformes; el substrato MUG permite detectar la presencia de la enzima glucoronidasa, especifica en Escherichia coli. En cada uno de los tratamientos se hicieron cinco repeticiones.
Análisis estadísticos. Para cada uno de los tratamientos se hizo un análisis de varianza de la disminución del pH en relación a las cepas, tiempos y temperaturas. El experimento fue de tipo factorial A x B x C. El factor A consistió de 20 cepas (20 niveles), el factor B tuvo tres niveles (tres temperaturas) y el factor C incluyó los cinco tiempos de medición del pH.
La combinación de niveles A x B generó un total de 60 tratamientos con cinco repeticiones, mismos que fueron alojados en un diseño completamente al azar con arreglo en parcelas divididas en tiempo (split plot in time), las parcelas divididas consistieron en cinco tiempos de medición. La comparación de medias de tratamientos se realizó con la prueba de Tukey (Steele & Torrie 1988) en el programa estadístico SAS versión 8.4 (Anónimo 2001).
Un diseño completamente al azar se utilizó para el análisis estadístico de los datos de los ácidos grasos volátiles de las seis cepas seleccionadas.
Las curvas de acidificación, a tres temperaturas (30, 37 y 42 °C), se analizaron con el software Numerical Methods Toolkit (Chapra & Canale 2002) para calcular el área bajo la curva de pH para cada una de las temperaturas por cada cepa. La comparación de medias de tratamientos se realizó con la prueba de Tukey (Steele & Torrie 1988) en el programa estadístico SAS versión 8.4 (Anónimo 2001).
RESULTADOS
Aislamiento, selección y purificación de las bacterias ácido lácticas a partir del queso crema tropical. Se formó un banco de 20 cepas de bacterias lácticas, las cuales fueron aisladas a partir del queso crema artesanal elaborado en la región Chontalpa en el estado de Tabasco. Las 20 cepas tuvieron características del género Lactobacillus en relación a su morfología de bacilos, su tinción de Gram positivos, su capacidad de crecer en ausencia de O2, su reacción catalasa negativas, su ausencia de esporas y por producir predominantemente ácido láctico a partir de la fermentación de carbohidratos simples.
Características bioquímicas de las BAL. Las cepas 3L, 5L, 6L, 19L y 20L tuvieron un perfil de fermentación similar a Lactobacillus fermentum. Esto por producir CO2, su capacidad de fermentar gluconato, D rafinosa, melibiosa, sacarosa, lactosa, glucosa, galactosa y ribosa. En tanto que la 14L tuvo un perfil de fermentación similar a Lactobacillus pentosus (Tabla 1).
Identificación molecular de las BAL mediante la secuenciación del gen 16S DNAr y RAPD–PCR. Los iniciadores diseñados fueron funcionales para amplificar el fragmento de 658 pb del gen 16S DNAr para las seis cepas de lactobacilos (Figura 1).

Para las cepas 3L, 5L, 6L, 19L y 20L, las secuencias de esos fragmentos fueron homólogas a las del gen 16S DNAr de L. fermentum; mientras que para la cepa 14L fueron homólogas a la del gen 16S DNAr de L. pentosus y de L. plantarum (Tabla 1).
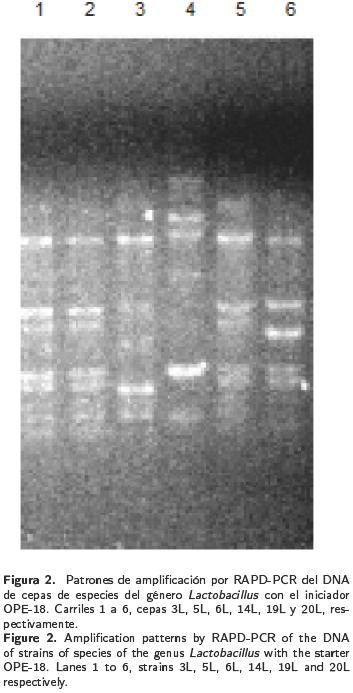
Las seis cepas de Lactobacillus seleccionadas, mostraron que L. fermentum y L. pentosus tuvieron diferentes patrones de amplificación en los geles de electroforesis (Figura 2).
Evaluación de la reducción del pH de la leche por efecto de la temperatura, tiempo de incubación y cepas. La fermentación de la leche (reducción de pH) tuvo variaciones significativas debido a los siguientes factores: el tipo de cepas utilizadas (ANDEVA F = 5,88; P = 0.0001), la temperatura de incubación (ANDEVA F = 43.91; p = 0.0001), el tiempo en horas de acidificación de la leche utilizadas (ANDEVA F = 1884.83; p = 0.0001) y las interacciones respectivas entre esos factores, que fueron cepa x temperatura (ANDEVA F = 3.54; p = 0.0001), cepa x hora (ANDEVA F = 1.98; p = 0.0001), temperatura x hora (ANDEVA F = 9.14; p = 0.0001). La interacción cepa x temperatura x hora no resultó significativamente diferente (ANDEVA F = 0.84; p = 0.906).
En las comparaciones posthoc, la caída del pH a 30 °C fue significativamente menor que a 37 y 42 °C . Entre 37 y 42 °C no se observaron diferencias significativas (Tukey; p < 0.05). En relación al tiempo de incubación, el pH alcanzo niveles más bajos a 72, 48, 24 6h respectivamente (Tukey; p < 0.05). En las comparaciones posthoc, la caída del pH por efecto de las cepas, se observaron 3 grupos. Las cepas que disminuyeron significativamente el pH con los valores más bajos fueron las tres siguientes: 3, 19, 20; que fueron identificadas como L. fermentum 3L, L. fermentum 19L, L. fermentum 20L (Tukey; p < 0.05). Las bacterias que siguieron en disminuir el pH a los valores mas bajos fueron las tres siguientes 5, 14 y 6, que fueron identificadas como L. fermentum 5L, L. pentosus 14L y L. fermentum 6L; las cuales pertenecen al grupo de las bacterias heterofermentativas obligadas y la cepa L. pentosus 14L del grupo de las heterofermentativas facultativas. Mediante métodos numéricos se calculo el área bajo la curva para cada una de las seis cepas a tres temperaturas), se comprobó la temperatura óptima para cada cepa (Figura 3).
Acido láctico y ácidos grasos volátiles de cadena corta en la leche acidificada a partir de las cepas seleccionadas. Estadísticamente no se presentaron diferencias significativas en cuanto a la producción de acidez, ácido láctico, acético, propiónico y butírico en las leches fermentadas con las seis cepas de BAL identificadas (ANDEVA F = 2.1; p = 0.76).
El ácido láctico fue el metabolito de mayor presencia en la leche fermentada con todas las cepas (Tabla 2).
Evaluaciones fisicoquímicas para el queso crema tropical con leche bronca y queso crema tropical con leche pasteurizada. Los quesos crema tropical elaborados con leche pasteurizada empleando L. fermentum 3L, L. fermentum 5L, L. fermentum 6L, L. pentosus 14L, L. fermentum 19L y L. fermentum 20L no presentaron diferencias significativas en las evaluaciones fisicoquímicas (pH, acidez y humedad) con respecto al queso crema tropical artesanal (ANDEVA F = 1.3; p = 0.88) (Tabla 3) pero si se presentaron diferencias significativas en la densidad (ANDEVA F = 14.3; p = 0.001).
Pruebas microbiológicas obtenidas de la leche bronca y leche pasterizada. Con el tratamiento térmico se redujo la carga microbiana inicial que contenía la leche bronca (cuenta total, coliformes, hongos y levaduras) y al mismo tiempo se observó que la leche fue un medio de crecimiento adecuado para el crecimiento de las BAL inoculadas como cultivos lácticos iniciadores (Tabla 4) para dar lugar a una fermentación láctica pura.
Calidad microbiológica de los quesos fabricados con leche bronca y leche pasterizada. En los análisis microbiológicos efectuados para ambos tipos de quesos, la cuenta total, hongos y levaduras y coliformes totales y E. coli fueron significativamente mas altos en los quesos manufacturados con leche sin pasterizar que en los quesos manufacturados con leche pasterizada (ANDEVA F = 14.3; p = 0.01, Tukey; p < 0.05), pero no se encontró diferencia estadística en la determinación de cuenta de BAL (ANDEVA F = 1.2; p = 0.46) (Tabla 4).
Las poblaciones más bajas de esos microorganismos se encontraron en el queso fabricado con leche pasterizada (p < 0.05). El número de bacterias lácticas no resultó diferente entre ambos tipos de queso (p > 0.05).
DISCUSIÓN
El propósito de este estudio fue aislar 20 BAL, y caracterizar e identificar las seis BAL con mayor potencial acidificante, característica importante para la elaboración del "queso crema tropical" tradicionalmente fabricado con leche bronca. Por sus características morfológicas y bioquímicas, estas bacterias pudieron considerarse como pertenecientes al género Lactobacillus (Walstra et al. 2001; Axelsson 2004).
La caracterización de las BAL se hizo con una combinación de las pruebas morfológicas, metabólicas y fisiológicas (Axelsson 2004) y por métodos moleculares, que estuvieron basados en la secuenciación del 16s rDNA (Holzapfel et al. 2001).
Dos iniciadores de PCR fueron diseñados a partir de secuencias de nucleótidos de genes que codifican para regiones conservadas del 16S rDNA del material genético de especies del género Lactobacillus. Esos iniciadores permitieron obtener los amplicones de las seis cepas de BAL de este estudio. Un análisis de las secuencias de nucleótidos de esos fragmentos, mostró que cinco cepas 3L, 5L, 6L, 19L y 20L contenían secuencias de pares de bases homólogas a L. fermentum. Esta identidad fue confirmada con las pruebas bioquímicas.
En el caso de la cepa Lactobacillus 14L, el análisis de la secuencia de nucleótidos mostró similitud con L. plantarum y L. pentosus, pues ambas especies tienen secuencias completamente homólogas en el segmento del 16s rDNA estudiado. Esas similitudes entre L. plantarum y L. pentosus fueron anteriormente observadas por Sui et al. (2002). Un criterio bioquímico que se usa para diferenciar entre estas especies, consiste en la capacidad de fermentar D–xilosa y glicerol por L. pentosus y no por L. plantarum (Zanoni et al. 1987). Por lo tanto para decidir a cuál de esas especies pertenecía la cepa 14 L, se consideró dicho criterio (Tabla 1), quedando Lactobacillus 14L como L. pentosus 14L.
Una caracterización de otras secciones del DNA ribosomal, como son el 23S o el 5S, además del 16S, han permitido distinguir entre algunas cepas muy parecidas como lo es L. plantarum y L. pentosus (Bringel et al. 1996).
Inicialmente no se sabía que L. plantarum/L. pentosus podría estar entre las cepas de la colección, por lo cual no se consideraron los mencionados genes y las regiones interespaciadoras para hacer los estudios de secuenciación.
La técnica de RAPD con el iniciador OPE–18 fue útil para distinguir entre L. fermentum y L. pentosus en los geles de electroforesis. Como actividad queda pendiente evaluar la formación de bandas de electroforesis para especies filogenéticamente relacionadas como L. plantarum y L. pentosus, que utilizando otros iniciadores han podido ser distinguidos utilizando el RAPD–PCR (van Reenen & Dicks 1996).
L. fermentum es una especie de Lactobacillus heterofermentativa obligada que ha sido aislada de productos lácteos, masa de pan, ensilados, compostas, heces y saliva de humanos (Dellaglio et al. 2004). En tanto que L. pentosus es una especie heterofermentativa facultativa que ha sido aislado predominantemente de productos vegetales, carnes, del tracto gastrointestinal (Sui et al. 2002) y también de productos lácteos (Ayad et al. 2004). No obstante, al parecer esta especie no se usa como cultivo iniciador para productos lácteos (Coeuret et al. 2003).
La cinética de disminución de pH fue un factor que se utilizó para la selección de las BAL, pues el queso artesanal presenta un pH bajo. El pH bajo inhibe el crecimiento de microorganismos indeseables y patógenos (Collins 1977), que se han detectado en algunos quesos en Tabasco (Borbolla et al. 2004). Un bajo pH lleva a la inhibición del grupo de coliformes, especialmente cuando hay otros mecanismos de estrés como son la reducción de la actividad de agua y la presencia de sal (Jay 2000). Los coliformes totales y Escherichia coli son los microorganismos indicadores de la calidad sanitaria de los procesos de pasteurización e higiene de equipos y materiales empleados (Monsalve & Gónzales 2005).
En el queso elaborado con las bacterias seleccionadas, la acidificación consistió en dos partes, en las primeras 12–18 horas durante el reposo de la cuajada para acidificar la leche, posteriormente durante el desuerado y escurrido, en la que siguió el proceso de acidificación, hasta el amasado, en la cual se interrumpe con la adición de sal y con el prensado, con la eliminación de agua.
La pasterización de la leche redujo la carga microbiana inicial en la leche y permitió obtener un producto terminado conteniendo casi exclusivamente bacterias lácticas, a diferencia del queso artesanal, en que la cuenta total, los hongos y levaduras y los coliformes y E. coli fue más alta que en los quesos elaborados con leche pasterizada. Sería conveniente evaluar que tanto la presencia de patógenos podría ser en realidad un problema a nivel epidemiológico para este tipo de quesos manufacturados con leche no pasterizada, pues puede decirse que el número de coliformes en el queso artesanal fue bajo, lo cual es consecuencia del bajo pH del queso, que tiene efectos antimicrobianos contra esos microorganismos (Booth & Kroll 1989).
La disminución del pH es consecuencia de la fermentación de lactosa en ácido láctico; lo cual además de reducir los riesgos de presencia de patógenos, tiene otros efectos como la gelificación de la leche, la sinéresis y la formación de sabor y aroma (Walstra et al., 2001). El ácido que se produjo fue predominantemente ácido láctico, que además de aumentar la vida útil del producto, imparte un sabor ácido suave y agradable, que no domina sobre otros compuestos aromáticos y más bien realza el sabor (Pszczola 2007).
Los ácidos acético, propiónico y butírico por las seis cepas de BAL identificadas, estuvieron en muy bajas concentraciones. Estos resultados concuerdan con el hecho de que las BAL producen predominantemente ácido láctico como resultado de la fermentación de carbohidratos simples como la glucosa y la lactosa (Holzapfel et al. 2001).
Este estudio presenta información acerca de que los lactobacilos son un grupo importante en la manufactura del queso crema tropical, tanto por su abundancia en el producto como por la presencia de ácido láctico, que es resultado de su metabolismo de la lactosa (Coeuret et al. 2003).
L. fermentum y L. pentosus fueron las especies identificadas en este estudio. Es posible que algunas de las cepas puedan utilizarse para manufacturar el queso crema tropical con leche pasterizada, pues la calidad microbiológica del queso elaborado con leche pasteurizada es mejor al queso crema tropical artesanal, además de asegurar la inocuidad hacia el consumidor final y cumplir con la normatividad en relación a la necesidad de usar leche pasterizada. En una evaluación preliminar de fabricación de queso con L. pentosus y L. fermentum se obtuvo una puntuación de sabor típico de 8.8 y de sabor agradable de 9.15 por diez consumidores al azar. Cabe señalar que los estudios sensoriales del queso producido con las cepas se están llevando a cabo para cumplir con expectativas de sabor, olor y textura que requieren los consumidores.
AGRADECIMIENTOS
Los autores agradecen a la Fundación Produce Tabasco A.C. por el financiamiento del proyecto FP5–06 "Selección de cultivos lácticos para la manufactura del queso Tabasco" y al Consejo Nacional de Ciencia y Tecnología (CONACYT), México por la beca otorgada al primer autor para estudios de postgrado.
LITERATURA CITADA
Anónimo (1994) Norma Oficial Mexicana Nom–121–SSA1–1994. Bienes y Servicios. Quesos: Frescos, Madurados y Procesados. Especificaciones Sanitarias. www.salud.gob.mx/unidades/cdi/nom/121ssa14. html. [ Links ]
Anónimo (2001) SAS Users Guide Basic Version 8.2. SAS Institute Inc., Cary, NYC. USA. [ Links ]
Anónimo (2009) National Institutes of Health, U.S. National Library of Medicine, National Center for Biotechnology Information. www.ncbi.nlm.nih.gov. [ Links ]
Ausebel FM, Brent R, Kingston RE, Moore DD, Seidman JG, Smith JA. Struhl, K (1989). Current protocols in molecular biology. Greene publishing associates and Wiley–Interscience, New York, N.Y. [ Links ]
Axelsson L (2004) Lactic acid bacteria: classification and physiology. En: Salminen S, von Wright A , Ouwehand A (eds). Lactic Acid Bacteria. CRC, Inc. New York, USA. 603 pp. [ Links ]
Ayad EHE, Nashat S, El–Sadek N, Metwaly H, El–Soda M (2004) Selection of wild lactic acid bacteria isolated from traditional Egyptian dairy products according to production and technological Criteria. Food Microbiology. 21: 715– 725. [ Links ]
Beuvier E, Berthaud K, Cegarra S, Dasen A, Pochet S, Buchin S, Duboz, G (1997) Ripening and quality of swiss type cheese made from raw, pasteurized or microfiltered milk. International Dairy Journal 7: 311–323. [ Links ]
Boldo, XM, Villa–Tanaca L, Zúñiga G, Hernández–Rodríguez C (2003) Genetic diversity among clinical isolates of Candida glabrata analyzed by randomly amplified polymorphic DNA and multilocus enzyme electrophoresis analyses. Journal of Clinical Microbiology 41: 4799–4804. [ Links ]
Booth IR, Kroll, RG (1989) The preservation of foods at low pH. En: Gould GW (ed) Mechanisms of action of food preservation procedures, Elsevier Applied Science, London, UK. 441 pp. [ Links ]
Borbolla E, Vidal PM, Piña GO, Ramírez MI, Vidal VJ (2004) Contaminación de los alimentos por Vibrio cholerae, coliformes fecales, Salmonella, hongos, levaduras y Staphylococcus aureus en Tabasco durante 2003. Salud en Tabasco 10: 221–232. [ Links ]
Bringel F, Curk MC, Hubert JC. (1996) Characterization of lactobacilli by southern–type hybridization with a Lactobacillus plantarum pyrDFE probe. International Journal of Systematic Bacteriology 46: 588–594. [ Links ]
Centeno JA, Cepeda A, Rodríguez JL (1996) Lactic Acid Bacteria isolated from Arzúa Cheese cow's milk cheese. International Dairy Journal 6: 65–78. [ Links ]
Chapra SC, Canale RP (2002) Numerical Methods for Engineers with Programming and Software and Application. Mc Graw Hill. New York. USA. 734 pp. [ Links ]
Coeuret V, Dubernet S, Bernardeau M, Gueguen M, Vernoux JP (2003) Isolation, characterisation and identification of lactobacilli focusing mainly on cheeses and other dairy products. Lait 83: 269–306. [ Links ]
Collins EB (1977) Influence of medium and temperature on end products and growth. Journal of Dairy Science 60: 799–804. [ Links ]
Dellaglio F, Torriani S, Felis GE (2004) Reclassification of Lactobacillus cellobiosus Rogosa et al. 1953 as a later synonym of Lactobacillus fermentum Beijerinck 1901. International Journal System Evolution Microbiology 54: 809–812. [ Links ]
Erwin E, Marco GJ, Emery E (1961) Volatile fatty acid analysis of blood and rumen fluid by gas chromatography. Journal of Dairy Science 44: 1768–1771. [ Links ]
González J, Mas M, Tabla R, Casares P (2003) Autochthonous starter effect on the microbiological, physi–cochemical and sensorial characteristics of Ibores goat's milk cheese. Lait 83: 193–202. [ Links ]
Holzapfel WH, Haberer P, Geisen R, Bjorkroth J, Schillinger U (2001) Taxonomy and important features of probiotic microorganisms in food nutrition. American Journal of Clinical Nutrition 73: 365S–373S. [ Links ]
Jay JM (2000) Modern Food Microbiology, 6th edition. An Aspen Publication, Aspen Publishers, Inc. Gaithersburg, USA. pp.720. [ Links ]
Kumar S, Tamura K, Jakobsen IB, and Nei M (2001) MEGA2: Molecular evolutionary genetics analysis software. Bioinformatics 17: 1244–12245. [ Links ]
Lockhart SR, Joly S, Pujol C, SobelJD, Pfaller MA, Soll DR (1997) Development and verification of fingerprinting probes for Candida glabrata. Microbiology: 143: 3733–3746. [ Links ]
Madrid J, Martínez–Teruel AM, Hernández F, Mejías MD (1999) A comparative study on the determination of lactic acid in silage juice by colorimetric, High–performance liquid chromatography and enzymatic methods. Journal of the Science of Food and Agriculture. 79: 1722–1726. [ Links ]
Méndez S, Centeno JA, Godínez R, Rodríguez JL (1998) Propiedades tecnológicas y actividades enzimáticas de cepas de Lactococcus lactis, aisladas del queso Arzúa Ulloa, Alimentaria 296: 59–64. [ Links ]
Monsalve J, Gónzales D (2005) Elaboración de un queso tipo ricotta a partir de suero lácteo y leche fluida. Revista Científica, FCV–LUZ 15: 543– 550. [ Links ]
Nicholas KB, Nicholas HB (1997) GeneDoc: A tool for editing and annotating multiple sequence alignments. Distribuido por los autores. [ Links ]
Pszczola DE (2007) Meeting the acid test. Food Technology 5: 47–59. [ Links ]
Sánchez I, Palop LL, Ballesteros C (2000) Biochemical characterization of lactic acid bacteria isolated from spontaneous fermentation of "Almagro" eggplants. International Journal of Food Microbiology 59: 9–17. [ Links ]
Steel RG, Torrie JH (1988) Bioestadística. Principios y procedimientos. 2ª, ed McGraw–Hill, D.F. 371 pp. [ Links ]
Sui J, Leighton S, Busta F, Brady L (2002) 16S ribosomal DNA analysis of the faecal lactobacilli composition of human subjects consuming a probiotic strain Lactobacillus acidophilus NCFM. Journal of Applied Microbiology 93: 907–912. [ Links ]
Thompson JDT, Gibson J, Plewniak F, Jeanmougin F, Higgins DG (1997) The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acid Research 24: 4876–4882. [ Links ]
van Reenen CA, Dicks LMT (1996) Evaluation of numerical analysis of Random Amplified Polymorphic DNA (RAPD)–PCR as a method to differentiate Lactobacillus plantarum and Lactobacillus pentosus. Current Microbiology 32: 183–187. [ Links ]
Villegas de Gante A (1989) Los quesos mexicanos. Universidad Autónoma de Chapingo. CIESTAAM. Edo de México. 251 pp [ Links ]
Walstra P, Geurts TJ, Normen A, Jellema A, Van Boekel MAJS (2001) Ciencia de la leche y tecnología de los productos lácteos. Editorial Acribia S.A. España. 2001. 730p. [ Links ]
Zanoni P, Farrow J, Phillips B, Collins M (1987) Lactobacillus pentosus (Fred, Peterson, and Anderson) International Journal of Systematics Bacteriology 37: 339–341. [ Links ]














