Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Ciencias marinas
versión impresa ISSN 0185-3880
Cienc. mar vol.47 no.4 Ensenada oct./dic. 2021 Epub 09-Dic-2022
https://doi.org/10.7773/cm.v47i4.3095
Artículos
Amplificación isotérmica mediada por bucle para diagnosticar patógenos marinos en tejidos de Crassostrea spp. y camarón blanco, Litopenaeus vannamei, cultivados en México
1Departamento Académico de Ciencias Marinas y Costeras, Universidad Autónoma de Baja California Sur, Carretera al Sur km 5.5 s/n, 23080, La Paz, Baja California Sur, Mexico.
2Programa de Acuicultura, Centro de Investigaciones Biológicas del Noroeste, Av. IPN 195, 23096, La Paz, Baja California Sur, Mexico.
3Programa Cátedras Consejo Nacional de Ciencia y Tecnología, Centro de Investigaciones Biológicas del Noroeste, Av. IPN 195, 23096, La Paz, Baja California Sur, Mexico.
La amplificación isotérmica mediada por bucle (LAMP, por sus siglas en inglés) es un método preciso, sensible, rápido y fácil de realizar para la amplificación de genes en condiciones isotérmicas, y ha servido como una herramienta de diagnóstico poderosa. En este estudio, desarrollamos un protocolo de diagnóstico con LAMP para la detección de Vibrio parahaemolyticus y el virus de la mancha blanca en camarón blanco (Litopenaeus vannamei) y de Perkinsus spp. en Crassostrea spp. en México. Estos patógenos están asociados a distintas enfermedades y se consideran una amenaza en la industria de la acuicultura. Se obtuvieron ostiones y camarones infectados y no infectados de granjas acuícolas en la costa noroeste de México para estandarizar la técnica de LAMP. Determinamos los genes candidatos a través de un primer análisis de muchos conjuntos de cebadores, y luego elegimos un conjunto de cebadores que amplificaron exitosamente con muestras mexicanas. Optimizamos las reacciones de LAMP para cada patógeno con los cebadores seleccionados usando gradientes de temperatura de 61 a 65 ºC, concentraciones de ADN de 2.5 pg a 250.0 ng y tiempos de reacción de 10 a 60 min. Este estudio estableció un procedimiento diagnóstico para la detección de patógenos de ostiones y camarones de México. El diagnóstico y el tratamiento temprano de patógenos pueden reducir enormemente la transmisión de enfermedades en granjas acuícolas.
Palabras clave: Crassostrea spp.; Litopenaeus vannamei; amplificación isotérmica mediada por bucle; acuicultura; patógenos
Loop-mediated isothermal amplification (LAMP) is an accurate, sensitive, rapid, and easy-to-perform method for gene amplification under isothermal conditions, and it has served as a powerful diagnostic tool. In this study, we used LAMP to develop a diagnostic protocol for detecting Vibrio parahaemolyticus and white spot syndrome virus in whiteleg shrimp (Litopenaeus vannamei), and Perkinsus spp. in Crassostrea spp. in Mexico. These pathogens are associated with different diseases and are considered a threat in the aquaculture industry. Infected and uninfected oysters and shrimp were obtained from farms in the northwest coast of Mexico to standardize the LAMP assay. We determined the candidate target genes in the first-round analysis of many sets of primers, and then we chose a set of primers that successfully amplified with Mexican samples. We optimized the LAMP reactions for each pathogen with the chosen primer sets using temperature gradients from 61 to 65 ºC, DNA concentrations from 2.5 pg to 250.0 ng, and reaction times from 10 to 60 min. This study established a diagnostic procedure for detecting pathogens in oysters and shrimp from Mexico. Early diagnosis and treatment of pathogens can immensely reduce disease transmission in aquaculture farms.
Key words: Crassostrea spp.; Litopenaeus vannamei; loop-mediated isothermal amplification; aquaculture; pathogens
Introducción
Ante el deterioro de las pesquerías a nivel mundial, la acuicultura ha sido la alternativa con mayor viabilidad económica para resolver la demanda de alimentos (FAO 2019). En México, la acuicultura ha experimentado avances significativos en las últimas décadas, particularmente en la región noroeste, donde la productividad biológica, que incluye peces, moluscos y crustáceos, es la más alta del país. Los ostiones del género Crassostrea y el camarón blanco, Litopenaeus vannamei (Boone, 1931), representan uno de los valores de acuicultura marina más altos en México (SAGARPA 2017). El aumento de la comercialización nacional y de exportación de productos de origen acuícola ha favorecido la introducción, la propagación y la transmisión de enfermedades (OIE 2019). En las granjas de ostiones, Perkinsus spp. han amenazado la producción de estos (Cáceres-Martínez et al. 2008, Enríquez-Espinoza et al. 2010). Las granjas camaroneras han sido afectadas severamente por episodios de mortalidad debido a enfermedades provocadas por bacterias pertenecientes al género Vibrio (Soto-Rodríguez et al. 2010) y el virus del síndrome de la mancha blanca (VSMB) (Hasson et al. 2006). Es necesario un control adecuado de enfermedades dentro de los sistemas de cultivo acuícola para detener la propagación de patógenos infecciosos. El monitoreo de enfermedades en términos de vigilancia y diagnóstico rápido es una parte integral del proceso de acuicultura. Permite tomar las medidas adecuadas cuando se detectan patógenos y se diagnostican enfermedades antes de convertirse en un problema importante para el acuicultor.
El diseño y el desarrollo de métodos para la identificación de patógenos en organismos acuáticos cultivados ha progresado. En la última década se han desarrollado técnicas de detección molecular, y la identificación de microorganismos se basa principalmente en el análisis de los ácidos nucleicos. Las ventajas de estas técnicas incluyen la especificidad, la sensibilidad, la rapidez y la automatización (Palomino-Camargo et al. 2014). La amplificación isotérmica mediada por bucle (LAMP, por sus siglas en inglés) es una técnica molecular que amplifica el ADN con alta especificidad, eficiencia y rapidez en condiciones isotérmicas sin requerir equipo especializado costoso (Notomi et al. 2000). La amplificación requiere 2 cebadores externos (F3 y B3), 2 cebadores internos (cebador interno directo, FIP, y cebador interno inverso, BIP) y 2 cebadores de bucle (cebador de bucle directo, LF, y cebador de bucle inverso, LB) (Notomi et al. 2000). Estos cebadores son complementarios a 6 regiones de la secuencia de ADN, lo que asegura una alta especificidad (Notomi et al. 2000, Nagamine et al. 2002). Los cebadores se unen lateralmente a diferentes sitios utilizando la ADN polimerasa Bst de desplazamiento de cadena, lo que proporciona una alta eficiencia de amplificación, y el ADN se amplifica a 109-1010 copias en 15-60 min (Notomi et al. 2000, Nagamine et al. 2002, Tomita et al. 2008, Mori y Notomi 2009, Li y Macdonald 2015). Este método no requiere un molde de ADN desnaturalizado (Nagamine et al. 2001), lo que permite a los usuarios evitar complicados protocolos para procesar el ADN, y también ha demostrado una alta tolerancia a las sustancias inhibidoras presentes en muestras biológicas (Boehme et al. 2007, Kaneko et al. 2007, Mori y Notomi 2009, Francois et al. 2011). Los productos amplificados pueden detectarse por medio de electroforesis en gel, turbidímetro, dispositivo de flujo lateral o indicadores de color o fluorescentes (Mori et al. 2001, Iwamoto et al. 2003, Mori et al. 2004, Tomita et al. 2008, Goto et al. 2009).
El método LAMP se ha utilizado para la detección de patógenos de mariscos en muchos países (Kono et al. 2004, Mekata et al. 2006, Yamazaki et al. 2008, Jaroenram et al. 2009, Mekata et al. 2009, Chen y Ge 2010, Arunrut et al. 2011, Maralit et al. 2012, Xu et al. 2012, Feng et al. 2013, Kongrueng et al. 2015). Diversos informes muestran inconsistencias en los protocolos LAMP para detectar patógenos de diferentes ubicaciones geográficas. Las secuencias de genes de agentes infecciosos pueden variar según la región geográfica, como resultado de un conjunto de variables que favorecen la dispersión y las posibles modificaciones en su genotipo. Es necesario elegir cebadores y sondas específicos de los genomas de cada región para evitar diagnósticos incorrectos. En el presente estudio, nosotros proponemos identificar patógenos potencialmente peligrosos específicos de México en L. vannamei y Crassostrea spp. con LAMP. La tecnología LAMP tiene un gran potencial como técnica para la detección rápida, sensible y específica de patógenos que se puede usar regularmente en granjas o laboratorios porque no necesita equipos costosos o personal altamente capacitado.
Materiales y métodos
Recolección de muestras y extracción del ADN
Tejidos de L. vannamei infectado con el VSMB y Vibrio parahaemolyticus, y tejidos del ostión de Cortéz (Crassostrea corteziensis) y el ostión del Pacífico (Crassostrea gigas) infectados con Perkinsus spp. se obtuvieron de granjas en el noroeste de México. Seleccionamos los tejidos de acuerdo con el manual de la Organización Mundial de Sanidad Animal (OIE 2019). También utilizamos organismos no infectados como controles negativos. El ADN se extrajo de los tejidos mediante el método de extracción orgánica, como lo describen Sambrook et al. (1989), y se resuspendió mediante la adición de tampón TE (10 mM, pH 8.0). La pureza y la concentración del ADN se estimaron utilizando el espectrofotómetro Nanodrop 1000 (Thermo Fisher Scientific; MA, EUA). La integridad del ADN se visualizó por electroforesis en gel de agarosa al 1.5% utilizando el sistema de documentación de imágenes UVP Biodoc-It 2 (Analytik Jena; Jena, Alemania) y el colorante fluorescente GelRed (Biotium; Fremont, CA).
Muestras positivas
El ADN de los organismos infectados y no infectados fue extraído y utilizado como molde para la amplificación por PCR de punto final para obtener tejidos de control positivos y negativos con los cebadores externos de LAMP (F3 y B3). Los tamaños de los amplicones de la PCR fueron 208 pb para el espaciador transcrito interno 2 (ITS2) (Perkinsus spp.), 213 pb para orf191 (VSMB), 231 pb para orf36 (VSMB), 229 pb para hemolisina termoestable (tlh) (V. parahaemolyticus) y 183 pb para tox-R (V. parahaemolyticus). Se limpiaron los productos de la PCR para su secuenciación con la empresa Genewiz (South Plainfield, NJ). Los fragmentos de secuencias obtenidos se analizaron utilizando la herramienta de análisis de secuencias NCBI BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi, 20 de noviembre de 2021 ) para corroborar la identidad del patógeno.
Condiciones de reacción de LAMP
Para LAMP, determinamos los genes diana candidatos en una primera ronda de análisis de varios conjuntos de cebadores. Luego elegimos aquellos que se amplificaron exitosamente con muestras mexicanas (Tabla 1) con alta repetibilidad, eficiencia y especificidad. Los cebadores fueron sintetizados por T4 Oligo (Irapuato, México). El ensayo de LAMP se llevó a cabo en un volumen total de 25 µL que contenía 1 µL (40 µM) de cada uno de los cebadores internos (FIP y BIP), 1 µL (5 µM) de cada uno de los cebadores externos (F3 y B3), 1 µL (20 µM) de cada uno de los cebadores de bucle (LF y LB), 4 µL (0.6 mM de cada nucleotido) de mezcla de dNTP (Thermo Fisher Scientific), 1 µL (5 M) de betaína (Sigma-Aldrich), 1.5 µL (100 mM) de MgSO4 (New England Biolabs), 1 µL (8 U·µL ) de ADN polimerasa Bst fragmento largo (New England Biolabs), 2.5 µL de tampón ThermoPol 10× (New England Biolabs), 1.0 µL de ADN diana y 8 µL de agua Milli-Q. Los cebadores de tox-R (Chen y Ge 2010) tienen solo un cebador de bucle, y se agregaron como se describió anteriormente para la concentración, con la adición de 1 µL más de agua Milli-Q. Posteriormente, se llevó a cabo la incubación a 61, 62, 63, 64 y 65 ºC durante 60 min, y se terminó la reacción calentando a 90 ºC durante 2 min. Las reacciones de LAMP se realizaron utilizando el termociclador T100 BioRad (CA, EUA). Los productos (10 µL) se sometieron a electroforesis en geles de agarosa al 2% para determinar las condiciones óptimas. Cuando los productos de LAMP se visualizan usando electroforesis en gel de agarosa, se documentan bandas de diferentes tamaños, hasta el pozo de carga. Las mezclas de amplicones resultantes se confirmaron con 10 µL de los productos por fluorescencia, con 1 µL de SYBR safe (Thermo Fisher Scientific; dilución 1:500 en agua Milli-Q), y se añadieron a la solución una vez completada la reacción. Los geles y los tubos fluorescentes se documentaron utilizando el sistema de imágenes UVP Biodoc-It 2 (Analytik Jena). Los extractos de ADN de las muestras de ostiones y camarones no infectados se incluyeron como controles negativos.
Tabla 1 Secuencias de cebadores de LAMP.
| Pathogen | Genes | Primer | Sequence (5′3′) | Source |
| Perkinsus spp. | ITS2 (FJ841985) | F3 | TGGATTTTGGTATTTCAAAACGA | Feng et al. (2013) |
| B3 | ATGTTTGTATCGGGAAGAAGAG | |||
| FIP | AGTGCTTATCGCACTTCGCTCAAACTCTCAACGATGGATGC | |||
| BIP | GATTTGCAGAATTCCGTGAACCAGCACTGATATGTATGTACAAAGAGG | |||
| LF | TCCTTCATCGATTCTCGAGC | |||
| LB | TCAACGCATACTGCACAAAG | |||
| WSSV | orf191 (AF332093) | F3 | AAAGTCTCATTTTAACAAGAGGA | Jaroenram et al. (2009) |
| B3 | ATTGGTCCAGTCTCAGCC | |||
| FIP | CCATCTCTCTGAAGCGAGGAAAATTTTAATTTCTTTGCTCGAGGCC | |||
| BIP | TGCTCTAGAGAATGCAGTACCTCTTTTGCGTAGTTCTTGCACGATT | |||
| LF | TCTCCAGCCGGAGCCAAGT | |||
| LB | ACAGAACCATAACAATGGTGAATGG | |||
| WSSV | orf36 (AF369029) | F3 | AAACACCGGATGGGCTAA | Mekata et al. (2009) |
| B3 | CAAGGCAATACAGAATGCG | |||
| FIP | TCCTGTCTTCAGGGAATACATATGCTCAGGGAAGAAATAGACCATG | |||
| BIP | GGACCCAAATCGAAATATAAGGCCTATGTTGCCCAAGATCCAC | |||
| LF | GTTAAGAATGATGCATCTAGTGCGA | |||
| LB | TGGAACAAAAGATGCTGCTCA | |||
| Vibrio parahaemolyticus | tlh (M36437) | F3 | AGCTACTCGAAAGATGATCC | Yamazaki et al. (2008) |
| B3 | GGTTGTATGAGAAGCGATTG | |||
| FIP | ATGTTTTTAAATGAAACGGAGCTCCGGCAAAAAACGAAGATGGT | |||
| BIP | ACGTCGCAAAACGTTATCCGGCGAAGAACGTAATGTCTG | |||
| LF | ACCAGTAGCCGTCAATG | |||
| LB | TTAGATTTGGCGAACGAGA | |||
| Vibrio parahaemolyticus | tox-R (L11929) | F3 | TTGGATTCCACGCGTTAT | Chen and Ge. (2010) |
| B3 | CGTTCAATGCACTGCTCA | |||
| FIP | TGAGATTCCGCAGGGTTTGTAATTATTTTTGGCACTATTACTACCG | |||
| BIP | GTTCCGTCAGATTGGTGAGTATCTAGAAGGCAACCAGTTGTT | |||
| Loop | AGAACGTACCAGTGATGACACC |
WSSV, white spot syndrome virus; FIP, forward inner primer; BIP, backward inner primer; LF, forward loop primer; LB, backward loop primer.
La temperatura, la sensibilidad, el tiempo de reacción y la especificidad de LAMP se optimizaron para cada patógeno. Para evaluar la sensibilidad, diluciones seriadas con un factor de dilución de 10 del ADN genómico total, que fue extraído de tejidos infectados, fueron utilizadas como moldes para seleccionar la mejor dilución con la cual trabajar, y la reacción de LAMP se llevó a cabo en un tiempo predeterminado (63 min). Para determinar el tiempo óptimo de amplificación, las reacciones de LAMP se llevaron a cabo a la temperatura y concentración de ADN previamente elegidas para cada patógeno durante 10, 20, 30, 40, 50 y 60 min; el tiempo en el control negativo se realizó a los 60 min. La especificidad de LAMP se examinó utilizando el ADN molde de otros patógenos y muestras no infectadas: para identificar la reactividad cruzada con el gen ITS2 para Perkinsus spp., utilizamos los dinoflagelados tóxicos Gymnodinium catenatum, Alexandrium tamiyavanichii y Alexandrium catenella. Para el diagnóstico de especificidad para tlh y tox-R con V. parahaemolyticus, usamos Vibrio campbelli, Photobacterium damselae, Pseudomonas aeruginosa, Providencia rettgeri y Acinetobacter baumannii. Para el diagnostico de especificidad para orf36 y orf191 para evaluar VSMB, usamos el virus de la necrosis hipodérmica y hematopoyética del camarón, el herpes virus de ostión y el virus del rizado de la hoja de la calabaza. La extracción del ADN se realizó como se mencionó anteriormente, y la reacción de LAMP se realizó con las condiciones óptimas obtenidas anteriormente.
Resultados
Los productos de PCR representativos de muestras positivas de tejidos infectados con los patógenos estudiados se pueden observar en la Figura 1. Estos resultados se obtuvieron con los cebadores F3 y B3. Las Figuras 2-6 muestran la electroforesis en gel y los tubos fluorescentes de los ensayos de LAMP de los patógenos probados. El ensayo de LAMP para detectar Perkinsus spp. en tejidos de Crassostrea spp. utilizando cebadores de la región del gen ITS2 mostraron condiciones óptimas de amplificación en cada temperatura probada (61 a 65 ºC). Los resultados fueron confirmados por los tubos con fluorescencia (Fig. 2a). El ensayo para examinar la sensibilidad de LAMP mostró que las concentraciones óptimas de ADN fueron 3.6 a 36.0 ng (Fig. 2b). El ensayo de LAMP para determinar el tiempo óptimo comenzó a los 30 min y se mantuvo hasta los 60 min (Fig. 2c). Para detectar V. parahaemolyticus en L. vannamei, utilizamos los genes tlh (Fig. 3) y tox-R (Fig. 4). Con los cebadores tlh, la optimización de temperatura mostró que la amplificación se realizó en todas las temperaturas probadas de 61 a 65 ºC (Fig. 3a). La Figura 3b muestra los resultados del ensayo de sensibilidad de LAMP, y la amplificación se realizó en todas las diluciones analizadas desde 2.5 pg a 250.0 ng de ADN total. La optimización del tiempo de la reacción LAMP mostró que la amplificación comenzó a los 40 min y continuó hasta los 60 min (Fig. 3c). La optimización de la temperatura de la reacción de LAMP con los cebadores tox-R mostró que la amplificación se realizó con éxito de 61 a 64 ºC (Fig. 4a). La sensibilidad de LAMP mostró amplificación a 25.0 a 250.0 ng de ADN (Fig. 4b), y la optimización del tiempo comenzó a los 40 min y continuó hasta los 60 min (Fig. 4c). Para detectar VSMB en L. vannamei, elegimos 2 conjuntos de cebadores, orf36 (Fig. 5) y orf191 (Fig. 6). Con el cebador orf36, la optimización de la temperatura del ensayo de LAMP mostró que la amplificación se realizó de 61 a 65 ºC (Fig. 5a), la sensibilidad mostró que el ADN se amplificó solo a una concentración de 42.0 ng (Fig. 5b) y el tiempo óptimo de amplificación fue de 30 a 60 min (Fig. 5c). Referente al gen orf191, la temperatura óptima mostró que hubo amplificación en todas las temperaturas probadas de 61 a 65 ºC (Fig. 6a), la sensibilidad mostró que la amplificación de LAMP se realizó desde 42.0 pg a 42.0 ng de ADN (Fig. 6b) y la optimización del tiempo de la reacción de LAMP mostró que la amplificación comenzó a los 30 min y se mantuvo durante 60 min (Fig. 6c).

Figura 1 Patógenos identificados en tejidos de Crassostrea spp. y Litopenaeus vannamei infectados, mediante la amplificación de ADN por PCR de punto final con los cebadores externos para la amplificación isotérmica mediada por bucle (F3 y B3). Carril M, marcador (escalera 2 log ADN; kb, kilobases); carril 1 Perkinsus spp. (ITS2); carril 2, virus del síndrome de la mancha blanca (VSMB) (orf191); carril 3, VSMB (orf36); carril 4, Vibrio parahaemolyticus (hemolisina termoestable [tlh]); carril 5, V. parahaemolyticus (tox-R). Los productos de la PCR se visualizaron en electroforesis en gel de agarosa al 2%.

Figura 2 Optimización del gen ITS2 con la amplificación isotérmica mediada por bucle para detectar Perkinsus spp. en tejidos de Crassostrea spp. (a) Temperatura (ºC). (b) Sensibilidad (pg). (c) Tiempo de reacción (min). (d) Especificidad: carril (+), Perkinsus marinus; carril 1, Gymnodinium catenatum; carril 2, Alexandrium tamiyavanichii; carril 3, Alexandrium catenella. Carril M, marcador (escalera 2 logADN; kb, kilobases); carril (-), control negativo (ADN de tejido no infectado). Los productos se visualizaron en electroforesis en gel de agarosa al 2% y tubos fluorescentes.
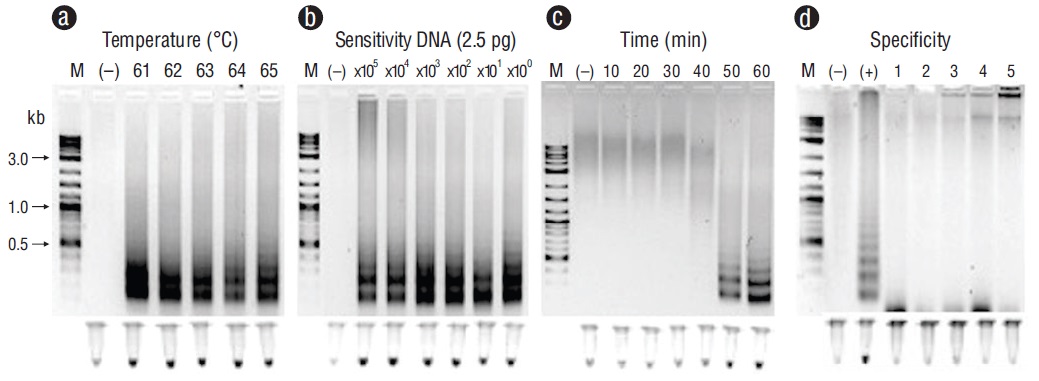
Figura 3 Optimización del gen hemolisina termoestable (tlh) con la amplificación isotérmica mediada por bucle para detectar Vibrio parahaemolyticus en tejidos de Litopenaeus vannamei. (a) Temperatura (ºC). (b) Sensibilidad (pg). (c) Tiempo de reacción (min). (d) Especificidad: carril (+), V. parahaemolyticus; carril 1, Vibrio campbelli; carril 2, Photobacterium damselae; carril 3, Pseudomonas aeruginosa; carril 4, Providencia rettgeri; carril 5, Acinetobacter baumannii. Carril M, marcador (escalera 2 log ADN; kb, kilobases); carril (-), control negativo (ADN de tejido no infectado). Los productos se visualizaron en electroforesis en gel de agarosa al 2% y tubos fluorescentes.

Figura 4 Optimización del gen tox-R con la amplificación isotérmica mediada por bucle para detectar Vibrio parahaemolyticus en tejidos de Litopenaeus vannamei. (a) Temperatura (ºC). (b) Sensibilidad (pg). (c) Tiempo de reacción (min). (d) Especificidad: carril (+), V. parahaemolyticus; carril 1, Vibrio campbelli; carril 2, Photobacterium damselae; carril 3, Pseudomonas aeruginosa; carril 4, Providencia rettgeri; carril 5, Acinetobacter baumannii. Carril M, marcador (escalera 2 log ADN; kb, kilobases); carril (-), control negativo (ADN de tejido no infectado). Los productos se visualizaron en electroforesis en gel de agarosa al 2% y tubos fluorescentes.

Figura 5 Optimización del gen orf36 con la amplificación isotérmica mediada por bucle para detectar el virus del síndrome de la mancha blanca (VSMB) en tejidos de Litopenaeus vannamei. (a) Temperatura (ºC). (b) Sensibilidad (pg). (c) Tiempo de reacción (min). (d) Especificidad: carril (+), VSMB; carril 1, virus de la necrosis hipodérmica y hematopoyética del camarón; carril 2, herpes virus de ostión; carril 3, virus del rizado de la hoja de la calabaza. Carril M, marcador (escalera de ADN de 2 log; kb, kilobases); carril (-), control negativo (ADN de tejido no infectado). Los productos se visualizaron en electroforesis en gel de agarosa al 2% y tubos fluorescentes.

Figura 6 Optimización del gen orf191 con la amplificación isotérmica mediada por bucle para detectar el virus del síndrome de la mancha blanca (VSMB) en tejidos de Litopenaeus vannamei. (a) Temperatura (ºC). (b) Sensibilidad (pg). (c) Tiempo de reacción (min). (d) Especificidad: carril (+), VSMB; carril 1, virus de la necrosis hipodérmica y hematopoyética del camarón; carril 2, herpes virus de ostión; carril 3, virus del rizado de la hoja de la calabaza. Carril M, marcador (escalera de ADN de 2 log; kb, kilobases); carril (-), control negativo (ADN de tejido no infectado). Los productos se visualizaron en electroforesis en gel de agarosa al 2% y tubos fluorescentes.
Probamos la reacción cruzada con otros de virus y bacterias para determinar la especificidad del método de LAMP (Figs. 2d, 3d, 4d, 5d, 6d). No se encontraron resultados positivos para LAMP para ninguna de las muestras, lo que indica que el método LAMP tiene una alta especificidad para los patógenos analizados.
Discusión
En este estudio, evaluamos el uso de LAMP para detectar patógenos en tejidos de camarones y ostiones cultivados en México. Describimos un protocolo de diagnóstico de LAMP para Perkinsus spp., VSMB y V. parahaemolyticus. Estos patógenos provocan graves pérdidas mundiales en la producción de ostiones y camarones, lo que resulta en graves daños económicos. La Organización Mundial de Sanidad Animal considera al VSMB como el virus más frecuente en el cultivo de camarón; además, otro tipo de patógeno que afecta gravemente a los cultivos de camarón es la bacteria V. parahaemolyticus, que actúa como agente oportunista y provoca pérdidas masivas (OIE 2019). Los protozoos Perkinsus marinus y Perkinsus olsenii son organismos que afectan los cultivos de ostiones, principalmente Crassostrea spp. (OIE 2019). Las enfermedades causadas por estos patógenos deben ser declaradas obligatoriamente ante la Organización Mundial de Sanidad Animal tan pronto como se detecten.
La detección precisa de patógenos es un paso crucial en el proceso de manejo de enfermedades. Se han desarrollado varios métodos de detección para monitorear y controlar estos patógenos en cultivos. Las técnicas basadas en la amplificación del ADN tienen ventajas de sensibilidad y especificidad que las convierten en valiosas herramientas de detección de patógenos. LAMP es un método sensible que puede amplificar algunas copias de ADN en menos de una hora en condiciones isotérmicas (Notomi et al. 2000, Mori et al. 2001, Nagamine et al. 2002, Mori y Notomi 2009). Es utilizado para detectar patógenos de mariscos en muchos países; sin embargo, muchos reportes muestran inconsistencias en los protocolos con LAMP para la detección de patógenos de diferentes ubicaciones geográficas, consecuencia de un conjunto de variables que favorecen la dispersión y posibles modificaciones en los genotipos de los patógenos. Evaluamos muchos conjuntos diferentes de cebadores para cada patógeno, y desarrollamos el protocolo con los cebadores y las condiciones de ensayo para reacciones de LAMP más adecuados para patógenos en sistemas de acuicultura mexicanos. Nuestro ensayo también podría ser utilizado con organismos de tipo silvestre, diferentes etapas de desarrollo y otras especies de mariscos cuando el patógeno está presente.
El protocolo LAMP que describimos en este estudio representa un método sensible, específico y rápido para detectar patógenos de mariscos. Muchos informes han evaluado el método de LAMP y argumentan que tiene muchas ventajas sobre otras técnicas moleculares. LAMP es un procedimiento de un solo paso que requiere de 4 a 6 cebadores que se unen lateralmente a diferentes sitios utilizando la ADN polimerasa Bst de desplazamiento de cadena, lo que permite una amplificación extremadamente específica en condiciones isotérmicas (Notomi et al. 2000). No requiere un molde de ADN desnaturalizado, lo que evita los métodos de extracción de ADN que consumen mucho tiempo, y tiene una baja susceptibilidad a los compuestos inhibidores; los resultados se pueden visualizar a simple vista con tinciones de ADN coloridas o fluorescentes. Como resultado, permite el uso de equipos simples de bajo costo y protocolos simplificados (Notomi et al. 2000, Nagamine et al. 2001, Kaneko et al. 2007, Tomita et al. 2008, Goto et al. 2009, Mori y Notomi 2009, Francois et al. 2011, Li y Macdonald 2015, Tanner et al. 2015).
El protocolo que mostramos en este trabajo es el método LAMP básico, diseñado principalmente para ser llevado a cabo en el laboratorio. LAMP ha demostrado ser ampliamente funcional para la detección in situ de patógenos debido a su adaptabilidad a diversos enfoques y entornos de detección, incluidos muchos kits portátiles comerciales, para los cuales el flujo de trabajo no requiere personal altamente calificado (Kiatpathomchai et al. 2008, Jaroenram et al. 2009, Khunthong et al. 2013, Mori et al. 2013, Tomlinson 2013, Dhama et al. 2014, Notomi et al. 2015, Besuschio et al. 2017, Khan et al. 2017, Wong et al. 2018, Nzelu et al. 2019, Becherer 2020). La detección rápida y precisa de infecciones latentes puede proporcionar a los acuicultores información confiable sobre el estado de la infección, para la prevención temprana y el control de brotes de enfermedades. A medida que aumentan los esfuerzos por aumentar los niveles de producción y el interés por la investigación en la acuicultura, también se está expandiendo el desarrollo de los métodos de biovigilancia. Las instalaciones de investigación pueden utilizar la tecnología, las universidades, las agencias gubernamentales y las granjas de mariscos para convertirse en un elemento básico para la detección de patógenos en la acuicultura en México y otros países.
Agradecimientos
Esta investigación fue apoyada por el Consejo Nacional de Ciencia y Tecnología (CONACYT, México; subvención no. 247567 a NE). IMA recibió una beca (no. 26089) del CONACYT para el desarrollo de este trabajo. Agradecemos a los Comités de Sanidad Acuícola de Baja California Sur, Nayarit, Sonora y Sinaloa los avisos de la presencia de patógenos en las granjas acuícolas y la recolección de muestras.
REFERENCIAS
Arunrut N, Prombun P, Saksmerprome V, Flegel TW, Kiatpathomchai W. 2011. Rapid and sensitive detection of infectious hypodermal and hematopoietic necrosis virus by loop-mediated isothermal amplification combined with a lateral flow dipstick. J Virol Methods. 171(1):21-25. https://doi.org/10.1016/j.jviromet.2010.09.022 [ Links ]
Becherer L, Borst N, Bakheit M, Frischmann S, Zengerle R, von Stetten F. 2020. Loop-mediated isothermal amplification (LAMP) - review and classification of methods for sequence-specific detection. Anal Methods. 12(6):717-746. https://doi.org/10.1039/c9ay02246e [ Links ]
Besuschio SA, Llano-Murcia M, Benatar AF, Monnerat S, Cruz I, Picado A, Curto MA, Kubota Y, Wehrendt DP, Pavia P, et al. 2017. Analytical sensitivity and specificity of a loop-mediated isothermal amplification (LAMP) kit prototype for detection of Trypanosoma cruzi DNA in human blood samples. PLoS Negl Trop Dis. 11(7):e0005779. https://doi.org/10.1371/journal.pntd.0005779 [ Links ]
Boehme CC, Nabeta P, Henostroza G, Raqib R, Rahim Z, Gerhardt M, Sanga E, Hoelscher M, Notomi T, Hase T, et al. 2007. Operational feasibility of using loop-mediated isothermal amplification for diagnosis of pulmonary tuberculosis in microscopy centers of developing countries. J Clin Microbiol. 45(6):1936-1940. https://doi.org/10.1128/JCM.02352-06 [ Links ]
Cáceres-Martínez J, Vásquez-Yeomans R, Padilla-Lardizábal G, del Río Portilla MA. 2008. Perkinsus marinus in pleasure oyster Crassostrea corteziensis from Nayarit, Pacific coast of México. J Invert Pathol. 99(1):66-73. https://doi.org/10.1016/j.jip.2008.03.005 [ Links ]
Chen S, Ge B. 2010. Development of a toxR-based loop-mediated isothermal amplification assay for detecting Vibrio parahaemolyticus. BMC Microbiol. 10(1):41. https://doi.org/10.1186/1471-2180-10-41 [ Links ]
Dhama K, Karthik K, Chakraborty S, Tiwari R, Kapoor S, Kumar A, Thomas P. 2014. Loop-mediated isothermal amplification of DNA (LAMP): a new diagnostic tool lights the world of diagnosis of animal and human pathogens: a review. PJBS. 17(2):151-166. https://doi.org/10.3923/pjbs.2014.151.166 [ Links ]
Enríquez-Espinoza TL, Grijalva-Chon JM, Castro-Longoria R, Ramos-Paredes J. 2010. Perkinsus marinus in Crassostrea gigas in the Gulf of California. Dis Aquat Organ. 89(3):269-273. https://doi.org/10.3354/dao02199 [ Links ]
[FAO] Food and Agriculture Organization of United Nations. 2019. FAO Yearbook of fishery and aquaculture statistics. Rome (Italy): FAO. 84 p. ISBN 978-92-5-131669-6. [ Links ]
Feng C, Wang C, Lin X, Zhang Y, Lv J, Deng J, Yuan X, Mei L, Wu S. 2013. Development of a loop-mediated isothermal amplification method for detection of Perkinsus spp. in mollusks. Dis Aquat Organ. 104(2):141-148. https://doi.org/10.3354/dao02591 [ Links ]
Francois P, Tangomo M, Hibbs J, Bonetti EJ, Boehme CC, Notomi T, Perkins MD, Schrenzel J. 2011. Robustness of a loop-mediated isothermal amplification reaction for diagnostic applications. FEMS Immunol Med Mic. 62(1):41-48. https://doi.org/10.1111/j.1574-695x.2011.00785.x [ Links ]
Goto M, Honda E, Ogura A, Nomoto A, Hanaki KI. 2009. Colorimetric detection of loop-mediated isothermal amplification reaction by using hydroxy naphthol blue. BioTechniques. 46(3):167-172. https://doi.org/10.2144/000113072 [ Links ]
Hasson KW, Fan Y, Reisinger T, Venuti J, Varner PW. 2006. White-spot syndrome virus (WSSV) introduction into the Gulf of Mexico and Texas freshwater systems through imported, frozen bait-shrimp. Dis Aquat Organ. 71(2):91-100. https://doi.org/10.3354/dao071091 [ Links ]
Iwamoto T, Sonobe T, Hayashi K. 2003. Loop-mediated isothermal amplification for direct detection of Mycobacterium tuberculosis complex, M. avium, and M. intracellulare in sputum samples. J Clin Microbiol. 41(6):2616-2622. https://doi.org/10.1128/JCM.41.6.2616-2622.2003 [ Links ]
Jaroenram W, Kiatpathomchai W, Flegel TW. 2009. Rapid and sensitive detection of white spot syndrome virus by loop-mediated isothermal amplification combined with a lateral flow dipstick. Mol Cell Probe. 23(2):65-70. https://doi.org/10.1016/j.mcp.2008.12.003 [ Links ]
Kaneko H, Kawana T, Fukushima E, Suzutani T. 2007. Tolerance of loop-mediated isothermal amplification to a culture medium and biological substances. J Biochem Biophys Methods. 70(3):499-501. https://doi.org/10.1016/j.jbbm.2006.08.008 [ Links ]
Khan M, Li B, Jiang Y, Weng Q, Chen Q. 2017. Evaluation of different PCR-based assays and LAMP method for rapid detection of Phytophthora infestans by targeting the ypt1 gene. Front Microbiol. 8:1920. https://doi.org/10.3389/fmicb.2017.01920 [ Links ]
Khunthong S, Jaroenram W, Arunrut N, Suebsing R, Mungsantisuk I, Kiatpathomchai W. 2013. Rapid and sensitive detection of shrimp yellow head virus by loop-mediated isothermal amplification combined with a lateral flow dipstick. J Virol Methods. 188(1-2):51-56. https://doi.org/10.1016/j.jviromet.2012.11.041 [ Links ]
Kiatpathomchai W, Jaroenram W, Arunrut N, Jitrapakdee S, Flegel TW. 2008. Shrimp Taura syndrome virus detection by reverse transcription loop-mediated isothermal amplification combined with a lateral flow dipstick. J Virol Methods. 153(2):214-217. https://doi.org/10.1016/j.jviromet.2008.06.025 [ Links ]
Kongrueng J, Tansila N, Mitraparp-arthorn P, Nishibuchi M, Vora, GJ, Vuddhakul V. 2015. LAMP assay to detect Vibrio parahaemolyticus causing acute hepatopancreatic necrosis disease in shrimp. Aquac Int. 23(5):1179-1188. https://doi.org/10.1007/s10499-014-9874-3 [ Links ]
Kono T, Savan R, Sakai M, Itami T. 2004. Detection of white spot syndrome virus in shrimp by loop-mediated isothermal amplification. J Virol Methods. 115(1):59-65. https://doi.org/10.1016/j.jviromet.2003.09.015 [ Links ]
Li J, Macdonald J. 2015. Advances in isothermal amplification: novel strategies inspired by biological processes. Biosens Bioelectron. 64:196-211. https://doi.org/10.1016/j.bios.2014.08.069 [ Links ]
Maralit BA, Caipang CMA, Santos MD, Calpe AT, Maningas MBB. 2012. Development of primers for loop-mediated isothermal amplification of Philippine white spot syndrome virus isolates. AACL Bioflux. 5(4):274-281. [ Links ]
Mekata T, Kono T, Savan R, Sakai M, Kasornchandra J, Yoshida T, Itami T. 2006. Detection of yellow head virus in shrimp by loop-mediated isothermal amplification (LAMP). J Virol Methods. 135(2):151-156. https://doi.org/10.1016/j.jviromet.2006.02.012 [ Links ]
Mekata T, Sudhakaran R, Kono T, Supamattaya K, Linh NTH, Sakai M, Itami T. 2009. Real-time quantitative loop-mediated isothermal amplification as a simple method for detecting white spot syndrome virus. Lett Appl Microbiol. 48(1):25-32. https://doi.org/10.1111/j.1472-765x.2008.02479.x [ Links ]
Mori Y, Kanda H, Notomi T. 2013. Loop-mediated isothermal amplification (LAMP): recent progress in research and development. J Infect Chemother. 19(3):404-411. https://doi.org/10.1007/s10156-013-0590-0 [ Links ]
Mori Y, Kitao M, Tomita N, Notomi T. 2004. Real-time turbidimetry of LAMP reaction for quantifying template DNA. J Biochem Biophys Methods. 59(2):145-157. https://doi.org/10.1016/j.jbbm.2003.12.005 [ Links ]
Mori Y, Nagamine K, Tomita N, Notomi T. 2001. Detection of loop-mediated isothermal amplification reaction by turbidity derived from magnesium pyrophosphate formation. Biochem Biophys Res Commun. 289(1):150-154. https://doi.org/10.1006/bbrc.2001.5921 [ Links ]
Mori Y, Notomi T. 2009. Loop-mediated isothermal amplification (LAMP): a rapid, accurate, and cost-effective diagnostic method for infectious diseases. J Infect Chemother. 15(2):62-69. https://doi.org/10.1007/s10156-009-0669-9 [ Links ]
Nagamine K, Hase T, Notomi T. 2002. Accelerated reaction by loop-mediated isothermal amplification using loop primers. Mol Cell Probe. 16(3):223-229. https://doi.org/10.1006/mcpr.2002.0415 [ Links ]
Nagamine K, Watanabe K, Ohtsuka K, Hase T, Notomi T. 2001. Loop-mediated isothermal amplification reaction using a nondenatured template. Clin Chem. 47(9):1742-1743. https://doi.org/10.1093/clinchem/47.9.1742 [ Links ]
Notomi T, Mori Y, Tomita N, Kanda H. 2015. Loop-mediated isothermal amplification (LAMP): principle, features, and future prospects. J Microbiol. 53(1):1-5. https://doi.org/10.1007/s12275-015-4656-9 [ Links ]
Notomi T, Okayama H, Masubuchi H, Yonekawa T, Watanabe K, Amino N, Hase T. 2000. Loop-mediated isothermal amplification of DNA. Nucleic Acids Res. 28:(12):e63. https://doi.org/10.1093/nar/28.12.e63 [ Links ]
Nzelu CO, Kato H, Peters NC. 2019. Loop-mediated isothermal amplification (LAMP): An advanced molecular point-of-care technique for the detection of Leishmania infection. PLoS Negl Trop Dis. 13(11):e0007698. https://doi.org/10.1371/journal.pntd.0007698 [ Links ]
[OIE] World Organisation for Animal Health. 2019. Manual of Diagnostic Tests for Aquatic Animals = Manual de las Pruebas de Diagnóstico para los Animales Acuáticos. [place unknown]: OIE; accessed Nov 2019. https://www.oie.int/es/normas/manual-acuatico/acceso-en-linea/ [ Links ]
Palomino-Camargo C, González-Muñoz Y. 2014. Técnicas moleculares para la detección e identificación de patógenos en alimentos: Ventajas y limitaciones = Molecular techniques for detection and identification of pathogens in food: advantages and limitations. Rev Peru Med Exp. Salud Publica. 31(3):535-546. [ Links ]
[SAGARPA] Secretaria de Agricultura, Ganadería, Desarrollo rural, Pesca y Alimentación. 2017. Anuario Estadístico de Acuicultura y Pesca 2017. México: CONAPESCA; accessed Jan 2019. https://www.conapesca.gob.mx/work/sites/cona/dgppe/2017/ANUARIO_ESTADISTICO_2017.pdf [ Links ]
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular Cloning: A Laboratory Manual Vol. 1, 2 and 3. 2nd. ed. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. 1669 p. [ Links ]
Soto-Rodriguez SA, Gomez-Gil B, Lozano R, Roque A. 2010. Density of vibrios in hemolymph and hepatopancreas of diseased Pacific white shrimp, Litopenaeus vannamei, from northwestern Mexico. J World Aquacult Soc. 41(S1):76-83.https://doi.org/10.1111/j.1749-7345.2009.00335.x [ Links ]
Tanner NA, Zhang Y, Evans TC. 2015. Visual detection of isothermal nucleic acid amplification using pH-sensitive dyes. BioTechniques. 58(2):59-68. https://doi.org/10.2144/000114253 [ Links ]
Tomita N, Mori Y, Kanda H, Notomi T. 2008. Loop-mediated isothermal amplification (LAMP) of gene sequences and simple visual detection of products. Nat Protoc. 3(5):877-882. https://doi.org/10.1038/nprot.2008.57 [ Links ]
Tomlinson J. 2013. In-field diagnostics using loop-mediated isothermal amplification. In: Dickinson M, Hodgetts J (eds.), Phytoplasma. Methods in Molecular Biology (Methods and Protocols), vol. 938. Totowa (NJ): Humana Press. https://doi.org/10.1007/978-1-62703-089-2_25 [ Links ]
Wong YP, Othman S, Lau YL, Radu S, Chee HY. 2018. Loop-mediated isothermal amplification (LAMP): a versatile technique for detection of micro-organisms. J Appl Microbiol. 124(3):626-643. https://doi.org/10.1111/jam.13647 [ Links ]
Xu H, He L, Lv S, Gong Q, Li S. 2012. Establishment of universal loop-mediated isothermal amplification method (LAMP) for rapid detection of pathogenic Vibrio spp. in aquatic organisms. African J Microbiol Res. 6(14):3447-3454. https://doi.org/10.5897/AJMR11.1417 [ Links ]
Yamazaki W, Ishibashi M, Kawahara R, Inoue K. 2008. Development of a loop-mediated isothermal amplification assay for sensitive and rapid detection of Vibrio parahaemolyticus. BMC Microbiol. 8(1):163. https://doi.org/10.1186/1471-2180-8-163 [ Links ]
Recibido: 23 de Enero de 2020; Aprobado: 16 de Marzo de 2021











 texto en
texto en 


