Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Ciencias marinas
versión impresa ISSN 0185-3880
Cienc. mar vol.42 no.4 Ensenada dic. 2016
https://doi.org/10.7773/cm.v42i4.2662
Artículos
Rastreabilidad del pedigrí en camarón blanco ( Litopenaeus vannamei ) mediante marcadores genéticos: Una comparación entre microsatélites y SNP
1 Centro de Investigaciones Biológicas del Noroeste (CIBNOR), Calle IPN 195, La Paz, Baja California Sur, México.
El mejoramiento genético en el camarón cultivado es de amplio interés para incrementar la productividad. La evaluación de parámetros genéticos (e.g., heredabilidad, interacción entre genotipo y ambiente, endogamia) y el diseño de planes de apareamiento son elementos necesarios para el mejoramiento genético. La rastreabilidad del pedigrí con marcadores genéticos es importante para estos objetivos. El objetivo de este trabajo fue comparar el desempeño de 2 paneles de marcadores genéticos (microsatélites y polimorfismos de nucleótido sencillo [SNP]) para rastrear el pedigrí en una población de camarón blanco (Litopenaeus vannamei) de cultivo. Se determinó el pedigrí de la progenie de 81 familias de hermanos completos mediante el uso de microsatélites y SNP como marcadores genéticos. Un panel de 76 SNP probó tener un mejor desempeño que los microsatélites para la asignación de parentesco, obteniéndose un 94-96% de asignación en una muestra de la progenie (n = 192). Un número mínimo de 50 SNP con una proporción de 60% de loci con una frecuencia alélica mínima de 0.3 es adecuado para una asignación exitosa de pedigrí. Se sugiere utilizar los marcadores SNP para determinar el parentesco de la progenie de camarón proveniente de padres conocidos.
Palabras clave: cultivo de camarón; asignación de parentesco; identificación genética; probabilidad de asignación; probabilidad de exclusión
To increase productivity, genetic improvement in cultivated shrimp is of much interest. Evaluation of genetic parameters (e.g., heritability, genotype-environment interaction, inbreeding) and the design of appropriate breeding plans are necessary steps towards genetic improvement. Pedigree traceability by genetic markers is relevant to these objectives. The aim of this study was to compare the performance of 2 genetic marker panels (microsatellites and single nucleotide polymorphisms [SNPs]) for pedigree traceability of a cultivated stock of whiteleg shrimp (Litopenaeus vannamei). Pedigree of a stock from 81 full-sib families reared in a common environment was assessed with microsatellites and SNPs as genetic markers. The panel of 76 SNPs performed better than microsatellites, allowing 94-96% of parentage assignment of the tested progeny (n = 192). A minimum number of 50 SNPs with a proportion of 60% loci with a minimum allele frequency of 0.3 is suitable for successful pedigree assignment. SNP markers are suggested for confidently testing the pedigree of shrimp from known parental broodstock.
Key words: shrimp farming; parentage assignment; genetic identification; assignment likelihood; exclusion probability
INTRODUCCIÓN
Se estima que sólo el 10% de la producción acuícola mundial está asociada a programas de mejoramiento genético, por lo que existe un alto potencial para el incremento de la producción a través de esta metodología (Gjedrem et al. 2012). El camarón blanco, Litopenaeus vannamei, es una de las especies para la cual existe un esfuerzo creciente en la instrumentación de programas de selección genética (Andriantahina et al. 2013). Ésta es la especie más importante en la acuacultura de camarones marinos en el mundo, ya que contribuye con más del 71% del total del volumen de producción (FAO 2014).
Rastrear el pedigrí en los núcleos genéticos es necesario para la estimación de parámetros genéticos como la heredabilidad, la interacción entre genotipo y ambiente (G × A) y la endogamia (Gjedrem 2010). Además, la determinación de las relaciones de parentesco entre reproductores es útil para el diseño de las cruzas que minimicen la pérdida de diversidad genética y el incremento de la endogamia y maximicen la productividad de la siguiente generación (Gjedrem 2010).
La rastreabilidad del pedigrí en los programas de mejoramiento genético se lleva a cabo mediante la crianza de familias por separado y/o mediante el marcaje físico con elastómeros de color. Sin embargo, dado que esta estrategia tiene limitaciones respecto a la identificación individual de los ejemplares y la asignación de parentesco, el marcaje genético se ha sugerido como una alternativa (Gjedrem 2010). Yue y Xia (2014) resumen varias consideraciones prácticas para este tipo de análisis particularizando en microsatélites y polimorfismos de nucleótido sencillo (SNP).
La factibilidad de los microsatélites para el análisis de parentesco se ha estudiado en algunas especies de camarón, tales como Marsupenaeus japonicus (Jerry et al. 2004), Penaeus monodon (Jerry et al. 2006) y Fenneropenaeus chinensis (Dong et al. 2006). Sin embargo, se ha reportado que 10% o más de los ejemplares no son asignados adecuadamente a sus padres putativos (Jerry et al. 2004, Dong et al. 2006, Jerry et al. 2006). El índice de falta de asignaciones con el uso de microsatélites ha sido también reportado para peces (e.g., Perez-Enriquez et al. 1999, Christie et al. 2010).
Con el desarrollo de la secuenciación masiva de nueva generación, se abrió la posibilidad de utilizar los SNP en estudios de parentesco. Lapègue et al. (2014) utilizaron marcadores SNP para identificar el pedigrí de 2 especies de ostión cultivado y sugirieron utilizar un panel de al menos 150 SNP para poder identificar el parentesco de más del 95% de la progenie. Para el camarón tigre, P. monodon, Sellars et al. (2013) obtuvieron asignaciones adecuadas con paneles de 56 y 63 SNP. En salmón, se ha demostrado que con 60 loci de SNP es posible diferenciar genéticamente entre salmón silvestre y salmón de cultivo (Karlsson et al. 2011).
En el camarón blanco, L. vannamei, los marcadores genéticos se han utilizado principalmente para la caracterización de la diversidad y composición genética de poblaciones silvestres o lotes de cultivo (Cruz et al. 2004, Freitas y Galetti 2005, Perez-Enriquez et al. 2009, Vela-Avitúa et al. 2013). Son limitados los estudios dirigidos a la identificación individual o asignación de parentesco de individuos cultivados (Perez-Enriquez R, datos no publicados; Perez F com. pers.). El objetivo de este trabajo fue probar y comparar el desempeño de 2 paneles de marcadores genéticos (microsatélites y SNP) para rastrear el pedigrí en una población de camarón blanco de cultivo.
MATERIALES Y MÉTODOS
Obtención de las muestras
Se seleccionó un lote de quinta generación del núcleo genético de la empresa Acuacultura Mahr para la obtención de 81 familias de hermanos completos mediante inseminación artificial. Se sembraron cantidades homogéneas de nauplios por familia en un tanque común, y se siguieron los procedimientos estándar de engorda hasta que los organismos alcanzaron un peso promedio de 20 g. Luego, se obtuvo una muestra aleatoria de 192 individuos de la progenie a este peso. Los pleópodos de los reproductores (n = 162) y la progenie se preservaron en etanol al 80%.
Obtención de los genotipos
El ADN se obtuvo de los pleópodos utilizando el Wizard SV 96 Genomic DNA Purification System (A2371, Promega). Se obtuvieron perfiles genéticos individuales con los microsatélites y SNP. Se utilizó un lote de 5 microsatélites previamente publicados: Pvan1758 y Pvan1815 (Cruz et al. 2002), Lvan05 (Perez-Enriquez et al. 2009) y TumxLv8.256 y TumxLv10.312 (Meehan et al. 2003). Los microsatélites Pvan1758, Lvan05 y TumxLv8.26 se amplificaron mediante PCR en un volumen de reacción final de 11 µL (1 µL de ADN a 10-95 ng µL-1, amortiguador 1×, 3.5 mM de MgCl2, 0.25 mM de dNTP, 0.39 µM de cada iniciador sentido y antisentido, 0.025 U µL-1 de Taq polimerasa) bajo las condiciones reportadas por los respectivos autores. Para el locus Pvan1815, se utilizó un protocolo de descenso gradual de temperatura (95 ºC por 3 min, 30 ciclos a 94 ºC por 45 s, temperatura de acoplamiento por 45 s [iniciando en 60 ºC y disminuyendo cada ciclo 0.3 ºC hasta alcanzar 51 ºC], 65 ºC por 1 min y extensión final a 65 ºC por 20 min). Para el locus TumxLv10.312, se utilizó el protocolo modificado de Yoshiday y Awaji (2000) (94 ºC por 2 min; 5 ciclos a 94 ºC por 5 s, 60 ºC por 1 min y 72 ºC por 1 min; 20 ciclos a 94 ºC por 1 s, 60 ºC por 1 min y 72 ºC por 1 min; y extensión final a 72 ºC por 10 min). Además, se utilizó un lote adicional de 5 loci (Livan04, Livan13, Livan44, Livan51, Livan60; Tabla 1), para los cuales se usaron 1.5 mM de MgCl2 y la misma cantidad de los otros componentes mencionados arriba con las condiciones de amplificación siguientes: 95 ºC por 3 min; 30 ciclos a 94 ºC por 35 s, 57 ºC por 40 s y 72 ºC por 40 s; y extensión final a 72 ºC por 5 min. Los productos de la PCR fueron analizados en un secuenciador (ABI3130, Applied Biosystems). Los genotipos se obtuvieron con el programa GeneMapper 4.0.
Los genotipos de SNP de un panel de 76 loci se obtuvieron en el Center for Aquaculture Technologies (San Diego, California, http://aquatechcenter.com/) utilizando ensayos de PCR específicos para cada alelo en una plataforma instrumental EP1 (Fluidigm Corporation, San Francisco, CA). Los genotipos se analizaron en un archivo Excel.
Análisis del pedigrí
Para ambos grupos (reproductores y progenie), se estimó la diversidad genética (número de alelos por locus y heterocigosidad observada y esperada) con ambos marcadores genéticos (microsatélites y SNP). Además, se calculó la probabilidad de identidad (probabilidad de que 2 individuos tengan exactamente el mismo genotipo múltiple) y la probabilidad combinada de no exclusión (probabilidad de no excluir a un padre no verdadero) mediante el programa Cervus 3.0.3 (Kalinowski et al. 2007). La presencia de alelos nulos y otras irregularidades de genotipificación se analizaron con el programa Microchecker 2.2.3 (Van Oosterhout et al. 2004).
Con ambos paneles, se realizaron análisis de paternidad para determinar el pedigrí (la familia de origen) de cada individuo de la progenie. Para estos análisis sólo se utilizaron ejemplares con un mínimo de 80% de genotipos completos (i.e., 8 microsatélites o 60 SNP).
Los análisis de asignación de parentesco se realizaron mediante exclusión directa y probabilística. La exclusión directa, que implica la eliminación de progenitores potenciales que tengan más de 2 loci sin coincidencia con los alelos de la progenie, se llevó a cabo con el programa Vitassign (Vandeputte et al. 2006). Este programa también realiza una simulación para determinar el poder de la asignación calculando el porcentaje de asignaciones únicas. Para la asignación probabilística se utilizó el programa Cervus 3.0.3 (Kalinowski et al. 2007), el cual otorga un valor mínimo de asignación (denominado LOD) a la pareja más probable en función de parámetros definidos (0.90 de proporción de padres muestreados, 0.01 de error para cálculos de probabilidad, número mínimo de loci genotipificados y 95% de nivel de confianza). Se realizó un análisis comparativo del porcentaje de asignación de paternidad obtenido con los microsatélites y SNP tanto a partir de simulaciones como de los datos reales.
Se elaboraron curvas de rarefacción para estimar el número mínimo de loci de SNP necesario para obtener más del 98% de asignaciones correctas. Para ello, los loci de los SNP se clasificaron en 2 grupos: el grupo con frecuencia alélica mínima (FAM) ≥ 0.3 (ambos alelos presentaron una frecuencia entre 0.3 y 0.7) y el grupo con FAM < 0.3 (uno de los alelos presentó una frecuencia menor que 0.3). Se elaboraron 4 curvas de rarefacción en función de la proporción (20%, 40%, 50% y 60%) de loci con FAM ≥ 0.3. Para cada curva, se realizaron análisis de paternidad con el programa Vitassign (permitiendo 2 no coincidencias) y con el programa Cervus (1.0 de proporción de padres muestreados, 0.001 de error para cálculos de probabilidad y 95% de nivel de confianza). Las curvas se construyeron obteniendo las asignaciones con 10, 20, 30, 40, 50, 60 y 70 loci, seleccionados al azar del lote original de 76 loci. Únicamente se utilizaron los ejemplares que tuvieron asignación correcta de acuerdo con el análisis de pedigrí descrito arriba.
RESULTADOS
De los 192 individuos de la progenie analizados, se obtuvieron lecturas completas de 168 con los microsatélites (87.5%) y 186 con los SNP (96.9%). Los individuos con genotipos incompletos no se consideraron para los análisis de parentesco.
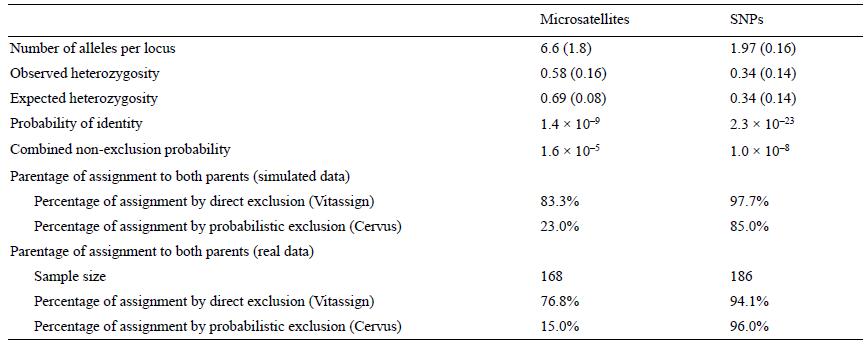
Los valores de diversidad genética (número de alelos por locus, heterocigosidad) fueron mayores con los microsatélites que con los SNP; sin embargo, la probabilidad de no exclusión fue 3 órdenes de magnitud más potente (i.e., un valor de probabilidad más bajo) con los SNP (Tabla 2). Esta característica se reflejó en un porcentaje mayor de asignación de paternidad al utilizar el panel de SNP con los 2 métodos de exclusión (directa y probabilística), tanto en la simulación como con los datos reales (Tabla 2). El uso del panel de 76 SNP resultó en una asignación de 94% a 96% de la progenie a una pareja única de padres, ampliamente mayor que lo obtenido con los microsatélites. Cabe mencionar que el 4-6% restante no se refiere a ejemplares con asignaciones múltiples, sino a una falta de asignación, lo que indica que probablemente esos ejemplares pertenecen a otros lotes de desove. Se detectaron alelos nulos en 3 loci (Lvan05, Livan13 y Livan60), que pueden explicar la reducida proporción de asignaciones con estos marcadores.
Tabla 2 Valores promedio de diversidad genética (desviación estándar en paréntesis), probabilidad de identidad, probabilidad combinada de no exclusión de ambos padres y porcentaje de asignaciones de parentesco de la progenie (datos simulados y reales) obtenidos con los paneles de microsatélites y SNP por exclusión directa y probabilística.
Las curvas de rarefacción indican que las pruebas de paternidad por exclusión directa usando 50 loci alcanzaron un 100% de asignaciones cuando el 60% de los loci tuvieron una FAM ≥ 0.3, y entre 98.3% y 99.4% de asignaciones para las proporciones de FAM ≥ 0.3 de 20%, 40% y 50% (Fig. 1). Las asignaciones probabilisticas con 50 loci alcanzaron el 100% en 3 proporciones de FAM ≥ 0.3 (40%, 50% y 60%; Fig. 1).

Figura 1 Curvas de rarefacción de las asignaciones de parentesco con el panel de SNP por exclusión directa (programa Vitassign) y exclusión probabilística (programa Cervus), de acuerdo con la proporción de loci con frecuencia alélica mínima (MAF) ≥ 0.3.
Considerando 50 loci como el número mínimo para un análisis de asignación de paternidad, el error estimado (la diferencia en las parejas de padres obtenidas entre métodos) mostró una tendencia de disminución del error en función de la proporción de loci con FAM ≥ 0.3, y la proporción del 60% fue la proporción con la que se obtuvo el valor más bajo (2.3%; Fig. 2).
DISCUSIÓN
Éste es el primer trabajo del conocimiento de los autores acerca de la utilidad de un panel de SNP en L. vannamei. Se demostró que la utilización del panel de 76 SNP es adecuado para la identificación de la genealogía o pedigrí del camarón blanco de cultivo. A pesar de una menor variabilidad en comparación con los microsatélites, el panel de SNP presenta mayor certidumbre en las asignaciones de parentesco. La menor resolución de los microsatélites en comparación con los SNP ha sido descrita también para otras especies acuáticas como la tilapia (Trọng et al. 2013) y el camarón tigre, P. monodon (Sellars et al. 2014). Las diferencias estriban en errores de genotipificación como resultado de la presencia de alelos nulos, la supresión de alelos y las mutaciones, estas últimas derivadas de una relativa alta tasa de mutación de los microsatélites (Ellegren 2000, Pompanon et al. 2005, Trọnget al. 2013, Lapègue et al. 2014). Sin embargo, de manera similar a lo reportado por Trọng et al. (2013), la eliminación de loci con frecuencias elevadas de alelos nulos no contribuyó a incrementar la proporción de asignación.
El análisis de exclusión directa con un error permitido de genotipificación de los SNP del 2.6% (2 no coincidencias) resultó en más del 96% de la progenie con asignación a una sola pareja, lo cual brinda una alta certidumbre en la asignación. Este nivel de precisión requiere del conocimiento del plan de apareamiento bajo el cual se obtuvo la progenie, es decir, qué hembra fue fecundada por qué macho. Sin embargo, aun si se desconoce el plan de apareamiento, el uso del panel de SNP es factible ya que el análisis probabilístico es capaz de identificar las familias con un 95% de confianza y un error menor que 3%. Trọng et al (2013) obtuvieron niveles similares de asignación de parentesco con ambos métodos analíticos (exclusión directa y probabilística) para una población de cultivo de tilapia.
Aun cuando el número de loci del panel de SNP (76) es ligeramente mayor que el número utilizado por Sellars et al. (2014) para P. monodon (56 y 63 SNP), los resultados indican que al utilizar un panel con una proporción de loci con FAM ≥ 0.3 de 60%, 50 loci serían suficientes para obtener una asignación correcta para más del 98% de la progenie. Estos resultados difieren de lo obtenido por Lapègue et al. (2014) para ostras y Trọng et al. (2013) para tilapia, quienes utilizaron paneles de entre 122 y 384 loci. De hecho, Lapègue et al. (2014) recomiendan un mínimo de 150 loci con un FAM promedio de 0.3 para obtener una asignación suficientemente potente.
La probabilidad de asignación está en función de las frecuencias alélicas de la población (Kalinowski et al. 2007); por lo tanto, los individuos emparentados dentro del lote de reproductores, los cuales comparten más alelos que individuos no emparentados, disminuirán la probabilidad de asignación correcta. En este sentido, Trọng et al. (2013) señalan que una de las razones de obtener una baja a moderada asignación en la población de tilapia que analizaron se debe a los altos niveles de parentesco entre individuos derivados de un programa de selección por talla por más de 8 generaciones. En el presente estudio, el lote se encontraba en la quinta generación de selección, para la cual la endogamia se ha mantenido en un nivel reducido de 1.1-2.4% (Perez-Enriquez et al., datos no publicados). Aunque esta baja endogamia pudiera no tener efecto en la precisión de asignación con los SNP, algunos microsatélites parecen haber sido afectados, ya que incrementó la frecuencia de alelos nulos. Cuando se realizan análisis de pedigrí es recomendable mantener registros adecuados del manejo histórico del lote de reproductores.
La certidumbre en la asignación de pedigrí con SNP abre posibilidades para realizar estimaciones de heredabilidad, interacción G × A y manejo reproductivo en condiciones de cultivo en ambientes comunes y bajo condiciones en las cuales el marcaje físico es inadecuado o ineficiente. Con las plataformas de análisis de este estudio, el uso de SNP en camarón se sugiere como alternativa de mayor beneficio- costo (costo por muestra, tasa de genotipificación, tiempo de análisis) que los microsatélites.
AGRADECIMIENTOS
Este estudio fue financiado por el CIBNOR (proyecto 985-0). Se agradece a Acuacultura Mahr SA de CV y Blue Genetics Mexico SA de CV el suministro de las muestras de reproductores y progenie. Ira Fogel de CIBNOR realizó la edición de la versión en inglés.
REFERENCIAS
Andriantahina F, Liu X, Feng T, Xiang J. 2013. Current status of genetics and genomics of reared penaeid shrimp: Information relevant to access and benefit sharing. Mar. Biotechnol. 15(4): 399-412. http://dx.doi.org/10.1007/s10126-013-9500-9 [ Links ]
Christie MR, Johnson DW, Stallings CD, Hixon MA. 2010. Self- recruitment and sweepstakes reproduction amid extensive gene flow in a coral-reef fish. Mol. Ecol. 19(5):1042-1057. http://dx.doi.org/10.1111/j.1365-294X.2010.04524.x [ Links ]
Cruz P, Ibarra AM, Mejía-Ruiz H, Gaffney PM, Perez-Enriquez R. 2004. Genetic variability assessed by microsatellites in a breeding program of Pacific white shrimp (Litopenaeus vannamei). Mar. Biotechnol. 6(2): 157-164. http://dx.doi.org/10.1007/s10126-003-0017-5 [ Links ]
Cruz P , Mejía-Ruiz CH, Perez-Enriquez R , Ibarra AM . 2002. Isolation and characterization of microsatellites in Pacific white shrimp Penaeus (Litopenaeus) vannamei. Mol. Ecol. Notes 2(3): 239-241. http://dx.doi.org/10.1046/j.1471-8286.2002.00211.x-i2 [ Links ]
Dong S, Kong J, Zhang T, Meng X, Wang R. 2006. Parentage determination of Chinese shrimp (Fenneropenaeus chinensis) based on microsatellite DNA markers. Aquaculture 258(1-4): 283-288. http://dx.doi.org/10.1016/j.aquaculture.2006.04.044 [ Links ]
Ellegren H. 2000. Microsatellite mutations in the germline: Implications for evolutionary inference. Trends Genet. 16(12): 551-558. http://dx.doi.org/10.1016/S0168-9525(00)02139-9 [ Links ]
[FAO] Food and Agriculture Organization of the United Nations. 2014. The State of World Fisheries and Aquaculture 2014. Rome, 223 pp. [ Links ]
Freitas PD, Galetti Jr PM. 2005. Assessment of the genetic diversity in five generations of a commercial broodstock line of Litopenaeus vannamei shrimp. Afr. J. Biotechnol. 4: 1362-1367. [ Links ]
Gjedrem T. 2010. Selection and Breeding Programs in Aquaculture. Springer, Dordrecht, The Netherlands. [ Links ]
Gjedrem T , Robinson N, Rye M. 2012. The importance of selective breeding in aquaculture to meet future demands for animal protein: A review. Aquaculture 350-353: 117-129. http://dx.doi.org/10.1016/j.aquaculture.2012.04.008 [ Links ]
Jerry DR, Evans BS, Kenway M, Wilson K. 2006. Development of a microsatellite DNA parentage marker suite for black tiger shrimp Penaeus monodon. Aquaculture 255(1-4): 542-547. http://dx.doi.org/10.1016/j.aquaculture.2005.11.026 [ Links ]
Jerry DR, Preston NP, Crocos PJ, Keys S, Meadows JRS, Li Y. 2004. Parentage determination of Kuruma shrimp Penaeus (Marsupenaeus) japonicus using microsatellite markers (Bate). Aquaculture 235(1-4): 237-247. http://dx.doi.org/10.1016/j.aquaculture.2004.01.019 [ Links ]
Kalinowski ST, Taper ML, Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Mol. Ecol. 16(5): 1099-1106. http://dx.doi.org/10.1111/j.1365-294x.2007.03089.x [ Links ]
Karlsson S, Moen T, Lien S, Glover KA, Hindar K. 2011. Genetic differences between farmed and wild Atlantic salmon identified from a 7K SNP-chip. Mol. Ecol. Resources 11(S1): 247-253. http://dx.doi.org/10.1111/j.1755-0998.2010.02959.x [ Links ]
Lapègue S, Harrang E, Heurtebise S, Flahauw E, Donnadieu C, Gayral P, Ballenghien M, Genestout L, Barbotte L, Mahla R, Haffray P, Klopp C. 2014. Development of SNP genotyping arrays in two shellfish species. Mol. Ecol. Resources 14(4): 820-830. http://dx.doi.org/10.1111/1755-0998.12230 [ Links ]
Meehan D, Xu Z, Zuniga G, Alcivar-Warren A. 2003. High frequency and large number of polymorphic microsatellites in cultured shrimp, Penaeus (Litopenaeus) vannamei (Crustacea: Decapoda). Mar. Biotechnol. 5(4): 311-330. http://dx.doi.org/10.1007/s10126-002-0092-z [ Links ]
Perez-Enriquez R , Hernández-Martínez F, Cruz P . 2009. Genetic diversity status of white shrimp Penaeus (Litopenaeus) vannamei broodstock in Mexico. Aquaculture 297(1-4): 44-50. http://dx.doi.org/10.1016/j.aquaculture.2009.08.038 [ Links ]
Perez-Enriquez R , Takagi, M, Taniguchi N. 1999. Genetic variability of a hatchery-reared stock of red sea bream (Pagrus major) used for stock enhancement, based on microsatellite DNA markers. Aquaculture 173(1-4): 413-423. http://dx.doi.org/10.1016/S0044-8486(98)00469-4 [ Links ]
Pompanon F, Bonin A, Bellemain E, Taberlet P. 2005. Genotyping errors: Causes, consequences and solutions. Nat. Rev. Genet. 6(11): 847-859. http://dx.doi.org/10.1038/nrg1707 [ Links ]
Sellars MJ, Dierens L, McWilliam S, Little B, Murphy B, Coman G, Barendse W, Henshall J. 2014. Comparison of microsatellite and SNP DNA markers for pedigree assignment in black tiger shrimp, Penaeus monodon. Aquacult. Res. 45(3): 417-426. http://dx.doi.org/10.1111/j.1365-2109.2012.03243.x [ Links ]
Trọng TQ, van Bers N, Crooijmans R, Komen H. 2013. A comparison of microsatellites and SNPs in parental assignment in the GIFT strain of Nile tilapia (Oreochromis niloticus): The power of exclusion. Aquaculture 388-391: 14-23. http://dx.doi.org/10.1016/j.aquaculture.2013.01.004 [ Links ]
Van Oosterhout C, Hutchinson WF, Wills DPM, Shipley P. 2004. MICRO-CHECKER: Software for identifying and correcting genotyping errors in microsatellite data. Mol. Ecol. Notes 4(3): 535-538. http://dx.doi.org/10.1111/j.1471-8286.2004.00684.x [ Links ]
Vandeputte M, Mauger S, Dupont-Nivet M. 2006. An evaluation of allowing for mismatches as a way to manage genotyping errors in parentage assignment by exclusion. Mol. Ecol. Resources 6(1): 265-267. http://dx.doi.org/10.1111/j.1471-8286.2005.01167.x [ Links ]
Vela-Avitúa S, Montaldo HH, Márquez-Valdelamar L, Campos- Montes GR, Castillo-Juárez H. 2013. Decline of genetic variability in a captive population of Pacific white shrimp Penaeus (Litopenaeus) vannamei using microsatellite and pedigree information. Electronic J. Biotechnol. 16(4). http://dx.doi.org/10.2225/vol16-issue4-fulltext-11 [ Links ]
Yoshida K, Awaji M. 2000. Improvement of polymerase chain reaction condition to detect polymorphic dinucleotide repeat microsatellite DNA marker in the puffer fish Fugu rubripes. Fish. Sci. 66(2): 397-399. http://dx.doi.org/10.1046/j.1444-2906.2000.00061.x [ Links ]
Yue GH, Xia JH. 2014. Practical considerations of molecular parentage analysis in fish. J. World Aquacult. Soc. 45(2): 89-103. http://dx.doi.org/10.1111/jwas.12107 [ Links ]
Recibido: Junio de 2016; Aprobado: Septiembre de 2016











 texto en
texto en 




