Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Ciencias marinas
versión impresa ISSN 0185-3880
Cienc. mar vol.38 no.1a Ensenada mar. 2012
Caracterización genética mediante microsatélites de Solea senegalensis (Soleidae, Pleuronectiformes) en poblaciones naturales de la costa atlántica del suroeste de la Península Ibérica*
Genetic characterization, based on microsatellite loci, of Solea senegalensis (Soleidae, Pleuronectiformes) in Atlantic coast populations of the SW Iberian Peninsula
E Díaz–Ferguson1,3, I Cross1, M Barrios1, A Pino2, J Castro2, C Bouza2, P Martínez2, L Rebordinos1*
1 Laboratorio de Genética, Universidad de Cádiz, Facultad de Ciencias del Mar, Polígono Río San Pedro s/n, Puerto Real 11510 Cádiz, Spain. *Corresponding author. E–mail: laureana.rebordinos@uca.es
2 Departamento de Genética, Universidad de Santiago de Compostela, Facultad de Veterinaria, 27002 Lugo, Spain.
3 Present address: Department of Biology, University of Florida, Gainesville FL, 32611–8525, USA.
Received March 2010
Received in revised form May 2011
Accepted August 2011
RESUMEN
El lenguado senegalés, Solea senegalensis, es una especie de pez plano importante desde el punto de vista comercial, cuyas pesquerías principales están localizadas en las costas atlánticas del suroeste de la Península Ibérica. La información existente sobre la estructura genética y conectividad entre poblaciones naturales de esta especie es muy reducida y está limitada a la utilización de marcadores de ADN mitocondrial. Este trabajo presenta el análisis de ocho poblaciones naturales del Atlántico situadas entre Lisboa y el golfo de Cádiz, usando nueve loci microsatélites, mediante los que se detecta una alta variabilidad genética intrapoblacional. De un total de 72 pruebas realizadas, 19 mostraron desviaciones estadísticamente significativas de las condiciones de equilibrio de Hardy–Weinberg (24%) y 7 después de aplicar las correcciones de Bonferroni (9%) en las poblaciones analizadas. La mayoría de las desviaciones en las poblaciones fueron debidas al déficit de heterocigotos (valores de Fis positivos) atribuidos a alelos nulos observados principalmente en dos loci (Sol GA12 y Sol MIJ) y una población (Río San Pedro). Solamente se detectó diferenciación genética entre parejas de poblaciones que incluían muestras de Río San Pedro (cinco de siete comparaciones) y Barbate (dos de siete comparaciones). La estructura genética observada complementa la información existente con datos mitocondriales y debería contribuir a la futura gestión de pesquerías de esta especie, puesto que algunas pesquerías no son ambientalmente sostenibles.
Palabras clave: pesquerías de peces planos, caracterización genética, microsatélites, lenguado, Solea senegalensis.
ABSTRACT
The Senegalese sole, Solea senegalensis, is an important commercial flatfish species with major fisheries located off the Atlantic coast of the SW Iberian Peninsula. Past information about the genetic structure and connectivity among natural populations of this species has been restricted to mitochondrial DNA analysis. The present analysis of eight natural Atlantic populations fished from Lisbon to the Gulf of Cádiz, using nine microsatellite loci, identifies high intrapopulation genetic variability. Out of 72 tests performed, a total of 19 statistically significant deviations from Hardy–Weinberg (H–W) expectations (24%), and 7 after Bonferroni corrections (9%), were observed in the populations examined. Most deviations from H–W expectations were caused by heterozygote deficiencies (positive Fis values) attributed to null alleles predominantly occurring in two loci (Sol GA12 and Sol MIJ) and one population (Río San Pedro). Pairwise genetic differentiation among populations was only found in comparisons involving Río San Pedro samples (five out of seven comparisons) and Barbate samples (two out of seven comparisons). The observed genetic structure complements existing information from mitochondrial data and should contribute to future management of fisheries of this species, since some fisheries are not environmentally sustainable.
Key words: flatfish fisheries, genetic characterization, microsatellites, sole, Solea senegalensis.
INTRODUCCIÓN
Solea senegalensis (Kaup 1858) es un pez plano común que se encuentra en la costa atlántica desde el norte de Senegal hasta Perthuis Charantais (Francia) y en el mar Mediterráneo desde el estrecho de Gibraltar hasta la costa de Túnez. El lenguado senegalés es una especie gonocórica, bien adaptada a climas cálidos, y comúnmente criada en extensos estanques de tierra en la costa sur de Portugal y España. En esta región, es considerada la especie de mayor importancia comercial por su alto valor económico y la elevada calidad nutricional de su carne (Imsland et al. 2003, APROMAR 2010). Esta especie alcanza la madurez sexual entre los tres y cuatro años de edad (Cabral et al. 2007). Los peces desovan en zonas estuarinas donde permanecen durante unos dos años antes de que las larvas migren a zonas costeras caracterizadas por fondos arenosos (Dinis et al. 1999). En vista de que las zonas de desove se encuentran alejadas de las zonas de crianza, el transporte de huevos y larvas es un aspecto important. Al igual que en otros peces pelágicos, factores físicos (corrientes, profundidad, temperatura, límites de salinidad, giros hidrodinámicos y topografía del fondo marino) y biológicos (depredación y competencia) ejercen una gran influencia en la distribución y estructura genética de sus poblaciones (Wegner et al. 2003). Además, factores antropogénicos como la contaminación, degradación de hábitat y sobrepesca también afectan la distribución de peces y la estructura poblacional (Mustafa 1999, Cabral et al. 2007).
El estudio de la estructura genética y la conectividad de las poblaciones marinas es esencial para comprender el potencial evolutivo, la salud y la sostenibilidad de éstas; sin embargo, a pesar de mucho trabajo previo, existe poco conocimiento acerca de la conectividad entre los hábitats ocupados por esta especie a través de su distribución geográfica (Feral 2002, Vinagre et al. 2008). En peces planos, se han aplicado varios marcadores para estudiar la estructura genética y conectividad, incluyendo isoenzimas, proteínas sanguíneas, fragmentos de ADN polimórfico amplificados al azar, microsatélites y polimorfismos de la longitud de fragmentos amplificados (AFLP por sus siglas en inglés) (Bouza et al. 2002, Guarniero et al. 2002, Exadactylos et al. 2003, Florin y Höglund 2007, Garoia et al. 2007). A pesar de patrones de historia de vida similares entre las especies de peces planos, se han obtenido resultados opuestos en cuanto al flujo de genes y la diferenciación y diversidad genética (Borsa et al. 1997, Garoia et al. 2007).
Se han observado diferentes patrones de diferenciación a diversas escalas geográficas entre especies. Por ejemplo, la falta de diferenciación genética en el rodaballo de las poblaciones del Atlántico contrasta con la alta divergencia observada en el mar Báltico (Bouza et al. 2002, Nielsen et al. 2004). Asimismo, se han documentado diferencias en la estructura poblacional entre los desovadores demersales y pelágicos de la platija europea (Florin y Höglund 2008).
Los estudios genéticos en S. senegalensis se han desarrollado usando alozimas y ADN mitocondrial (ADNmt) (Díaz–Ferguson et al. 2002, 2007; Cabral et al. 2003; Díaz–Ferguson 2004). Los resultados de estudios de polimorfismo de la longitud de fragmentos de restricción (RFLP por sus siglas en inglés) del ADNmt han mostrado una baja diversidad genética y alta diferenciación entre poblaciones del suroeste de la Península Ibérica y el golfo de Cádiz, especialmente en la costa portuguesa; los resultados sugieren que esta especie muestra un comportamiento "hogareño" (homing behavior) (Díaz–Ferguson et al. 2007) y que la estructura genética está ausente y la conectividad es alta dentro del área del golfo de Cádiz.
A pesar de que secuencias microsatelitales han sido desarrolladas y probadas en individuos cultivados y salvajes, a la fecha no se han realizado estudios genéticos poblacionales con base en estos marcadores en poblaciones naturales de S. senegalensis (Funes et al. 2004, Castro et al. 2006, Porta et al. 2006). Este estudio es el primero en aplicar estos marcadores nucleares muy variables a poblaciones naturales de esta especie, a microescala espacial, en una de las principales zonas pesqueras de esta especie, con el objetivo de contribuir a la futura gestión de sus pesquerías, ya que algunas pesquerías existentes no se consideran ambientalmente sostenibles (Baeta et al. 2005).
MATERIALES Y MÉTODOS
Muestras y extracción de ADN
Se recolectaron un total de 265 adultos de S. senegalensis de dos poblaciones portuguesas y seis españolas localizadas en el golfo de Cádiz (fig. 1). Los sitios de muestreo se seleccionaron porque representan las principales áreas de distribución de la especie. El estuario del Tajo es una zona de crianza de S. senegalensis (Vasconcelos et al. 2010). Los ejemplares de este sitio (Lisboa) y Portimão fueron adquiridos directamente de barcos pesqueros comerciales después de confirmar el origen de los peces. Se establecieron seis sitios de muestreo en la costa de España, dada sus características conocidas. Esta zona es parte del golfo de Cádiz y es influenciada por la corriente superficial del Atlántico; el efecto de tal corriente aumenta debido a su proximidad al estrecho de Gibraltar, el cual promueve un flujo de agua importante entre el océano Atlántico y el mar Mediterráneo. Además, la topografía de la costa puede representar una barrera física entre las poblaciones. La población 3 fue muestreada por E Díaz–Ferguson durante un crucero científico, mientras que las poblaciones 4, 5, 6, 7 y 8 fueron muestreadas durante cruceros contratados para este proyecto.
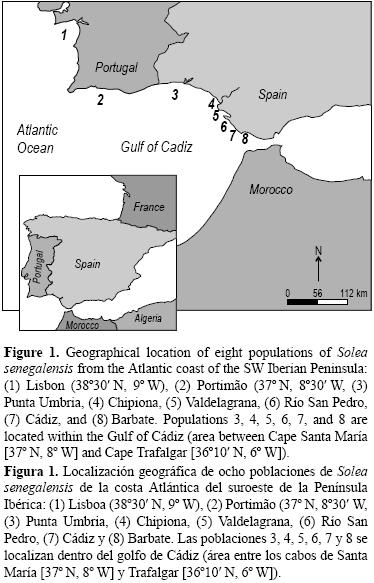
El ADN se obtuvo de una muestra pequeña de la aleta caudal, usando el procedimiento con resina Chelex 100 (Walsh et al. 1991).
Amplificación de loci microsatélites y genotipado
Las secuencias de los cebadores y las temperaturas de fusión de los nueve loci microsatélites se tomaron de la base de datos GenBank (Coughlan et al. 1996, Iyengar et al. 2000, Funes et al. 2004, Porta y Alvarez 2004) (tabla 1). Se realizaron reacciones en cadena de la polimerase en una mezcla de 15 µL conteniendo 30 ng de ADN genómico total, 1.5 mM de MgCl2, 0.1 mM de dNTP, 1 µM de cada partidor, 0.5 unidades de AmpliTag Gold DNA Polymerase y 1x de amortiguador de polimerasa (proporcionado por el fabricante). Las amplificaciones se llevaron a cabo según el siguiente programa: predenaturación a 95 °C por 10 min, seguido de 35 ciclos a 94 °C por 45 s, 50 s a la temperatura de fusión específica, y extensión a 72 °C por 50 s; la extensión final se realizó a 72 °C por 10 min. El tamaño de los fragmentos de los loci microsatélites se determinó en un secuenciador automático ABI Prism 3100 (Applied Biosys–tems). El tamaño de los alelos se determinó por comparación con un estándar interno de peso molecular de 500 pares de bases (pb) y el programa GeneScan Analysis Software 3.7. Los genotipos de cada locus se formatearon según programas estadísticos específicos para su posterior análisis.
Variabilidad genética y expectativas de Hardy–Weinberg
Se estimaron la frecuencia de alelos, el número de alelos (Na), y la heterocigosidad observada (Ho) y esperada (He) por locus por población y la heterocigosidad total (Ht) con el programa de cómputo GENETIX 4.01 (Belkhir et al. 2001). Se realizaron pruebas de Kruskal–Wallis (SYSTAT 11.0) para determinar las diferencias en los parámetros de diversidad genética (valores promedio de alelos, He y Ho entre los loci) entre las poblaciones. Para evaluar la conformidad con las expectativas de Hardy–Weinberg (H–W) por población y locus, se realizaron pruebas exactas con el método de la cadena de Markov implementado en el programa GENEPOP 3.1 (Raymond y Rousset 1995). Los parámetros de la cadena de Markov fueron 5000 pasos y un valor de demorización de 1000. Se realizó una prueba global de los loci dentro de las poblaciones con el método de Fisher. Las estimaciones de Fis para examinar la magnitud y el signo de las desviaciones en cada locus fueron obtenidas siguiendo a Weir y Cockerham (1984) con FSTAT 2.9.3 (Goudet 1995). Se realizaron pruebas para la presencia de alelos nulos y errores de puntuación con MICROCHECKER (Van Oosterhout et al. 2004). Se aplicó el ajuste de Bonferroni para comparaciones múltiples (Rice 1989).
Diferenciación génetica entre las poblaciones
Se realizaron pruebas de probabilidad exacta para determinar la homogeneidad entre todos los pares de muestras y en todas las muestras y los loci mediante GENEPOP 3.1 (Raymond y Rousset 1995). Los componentes relativos de la diversidad genética (prueba estadística global F) fueron calculados con FSTAT 2.9.3: Gst (Nei 1987) y Rst (Slatkin 1995) con base en el modelo de alelos infinitos y el modelo de pasos (stepwise), respectivamente. Para determinar la diferenciación genética entre sitios, se calculó Fst por pares, y la significación de los resultados se probó mediante pruebas de permutación con FSTAT 2.9.3. Además, también se examinó el nivel de estructura mediante una estimación del número de población (K) con el programa STRUCTURE 2.2 (Pritchard et al. 2000). El programa se corrió con seis réplicas independientes para K (i.e., poblaciones distintas o grupos de genes con K de uno a ocho). El periodo de descarte fue 10,000 réplicas seguido de 50,000 simulaciones de Monte Carlo en un modelo que supone que no hay mezclas y frecuencias de alelos independientes. El número más probable de grupos se determinó mediante el método propuesto por Evanno et al. (2005).
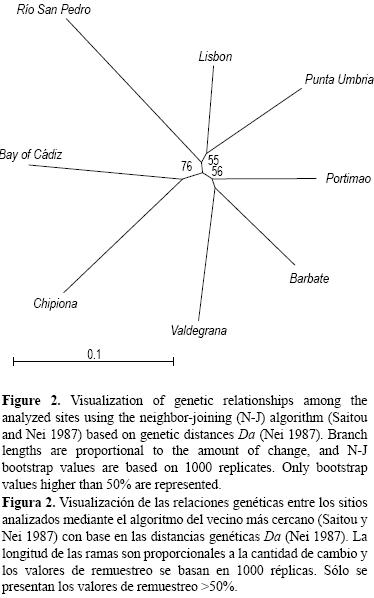
Las relaciones genéticas entre sitios se visualizaron mediante un dendrograma del vecino más próximo (neighbor–joining; Saitou y Nei 1987) generado mediante el programa POPULATIONS 1.2.28, con 1000 réplicas de remuestreo (bootstrapping) (Langella 1999, obtenido de http://www.pge.cnrs–gif.fr/bioinfo/populations). Las réplicas se obtuvieron de la matriz de distancia genética Da (Nei 1987, Takezaki y Nei 1996).
RESULTADOS
Diversidad genética y expectativas de Hardy–Weinberg
De las 72 pruebas realizadas, 19 mostraron desviaciones significativas de las expectativas de H–W y 7 de éstas persistieron después de aplicar la corrección de Bonferroni (no se muestran los datos). La mayoría de las 19 desviaciones se debieron a un déficit de heterocigotos (tabla 2; Fis promedio = 0.076 ± 0.021), asociado principalmente con los loci Sol GA12 (Fis = 0.135 ± 0.022) y Sol MIJ (Fis = 0.119 ± 0.040), y fueron particularmente altas en la población de Río San Pedro (Fis promedio = 0.188 ± 0.072); 66.7% de las pruebas fueron significativas en esta población (tabla 2, apéndice). El análisis con MICROCHECKER mostró la presencia de alelos nulos en diferentes loci, pero principalmente en los que presentaron un déficit de heterocigotos (Sol GA12 y Sol MIJ), e indicó que la muestra de Río San Pedro tenía el mayor número de loci con alelos nulos (Sse 38, Sol 9A, sol MII, Sol MIJ y Sol GA12). Con respecto a los errores de genotipado, el análisis sugirió un error de tartamudeo en el locus Sse 38 de la muestra de Río San Pedro.
Se obervó una alta variabilidad genética, tanto del número de alelos y la heterocigosidad por locus, en los nueve microsatélites analizados después de la caracterización genética de 265 individuos de ocho poblaciones naturales. Se obtuvo un promedio de 14.65 ± 0.62 alelos por locus por población (tablas 2, 3). El número de alelos varió de 9 en Sol 19 a 68 en Sol MJI, la mayoría de los loci mostrando entre 20 y 30 alelos. Se observaron diferencias ligeras y no significativas (P > 0.05) en el número de alelos entre poblaciones; el valor más bajo correspondió a la muestra de Río San Pedro (11.22) y los valores más altos a las poblaciones portuguesas (16.11 y 17.00 para Portimão y Lisboa, respectivamente). Las diferencias entre la heterocigosidad esperada de cada población no fueron significativas y variaron de 0.8 a 0.9 para la mayoría de los loci, con un promedio de 0.860 ± 0.005 por locus por población.
Diferenciación genética entre las poblaciones
Se observó una diferenciación genética reducida pero significativa (P < 0.001) para todos los loci y para sus promedios globales (Rst = 0.009 y Gst = 0.005) en todas las poblaciones (tabla 3); sin embargo, las comparaciones de pares de Fst sólo mostraron valores pequeños pero significativos para las comparaciones incluyendo las poblaciones de Barbate y, particularmente, Río San Pedro (tabla 4). No se encontró evidencia de separación entre las poblaciones de Portugal y el golfo de Cádiz. Esto se reflejó en el árbol de consenso sin raíz, basado en las distancias genéticas Da; el árbol mostró ramas largas para todas las poblaciones, y una robustez baja o moderada para la mayoría de las agrupaciones. Este árbol de consenso sin raíz no mostró ninguna tendencia geográfica específica; sólo dos poblaciones geográficamente cercanas, bahía de Cádiz y Chipiona, constituyeron un grupo razonablemente robusto (valor de remuestreo = 76%) (fig. 2). No obstante, la estimación de K (número de poblaciones) con el programa STRUCTURE 2.2 no encontró ninguna estructura grupal en los datos analizados, y los valores (K = 1) corroboraron la existencia de una sola población panmíctica para la especie, extendiéndose a lo largo de la costa de la Península Ibérica cubierta por el estudio.
DISCUSIÓN
Variabilidad genética y expectativas de Hardy–Weinberg
La diversidad genética proporciona estimaciones precisas del grado de salud y sostenibilidad de las poblaciones marinas (Díaz–Ferguson et al. 2010). Los valores de diversidad genética por locus por población presentados aquí para S. senegalensis son similares a los documentados previamente para otras especies de peces planos como el rodaballo Scophthalmus maximus (Bouza et al. 2002), el lenguado japonés Paralichthys olivaceus (Sekino y Hara 2001), la platija Pleuronectes platessa (Hourau et al. 2002) y el lenguado común Solea solea (Iyengar et al. 2000) usando loci microsatélites. Además, estos valores resultaron homogéneos entre las poblaciones del lenguado senegalés a través de la extensión geográfica analizada. A pesar de su condición estuarina, los valores de diversidad genética registrados para S. senegalensis fueron similares a los documentados para especies marinas de acuerdo con el gradiente de diversidad genética relacionado con el hábitat propuesto por DeWoody y Avise (2000). Estos autores sugieren que la diversidad genética incrementa gradualmente de valores menores en especies de agua dulce, a intermedios en especies estuarinas, a máximos en especies marinas.
El marcador seleccionado obviamente afecta la diversidad genética observada en estas poblaciones. Estudios previos con isozimas y segmentos de ADNmt encontraron una diversidad genética baja para S. senegalensis en poblaciones naturales (Vega et al. 2002, Díaz–Ferguson 2004, Díaz–Ferguson et al. 2007) y cultivadas (Díaz–Ferguson et al. 2002, Vega et al. 2002), así como en otras especies de lenguado como Solea vulgaris (Kotoulas et al. 1995, Cabral et al. 2003). Esto se podría atribuir a la existencia de diversas tasas de mutación entre los marcadores y los polimorfismos intraespecíficos dependiendo del número de pares de bases de nucleótidos examinados (Bouza et al. 2002, Díaz–Ferguson et al. 2007). En las poblaciones estudiadas se detectó una proporción alta de desviaciones significativas del equilibrio de H–W (26.4%, P < 0.05; 9.7% después de aplicar la corrección de Bonferroni). La mayoría de las desviaciones se debieron al déficit de heterocigotos y, a pesar de estar esparcidas a través de diferentes loci y poblaciones (todos los valores promedio de Fis de los loci y las poblaciones fueron positivos), estas desviaciones estuvieron particularmente asociadas con dos loci (SolGA12 y SolMIJ) y una población (Río San Pedro). Se ha observado un déficit de heterocigotos en otros peces planos con comportamiento estuarino, como por ejemplo P. platessa (Hourau et al. 2002), Solea lascaris y S. solea (Kotoulas et al. 1995, Pinheiro et al. 2005). La alta incidencia del déficit de heterocigotos en loci específicos (Sol GA12: Fis promedio = 0.135; SolMIJ: Fis promedio = 0.120) podría indicar la presencia de alelos nulos a frecuencias moderadas, como ha sido confirmado mediante el análisis con MICROCHECKER. Castro et al. (2006) encontraron evidencia de la presencia de alelos nulos en el locus Sol GA12 utilizando datos de familia, considerado el mejor enfoque para abordar esta cuestión. Los mismos autores documentaron dificultades técnicas para Sol MIJ, que presentó tres series alélicas solapadas difiriendo por 1 pb en algunas partes. En este caso, no fue posible comprobar que hubo errores de genotipado en los heterocigotos para alelos consecutivos difiriendo por 1 pb ya que el análisis con MICROCHECKER mostró sólo bandas de tartamudeo en el locus Sse 38 de una muestra (Río San Pedro). A nivel pobla–cional, los déficits altos y heterogéneos entre los loci de las muestras de Río San Pedro (Fis = 0.188) podrían ser explicados por la presencia de alelos nulos en varios loci.
Diferenciación genética entre las poblaciones
La diferenciación genética en S. senegalensis fue baja pero significativa, con base en estimadores relativos globales de divergencia genética (Gst = 0.005; Rst = 0.009). Entre las poblaciones se observó una diferenciación ligera pero significativa en las comparaciones por pares, especialmente para la población de Río San Pedro, la cual mostró valores significativos de Fst con respecto a la mayoría de las poblaciones, así como para la población de Barbate, la cual se diferenció significativamente de las otras dos poblaciones. En el presente estudio también se demostró una heterogeneidad genética altamente significativa en las frecuencias alélicas entre las muestras mediante pruebas de exactitud (P < 0.001), coincidiendo con la presencia de un gran número de alelos privados, especialmente en la población de Chipiona.
Se ha recomendado el uso de medidas absolutas de divergencia para marcadores altamente variables, como los microsatélites, ya que las medidas relativas de diferenciación entre poblaciones dependen del grado de diversidad intrapo–blacional (Hedrick 1999). Se observaron cifras moderadas pero sustanciales de la distancia genética entre todos los pares poblacionales (Da promedio = 0.1897 ± 0.0059) para el lenguado senegalés, mucho más altas que las documentadas para otros peces planos con una distribución geográfica similar (Bouza et al. 2002). De hecho, el dendrograma obtenido a partir de las distancias genéticas mostró ramas largas sin ninguna tendencia geográfica. Aparentemente las poblaciones de esta especie muestran algún grado de aislamiento, pero también un alto grado de intercambio de migrantes con una o más poblaciones de forma aleatoria. Es posible que el efecto de la corriente superficial del Atlántico promueva suficiente flujo de genes para homogeneizar las frecuencias alélicas entre la mayoría de las poblaciones analizadas, pero sin ninguna tendencia geográfica. La discrepancia observada entre los valores relativos y absolutos puede ser explicada por los altos valores de heterocigosidad intrapoblacional registrados en este estudio (cerca de 0.90), lo cual limitaría la fiabilidad de estas cifras para mostrar una diferenciación relativa significativa, como ha sido sugerido para loci muy variables (Hedrick 1999).
Estudios poblacionales anteriores del lenguado senegalés han sugerido la existencia de un modelo panmíctico para los sistemas estuarinos de Portugal, con una ligera diferenciación entre las poblaciones de Lisboa y el golfo de Cádiz; un estudio previo también muestra la ausencia de una estructura genética aparente entre las poblaciones de la región del golfo de Cádiz (Díaz–Ferguson et al. 2007). La baja diferenciación genética observada aquí concuerda más con la estructura poblacional del lenguado común, S. solea, en donde las poblaciones del Atlántico desde Dinamarca hasta Portugal se consideran representativas de una sola unidad panmíctica (Rolland et al. 2007). No obstante, un análisis comparativo de marcadores AFLP y SSR en las poblaciones mediterráneas de S. vulgaris arrojó datos de diferenciación contradictorios, mostrando una homogeneidad genética entre las muestras de los mares Adriático y Tirreno con marcadores microsatélites, pero valores de Fst altamente significativos con marcadores AFLP (Garoia et al. 2007). En otros peces planos costeros como el lenguado japonés P. olivaceus, se han encontrado valores similares de Fst para especies que habitan zonas costeras en el mismo rango del golfo de Cádiz (Sekino y Hara 2001). A pesar de los valores bajos de Fst, estos autores sugieren que las poblaciones de esta especie no representan una sola población panmíctica, ya que las comparaciones por pares mostraron diferencias significativas para por lo menos un locus y una población. Así, en nuestro estudio a microescala (con marcadores de escala más fina), el análisis integrado de datos globales y de pares con estimadores absolutos y relativos de divergencia genética indicó la existencia de una diferenciación genética ligera pero significativa. Este estudio no apoya previos datos de ADNmt que sugieren una separación genética entre las poblaciones de Portugal y el golfo de Cádiz del lenguado senegalés (Díaz–Ferguson et al. 2007). Al contrario, los resultados apoyan la existencia de panmixia para la Península Ibérica. La existencia de diferentes tasas de migración por sexo, menor para hembras que machos, podría explicar parcialmente la discrepancia entre los marcadores mitocondriales y nucleares.
En conclusión, los resultados obtenidos en este estudio mostraron una baja diferenciación genética global y ausencia de una estructura genética para el lenguado senegalés. Estos resultados son similares a los de estudios previos realizados con otros peces planos distribuidos en la costa de la Península Ibérica. La existencia de una sola unidad panmíctica demostró que estos resultados deberían ser tomados en cuenta para futuros planes de manejo y conservación para la especie en toda su distribución geográfica.
AGRADECIMIENTOS
El primer autor recibió una beca doctoral de la Agencia Española de Cooperación Internacional (AECI) durante los primeros tres años de la investigación. Los autores agradecen el apoyo técnico y logístico proporcionado por el proyecto OPAM (INTERREG IIIA) durante el último año de estudio, y a FM Utter y C Figiel Jr. sus comentarios y sugerencias para la versión final del artículo.
REFERENCIAS
APROMAR. 2010. Informe APROMAR. Asociación Empresarial de Productores de Cultivos Marinos, http://www.apromar.es. [ Links ]
Baeta F, Pinheiro P, Corte–Real M, Costa J, de Almeida P, Cabral H, Costa M. 2005. Are the fisheries in Tagus sustainable? Fish. Res. 76: 243–251. [ Links ]
Borsa P, Blanquer A, Barrebi P. 1997. Genetic structure of the flounder Platichthys flesus and P. stellatus at different geographic scales. Mar. Biol. 129: 233–246. [ Links ]
Bouza C, Presa P, Castro J, Sánchez M, Martínez P. 2002. Allozyme and microsatellite diversity in natural and domestic populations of turbot (Scophthalmus maximus) in comparison with other pleuronectiforms. Can. J. Fish. Aquat. Sci. 59: 1460–1473. [ Links ]
Belkhir K, Borsa P, Chikhi L, Raufaste N, Bonhomme F. 2001. Genetix: Logiciel sous Windows™ pour la génétique des populations. Laboratoire Génome, Populations, Interactions. CNRS (France) UPR 9060. Université de Montpellier II, Montpellier, France. [ Links ]
Cabral H, Castro F, Linhares D, Alexandrinho P. 2003. Genetic differentiation of Solea solea (Linnaeus 1758) and Solea senegalensis Kaup 1858 (Pisces: Pleuronectiformes) from several estuarine systems of the Portuguese coast. Sci. Mar. 67: 43–52. [ Links ]
Cabral NH, Vasconcelos RP, Vinagre C, Franc A, Fonseca S, Maia V, Reis–Santos A, Lopes P, Ruano M, Campos M, Freitas J, Santos V, Costa MJ. 2007. Relative importance of estuarine flatfish nurseries along the Portuguese coast. J. Sea Res. 57: 209–217. [ Links ]
Castro J, Pino A, Hermida M, Bouza C, Riaza A, Ferreiro I, Sanchez L, Martinez P. 2006. A microsatellite marker tool for parentage analysis in Senegal sole (Solea senegalensis): Genotyping errors, null alleles and conformance to theoretical assumptions. Aquaculture 261: 1194–1203. [ Links ]
Coughlan J, McCarthy E, McGregor D, O'Donoghue P, Galvin P, Fitzgerald R, Daemen E, Imsland A, Stefansson S, Cross T. 1996. Four polymorphic microsatellites in turbot Scophthalmus maximus. Anim. Genet. 27: 441. [ Links ]
DeWoody JA, Avise JC. 2000. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. J. Fish Biol. 56 : 461–473. [ Links ]
Díaz–Ferguson E. 2004. Development of genetic markers and genetic structure of natural populations of Solea senegalensis (Soleidae, Pleuronectiformes) from the Gulf of Cádiz (in Spanish). PhD thesis, University of Cádiz, Spain. 118 pp. [ Links ]
Díaz–Ferguson E, Vega L, Cross I, Rebordinos L. 2002. Genetic characterization of a stock of breeders of Solea senegalensis (Pleuronectiformes: Soleidae) using haemoglobin and transferrin. 7th World Congress on Genetics Applied to Livestock Production, Montpellier, France. [ Links ]
Díaz–Ferguson E, Cross I, Barrios MM, Rebordinos L. 2007. Genetic relationships among populations of the Senegalese sole Solea senegalensis in the southwestern Iberian Peninsula detected by mitochondrial DNA–restriction fragment length polymorphisms. Trans. Am. Fish. Soc. 136: 484–491. [ Links ]
Díaz–Ferguson E, Haney RA, Wares JP, Silliman BR. 2010. Population genetics of a trochid gastropod broadens picture of Caribbean Sea connectivity. PLos ONE 5: e12675. [ Links ]
Dinis MT, Ribeiro L, Soares F, Sarasquete C. 1999. A review on the cultivation potential of Solea senegalensis in Spain and in Portugal. Aquaculture 176: 27–38. [ Links ]
Evanno G, Regnaut S, Goudet J. 2005. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study. Mol. Ecol. Notes 14: 2611–2620. [ Links ]
Exadactylos A, Geffen A, Panagiotaki P, Thorpe J. 2003. Population structure of Dover sole Solea solea: RAPD and allozyme data indicate divergence in European stocks. Mar. Ecol. Prog. Ser. 246: 253–264. [ Links ]
Feral JP. 2002. How useful are the genetic markers in attempts to understand and manage marine biodiversity? J. Exp. Mar. Biol. Ecol. 268: 121–145. [ Links ]
Florin AB, Höglund J. 2007. Absence of population structure of turbot (Psetta maxima ) in the Baltic Sea. Mol. Ecol. 16: 115–126. [ Links ]
Florin AB, Höglund J. 2008. Population structure of flounder (Platichthys flesus) in the Baltic Sea: Differences among demersal and pelagic spawners. Heredity 101: 27–38. [ Links ]
Funes V, Zuasti E, Catanese G, Infante C, Manchado M. 2004. Isolation and characterization of ten microsatellite loci for Senegal sole (Solea senegalensis Kaup). Mol. Ecol. Notes 4: 339–341. [ Links ]
Garoia F, Guarniero I, Grifoni D, Marzola S, Tinti F. 2007. Comparative analysis of AFLP and SSRs efficiency in resolving population genetic structure of Mediterranean Solea vulgaris. Mol. Ecol. 16: 1377–1387. [ Links ]
Goudet J. 1995. Fstat (Version 1.2): A computer program to calculate F–statistics. J. Hered. 86: 485–486. [ Links ]
Guarniero I, Franzellitti S, Ungaro N, Tommasini S, Piccinetti C, Tinti F. 2002. Control region haplotype variation in the central Mediterranean common sole indicates geographical isolation and population structuring in Italian stocks. J. Fish Biol. 60: 1459–1474. [ Links ]
Hedrick PW. 1999. Genetics of Populations. Jones & Bartlett Publishers, London. 658 pp. [ Links ]
Hourau G, Rijnsdorp D, Van Der Veer HW, Stam T, Olsen JL. 2002. Population structure of plaice (Pleuronectes platessa L.) in northern Europe: Microsatellites revealed large–scale spatial and temporal homogeneity. Mol. Ecol. 11: 1165–1176. [ Links ]
Imsland AK, Foss A, Conceiçao LEC, Dinis MT, Delbare D, Schram E, Kamstra A, Rema P, White P. 2003. A review of the culture potential of Solea solea and S. senegalensis. Rev. Fish Biol. Fish. 13: 379–407. [ Links ]
Iyengar A, Piyapattanakorn S, Stone D. 2000. Identification of microsatellite repeats in turbot (Scophthalmus maximus) and Dover sole (Solea solea) using a RAPD–based technique: Characterization of microsatellite markers in Dover sole. Mar. Biotechnol. 2: 49–56. [ Links ]
Kotoulas G, Bonhomme F, Borsa P. 1995. Genetic structures of the common sole Solea vulgaris at different geographic scales. Mar. Biol. 122: 361–375. [ Links ]
Langella O. 1999. Populations 1.2.28. Centre National de la Recherche Scientifique (France) UPR9034. Available at http://www.cnrs–gif.fr/pge/bioinfo/lea/index. [ Links ]
Mustafa S (ed.). 1999. Genetics in Sustainable Fisheries Management. Wiley–Blackwell, London, 223 pp. [ Links ]
Nei M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York. 512 pp. [ Links ]
Nielsen E, Nielsen P, Meldrup D, Hansen M. 2004. Genetic population structure of turbot (Scophthalmus maximus L.) supports the presence of multiple hybrid zones for marine fishes in the transition zone between the Baltic Sea and the North Sea. Mol. Ecol. 13: 585–595. [ Links ]
Pinheiro A, Teixeira CM, Rego AL, Marques JF, Cabral HN. 2005. Genetic and morphological variation of Solea lascaris (Risso 1810) along the Portuguese coast. Fish. Res. 73: 67–78. [ Links ]
Porta J, Alvarez MC. 2004. Development and characterization of microsatellites from Senegal sole (Solea senegalensis). Mol. Ecol. Notes 4: 277–279. [ Links ]
Porta J, Porta JM, Martinez–Rodriguez G, Alvarez MC. 2006. Development of a microsatellite multiplex PCR for Senegalese sole (Solea senegalensis) and its application to broodstck management. Aquaculture 256: 159–166. [ Links ]
Pritchard JK, Stephens M, Donnelly P. 2000. Inference of population structure using multi–locus genotype data. Genetics 155: 945–959. [ Links ]
Raymond M, Rousset F. 1995. GENEPOP (Version 3.1): Population genetics software for exact test and ecumenecism. J. Hered. 86: 248–249. [ Links ]
Rice WR. 1989. Analysing tables of statistical tests. Evolution 43: 223–225. [ Links ]
Rolland JL, Bonhomme F, Lagardere F, Hassan M, Guinand B. 2007. Population structure of the common sole (Solea solea) in the northeastern Atlantic and the Mediterranean Sea: Revisiting the divide with EPIC markers. Mar. Biol. 151: 327–341. [ Links ]
Saitou N, Nei N. 1987. The neighbor–joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406–425. [ Links ]
Sekino M, Hara M. 2001. Application of microsatellite markers to population genetics studies of Japanese flounder Paralichthys olivaceous. Mar. Biotech. 3: 572–589. [ Links ]
Slatkin M. 1995. A measure of population subdivision based on microsatellite allele frequencies. Genetics 89: 583–589. [ Links ]
Takezaki N, Nei M. 1996. Genetic distances and reconstruction of phylogenetic trees from microsatellite DNA. Genetics 144: 389–399. [ Links ]
Van Oosterhout C, Hutchinson WF, Wills DPM, Shipley P. 2004. MICROCHECKER: Software for identifying and correcting genotyping errors in microsatellite data. Mol. Ecol. Notes 4: 535–538. [ Links ]
Vega L, Díaz–Ferguson E, Cross I, Rebordinos L. 2002. Caracterizaciones citogenética e isoenzimática del lenguado Solea senegalensis Kaup 1858. Bol. Inst. Esp. Oceanogr.18: 245–250. [ Links ]
Vasconcelos RP, Reis–Santos P, Maia A, Fonseca A, Fraça S, Wouters N, Costa MJ, Cabral HN. 2010. Nursery use patterns of commercially important marine fish species in estuarine systems along the Portuguese coast. Estuar. Coast. Shelf Sci. 86: 613–624. [ Links ]
Vinagre C, Fonseca A, Maia A, Amara R, Cabral H. 2008. Habitat specific growth rates and condition indices for the sympatric soles Solea solea (Linnaeus 1758) and Solea sonegalensis (Kaup 1858) in the Tagus Estuary, Portugal, based on otolith daily increments and RNA–DNA ratio. J. Appl. Ichthyol. 24: 163–169. [ Links ]
Walsh PS, Metzger DA, Higuchi R. 1991. Chelex 100 as a medium for simple extraction of DNA for PCR–based typing from forensic material. Biotechniques 10: 506–513. Wegner GW, Damm U, Purps M. 2003. Physical influences on the stock dynamics of plaice and sole in the North Sea. Sci. Mar. 67: 219–234. [ Links ]
Weir BS, Cockerham CC. 1984. Estimating F–Statistics for the analysis of population structure. Evolution 38: 1358–1370. [ Links ]
*Descargar versión bilingüe (Inglés–Español) en formato PDF.
Traducido al español por Christine Harris.














