Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Ciencias marinas
versión impresa ISSN 0185-3880
Cienc. mar vol.37 no.3 Ensenada sep. 2011
Caracterización de bacterias cultivables presentes en sistemas de cultivo de larvas de ostión chileno Argopecten purpuratus*
Characterization of culturable bacteria in larval cultures of the Chilean scallop Argopecten purpuratus**
FA Godoy1, 3*, M Espinoza1, G Wittwer 1, I Uriarte1, 2, C Aranda3
1 CIEN Austral, Universidad Austral de Chile, Casilla 1327, Avda. Los Pinos s/n, Pelluco, Puerto Montt, Chile.
2 Instituto de Acuicultura, Universidad Austral de Chile, Casilla 1327, Avda. Los Pinos s/n, Pelluco, Puerto Montt, Chile.
3 Centro i–mar, Universidad de los Lagos, Camino a Chinquihue Km 6, Casilla 557, Puerto Montt, Chile.
* Corresponding author.
E–mail: felix.godoy@ulagos.cl
Received October 2010
Accepted April 2011
RESUMEN
Las etapas tempranas del cultivo comercial de Argopecten purpuratus son afectadas por la mortalidad ocasionada por la presencia de patógenos bacterianos. El propósito de este estudio fue caracterizar las bacterias cultivables presentes en los sistemas de cultivo de larvas de A. purpuratus en presencia y ausencia de un antibiótico (Tetraciclina). Se efectuaron recuentos bacterianos en medio agar marino a partir de muestras de agua, de microalgas y de larvas de las unidades de cultivo. Estos recuentos se realizaron cada tres días durante los 20 días de incubación. Las cepas aisladas se identificaron mediante la secuenciación del gen ADNr 16S. Además, se realizaron recuentos de bacterias totales del agua de cultivo mediante microscopía de epifluorescencia. Los recuentos de bacterias cultivables de las muestras de agua fluctuaron entre 8 × 103 y 3 × 104 UFC mL–1. Estos recuentos representaron entre 3% y 18% de los recuentos de bacterias totales. Los recuentos de las muestras de larvas fluctuaron entre 3 × 107 y 3 × 108 UFC g–1 y fueron en promedio mayores en ausencia del antibiótico (P < 0.05). Los recuentos bacterianos de las muestras de microalgas fluctuaron entre 1 × 104 y 9 × 104 UFC mL–1. De las 46 cepas aisladas e identificadas, 25 (54%) pertenecieron a la clase Gammaproteobacteria, 11 (24%) a Bacilli, 5 (11%) a Actinobacteria, 4 (9%) a Alfaproteobacteria y 1 (2%) a Flavobacteria. Los géneros más abundantes en las muestras de agua fueron Alteromonas, Brevibacterium, Bacillus, Microbacterium y Marinobacter, mientras que en las de larvas fueron Pseudoalteromonas, Bacillus, Marinobacterium, Alteromonas y Vibrio, de un total de 15 diferentes géneros bacterianos identificados. La supervivencia de las larvas disminuyó durante el tiempo de incubación, sin existir diferencias por la presencia del antibiótico. Los resultados indican que existe una diversidad alta de géneros bacterianos presentes en las larvas, que en su mayoría son diferentes a los encontrados en el agua y las microalgas suministradas como alimento.
Palabras clave: bacterias cultivables, Argopecten purpuratus, cultivos larvales, ostión chileno.
ABSTRACT
The early stages of culture of the Chilean scallop Argopecten purpuratus are affected by the mortality attributed to the presence of pathogenic bacteria. The purpose of this study was to characterize the culturable bacteria present in larval cultures of A. purpuratus in the presence and absence of antibiotic (Tetracycline). Bacterial counts on marine agar from water, algal, and larval samples from the culture units were carried out. These counts were performed every three days during 20 days of incubation. The bacterial strains isolated from count plates were identified by 16S rDNA gene sequence analysis. Additionally, the total bacterial counts from culture water were determined by epifluorescence microscopy. Culturable bacterial counts of water samples ranged from 8 × 103 to 3 × 104 CFU mL–1. These counts represent from 3% to 18% of the total bacterial counts. The culturable bacterial counts in larvae were between 3 × 107 and 3 × 108 CFU g–1 and were on average higher in the absence of antibiotic (P < 0.05). Microalgal bacterial counts ranged from 1 × 104 to 9 × 104 CFU mL–1. Of the 46 strains isolated and identified, 25 (54%) belonged to the Gammaproteobacteria, 11 (24%) to the Bacilli, 5 (11%) to the Actinobacteria, 4 (9%) to the Alphaproteobacteria, and 1 (2%) to the Flavobacteria. The most dominant genera in the water samples were Alteromonas, Brevibacterium, Bacillus, Microbacterium, and Marinobacter, while the most dominant genera in larvae were Pseudoalteromonas, Bacillus, Marinobacterium, Alteromonas, and Vibrio, from a total of 15 different bacterial genera identified. The survival of larvae decreased drastically during the 20 days of culture, showing no differences to antimicrobial treatments. The results indicate that there is a high diversity of culturable bacteria present in the larvae, and most are different from those found in the water and microalgae supplied as food.
Key words: culturable bacteria, Argopecten purpuratus, hatchery, Chilean scallop.
INTRODUCCIÓN
El ostión chileno Argopecten purpuratus (Lamark 1819) es uno de los principales recursos marinos de importancia comercial en Chile. El desarrollo del sistema de cultivo de esta especie es importante porque permite proveer el 30% de semillas en condiciones controladas cuando la recolección de semillas en el medio natural no es suficiente para sustentar la actividad comercial del recurso (Farías et al. 1998). No obstante, las etapas tempranas de desarrollo larval se ven afectadas, frecuentemente, por la mortalidad alta provocada por patógenos oportunistas (Riquelme et al. 1996). Se han asociado algunas bacterias patógenas, principalmente de los géneros Vibrio, Aeromonas y, recientemente, Halomonas, a eventos de mortalidad (Riquelme et al. 1995a, 1996; Rojas et al. 2009). Los tratamientos con antibióticos han sido usados para controlar la presencia de estos patógenos, aun cuando esto implica el riesgo de surgimiento de cepas bacterianas resistentes a los antibióticos. El cloranfenicol es el antibiótico que se utiliza con más frecuencia en sistemas de cultivo de organismos marinos, pero la investigación para nuevas alternativas (e.g., oxitetraciclina y ácido oxolínico) aún continúa (Torkildsen et al. 2002).
En los organismos marinos se han descrito dos tipos de floras bacterianas: la flora transitoria, que está presente de forma temporal y por un corto tiempo en el organismo, y la flora normal o residente, la cual está presente de forma permanente y estable en el organismo huésped. Según varios autores, la flora bacteriana en los bivalvos cambia con la ingesta, y el tránsito es muy rápido para constituir una flora residente real (Moriarty 1990, Jorquera et al. 2001). Sin embargo, las bacterias en bivalvos pueden colonizar el integumento, el intestino y la hemolinfa (Olafsen et al. 1993). Algunos estudios han demostrado la incorporación selectiva de bacterias desde el agua mediante la ingesta de microalgas cultivadas, lo que puede afectar el establecimiento y la dominancia de especies en la microbiota larval (Riquelme et al. 2000). También se ha descrito la presencia de bacterias de los géneros Chromobacterium, Flavobacterium, Moraxella y Vibrio en las gónadas de A. purpuratus silvestre y cultivada, y se ha determinado la transferencia de éstas desde los progenitores a las primeras etapas larvales (Riquelme et al. 1994, 1995b). La flora bacteriana de la larva también puede estar influenciada por las bacterias que se consumen con las microalgas suministradas como alimento y por el agua que ingresa a los sistemas de cultivo (Skjermo y Vadstein 1999, Sandaa et al. 2003).
En este trabajo se caracterizaron las comunidades de bacterias cultivables en larvas, agua de cultivo y microalgas, considerando que éstas son las principales fuentes que contribuyen a la conformación de la flora bacteriana en larvas de A. purpuratus durante el cultivo.
MATERIALES Y MÉTODOS
Condiciones de cultivo
Todas las larvas de A. purpuratus se obtuvieron del Hatchery de la Universidad Austral de Chile, sede Puerto Montt. Las larvas se cultivaron por 20 días (de larva D a larva con ojo) en estanques de 15 L a una temperatura promedio de 19 °C (±1) y salinidad promedio de 30. La densidad larval inicial fue de 1 larva mL–1. El recambio de agua de los cultivos larvarios se realizó de forma manual cada 4 días. El agua utilizada fue tratada con luz UV y filtrada. Los cultivos se alimentaron diariamente con una monodieta de la microalga Isochrysis galbana (T–ISO). El agua en los estanques adicionados con antibiótico fue tratada con 1 mL de Tetraciclina (1 mg mL–1) cada 36 h.
Toma de muestras de agua, larvas y microalgas
Las muestras de agua, larvas y microalgas se tomaron a los 0, 3, 8, 12, 16 y 20 días de incubación de cuatro estanques, dos de los cuales contenían agua tratada con el antibiótico. Las larvas se recolectaron mediante la filtración del agua de salida con un tamiz de nilón de 62 µm. Alrededor de 500 larvas fueron recolectadas y lavadas tres veces con agua de mar estéril, colocadas en frascos estériles de 100 mL y transportadas al laboratorio a 4 °C. Las muestras de agua para el análisis microscópico de epifluorescencia se fijaron inmediatamente en formaldehído (3% concentración final). Las muestras de microalgas suministradas como alimento se tomaron antes de que ingresaran a los sistemas de cultivo y se transportaron inmediatamente a 4 °C para su posterior análisis.
Recuento y aislamiento de cepas bacterianas
Para los recuentos bacterianos, se prepararon diluciones seriadas decimales con agua de mar estéril y se sembraron en medio agar marino 2216 (Difco). Los recuentos se realizaron después de 72 h de incubación a 20 °C. Las muestras de larvas se colocaron en tubos eppendorf y se centrifugaron a 4000 g por 7 min. Una vez eliminado el sobrenadante, las larvas fueron pesadas y resuspendidas en 1 mL de agua de mar estéril para luego ser maceradas mediante un sonicador ultrasónico con vástago. Se registraron el peso seco y los porcentajes de humedad de las muestras de larvas. A partir de este macerado, se realizaron diluciones seriadas en placas. Después de la incubación, se contaron aquellas placas que presentaban entre 30 y 300 unidades formadoras de colonias (UFC).
Recuentos de bacterias totales
Los recuentos de bacterias totales se determinaron por microscopía de epifluorescencia, según el método descrito por Fry (1990). Brevemente, las muestras se fijaron y tiñeron con 4'6–diamino–2 fenilindol (DAPI, Merck) (10 µg mL–1) por 15 min. Posteriormente, se pasaron por un filtro de policarbonato negro de 0.22 (Millipore) y se observaron bajo un microscopio Olympus BX61.
Aislamiento e identificación de cepas bacterianas
Las cepas cuyos morfotipos eran más abundantes en las muestras analizadas (>10% de representación) fueron aisladas de las placas de recuento. Estas cepas se replicaron tres veces mediante siembra para verificar su pureza. El porcentaje de representación morfológica de la colonia en las placas de recuento se consideró como un valor de abundancia relativa de la cepa posteriormente identificada.
El procedimiento de extracción de ADN se realizó utilizando el paquete comercial Wizard Genomics DNA Purification Kit (Promega), según las recomendaciones del fabricante. Las concentraciones de ADN se examinaron con un espectrofotómetro Eppendorf BioPhotometer plus y en gel de agarosa al 0.8%. El ADN extraído se amplificó mediante la reacción en cadena de la polimerasa (PCR, por sus siglas en inglés) con los partidores bacterianos universales 27F (5 '–AGAGTTTGATCCTGGCTCAG–3') y 1492R (5'– GGTTACCTTGTTACGACTT–3') del gen ADNr 16S descritos por Lane (1991). Los productos de amplificación del ADNr 16S de un tamaño aproximado de 1500 pb se purificaron y se enviaron a secuenciar a Macrogen Inc. (http://www.macrogen.com) utilizando el partidor universal 907r. La relación filogenética de los productos secuenciados se determinó por alineación de secuencias individuales del gen ADNr 16S disponibles en el banco de bases GenBank (http://www.ncbi.nlm.nih.gov/GenBank/index.html) y el Ribosomal Database Project II (RDP– II, http://rdp.cme.msu.edu/index.jsp). Las secuencias parciales del gen ADNr 16S pertenecientes a cada cepa se depositaron en la base de datos GenBank con números de acceso de HQ677191 a HQ677235.
Análisis estadístico
El análisis de varianza (ANOVA) se realizó con la paquetería Statistica 7 (Statsoft, Tulsa, EUA), seguido de una prueba a posteriori de diferencia significativa honesta (HSD) de Tukey.
RESULTADOS
Recuentos bacterianos
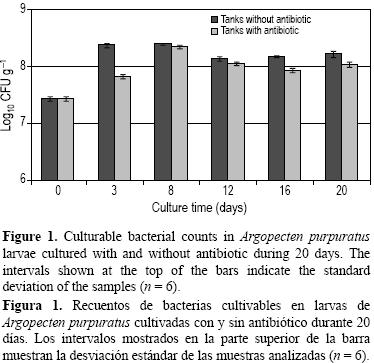
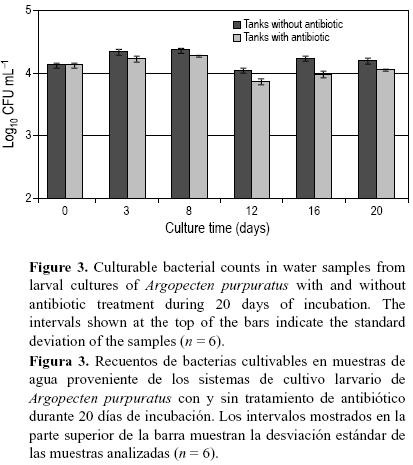
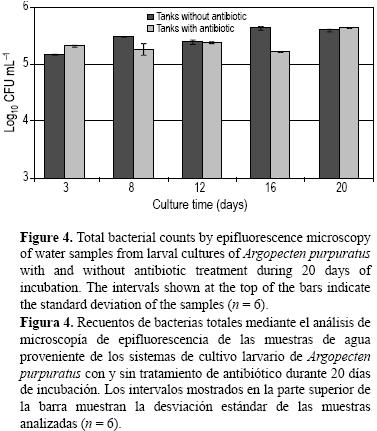
Los muestreos se realizaron a diferentes intervalos de tiempo y de los diferentes compartimentos que forman parte del sistema de cultivo: agua, microalgas suministradas como alimento y larvas. Los recuentos de bacterias en larvas (fig. 1) no variaron en más de un logaritmo en los diferentes días de incubación dado que los valores fluctuaron entre 7.5 y 8.4 log10 UFC g–1. Después de los primeros tres días de incubación, se observa un incremento significativo (P < 0.05) de la población bacteriana que tiende a mantenerse después del día 8. En general, y tomando el promedio de los recuentos sin considerar el día 0, los recuentos bacterianos de las muestras de larvas en los estanques sin antibiótico (1.9 × 108 UFC g–1) fueron mayores que en los estanques con antibiótico (1.1 × 108 UFC g–1). En promedio, hubo 40% menos bacterias cultivables en las larvas tratadas con Tetraciclina. Se observaron diferencias significativas (P < 0.05) al analizar los efectos del antibiótico por día: los recuentos fueron mayores en los estanques sin antibiótico (días 3, 12, 16 y 20), excepto el día 8 en que no existieron diferencias significativas. Los recuentos de bacterias cultivables a partir de las muestras de microalgas (fig. 2) se mantuvieron entre 103 y 104 UFC g–1. Los recuentos bacterianos a partir de las muestras del agua de cultivo (fig. 3) no variaron en más de un logaritmo durante los 20 días de incubación. Al igual que los recuentos bacterianos de larvas, los recuentos bacterianos de las muestras de agua sin antibiótico fueron significativamente mayores que los de las muestras de agua con antibiótico (P < 0.05), excepto al inicio del experimento. Además, estos recuentos fueron, en promedio, el 8% de los recuentos totales de agua medidos por microscopía de epifluorescencia (fig. 4). Los recuentos bacterianos totales de las muestras de agua no mostraron un patrón de comportamiento similar en presencia del antibiótico como lo encontrado en los recuentos de bacterias cultivables de las larvas y del agua. Por ejemplo, en el día 3 los recuentos totales de bacterias en el agua con antibiótico fueron significativamente (P < 0.05) mayores que en ausencia de éste, mientras que en el día 16 los recuentos totales fueron significativamente mayores en ausencia de antibiótico (P < 0.05), y en los días 12 y 20 no se observaron diferencias significativas (P > 0.05) por el efecto del antibiótico.

Aislamiento e identificación de cepas
En total, se aislaron e identificaron 46 cepas bacterianas. Cuatro de estas cepas fueron aisladas de las microalgas utilizadas como alimento, 10 del agua de cultivo y 32 de las muestras de larvas (tabla 1). Del total de cepas bacterianas, 25 (54%) correspondieron a la clase Gammaproteobacteria, de las cuales 7 (28%) y 6 (24%) pertenecieron a los géneros Marinobacter y Alteromonas, respectivamente. Otro grupo predominante consistió en 11 (24%) cepas que pertenecieron a la clase Bacilli (filo Firmicutes), 10 (91%) de las cuales pertenecieron al género Bacillus (tabla 2).
Se observaron diferencias en la composición de géneros bacterianos cultivables en larvas de A. purpuratus cultivadas con y sin tratamiento de antibiótico. En el caso de las larvas cultivadas con el tratamiento antibiótico, se observó una predominancia de cepas pertenecientes a los géneros Pseudoalteromonas, Bacillus, Marinobacterium y Alteromonas. Este último género comenzó a dominar hacia el final del tiempo de incubación según la representatividad observada de la cepa en las placas de recuento. En la microbiota presente en las larvas cultivadas sin tratamiento antibiótico, se observó la predominancia de Alteromonas, Bacillus y Vibrio. El género Bacillus fue abundante al principio de la incubación, pero en la medida que transcurrió el tiempo comenzó a ser menos dominante y fue desplazado por Vibrio, que comenzó a dominar la microbiota al final del tiempo de incubación. En el caso de la flora bacteriana asociada a las microalgas, se observó la presencia y dominancia de otros géneros no encontrados en el agua ni en las larvas. Los géneros bacterianos como Kangiella y Nesterenkonia se observaron dentro de la flora dominante, junto con Bacillus y Alteromonas. En las muestras de agua sin antibiótico, los géneros Alteromonas, Brevibacterium y Marinobacter fueron los más abundantes, mientras que en las muestras de agua con antibiótico los géneros con mayor abundancia relativa fueron Alteromonas, Marinobacter, Bacillus y Microbacterium. El 83% de los géneros identificados en las muestras de agua se encontraron presentes en las larvas; no obstante, la mayoría (65%) de los géneros de bacterias cultivables encontrados en las larvas no se encontraron en el agua.
Por otra parte, los estudios de la supervivencia de larvas de A. purpuratus mostraron que, en el día 12 de cultivo, la supervivencia fue de 7% y 10% en los dos estanques con antibiótico y de 22% y 33% en los dos estanques sin antibiótico. Al final del tiempo de incubación (20 días), la supervivencia de las larvas fue nula en todos los estanques.
DISCUSIÓN
En el presente estudio se analizaron las poblaciones bacterianas de los sistemas de cultivo larvario de A. purpuratus, considerando las bacterias presentes en el agua, las larvas y las microalgas suministradas como alimento. Este análisis se realizó durante 20 días de cultivo larvario con y sin tratamiento antibiótico. Los resultados obtenidos no indican un claro efecto de la Tetraciclina sobre los recuentos bacterianos y la supervivencia larval. Los recuentos de bacterias cultivables indican una carga bacteriana alta, que en su mayoría fue significativamente más alta en ausencia de antibiótico; no obstante, los porcentajes de supervivencia larval fueron menores en presencia del antibiótico (día 12).
La identificación de las bacterias cultivables presentes en las larvas mediante la secuenciación del gen ADNr 16S indica una alta diversidad de géneros bacterianos durante el tiempo de incubación, representada por 15 géneros bacterianos clasificados en cuatro filos. Según los datos obtenidos, al inicio del experimento ya existe una carga bacteriana importante (107 UFC g–1), con una predominancia de los géneros Bacillus y Pseudomonas. A medida que transcurrió el tiempo de cultivo esta dominancia y composición de géneros fue cambiando, sin observarse una tendencia clara en estas variaciones. Algunos géneros bacterianos, como Vibrio, estuvieron presentes sólo en los estanques de cultivo sin antibiótico. La presencia de este género y Alteromonas en los estanques con antibiótico comenzó a ser más abundante después del día 12 de cultivo, cuando la supervivencia de larvas en todos los estanques fue inferior a 35%. El aumento de mortalidad junto con el aumento de estos géneros podría indicar una posible relación patogénica entre estos géneros y las larvas en cultivo. No obstante, sólo Vibrio ha sido reportado como patógeno de larvas de A. purpuratus (Riquelme et al. 1996). Se requieren estudios adicionales de desafío de larvas de A. purpuratus para establecer las relaciones de patogenicidad atribuibles a estas cepas (Riquelme et al. 1996).
Los recuentos de bacterias cultivables heterotróficas en el agua fluctuaron entre 8 × 103 y 3 × 104 UFC mL–1. No se observó un aumento de la carga bacteriana durante el tiempo de incubación que pudiese vincularse a eventos de mortalidad larval. Estos recuentos representaron entre 3% y 18% de los recuentos de bacterias totales, lo que indica un alto porcentaje de bacterias cultivables en este estudio. Estos valores contrastan con los porcentajes de bacterias cultivables (0.04–1.92%) registrados por Jorquera et al. (2004) para diferentes sistemas de cultivo larvario de A. purpuratus. La diversidad de bacterias cultivables en el agua de cultivo, representada por seis géneros, resultó menor comparada con la encontrada en las larvas. Los géneros de Gamaproteobacterias (e.g., Alteromonas y Marinobacter) fueron dominantes durante el tiempo de cultivo larvario.
Estos géneros han sido aislados frecuentemente de organismos marinos (Hansen y Olafsen 1999, Dworkin et al. 2006). Además, en los estudios realizados con sondas especificas para Gamaproteobacterias también se han detectado porcentajes importantes (21% y 28%) de este grupo bacteriano en los recuentos totales de muestars de agua de sistemas de cultivo de A. purpuratus (Jorquera et al. 2004).
Los recuentos de bacterias cultivables en las muestras de microalgas estuvieron en el orden de 104 UFC mL–1 y fueron menor que los recuentos (de 106 a 108 UFC mL–1) registrados por Priuer (1981) para microalgas utilizadas para cultivos de bivalvos. Nicolas et al. (2004) registraron recuentos de bacterias cultivables que fluctuaron entre 1.3 × 105 y 5.3 × 108 UFC mL–1 asociadas a diferentes microalgas. La identificación molecular de cepas aisladas desde las placas de recuento permitió establecer la presencia de cuatro géneros: Bacillus, Alteromonas, Nesterenkonia y Kangiella. El género Bacillus ha sido encontrado típicamente en ambientes terrestres y existen pocas especies marinas descritas, mientras que Alteromonas es un género de bacterias heterotróficas propias del ambiente marino (Dworkin et al. 2006). Nesterenkonia es un género de organismos moderadamente halofílicos, de lagos y ambientes salinos (Li et al. 2004), y hasta la fecha no se ha descrito su asociación con el fitoplancton marino. El género Kengiella ha sido identificado recientemente en el ambiente marino (Yoon et al. 2004, Ahn et al. 2010). En general pocos autores han estudiado las bacterias asociadas con algas utilizadas para la alimentación en sistemas de cultivo (Prieur 1981, Fukami et al. 1997, Nicolas et al. 2004). Se ha empleado la técnica de electroforesis en gel con gradiente desnaturalizante (DGGE) para describir la presencia de Roseobacter y Rhodobacter asociada a cultivos de microalgas en los sistemas de cultivo de Pecten maximus (Sandaa et al. 2003). La presencia de determinadas bacterias asociadas a microalgas podría ser importante ya que pueden afectar positiva o negativamente el crecimiento de los cultivos de microalgas, e incluso la supervivencia de larvas de bivalvos en cultivo. Se necesitan estudios posteriores para determinar los efectos eventuales de estas bacterias sobre los cultivos.
En resumen, una diversidad importante de géneros bacterianos cultivables fue determinada en las larvas, la mayoría de los cuales no están presentes en el agua ni tampoco en las microalgas. Esto sugiere que las larvas pueden desarrollar una flora bacteriana cultivable propia independiente del agua que la rodea, y que ciertos mecanismos de selección pudiesen estar implicados en la conformación de esta flora.
AGRADECIMIENTOS
Este estudio fue financiado por el Fondo Nacional de desarrollo Científico y Tecnológico (FONDECYT No. 1070800, Chile).
REFERENCIAS
Ahn J, Park JW, McConnell AJ, Ahn YB, Haggblom MM. 2010. Kangiella spongicola sp. nov., a halophilic marine bacteriumisolated from the marine sponge Chondrilla nucula. Int. J. Syst. Evol. Microbiol. (in press). [ Links ]
Dworkin M, Falkow S, Rosenberg E, Schleifer KH, Stackebrandt E. 2006. The Prokaryotes. A Handbook on the Biology of Bacteria. 3rd ed. Vol.6. Proteobacteria: Gamma Subclass, 1240 pp. [ Links ]
Farías A, Uriarte I, Castilla JC. 1998. A biochemical study of the larval and postlarval stages of the Chilean scallop Argopecten purpuratus. Aquaculture 166: 37–47. [ Links ]
Fry JC. 1990. Direct methods and biomass estimations. Methods in Microbiol. 22: 41–124. [ Links ]
Fukami K, Nishijima T, Ishida Y. 1997. Stimulative and inhibitory effects of bacteria on the growth of micro–algae. Hydrobiologia 358: 185–191. [ Links ]
Hansen GH, Olafsen JA. 1999. Bacteria interactions in early life stages of marine cold water fish. Microb. Ecol. 38:1–26. [ Links ]
Jorquera MA, Silva FR, Riquelme CE. 2001. Bacteria in the culture of the scallop Argopecten purpuratus (Lamarck 1819). Aquacult. Int. 9: 285–303. [ Links ]
Jorquera M, Leyton L, Riquelme C. 2004. Bacteria of subclass γ–proteobacteria associated with commercial Argopecten purpuratus (Lamark 1819) hatcheries in Chile. Aquaculture. 236: 37–51. [ Links ]
Lane DJ. 1991. 16S/23S rRNA sequencing. In: Stackebrandt E, Goodfellow M (eds.), Nucleic Acid Techniques in Bacterial Systematics. Wiley, New York, pp. 115–175. [ Links ]
Li WJ, Chen HH, Zhang YQ, Schumann P, Stackebrandt E, Xu LH, Jiang CL. 2004. Nesterenkonia halotolerans sp. nov. and Nesterenkonia xinjiangensis sp. nov., actinobacteria from saline soils in the west of China. Int. J. Syst. Evol. Microbiol. 54: 837–841. [ Links ]
Moriarty D. 1990. Interactions of microorganisms and aquatic animals, particularly the nutritional role of the gut flora. In: Lésel R. (ed.), Microbiology in Poecilotherms. Elsevier, Paris. [ Links ]
Nicolas JL, Corre S, Cochard JC. 2004. Bacterial population association with phytoplankton cultured in a bivalve hatchery. Microb. Ecol. 48: 400–413. [ Links ]
Olafsen JA, Mikkelsen HV, Gieasver HM, Hansen GH. 1993. Indigenous bacteria in hemolymph and tissues of marine bivalves at low temperatures. Appl. Environ. Microbiol. 59: 1848–1854. [ Links ]
Prieur D. 1981. Les relations entre mollusques bivalves et bacteries heterotrophes en milieu marin. Thesis, Universite de Bretagne Occidentale, Brest, France, pp. 65–80. [ Links ]
Riquelme C, Chavez P, Morales Y, Hayashida G. 1994. Evidence for parental bacterial transfer to larvae in Argopecten purpuratus (Lamarck 1819). Biol. Res. 27: 129–134. [ Links ]
Riquelme C, Hayashida G, Toranzo A, Vilches J, Chavez P. 1995a. Pathogenicity studies on a Vibrio anguillarum–related (VAR) strain causing an epizootic in Argopecten purpuratus larvae cultured in chile. Dis. Aquat. Org. 22: 135–141. [ Links ]
Riquelme C, Hayashida G, Vergara N, Vasquez A, Morales Y, Chavez P. 1995b. Bacteriology of the scallop Argopecten purpuratus (Lamarck 1819) cultured in Chile. Aquaculture 138: 49–60. [ Links ]
Riquelme C, Toranzo A, Barja J, Vergara N, Araya R. 1996. Association of Aeromonas hydrophila and Vibrio alginolyticus with larval mortalities of scallop (Argopecten purpuratus). J. Invert. Pathol. 67: 213–218. [ Links ]
Riquelme C, Ruben A, Escribano R. 2000. Selective incorporation of bacteria by Argopecten purpuratus larvae: Implications for the use of probiotics in culturing systems of the Chilean scallop. Aquaculture 181: 25–36. [ Links ]
Rojas R, Miranda CD, Amaro AM. 2009. Pathogenicity of a highly exopolysaccharide–producing Halomonas strain causing epizootics in larval cultures of the Chilean scallop Argopecten purpuratus (Lamarck 1819). Microb. Ecol. 57: 129–139. [ Links ]
Sandaa R, Magnesen T, Torkildsen L, Bergh L. 2003. Characterisation of the bacterial community associated with early stages of great scallop (Pecten maximus), using denaturing gradient gel electrophoresis (DGGE). Syst. Appl. Microbiol. 26: 302–311. [ Links ]
Skjermo J, Vadstein O. 1999. Techniques for microbial control in the intensive rearing of the marine larvae. Aquaculture 177: 333–343. [ Links ]
Torkildsen L, Coyne R, Samuelsen OB, Magnesen T, Bergh O. 2002. Treatment of the early life stages of scallop (Pecten maximus) with antimicrobial agents; searching for an alternative to chloramphenicol. Aquacult. Int. 10: 399–109. [ Links ]
Yoon JH, Oh TK, Park YH. 2004. Kangiella koreensis gen. nov., sp. nov. and Kangiella aquimarina sp. nov., isolated from a tidal flat of the Yellow Sea in Korea. Int. J. Syst. Evol. Microbiol. 54: 1829–1835. [ Links ]
*English translation by Christine Harris.
**Descargar versión bilingüe (Inglés–Español) en formato PDF.














