Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Ciencias marinas
Print version ISSN 0185-3880
Cienc. mar vol.32 n.4 Ensenada Dec. 2006
Artículos
Diversidad de levaduras marinas productoras de fitasas
Diversity of phytase-producing marine yeasts
NY Hirimuthugoda1,2, Z Chi1*, X Li1, L Wang1, L Wu1
1 UNESCO Chinese Center of Marine Biotechnology, Ocean University of China, Yushan Road No. 5, Qingdao, China. * E-mail: zhenming@sdu.edu.cn
2 Department of Animal Science, Faculty of Agriculture, University of Ruhuna, Mapalana, Kamburupitiya, Sri Lanka.
Recibido en febrero de 2006
Aceptado en octubre de 2006.
Resumen
Se aislaron cepas de levaduras marinas de diferentes hábitats y se monitorearon las que mostraron ser productoras de fitasa. De las 327 cepas aisladas, 10 mostraron comparativamente mayor actividad fitasa. Se examinaron las características fisiológicas y bioquímicas de las levaduras productoras de fitasas, así como el pH y temperatura óptimos para su producción. También se analizaron secuencias parciales del gen 18S rARN. Con base en las características bioquímicas y fisiológicas y análisis filogenéticos, se encontró que las 10 cepas están estrechamente relacionadas con Hanseniaspora uvarum (cepa WZ1), Yarrowia lipolytica (cepa W2B), Kodamaea ohmeri (cepa BG3), Candida carpophila (cepa N12C), Issatchenkia orientalis (cepa YF04C), Candida tropicalis (cepa MA6), Yarrowia lipolytica (cepa YF08), Candida carpophila (cepa NY4E), Candida tropicalis (cepa YF12C), y Candidad tropicalis (cepa MB2). Estas cepas se obtuvieron de estómagos de peces no identificados, de vísceras de Holothuria scabra, de Hexagrammos otakii, de Hexagrammos otakii, de Synechogobius hasta, así como directamente de agua de mar. Es interesante resaltar que algunas levaduras marinas pueden producir fitasa alcalina. Ésta es la primera vez que se reportan cepas de levaduras marinas capaces de producir fitasa extracelular o unida a las células. Los resultados también indican que las levaduras marinas productoras de fitasa fueron lo suficientemente diversas como para ser utilizadas en la bioremediación de contaminación marina por fósforo.
Palabras clave: levaduras marinas, fitasa, diversidad genética, árbol filogenético.
Abstract
Marine yeast strains from different habitats were isolated and phytase-producing marine yeasts were screened. Of the 327 yeast strains isolated, 10 showed comparatively higher phytase activity. Physiological and biochemical characters of these 10 strains, and optimum pH and temperature of the crude phytases produced by them were examined. Partial sequences of the 18S rDNA were also analyzed. Based on the physiological and biochemical characters and phylogenetic analyses, the 10 strains were found to be closely related to Hanseniaspora uvarum (strain WZ1), Yarrowia lipolytica (strain W2B), Kodamaea ohmeri (strain BG3), Candida carpophila (strain N12C), Issatchenkia orientalis (strain YF04C), Candida tropicalis (strain MA6), Yarrowia lipolytica (strain YF08), Candida carpophila (strain NY4E), Candida tropicalis (strain YF12C), and Candida tropicalis (strain MB2). They were obtained from gut of unknown fish, gut of Holothuria scabra, gut of Hexagrammos otakii, gut of Hexagrammos otakii, gut of Synechogobius hasta, and seawater. It is interesting to note that some marine yeast strains could produce alkaline phytase. This is the first report of marine yeast strains capable of producing extracellular phytase or cell-bound phytase. The results also indicated that phytase-producing marine yeasts were diverse enough to be used in bioremediation of marine phosphorous pollution.
Key words: marine yeasts, phytase, genetic diversity, phylogenetic tree.
Introducción
La fitasa (mioinositol hexakisfosfato fosfohidrolasa, EC 3.1.3.8) cataliza la liberación de fosfato a partir del fitato (hexakifosfato de inositol), que es el principal tipo de fosfato presente en los granos de cereal, legumbres y semillas oleaginosas, por lo que suele incorporarse en el pienso de aves de corral, cerdo y pescado. Además, la fitasa tiene un amplio rango de aplicaciones en nutrición animal y humana debido a que puede reducir la excreción de fósforo en animales mono-gástricos si se utiliza como sustituto del fosfato inorgánico en la dieta animal, contribuyendo significativamente a la protección del medio ambiente. La fitasa también permite mejorar la disponibilidad de minerales, elementos traza, aminoacidos y energía en la dieta (Vats y Barnerjee 2004).
En las últimas décadas la industria de la maricultura en general, incluyendo cultivos de gambas, bivalvos, algas y pepinos de mar, se ha desarrollado rápidamente gracias a la gran demanda de alimentos marinos. Prácticas de gestión no orientadas a la protección ambiental, muy comunes en los sistemas de cultivo intensivo, tales como descargas de restos y aguas residuales de las plantas de tratamiento, parecen estar relacionadas con la contaminación por fósforo en el medio marino. Dada la falta de fitasa en los estómagos de animales marinos cultivados, ésta se ha venido incorporando en su alimentación para reducir este tipo de contaminación.
Debido a la facilidad de su cultivo y a su gran productividad enzimática, los microorganismos son las mejores fuentes para la producción comercial de fitasas. Actualmente todas las preparaciones de fitasa autorizadas en la UE como aditivos alimenticios se producen mediante recombinaciones de cepas de hongos filamentosos, y los genes que expresan la fitasa provienen de hongos de dos casos del género Aspergillus (Haefner et al. 2005). En la última década, las encimas que pueden degradar fitasas provenientes de levaduras terrestres como Schwanniomyces occidentalis (Nakamura et al. 1999), Pichia anomala (Vohara y Satyanarayana 2004), Arxula adeninivo-rans (Sano et al. 1999), Hansenula polymorph (Mayer et al. 1999) y Rhodotorula gracilis (Bindu et al. 1998) también han sido objeto de creciente atención, ya que pueden ser fácilmente incorporadas en dietas balanceadas y son ricas en nutrientes. Sin embargo, se conoce muy poco sobre levaduras marinas productoras de fitasa.
Recientemente se ha encontrado que el océano cuenta con un potencial enorme de fuentes biológicas, incluyendo levaduras marinas y sus genes. Se cree que las levaduras marinas productoras de fitasas pueden ser más adecuadas como aditivo para los piensos marinos que las fitasas de microorganismos de origen terrestre, considerando además que en el medio marino las levaduras son mas eficientes que los microorganismos terrestres. Por tanto, en este estudio se examinó la diversidad fisiológica y genética de las levaduras marinas productoras de fitasa provenientes de diferentes medios, con el objetivo de encontrar levaduras marinas que puedan producir grandes cantidades de fitasa extracelular de potencial aplicación en la industria de cultivos marinos.
Material y métodos
Muestreo
Durante la campaña Antártica de 2004 se recolectaron distintas muestras de agua y sedimentos en la parte sur del Mar de China y del Océano Pacífico. También se recolectaron muestras de agua hipersalina, sedimentos provenientes de salinas y diferentes especies de animales marinos y algas a lo largo de la costa de Qingdao, China y en el Océano Índico.
Aislamiento de levaduras marinas
De las diferentes muestras se obtuvieron 2 mL de agua de mar, 2 g de sedimento o bien 2 mL de intestino de animales marinos u homogeneizado de algas, que inmediatamente después del muestreo se mezclaron en una suspensión de 20 mL de medio YPD (2% glucosa, 2% polipeptona y 1% de extracto de levadura), preparado con agua de mar con un suplemento de 0.05% de cloranfenicol, dejando dicha suspensión a temperatura ambiente durante cinco días. Después de la dilución necesaria de los distintos cultivos, la mezcla resultante se introdujo en placas de medio YPD con 0.05% de cloranfenicol, que fueron incubadas a 20-25°C durante cinco días. De estas placas se extrajeron diferentes colonias que fueron transferidas a nuevas placas YPD.
Búsqueda de levaduras marinas productoras de fitasa
Cada cepa de levadura obtenida de las placas se cultivó en medio YPD líquido durante 24 h a 28°C. Se recolectaron 2 mL de los cultivos y las células obtenidas fueron limpiadas tres veces con agua destilada esterilizada por centrifugación. Las células limpias fueron inyectadas en placas con vitaminas, sales minerales, 1% de sulfato amónico, 0.5% de fitato sódico como única fuente de carbono y fosfato, y 2% de agar, todo ello tamponado a pH 5. Después de 24 h de incubación, cada colonia fue transferida a una segunda placa con el mismo medio. Todas las cepas que crecieron vigorosamente en la segunda placa fueron usadas para la estimación de fitasa.
Estimación de fitasa
Las cepas de levadura seleccionadas de la segunda placa fueron inoculadas en viales de 250 mL con 50 mL de medio sintético mínimo con vitaminas, sales minerales, 1% de sulfato de amonio, 0.5% de fitato sódico, y posteriormente fueron incubadas aeróbicamente durante cinco días a 28°C. Se centrifugó 1 mL del medio de cultivo a 5000 rpm durante 10 min. El sobrenadante obtenido fue usado como preparación cruda de fitasa extracelular. Las pelotillas fueron lavadas tres veces con agua esterilizada y destilada doblemente, y posteriormente resuspendidos en 1 mL de medio sintético. Esta suspensión se usó como preparación para la fitasa unida a la célula. La actividad fitasa fue estimada de la siguiente manera: se preincubo 0.8 mL de solución de fitato de sodio (5 mM fitato de sodio en 0.2 M acetato de sodio, pH 5) a 60°C durante 5 min, añadiendo o bien 0.2 mL de la solución cruda de fitasa extracelular o de la preparación de fitasa unida a la célula, mezclando bien la solución en ambos casos con la ayuda de una punta de pipeta. La mezcla fue incubada a 60°C durante 30 min. La reacción fue posteriormente detenida mediante la adición de 1 mL de TCA (ácido tricloroacético) al 5%. El fosfato inorgánico liberado fue determinado cuantitativamente mediante el método del molib-dato de amonio (Chi et al. 1999) en un espectrofotómetro a 700 nm. La unidad de actividad fitasa se definió como la cantidad de encima que provoca la liberación de 1 μM de fosfato inorgánico por minuto bajo las condiciones del experimento. Para poder examinar los efectos de la temperatura en la actividad fitasa, ésta última se midió a 37°C, 45°C, 50°C, 55°C, 60°C, 65°C y 70°C. El efecto del pH en la actividad enzimática se determinó incubando la mezcla entre pH 4 y 9, usando las condiciones de experimentación estándar. Los tampones usados fueron acetato 0.2 M (pH 4.0-6.0) y Na2B407 10 H2O-H3Br3 0.2 M (pH 7.0-9.0).
Extracción total del ADN genómico y PCR
Todo el ADN genómico de las cepas de levaduras se aisló y purificó utilizando los métodos descritos por Sambrook et al. (1989). Se utilizaron precursores universales para la amplificación del gen 18S ARNr de las levaduras. El precursor forward fue P1 (5׳-ATCTGGTTGATCCTGCCAGT-3׳) y el reverse fue P2 (5׳-GATCCTTCCGCAGGTTCACC-3׳) (Thanh et al. 2002). Veinticinco microlitros de la mezcla reactiva contenían 12.5 μL de 10 χ tampón, 2 μL de dNTP 10 mM, 1.5 μL de MgCl2 25 mM, 1 μL de 20 μg mL-1 de P1, 1 μL de 20 μg mL-1 de P2, 0.5 μL de 5 UμL-1 Taq ADN polimerasa, 0.5 μL de ADN templado y 17.75 μL de H2O. La amplificación por PCR se llevó a cabo mediante la desnaturalización a 94°C durante 5 min seguida por 30 ciclos de desnaturalización a 94°C durante 1 min, templado a 55°C durante 1 min, extensión a 72°C por 2 min, y una extensión final a 72°C por 10 min. El producto del PCR se separó por electroforesis en agarosa, y se recuperó usando una columna en gel UNIQ para ADN (BIOASIA, Shanghai). El producto PCR recuperado fue incluido en un vector pGEM-T e introducido en células competentes de Escherichia coli JM109. Los transformadores fueron seleccionados en placas con ampicilina. Los plásmidos en las células transformadas se extrajeron usando el método descrito en Sambrook et al. (1989). Para poder confirmar que los productos del PCR fueron incluidos en el vector, los plásmidos purificados se utilizaron como plantilla para la amplificación del gen 18S rARN de las cepas de levadura. El sistema de reactivos y las condiciones para la amplificación del PCR fueron las mismas descritas anteriormente. El fragmento del gen 18S rARN insertado en el vector fue secuenciado por la Compañía Shanghai Sangon.
Análisis filogenético
Las secuencias obtenidas fueron analizadas para detectar similitudes utilizando BLAST (http://www.ncbi.nlm.nih.gov/ BLAST). El árbol filogenético se construyó usando el paquete de software PHYLIP, versión 3.75c (Felsenstein, 1995). Las matrices de distancia se generaron usando el programa DNA-DIST, basado en el modelo de dos parámetros de Kimura (Kimura 1980). Se realizaron análisis tipo Neighbor-joining de los datos mediante el programa Neighbor del paquete PHYLIP.
Identificación rutinaria de las levaduras
La identificación rutinaria de las levaduras se realizó usando los métodos descritos por Kurtzman y Fell (2000).
Resultados
Levaduras marinas con actividad fitasa
Se obtuvo un total de 327 cepas de levadura a partir de agua de mar, intestinos de peces marinos y algas marinas, pero sólo 10 de estas cepas segregaron cantidades elevadas de fitasa en el medio o tuvieron actividad fitasa ligada a la célula (tabla 1).
Las cepas WZ1, W2B, BG3, N12C, YF04C, MA6, YF08, NY4E, YF12C y MB2 fueron aisladas a partir del intestino de peces marinos no identificados a lo largo de la línea de costa de Qingdao, intestino de Holothuria scabra en China, intestino de Hexagrammes otakii, agua de mar del Océano Pacífico, intestino de Hexagrammes otakii, agua de mar del Océano Índico, intestino de Synecogobius hasts, agua proveniente de salinas, intestino de Holothuria scabra de Sri Lanka y agua de mar del sur del Mar de China, respectivamente.
Caracterización fisiológica y bioquímica
La tabla 2 muestra los espectros de fermentación y asimilación de fuentes de carbono, y otras características fisiológicas y bioquímicas de las distintas levaduras marinas productoras de fitasa obtenidas en este estudio.
Con base en los espectros de fermentación y de asimilación de fuentes de carbono y otras características fisiológicas y bioquímicas de las distintas levaduras marinas productoras de fitasa y las cepas tipo listadas por Kurtzman y Fell (2000), encontramos que las cepas WZ1, W2B, BG3, YF04C y YF08 fueron similares a Hanseniaspora uvarum, Yarrowia lipoly-tica, Kodamaea ohmeri, Issatchenkia orientalis y Yarrowia lipolytica, respectivamente, mientras que N12C y NY4E fueron similares a Candida carpophila, y MA6, YF12C y MB2 lo fueron a Candida tropicalis .
Análisis filogenético de las secuencias parciales de los genes 18S rARN
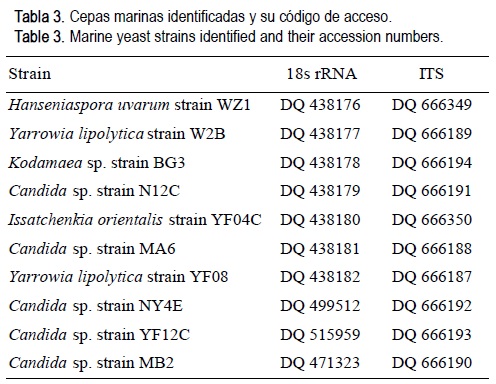
De acuerdo con Kurtzman y Fell (2006), los métodos tradicionales y la identificación rutinaria que dependen del fenotipo generalmente tienden a interpretaciones poco precisas de las interacciones entre especies. El análisis secuencial de la filogenia para realizar taxonomía microbiana es un método más preciso para determinar relaciones intra e interespecíficas. Por tanto, se analizaron secuencias parciales del ITS de 18S rARN de las cepas de levadura para los análisis comparativos usando un análisis BLAST (http://www.ncbi.nlm.nih.gov/BLAST) y se depositaron en el National Center for Biotechnological Information (NCBI), EUA, bajo los números de acceso que aparecen en la tabla 3. Cafetena roenbergensis (un flagelado heterótrofo) se usó como elemento fuera del grupo para construir el árbol filogenético. La búsqueda de similitudes entre las secuencias de 18S rARN aisladas y las que se encuentran en la base de datos del NCBI indica que las cepas de levadura encontradas en este análisis son similares a varias especies filogenéticamente relacionadas. En la figura 1 se muestran las relaciones filogenéticas de las secuencias de 18S rARN de las cepas identificadas de levaduras marinas, y sus números de acceso en Genbank se muestran en la tabla 3.
Discusión
Hasta donde sabemos, éste es el primer trabajo de investigación completo sobre un rango considerablemente diverso de levaduras marinas productoras de fitasa. Es importante destacar que ninguno de los animales marinos utilizados era de cultivo, sino que provenían de medios marinos naturales. Estos resultados muestran que muchas de las levaduras marinas productoras de fitasa fueron obtenidas de intestinos de animales marinos, sugiriendo que pueden existir en dichos ambientes. En la tabla 1 se puede observar que algunas de las fitasas de levaduras marinas pueden actuar de forma óptima en medios alcalinos. Por ejemplo, la fitasa extracelular en crudo producida por las cepas W2B, NY4E y YF12C, y la fitasa unida a células en WZ1 tuvieron un pH óptimo de 8, mientras la fitasa extracelular producida por la cepa YF04C tuvo un pH óptimo de 9. Sin embargo, el pH óptimo de las fitasas de levaduras ymohos terrestres es de 2.5 a 5.5. Se ha descrito que las fita-sas alcalinas de microorganismos pueden tener aplicaciones prometedoras para la industria (Haefner et al. 2005). Por lo tanto, es necesario realizar más trabajos sobre la fitasa alcalina proveniente de la cepa YF04C. En la tabla 1 se puede observar que Yarrowia lipolytica W2B y Kodamea ohmeri BG3 tuvieron la mayor actividad fitasa extracelular, y ambas fueron aisladas a partir de intestinos de animales marinos. Varios trabajos han mostrado que estas dos especies de levadura son comunes en ambientes marinos o de alta salinidad (Barnett et al. 2000, Jain et al. 2004).
La temperatura óptima de las fitasas de levaduras marinas estuvo en un rango entre 55°C y 65°C. Este resultado es idéntico que para las levaduras terrestres. En general, la fitasa de levaduras terrestres muestra una gran actividad en el rango de temperaturas entre 50°C y 70°C, mientras que la temperatura óptima se encuentra principalmente entre 45°C y 60°C (Haefner et al. 2005). También se puede observar en la tabla 1 que las cepas WZ1, N12C, NY4E y YF12C pueden producir tanto fitasa extracelular como fitasa ligada a la célula, pero ambas fitasas mostraron diferentes temperaturas y pH óptimos. Vohara y Satyanarayana (2004) informaron de ligadura de fitasa a la célula en cepas de levaduras terrestres. De acuerdo con estos autores está probado que un considerable porcentaje de la fitasa está ligado a la pared celular y, por tanto, no está disponible para la digestión por fitasa esperada en la alimentación de seres vivos. También se puede inferir de la tabla 1 que las cepas de levaduras marinas aisladas de la superficie de algas no produjeron ni fitasa extracelular ni ligada a la pared celular.
Con base en la topología del árbol filogenético parece que las 10 cepas aisladas pertenecen a seis especies genéticas. La topología del filograma en la figura 1 confirma que la cepa WZ1 está muy relacionada con Hanseniaspora uvarum, mientras que las cepas W2B y YF08 están muy relacionadas con Yarrowia lipolytica. La cepa BG3 fue asignada a Kodamaea ohmeri. Las cepas N12C y NY4E pueden estar estrechamente relacionadas con Candida carpophila mientras que la cepa YF04C pertenece a los miembros de Issatchenkia orientalis. Las secuencias de 18S rARN de las cepas MA6, YF12C y MB2 fueron idénticas que las de Candida tropicalis . A pesar de que los resultados de asimilación de carbono y fermentación de las cepas YF12C, MA6 y MB2 fueron similares a Candida tropicalis (tabla 2), el árbol filogenético indica que las cepas MA6, YF12C y MB2 pertenecen a dos genotipos (fig.1).
A pesar de que muchas especies de levaduras terrestres han sido identificadas como productoras de fitasa (Segueilha et al. 1992, Lambrechts et al. 1993, Bindu et al. 1998, Mayer et al. 1999, Nakamura et al. 1999), los resultados obtenidos en este estudio indican que todas las cepas encontradas fueron especies nuevas de levaduras marinas con actividad fitasa. Aunque el ambiente intestinal de los animales marinos es ácido, también pueden encontrarse cepas de levaduras productoras de fitasa alcalina en el intestino de animales marinos (tabla 1). Con base en los resultados obtenidos puede concluirse que cada una de las cepas puede ser objeto de análisis futuros tales como análisis de las propiedades de la enzima purificada, clonación de genes, etc. Por lo tanto, la purificación y caracterización de fitasas alcalinas a partir de levaduras marinas está siendo objeto de estudio en este laboratorio. La purificación y las propiedades de las enzimas purificadas de estas cepas serán discutidas en trabajos futuros.
Agradecimientos
Esta investigación ha sido financiada mediante el consejo de becas de China y las becas 3032802 de la Fundación Nacional de Ciencias Naturales de China.
Referencias
Barnett JA, Payne RW, Yarrow D. 2000. Yeasts: Characteristics and Identification. 3rd ed. Cambridge Univ. Press. [ Links ]
Bindu S, Somashekar D, Joseph R. 1998. A comparative study on permeabilization treatments for in situ determination of phytase of Rhodotorula gracilis. Lett. Appl. Microbiol. 27: 336-340. [ Links ]
Chi Z, Kohlwein SD, Paltauf F. 1999. Role of phosphatidylinositol (PI) in ethanol production and ethanol tolerance by highly ethanol producing yeasts. J. Indus. Microbiol. Biotechnol. 22: 58-63. [ Links ]
Felsenstein J. 1995. PHYLIP (Phylogenetic Inference Package), Version 3.75. Distributed by author, Department of Genetics, University of Washington, Seattle. [ Links ]
Haefner S, Knietsch A, Scholten E, Braun J, Lohscheidt M, Zelder O. 2005. Biotechnological production and applications of phytases. Appl. Microbiol. Biotechnol. 68: 588-597. [ Links ]
Jain MR, Zinjarde SS, Deobagkar DD, Deobagkar DN. 2004. 2,4,6- Trinitrotoluene transformation by a tropical marine yeast, Yarrowia lipolytica NCIM 3589. Mar. Pollut. Bull. 49: 783-788. [ Links ]
Kimura M. 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies on nucleotide sequences. J. Mol. Evol. 2: 87-90. [ Links ]
Kurtzman CP, Fell J. 2000. The yeasts. A taxonomic Study. 4th revised and enlarged ed. Elsevier, Amsterdam. Pp. 5-174. [ Links ]
Kurtzman CP, Fell JW. 2006. Yeast systematics and phylogeny: Implications of molecular identification methods for studies in Ecology. In: Rosa CA, Peter G (eds.), The Yeast Handbook. Springer-Verlag, Berlin, pp.11-30. [ Links ]
Lambrechts C, Boza H, Segueilha L, Moulin G, Galzy P. 1993. Influence of culture conditions on the biosynthesis of Schwanniomyces castellii phyatse. Biotechnol. Lett. 15: 399-404. [ Links ]
Mayer AF, Hellmuth K, Schlieker H, Lopez-Ulibarri R, Oertel, S, Dahlems U, Strasser AWM, Van-Loon APGM. 1999. An expression system matures: A highly efficient and cost-effective process for phytase production by recombinant strains of Hansenulapolymorpha. Biotechnol. Bioeng. 63: 373-381. [ Links ]
Nakamura T, Suzuki T, Tokuda J, Kato N, Sakai Y, Mochizuki D, Takhashi H. 1999. Secretory manufacture of Schwanniomyces occidentalis phytase using a Candida boidinii host. Eur. Patent Appl. Ep. 931, 837 July 28. [ Links ]
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular Cloning: A Laboratory Manual. 2nd ed. Cold Spring Harbor Laboratory Press, Beijing (Chinese translating ed. [ Links ]).
Sano K, Fukuhara H, Nakamura Y. 1999. Phytase of the yeast Arxula adeninivorans. Biotechnol. Lett. 21: 33-38. [ Links ]
Segueilha L, Lambrechts C, Boze H, Moulin G, Galzy P. 1992. Purification and properties of the phytase from Schwanniomyces castellii. J. Ferment. Bioeng. 74: 7-11. [ Links ]
Thanh VN, Van DMS, Wingfield MJ. 2002. Debaryomyces mycophilus sp. nov., a siderophore-dependent yeast isolated from woodlice. FEMS Yeast Res. 2: 415-427. [ Links ]
Vats P, Banerjee CU. 2004. Production studies and catalytic properties of phytases (myo-inositolhexakisphosphate Pposphohydrolases): An overview. Enzyme Microb. Technol. 35: 3-14. [ Links ]
Vohara A, Satyanarayana T. 2004. A cost effective molasses medium for enhanced cell bound phytase production by Pichia anomala. J.Appl. Microbiol. 97: 471-476. [ Links ]














