Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Ciencias marinas
versión impresa ISSN 0185-3880
Cienc. mar vol.32 no.3 Ensenada sep. 2006
Artículos
Variabilidad mitocondrial del dorado Coryphaena hippurus en poblaciones del Pacífico
Mitochondrial variability of dolphinfish Coryphaena hippurus populations in the Pacific Ocean
A Rocha-Olivares1*, M Bobadilla-Jiménez1, S Ortega-García2, N Saavedra-Sotelo1, JR Sandoval-Castillo1
1 Laboratorio de Ecología Molecular, Departamento de Oceanografía Biológica, Centro de Investigación Científica y de Educación Superior de Ensenada (CICESE), Km 107 Carretera Tijuana-Ensenada, Ensenada, CP 22860, Baja California, México. * E-mail: arocha@cicese.mx
2 Departamento de Pesquerías, Centro Interdisciplinario de Ciencias Marinas (CICIMAR-IPN), Playa El Conchalito s/n, La Paz, CP 23000, Baja California Sur, México.
Recibido en marzo de 2006
Aceptado en junio de 2006.
Resumen
Los patrones de estructura genética en las poblaciones marinas están asociados a una variedad de mecanismos de dispersión y de escalas espaciales. Las especies pelágicas migratorias, como el dorado Coryphaena hippurus, reafirman la noción de un medio ambiente marino abierto y continuo. Muchos estudios han mostrado que las especies pelágicas oceánicas tienden a ser homogéneas genéticamente a grandes escalas geográficas, sólo existiendo diferenciación entre sus límites de distribución o entre cuencas oceánicas. En este trabajo presentamos resultados que indican heterogeneidad genética del dorado a escalas geográficas menores a las predichas por las generalizaciones anteriores. Patrones de restricción (RFLPs) del gen mitocondrial NADH1 produjeron niveles de estructura genética altamente significativos (ΦST = 0.029, P = 0.004, AMOVA) entre peces de Baja California Sur (BCS), Sinaloa y Hawai, consistentes con frecuencias haplotípicas heterogéneas (P = 0.014, prueba exacta de diferenciación genética) y con una menor diversidad molecular en los peces muestreados en BCS.
Palabras clave: dorado, NADH1, ADN mitocondrial, estructura genética, RFLPs.
Abstract
Patterns of genetic structure among marine populations involve a variety of dispersal mechanisms and spatial scales. Pelagic species, such as the dolphinfish Coryphaena hippurus, epitomize the open and continuous nature of the marine environment due to their extensive migrations. Many studies have revealed that oceanic pelagic species tend to be genetically homogeneous over local and often extended geographic scales and only show levels of differentiation among extreme localities or ocean basins. Here we present genetic data suggesting genetic heterogeneity in the dolphinfish at geographic scales much smaller than those predicted by those generalizations. Mitochondrial NADH1 gene RFLPs revealed a highly significant (ΦST = 0.029, P = 0.004, AMOVA) molecular genetic structure among fish from Baja California Sur (BCS), Sinaloa and Hawaii, consistent with heterogeneous haplotype frequencies (P = 0.014, exact test of genetic differentiation) and a depressed molecular diversity in fish sampled off BCS.
Key words: dolphinfish, NADH1, mitochondrial DNA, genetic structure, RFLPs.
Introducción
La diversidad de hábitats ocupados por las poblaciones de peces marinos se extiende desde aguas costeras intermareales hasta los giros oceánicos oligotróficos. Como consecuencia, no existe un paradigma general que explique los patrones de conectividad entre poblaciones alopátricas (Smith y Fujio 1982). La genética poblacional ha sido una forma indirecta pero poderosa de estudiar la conectividad marina, particularmente en la evaluación de influencias históricas (Dawson et al. 2001) y en el descubrimiento de barreras crípticas a la dispersión y libre entrecruzamiento (Sandoval-Castillo et al. 2004). Se han hecho esfuerzos considerables para entender los procesos pasivos, activos e históricos que determinan la dispersión y retención de algunas especies relativamente sedentarias como los peces demersales (Buonaccorsi et al. 2002) y arrecifales (Taylor y Hellberg 2003). Dichos procesos son de suma importancia en el diseño e implementación de reservas marinas para la protección de poblaciones amenazadas por la sobreex-plotación y la degradación de hábitat (Palumbi 2003). Una perspectiva muy diferente emerge de los patrones de diferenciación genética de especies pelágicas altamente migratorias y con amplias distribuciones geográficas (Graves 1996). Aunque las fuerzas fundamentales que determinan su arquitectura genética no son diferentes de sus contrapartes sésiles, los peces migratorios y ampliamente distribuidos comparten características biológicas que pueden limitar la diferenciación genética entre localidades distantes tales como: (1) grandes grupos de organismos desovantes, lo que puede limitar los efectos de la deriva génica; (2) ausencia de barreras para la migración, que permite algunas veces movimientos transoceánicos e inclusive interoceánicos; y (3) capacidad intrínseca de dispersión a grandes distancias, permitiendo altos niveles de flujo genético por transporte pasivo (en etapas tempranas de vida) pero principalmente por migración activa de adultos a grandes distancias (Jorgensen et al. 2005). Sin embargo, estas expectativas a veces se encuentran con datos contradictorios que ponen en duda su aplicabilidad universal e implican la existencia de factores especie-específicos o regionales que limitan la conectividad y el flujo genético en especies pelágicas y que ocasionan la formación de una estructura genética poblacional (Graves 1996).
El dorado Coryphaena hippurus L. es un pez pelágico de distribución tropical y subtropical de gran importancia tanto para la pesca comercial como para la deportiva (Oxenford 1999). Los dorados son peces de rápido crecimiento (Massutí et al. 1999) y voraces depredadores visuales diurnos que se alimentan de otros teleósteos y cefalópodos (Oxenford y Hunte 1999). Están muy bien adaptados morfo-fisiológicamente para maniobrar y nadar rápidamente (Brill 1996). Su resistencia y agresividad la hacen una especie muy atractiva en la pesca deportiva (Harper et al. 2000). El dorado es también una especie valorada para consumo humano, por lo que se ha desarrollado tecnología de acuacultura para su producción en cautiverio (Lee y Ostrowski 2001). El hábitat del dorado son las aguas epipelágicas tropicales y subtropicales en donde tienden agregarse alrededor de objetos flotantes. Dicho comportamiento ha sido aprovechado en pesquerías comerciales en las que se utilizan artefactos flotantes para la agregación de peces (Deudero et al. 1999).
La abundancia y reproducción estacional de C. hippurus ha sido asociada a su conducta migratoria en diversas regiones del mundo, como en el Pacífico central (Hawai) (Kraul 1999), el Pacífico oriental (Lasso y Zapata 1999), el Atlántico occidental y el Caribe (Arocha et al. 1999, Rivera y Appeldoorn 2000), y el Mediterráneo (Massutí y Morales-Nin 1995). Estudios de marcado y recaptura realizados en Australia han mostrado el gran potencial de dispersión del dorado de hasta 440 km a una velocidad estimada de 20 km día-1. Sin embargo, dichos estudios han mostrado también que la especie exhibe rangos hogareños limitados, ya que la mayoría de los individuos fueron recapturados alrededor de los mismos objetos flotantes donde fueron originalmente marcados (Kingsford y Defries 1999). Además, si bien las hembras en cautiverio son capaces de desovar prolíficamente a lo largo del año, la abundancia estacional y desoves parciales documentados en el Pacifico oriental (e.g., García-Melgar 1995) sugieren que las frezas en vida libre están más limitadas temporal y espacialmente, o bien que las larvas están sujetas a periodos discretos de mayor supervivencia, de tal suerte que la abundancia estacional puede depender tanto de las tasas de supervivencia de cada cohorte como de los cambios en el régimen de temperatura superficial (Kraul 1999).
Tanto los rangos hogareños restringidos como los pulsos demográficos temporal y espacialmente localizados pueden favorecer la existencia de una estructura poblacional en stocks diferenciados y de heterogeneidad genética, lo cual ha sido sugerido por diferencias en la dinámica poblacional de la especie en aguas mexicanas (Beltrán-Pimienta 2000) así como en el Mediterráneo (Cimmaruta et al. 1998; pero ver Pla y Pujolar 1999), el sureste de los Estados Unidos de América (Oxenford 1999), y entre muestras de Hawai y Taiwán (Herzig 1990). En este artículo se evaluaron los niveles de variabilidad genética y conectividad entre poblaciones a dos escalas espaciales, una regional (Pacífico oriental) y una oceánica (entre Pacífico oriental y central), usando polimorfismos de longitud de fragmentos de restricción (RFLPs) del gen mitocondrial NADH1. Estos resultados podrán ser utilizados para diseñar medidas más adecuadas en el manejo de este recurso pelágico, actualmente reservado para la pesca deportiva en las aguas territoriales mexicanas.
Materiales y métodos
De la pesca deportiva en cuatro localidades de México y del mercado de mariscos de la isla Oahu (Hawai) se recolectaron muestras de tejido (muscular y hepático) de C. hippurus que se preservaron en etanol al 95% hasta su análisis en laboratorio (tabla 1). Se extrajo y purificó el ADN genómico total a partir de la digestión del tejido con proteinasa K, la precipitación proteica con cloruro de litio, y la extracción orgánica con cloroformo-alcohol isoamílico, para ser finalmente precipitado con etanol en presencia de acetato de amonio. El gen mitocondrial NADH1 se amplificó por la reacción en cadena de la polimerasa (PCR) con los cebadores L3324 5י GCTCTACGTGATCTGAGTTCAG 3י y H4716 5י TACATGTTTGGGGTATGGGC 3י (Wingrove 2000) utilizando HotStart Taq Mastermix® de acuerdo al protocolo del fabricante (Qiagen, Valencia, California). Entre los genes que codifican proteínas mitocondriales, éste es uno de los que poseen tasas de evolución mayores (Lopez et al. 1997). La PCR se realizó con el siguiente perfil de termociclado: activacion inicial de la Taq de 15 min a 95°C, seguida de 35 ciclos de 95°C por 55 segundos, 50°C por 45 segundos, y 72°C por 90 segundos, e incubación final a 72°C por 5 min. Posteriormente, las amplificaciones se confirmaron por medio de electroforesis en geles de agarosa al 1.5%. Los productos de la PCR fueron digeridos con cinco endonucleasas de restricción (Alu I, Hae III, Rsa I, ScrFI y Dpn II) siguiendo las indicaciones del fabricante (New England Biolabs, Ipswich, Massachusetts). Los fragmentos de restricción fueron separados por electroforesis en geles de poliacrilamida (29:1) al 6%, teñidos con bromuro de etidio (0.25 μg L-1) y posteriormente fotografiados para su análisis. Los tamaños de los fragmentos en pares de bases (pb) fueron determinados con el programa 1D versión 2.03 (Kodak, New Haven, Connecticut).
Los sitios de restricción fueron mapeados con secuencias ortólogas obtenidas del Genbank (AF272056, AF272058, AF290386, AF290388 y AF272059) y los patrones de restricción de cada enzima se combinaron en un haplotipo compuesto para cada organismo. Se calculó la distancia interhaplotípica a partir de la presencia o ausencia de los sitios de restricción de acuerdo a Nei y Miller (1990, ec. 4). Se estimaron las diversidades haplotípica y nucleotídica de acuerdo a Nei (Nei 1987, ec. 8.4 y 10.14) y la divergencia nucleotídica siguiendo a Nei y Tajima (Nei y Tajima 1981, ec. 22). Estos análisis se realizaron con el programa REAP 4.0 (McElroy et al. 1992). Se realizó una prueba exacta de homogeneidad en la distribución de haplotipos para la cual se estimó la significancia estadística mediante simulaciones Monte Carlo de acuerdo con Raymond y Rousset (1995). Se calcularon índices de fijación FST y su análogo molecular 0ST a partir de un análisis de varianza molecular (AMOVA), usando una matriz de distancias interha-plotípicas como distancias euclideanas (Excoffier et al. 1992). Se estimaron los niveles de flujo genético (Nm) a partir de los índices de fijación (Cockerham y Weir 1993, Slatkin 1995). Para estos análisis se utilizó el programa Arlequin 2.0 (Schneider et al. 1999) estimándose la significancía estadística por métodos exactos (Guo y Thompson 1992). Finalmente, se reconstruyeron árboles filogenéticos en la forma de una red de mínima expansión (MSN) y de un filograma "Neighbor-Joining" (NJ, Saitou y Nei 1987) usando el número de diferencias en sitios de restricción entre cada haplotipo y la distancia de Nei y Tajima con los programas Arlequin 2.0 y Mega 2.1 (Kumar et al. 2001), respectivamente.
Resultados
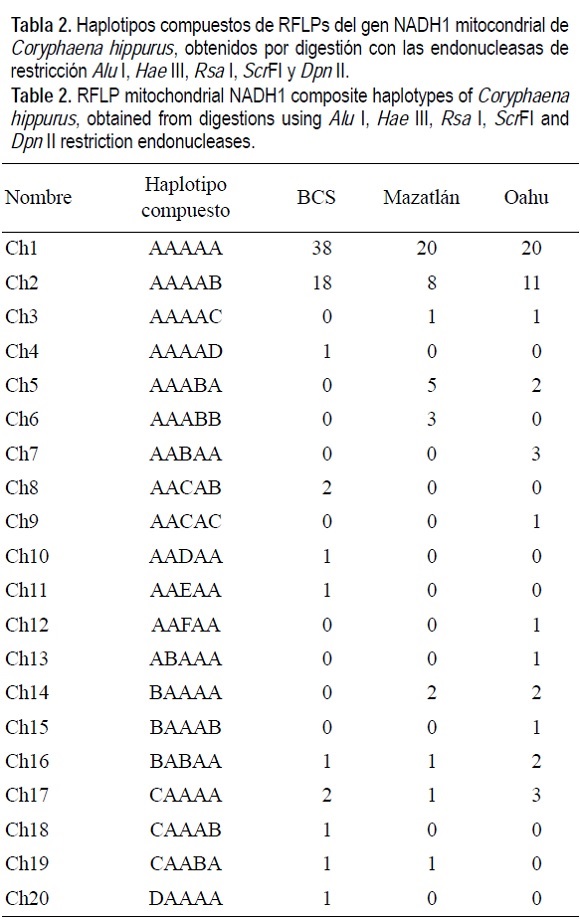
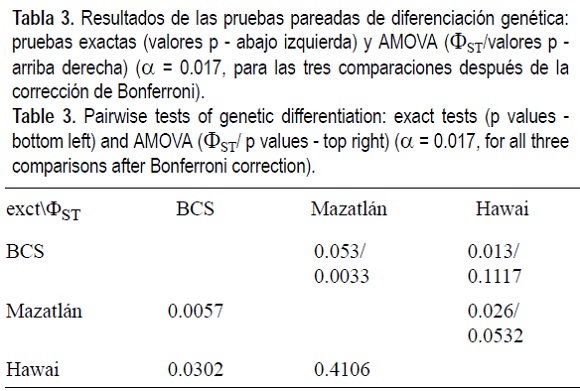
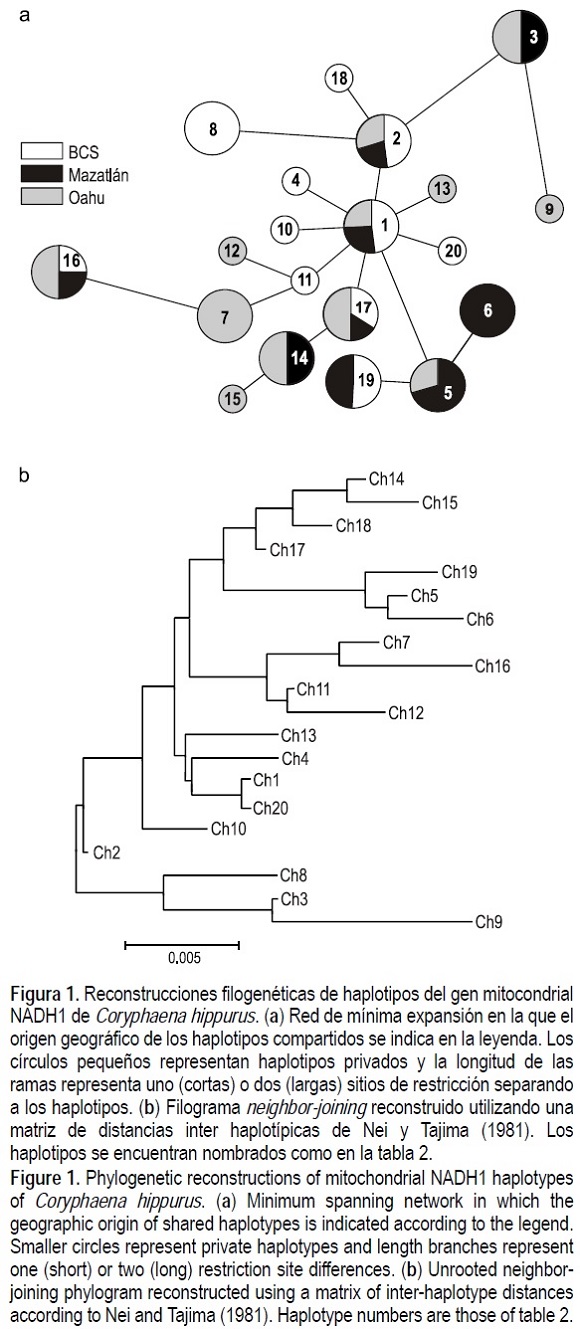
Se analizaron 157 muestras a las cuales se amplificó el gen NADH1 (alrededor de 1400 pb) y se digirió exitosamente. Se encontraron 38 sitios de restricción en todas las enzimas cuyo número varió entre 4 sitios para Dpn I y 11 para ScrFI. Los polimorfismos nucleotídicos produjeron 20 haplotipos compuestos del gen NADH1, uno de los cuales (Chl) se encontró en 78 peces (49.7%), predominando en todas las regiones (tabla 2). El siguiente haplotitpo más abundante, que difirió en sólo un sitio de restricción, ocurrió en 24% de las muestras. La mayoría de los haplotipos (60%) fueron privados, ocurriendo en sólo una de las regiones en bajas frecuencias (tabla 2). La combinación de dos haplotipos predominantes y muchos raros resultó en una alta diversidad haplotípica (tabla 1). Las muestras provenientes de Baja California Sur (BCS: La Paz, Los Cabos y Punta Lobos) fueron genéticamente indiferenciables (ΦST = -0.013, p = 0.66, AMOVA), por lo que se agruparon en una muestra de BCS en los análisis sucesivos para incrementar el tamaño de muestra y el poder estadístico de las pruebas de diferenciación (tabla 1). Las muestras de BCS fueron mucho menos diversas tanto a nivel haplotípico como nucleotídico (tabla 1), como resultado de una sobre dominancia (84%) de los dos haplotipos más abundantes en comparación con las otras dos localidades (65-67%) (tabla 2). La frecuencia de haplotipos entre las tres localidades fue significativamente heterogénea (P = 0.014, prueba exacta). Asimismo, la proporción de varianza genética molecular explicada por la estructura genética fue pequeña pero altamente significativa (ΦST = 0.029, P = 0.004, AMOVA); sin embargo, el índice de fijación basado en frecuencias haplotípicas no fue significativamente diferente de cero (FST = 0.005, P = 0.21). Las comparaciones pareadas consistentemente identificaron como significativa a la diferenciación entre BCS y Mazatlán, pero la diferencia entre BCS y Hawai sólo fue significativa en la prueba exacta mientras que el ΦST entre Hawai y Mazatlán no fue significativo (tabla 3). Los niveles de flujo genético entre las regiones fueron: Nm = 9 (ΦST = 0.053, BCS/Mazatlán), Nm = 37 (ΦST = 0.013, BCS/ Hawaii), y Nm = 19 (ΦST = 0.026, Mazatlán/Hawaii). Se detectaron diferencias topológicas entre las reconstrucciones filogenéticas MSN (fig. 1a) y el filograma NJ (fig. 1b). Por ejemplo, del clado [Ch3, 8, 9] conectado a Ch2 en el filograma, sólo [Ch3, 9] permanecen unidos y Ch8 se conecta directamente a Ch2 en el MSN. No obstante, la mayor parte de las relaciones son congruentes entre ambas reconstrucciones, en particular los haplotipos más abundantes (Ch1 y Ch2) aparecen como ancestrales. Otro aspecto relevante es la filogenia con forma de estrella del MSN (fig 1a). La distribución geográfica de los linajes mitocondriales reveló una ausencia de patrones filogeográficos, con la posible excepción de la preponderancia del linaje divergente que comprende los haplotipos Ch5, 6 y 19 y del cual nueve de las copias se encuentran en Mazatlán (fig. 1), aunque sólo el Ch6 es exclusivo de esta localidad. El MSN y el filograma también revelaron que la mayoría de los haplotipos de BCS, con excepción de Ch8 y 19, se encuentran poco diferenciados, difiriendo en sólo uno o dos sitios de restricción, lo cual explica la poca diversidad nucleotídica observada en estos peces y la singularidad genética de esta muestra.
Discusión
Los niveles de variabilidad y de estructura genética detectados con loci nucleares aloenzimáticos (Gyllensten 1985, Ward et al. 1994) y microsatelitales (DeWoody y Avise 2000) han revelado ciertas tendencias en peces marinos y dulceacuícolas. Los peces marinos poseen mayores niveles de heterocigosidad, particularmente en loci microsatelitales hipervariables (DeWoody y Avise 2000), pero niveles de estructura genética significativamente menores (expresados como GST multilocus) puesto que intercambian de 10 a 100 veces más migrantes por generación que los peces dulceacuícolas (Ward et al. 1994). Dichas tendencias son atribuibles a diferencias en tamaño efectivo poblacional y a la ausencia de barreras para la dispersión en el medio ambiente marino. El océano representa un hábitat continuo e ilimitado y pocas especies manifiestan este hecho mejor que las comunidades oceánicas pelágicas que son habitadas por peces migratorios como los escómbridos (atunes), istiofóridos (picudos) y corifénidos (dorado), entre otros.
Al igual que la gran mayoría de peces marinos, las especies pelágicas poseen larvas planctónicas que son acarreadas pasivamente por corrientes oceánicas. Esta dispersión, aunada al movimiento activo de adultos, genera altos niveles de conectividad a escalas espaciales mayores que las de especies costeras (Bowen y Grant 1997). No obstante, en el caso del dorado que puede desarrollarse en un juvenil de 16.5 cm de longitud furcal en alrededor de 50 días (Massutí et al. 1999), el rápido crecimiento permite suponer que su permanencia meroplanctónica es lo suficientemente corta para limitar su tiempo a la deriva. Por ello no se espera que las corrientes oceánicas tengan un papel preponderante en el flujo genético.
La movilidad de las especies de peces pelágicos refleja la aparente naturaleza abierta y sin barreras del océano, que se contrapone a la noción de poblaciones geográficamente estructuradas con niveles de flujo genético limitados. Varios estudios han revelado patrones de diferenciación en los que las especies oceánicas tienden a ser genéticamente homogéneas a escalas geográficas regionales y frecuentemente mucho mayores, mostrando diferenciación solamente entre poblaciones extremas o entre cuencas oceánicas (e.g., Alvarado-Bremer et al. 1998, Reeb et al. 2000, Broughton et al. 2002, Pujolar et al. 2003). Inclusive en ausencia de altos niveles de flujo genético, la existencia de poblaciones de gran tamaño puede impedir o retrasar la diferenciación por efecto de deriva génica (Ely et al. 2005). La evidencia de heterogeneidad insospechada puede responder a diferencias en tipos de análisis, tipos de marcador genético (Buonaccorsi et al. 2001) o inclusive a la posible influencia de caracteres no relacionados, como el sexo o la edad de los especimenes. Por ejemplo, Gold y Richardson (1998) encontraron altos niveles de flujo genético en la macarela Scomberomorus cavalla desde Yucatán (México) hasta Carolina del Norte (EUA), así como niveles bajos de heterogeneidad, pero no encontraron índices de fijación (FST) significativos entre haplotipos mitocondriales del Golfo de México y del Atlántico. Por otra parte, descubrieron evidencia discordante entre el ADNmt y un locus aloenzimático nuclear, encontrando que la variación mitocondrial era independiente del sexo y de la edad de los peces mientras que la del locus PEPA-2a se relacionaba con dichos factores. Estos resultados explicaron por qué esta proteína daba evidencias de heterogeneidad genética dentro del Golfo de México en respuesta a diferencias demográficas entre las poblaciones (Gold y Richardson 1998). Un estudio posterior basado en loci microsatelitales hipervariables corroboró dicha conclusión (Broughton et al. 2002).
Los genes mitocondriales con mayores tasas de evolución molecular (Lopez et al. 1997), que incluyen los que codifican las subunidades de la Nicotinamida Deshidrogenasa (NADH1, 2, 5 y 6), han sido exitosamente utilizados para elucidar relaciones filogenéticas y estructura poblacional en especies de peces pelágicos como macarelas (Banford et al. 1999) y anchovetas (Bembo et al. 1996), así como de otros teleosteos marinos y dulceacuícolas (Kocher et al. 1995, Toline y Baker 1995, Patton et al. 1997, Birstein et al. 2002, Klett y Meyer 2002, Mateos et al. 2002).
Es difícil identificar de inmediato cuáles factores son responsables de la heterogeneidad observada en el dorado; sin embargo, no hay razón para sospechar que los resultados aquí obtenidos se deban a sesgos en la naturaleza de las muestras. Todos los organismos muestreados poseen tallas similares y aunque no se tienen datos de los organismos de Hawai, éstos representan peces de tamaño comercial reclutados a la pesquería, al igual que los mexicanos. Desafortunadamente, pocos estudios han examinado la estructura genética de las poblaciones de dorado. En el Mediterráneo, Cimmaruta et al. (1998) encontraron evidencia de dos subpoblaciones diferenciadas utilizando electroforesis de aloenzimas. Estos resultados no pudieron ser corroborados por Pla y Pujolar (1999), quienes utilizaron también loci aloenzimáticos y encontraron una sola población panmíctica desde las islas de Mallorca (España) y Sicilia (Italia) en el Mediterráneo occidental hasta las Islas Canarias (España) en el Atlántico oriental. En el mar Caribe, Oxenford y Hunte (1986) utilizaron caracteres de la historia de vida para identificar dos stocks de dorado en la región, así como genotipos de 16 loci polimórficos aloenzimáticos, de un total de 55 analizados, de los cuales uno (IDHP-2*) mostró evidencia de diferenciación entre Miami, Florida (EUA), y Barbados, coincidiendo con la diferenciación fenotípica detectada (Oxenford y Hunte 1986, Oxenford 1999). Finalmente, en un estudio encaminado a desarrollar nuevos métodos de marcaje de dorado, Herzig (1990) secuenció 201 pb del gen mitocondrial citocromo b que revelaron diferencias entre peces capturados en Hawai y los provenientes de Taiwan. Aunque no fue un estudio de genética poblacional, los resultados de Herzig sugieren que existe un potencial real para la diferenciación genética entre las poblaciones de dorado del Pacífico. En México, estudios comparativos de biología pesquera sugieren esta posibilidad (Beltrán-Pimienta 2000). Los resultados del presenta trabajo apoyan este hecho, aunque requieren de mayor esfuerzo para ser corroborados con muestras adicionales y un mayor número de marcadores genéticos.
Agradecimientos
Agradecemos a M Zúñiga (CICIMAR), J Hyde, E Lynn y R Vetter (NMFS-NOAA) su ayuda en la recolección de muestras, y a R Chapman (SCDNR) por compartir generosamente cebadores e información no publicada. Este artículo deriva parcialmente de la tesis de licenciatura de M Bobadilla-Jiménez llevada a cabo en CICESE bajo la dirección de A Rocha-Olivares. Este proyecto recibió apoyo de la Sociedad Cooperativa Pesquera Ejidal Punta Lobos y del Consejo Estatal de Pesca y Acuacultura de Baja California Sur, mismo que agradecemos cordialmente. El financiamiento provino del proyecto sectorial SAGARPA- CONACYT-2003-C01-42. Agradecemos cordialmente las críticas constructivas de dos árbitros anónimos.
Referencias
Alvarado-Bremer JR, Stequert B, Robertson NW, Ely B. 1998. Genetic evidence for inter-oceanic subdivision of bigeye tuna (Thunnus obesus) populations. Mar. Biol. 132(4): 547-557. [ Links ]
Arocha F, Marcano LA, Larez A, Altuve D, Alio J. 1999. The fishery, demographic size structure and oocyte development of dolphinfish, Coryphaena hippurus, in Venezuela and adjacent waters. Sci. Mar. 63(3-4): 401-409. [ Links ]
Banford HM, Bermingham E, Collette BB, McCafferty SS. 1999. Phylogenetic systematics of the Scomberomorus regalis (Teleostei: Scombridae) species group: Molecules, morphology and biogeography of Spanish mackerels. Copeia (3): 596-613. [ Links ]
Beltrán-Pimienta R. 2000. Contribución al conocimiento de la pesquería y la biología poblacional del dorado (Coryphaena hippurus, Linnaeus 1758) de las costas de Sinaloa, Nayarit y Baja California Sur, México, durante 1997. Tesis de maestría, Facultad de Ingeniería Pesquera, Universidad Autónoma de Nayarit, México. [ Links ]
Bembo DG, Carvalho GR, Snow M, Cingolani N, Pitcher TJ. 1996. Stock discrimination among European anchovies, Engraulis encrasicolus, by means of PCR-amplified mitochondrial DNA analysis. Fish. Bull. 94(1): 31-10. [ Links ]
Birstein VJ, Doukakis P, DeSalle R. 2002. Molecular phylogeny of Acipenseridae: Nonmonophyly of Scaphirhynchinae. Copeia (2): 287-301. [ Links ]
Bowen BW, Grant WS. 1997. Phylogeography of the sardines (Sardinops spp.): Assessing biogeographic models and population histories in temperate upwelling zones. Evolution 51(5): 1601-1610. [ Links ]
Brill RW. 1996. Selective advantages conferred by the high performance physiology of tunas, billfishes, and dolphin fish. Comp. Biochem. Physiol. A 113(1): 3-15. [ Links ]
Broughton RE, Stewart LB, Gold JR. 2002. Microsatellite variation suggests substantial gene flow between king mackerel (Scomberomorus cavalla) in the western Atlantic Ocean and Gulf of Mexico. Fish. Res. 54(3): 305-316. [ Links ]
Buonaccorsi VP, McDowell JR, Graves JE. 2001. Reconciling patterns of inter-ocean molecular variance from four classes of molecular markers in blue marlin (Makaira nigricans). Mol. Ecol. 10(5): 1179-1196. [ Links ]
Buonaccorsi VP, Kimbrell C, Lynn EA, Vetter RD. 2002. Population structure of copper rockfish (Sebastes caurinus) reflects postglacial colonization and contemporary patterns of larval dispersal. Can. J. Fish. Aquat. Sci. 59(8): 1374-1384. [ Links ]
Cimmaruta R, Iaconelli M, Nascetti G, Bullini L. 1998. Genetic diversity in Mediterranean large pelagic fish populations. Biol. Mar. Mediterr. 5(3): 300-310. [ Links ]
Cockerham CC, Weir BS. 1993. Estimation of gene flow from F- statistics. Evolution 47(3): 855-863. [ Links ]
Dawson MN, Staton JL, Jacobs DK. 2001. Phylogeography of the tidewater goby, Eucyclogobius newberryi (Teleostei, Gobiidae), in coastal California. Evolution 55(6): 1167-1179. [ Links ]
Deudero S, Merella P, Morales-Nin B, Massutí E, Alemany F. 1999. Fish communities associated with FADs. Sci. Mar. 63 (3-4): 199-207. [ Links ]
DeWoody JA, Avise JC. 2000. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. J. Fish Biol. 56(3): 461-473. [ Links ]
Ely B, Viñas J, Alvarado-Bremer JR, Black D, Lucas L, Covello K, Labrie AV, Thelen E. 2005. Consequences of the historical demography on the global population structure of two highly migratory cosmopolitan marine fishes: the yellowfin tuna (Thunnus albacares) and the skipjack tuna (Katsuwonus pelamis). BMC Evol. Biol. 5(19): doi: 10.1186/1471-2148-1185-1119. [ Links ]
Excoffier L, Smouse PE, Quattro JM. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics 131(2): 479-191. [ Links ]
García-Melgar CG. 1995. Ciclo de reproducción del dorado Coryphaena hippurus (Linnaeus 1758, Pisces: Coryphaenidae) en el área de los Cabos, BCS, México. Tesis de licenciatura, Universidad Autónoma de Baja California Sur México. [ Links ]
Gold JR, Richardson LR. 1998. Mitochondrial DNA diversification and population structure in fishes from the Gulf of Mexico and western Atlantic. J. Hered. 89(5): 404-414. [ Links ]
Graves JE. 1996. Conservation genetics of fishes in the pelagic marine realm. In: Avise JC, Hamrick JL (eds.), Conservation Genetics: Case Histories from Nature. Chapman & Hall, New York, pp. 335-366. [ Links ]
Guo S, Thompson E. 1992. Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics 48: 361-372. [ Links ]
Gyllensten U. 1985. The genetic structure of fish: Differences in the intraspecific distribution of biochemical genetic variation between marine, anadromous and freshwater fishes. J. Fish Biol. 26: 691-699. [ Links ]
Harper DE, Bohnsack JA, Lockwood BR. 2000. Recreational fisheries in Biscayne National Park, Florida, 1976-1991. Mar. Fish. Rev. 62(1): 8-24. [ Links ]
Herzig CE. 1990. Mitochondrial DNA used to track the dispersal of a preferred menu item in Waikiki restaurants: Mahimahi as an example of genetic tagging with mtDNA. Pac. Sci. 44(2): 186-187. [ Links ]
Jorgensen HBH, Hansen MM, Bekkevold D, Ruzzante DE, Loeschcke V. 2005. Marine landscapes and population genetic structure of herring (Clupea harengus L.) in the Baltic Sea. Mol. Ecol. 14(10): 3219-3234. [ Links ]
Kingsford MJ, Defries A. 1999. The ecology of and fishery for Coryphaena spp. in the waters around Australia and New Zealand. Sci. Mar. 63(3-4): 267-275. [ Links ]
Klett V, Meyer A. 2002. What, if anything, is a Tilapia? Mitochondrial ND2 phylogeny of tilapiines and the evolution of parental care systems in the African cichlid fishes. Mol. Biol. Evol. 19(6): 865-883. [ Links ]
Kocher TD, Conroy JA, McKaye KR, Stauffer JR Jr., Lockwood SF. 1995. Evolution of NADH dehydrogenase subunit 2 in East African cichlid fish. Mol. Phylogenet. Evol. 4(4): 420-432. [ Links ]
Kraul S. 1999. Seasonal abundance of the dolphinfish, Coryphaena hippurus, in Hawaii and the Tropical Pacific Ocean. Sci. Mar. 63(3-4): 261-266. [ Links ]
Kumar S, Tamura K, Jakobsen IB, Nei M. 2001. MEGA 2: Molecular Evolutionary Genetic Analysis software. Bioinformatics 17(12): 1244-1245. [ Links ]
Lasso J, Zapata L. 1999. Fisheries and biology of Coryphaena hippurus (Pisces: Coryphaenidae) in the Pacific coast of Colombia and Panama. Sci. Mar. 63 (3-4): 387-399. [ Links ]
Lee CS, Ostrowski AC. 2001. Current status of marine finfish larviculture in the United States. Aquaculture 200(1-2): 89-109. [ Links ]
Lopez JV, Culver M, Stephens JC, Johnson WE, O'Brien SJ. 1997. Rates of nuclear and cytoplasmic mitochondrial DNA sequence divergence in mammals. Mol. Biol. Evol. 14(3): 277-286. [ Links ]
Massutí E, Morales-Nin B. 1995. Seasonality and reproduction of dolphin-fish (Coryphaena hippurus) in the western Mediterranean. Sci. Mar. 59(3-4): 357-364. [ Links ]
Massutí E, Morales-Nin B, Moranta J. 1999. Otolith microstructure, age, and growth patterns of dolphin, Coryphaena hippurus, in the western Mediterranean. Fish. Bull. 97(4): 891-899. [ Links ]
Mateos M, Sanjur OI, Vrijenhoek RC. 2002. Historical biogeography of the livebearing fish genus Poeciliopsis (Poeciliidae: Cyprinodontiformes). Evolution 56(5): 972-984. [ Links ]
McElroy D, Moran P, Bermingham E, Kornfield I. 1992. The restriction enzyme analysis package (REAP). Heredity 83: 157-158. [ Links ]
Nei M. 1987. Molecular Evolutionary Genetics. Columbia Univ. Press, New York, 512 pp. [ Links ]
Nei M, Tajima F. 1981. DNA polymorphism detectable by restriction endonucleases. Genetics 97: 145-163. [ Links ]
Nei M, Miller J. 1990. A simple method for estimating average number of nucleotide substitutions in and between populations from restriction data. Genetics 125: 873-879. [ Links ]
Oxenford HA. 1999. Biology of the dolphinfish (Coryphaena hippurus) in the western central Atlantic: A review. Sci. Mar. 63(3-4): 277-301. [ Links ]
Oxenford HA, Hunte W. 1986. A preliminary investigation of the stock structure of the dolphin, Coryphaena hippurus, in the western central Atlantic. Fish. Bull. 84(2): 451-460. [ Links ]
Oxenford HA, Hunte W. 1999. Feeding habits of the dolphinfish (Coryphaena hippurus) in the eastern Caribbean. Sci. Mar. 63(3- 4): 303-315. [ Links ]
Palumbi SR. 2003. Population genetics, demographic connectivity, and the design of marine reserves. Ecol. Appl. 13(1): S146-S158. [ Links ]
Patton JC, Gallaway BJ, Fechhelm RG, Cronin MA. 1997. Genetic variation of microsatellite and mitochondrial DNA markers in broad whitefish (Coregonus nasus) in the Colville and Sagavanirktok rivers in northern Alaska. Can. J. Fish. Aquat. Sci. 54(7): 1548-1556. [ Links ]
Pla C, Pujolar JM. 1999. Genetic homogeneity of dolphinfish (Coryphaena hippurus) in the western Mediterranean and the eastern Atlantic. Sci. Mar. 63(3-4): 337-341. [ Links ]
Pujolar JM, Roldan MI, Pla C. 2003. Genetic analysis of tuna populations, Thunnus thynnus thynnus and T. alalunga. Mar. Biol. 143(3): 613-621. [ Links ]
Raymond M, Rousset F. 1995. GENEPOP (version 1.2): Population genetics software for exact tests and ecumenicism. J. Hered. 86: 248-249. [ Links ]
Reeb CA, Arcangeli L, Block BA. 2000. Structure and migration corridors in Pacific populations of the swordfish Xiphias gladius, as inferred through analyses of mitochondrial DNA. Mar. Biol. 136(6): 1123-1131. [ Links ]
Rivera GA, Appeldoorn RS. 2000. Age and growth of dolphinfish, Coryphaena hippurus, off Puerto Rico. Fish. Bull. 98(2): 345-352. [ Links ]
Saitou N, Nei M. 1987. The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol. 4: 406-425. [ Links ]
Sandoval-Castillo JR, Rocha-Olivares A, Villavicencio-Garayzar C, Balart E. 2004. Cryptic isolation of Gulf of California shovelnose guitarfish evidenced by mitochondrial DNA. Mar. Biol. 145(5): 983-988. [ Links ]
Schneider S, Roessli D, Excoffier L. 1999. Arlequin ver 2.0: A software for population genetic data analysis. Genetics and Biometry Lab, Dept. of Anthropology, University of Geneva, Switzerland. [ Links ]
Slatkin M. 1995. A measure of population subdivision based on microsatellite allele frequencies. Genetics 139(1): 457-462. [ Links ]
Smith PJ, Fujio Y. 1982. Genetic variation in marine teleosts: High variability in habitat specialists and low variability in habitat generalists. Mar. Biol. 69(1): 7-20. [ Links ]
Taylor MS, Hellberg ME. 2003. Genetic evidence for local retention of pelagic larvae in a Caribbean reef fish. Science 299 (5603): 107-109. [ Links ]
Toline CA, Baker AJ. 1995. Mitochondrial DNA variation and population genetic structure of the northern redbelly dace (Phoxinus eos). Mol. Ecol. 4(6): 745-753. [ Links ]
Ward RD, Woodwark M, Skibinski DOF. (1994). A comparison of genetic diversity levels in marine, freshwater, and anadromous fishes. J. Fish Biol. 44(2): 213-232. [ Links ]
Wingrove RS. 2000. The population structure of dolphin, Coryphaena hippurus L. 1758, in the western central Atlantic, Gulf of Mexico and eastern Caribbean Sea inferred from mitochondrial DNA variation. M.Sc. thesis, Department of Biology, College of Charleston. [ Links ]














