Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Ciencias marinas
versão impressa ISSN 0185-3880
Cienc. mar vol.29 no.2 Ensenada Jun. 2003
Artículos
Diversidad mitocondrial y estructura genética en poblaciones alopátricas del huachinango del Pacífico Lutjanus peru
Mitochondrial diversity and genetic structure in allopatric populations of the Pacific red snapper Lutjanus peru
Axayácatl Rocha-Olivares1 y Jonathan R. Sandoval-Castillo1, 2
1 Departamento de Ecología, Centro de Investigación Científica y de Educación Superior de Ensenada, Apartado postal 2732, Ensenada, CP 22860, Baja California, México. E-mail: arocha@cicese.mx
2 Departamento de Biología Marina, Universidad Autónoma de Baja California Sur, Apartado postal 19B, La Paz, CP 23080, Baja California Sur, México.
Recibido en julio de 2002;
aceptado en enero de 2003.
Resumen
Analizamos la diversidad genética y la estructura de tres poblaciones alopátricas del huachinango del Pacífico Lutjanus peru (Nichols y Murphy, 1922) mediante análisis de fragmentos de restricción de toda la región de control mitocondrial (1350 pb). Encontramos altos niveles de diversidad haplotípica (h = 0.966) y nucleotídica (π = 3.23%) en los 100 organismos analizados provenientes de las costas de Baja California Sur, Sinaloa y Jalisco (México). La mayor fracción de la variabilidad fue detectada con sólo una de las cinco enzimas de restricción utilizadas (MseI). El análisis de heterogeneidad de las frecuencias haplotípicas no fue significativo (X2 = 125.1, P > 0.25); los valores de los índices de fijación de Wright (FST = 0.0062, P = 0.140) y su análogo molecular (ΦST = 0.0194, P = 0.056) tampoco lo fueron. Un análisis de variancia molecular (AMOVA) corroboró la ausencia de diferenciación entre poblaciones peninsulares y continentales, aunque arrojó un ΦST marginalmente significativo (ΦST = 0.0116, P = 0.048). No se detectó ningún patrón filogeográfico en un árbol génico reconstruido por Neighbor-Joining. Argumentamos que los altos niveles de diversidad genética molecular son consistentes con la existencia de un tamaño poblacional importante, mientras que la ausencia de estructura genética en el extremo norte de distribución de la especie sugiere la acción de mecanismos pasivos de transporte larvario entre poblaciones alopátricas, aunque la migración activa de organismos adultos no puede ser descartada del todo.
Palabras clave: huachinango, ADN mitocondrial, estructura poblacional, diversidad genética molecular, AMOVA.
Abstract
We analyzed the genetic diversity and structure of three allopatric populations of the Pacific red snapper Lutjanus peru (Nichols and Murphy, 1922) by means of restriction fragment length polymorphisms of the entire mitochondrial control region (1350 bp). We found high levels of haplotype (h = 0.966) and nucleotide (π = 3.23%) diversities among the 100 organisms analyzed from Baja California Sur, Sinaloa and Jalisco (Mexico). Most of the molecular variability was detected with only one of the five restriction enzymes used (MseI). Heterogeneity analysis of haplotype frequencies was not significant (X2 = 125.1, P > 0.25); neither were Wright's fixation index (FST = 0.0062, P = 0.140) nor its molecular analogue (Φst = 0.0194, P = 0.056). An analysis of molecular variance (AMOVA) confirmed the absence of differentiation between peninsular and continental populations, even though it produced a marginally significant Φst (Φst = 0.0116, P = 0.048). No phylogeographic pattern was detected in a Neighbor-Joining gene tree. We argue that high molecular diversity levels are consistent with a large population size and that the absence of an apparent genetic structure, in the extreme north of the species distribution, suggests the action of mechanisms favoring passive larval transport among allopatric populations; although active adult migration cannot be ruled out.
Key words: mitochondrial DNA, population structure, Pacific red snapper, molecular genetic diversity, AMOVA.
Introducción
El manejo sustentable de cualquier especie requiere del conocimiento de su estructura poblacional puesto que variaciones en la dinámica poblacional y demográfica dentro de su ámbito geográfico repercuten en su explotabilidad y sustentabilidad (Sinclair, 1988). La utilización de genética molecular para estimar el grado de aislamiento reproductivo entre poblaciones ha permitido tener un acceso más directo a la diferenciación intraespecífica heredable y evolutivamente relevante (Grant et al., 1999). Este tipo de información también ha dado origen a un nuevo concepto de unidad biológica: la Unidad Evolutivamente Significativa (UES), que se define como una población o grupo de poblaciones que se encuentran aisladas reproductivamente de otras y que representan un componente importante del legado evolutivo de la especie (Waples, 1995). Debido a sus características evolutivas que incluyen, entre otras, una alta tasa de sustitución nucleotídica, una herencia clonal matrilineal y la ausencia de recombinación (Avise, 1994), el ADN mitocondrial (ADNmt) ha cobrado un papel preponderante en estudios de genética intraespecífica de especies con prioridades de manejo y conservación. En México, los estudios de genética poblacional molecular se hacen cada vez más urgentes en la medida que el número de organismos marinos afectados por un manejo inadecuado aumenta a nivel nacional (Cisneros-Mata et al., 1995; Rosel y Rojas-Bracho, 1999; D'Agrosa et al., 2000). En este trabajo presentamos los primeros datos sobre la diversidad genética mitocondrial y diferenciación de poblaciones alopátricas de un pez óseo activamente explotado en el litoral del Pacífico mexicano: el huachinango del Pacífico Lutjanus peru (Nichols y Murphy, 1922).
Lutjanus peru se distribuye desde el norte de Perú hasta el sur del Estado de Baja California Sur, México (Allen, 1987). Son depredadores generalistas demersales que se alimentan principalmente durante la noche de crustáceos, moluscos, peces y urocordados (Allen, 1985; Díaz-Uribe y Elorduy-Garay, 1994; Santamaría, 1998). Habitan aguas tropicales a profundidades relativamente someras hasta por lo menos 90 m (Allen y Robertson, 1994). Su crecimiento es lento, alométrico y similar entre ambos sexos, llegando a alcanzar hasta 99 cm de longitud total (LT) (González-Ochoa, 1997; Rocha-Olivares, 1998); son gonocóricos y maduran entre los 25 y 40 cm LT, presentando generalmente una proporción de sexos cercana a 1 y más de un periodo de desove anual (Cruz-Romero et al., 1991; Reyna-Trujillo, 1994; Santamaría, 1998). Allen (1987) menciona que los adultos y juveniles de L. peru son esencialmente sedentarios, sin extensas migraciones y que están típicamente asociados con sustratos rocosos o coralinos. Sin embargo, aunque algunos estadios larvarios se han descrito recientemente (Watson y Brogan, 1996), no se cuenta con una descripción completa de su ciclo de vida y se desconoce totalmente la ecología larvaria de la especie. De acuerdo con las estadísticas oficiales, la producción pesquera de huachinango en el Pacífico mexicano ha oscilado entre 1739 y 4917 toneladas anuales durante los últimos 30 años (1968 a 1998), siendo de gran importancia las capturas realizadas en Baja California Sur, Jalisco y Sinaloa (Cruz-Romero et al., com. pers.). Al igual que otras especies demersales que durante su desarrollo comparten el hábitat con crustáceos penéidos, los juveniles del huachinango forman parte de la fauna acompañante en la pesca de camarón (Rocha-Olivares, 1998).
Si bien los datos biológicos existentes sobre L. peru sugieren una diferenciación fenotípica a lo largo del litoral mexicano, se carece totalmente de información genética para un manejo adecuado del recurso. Esta información es necesaria tanto para poder interpretar los datos biológicos y ecológicos desde una perspectiva evolutiva, como para identificar y comprender qué factores bióticos y abióticos determinan la estructura poblacional de la especie.
Materiales y métodos
Muestreo
Los muestreos se realizaron en los estados de Jalisco (enero 2000), Sinaloa (mayo 2000) y Baja California Sur (junio 2000), que sustentan una importante pesquería de la especie. Las muestras provinieron de la pesca comercial del recurso que se realiza en Barra de Navidad, Jalisco (n = 10), Mazatlán, Sinaloa (n = 45), y La Paz, Baja California Sur (n = 45). De cada organismo se extrajeron el corazón y un fragmento de hígado, evitando la contaminación cruzada entre muestras individuales. Los tejidos se preservaron en etanol (95%) y se refrigeraron hasta su procesamiento en el laboratorio.
Extracción y purificación de ADN
El ADN genómico total se extrajo de ca 10 mg de tejido que se maceraron e incubaron durante la noche a temperatura ambiente en 500 µL de DNAzol (Molecular Research Center, Inc.) y 100 µg de proteinasa K (Gibco BRL). Se siguió el protocolo del fabricante para la purificación del ADN, el cual se precipitó con 250 µL y se enjuagó con 800 µL de etanol al 100% y 75%, respectivamente. El ADN purificado se redisolvió en 50 µL de amortiguador TE (10 mM Tris-HCl, 1 mM EDTA, pH 8.0). La cantidad y calidad del ADN extraído se estimaron visualmente en geles de agarosa al 1% (0.5 x TBE) teñidos con bromuro de etidio (0.25 µg/L).
Amplificación de ADN
Para cada organismo se amplificó un fragmento del ADNmt que incluye la totalidad de la región de control, abarcando parte del gen del ARN de transferencia de prolina (ARNtPro) hasta el inicio del gen que codifica la subunidad pequeña del ARN ribosomal mitocondrial (12S ADNr). Se utilizaron promotores universales degenerados ProDG (YYACCNNYRRCNCCCAAAGC) y 12Sar (AAACTRGGATTAGATACCCYAYTAT) (Palumbi, 1996) en reacciones de 25 ó 50 µL (180 µM dNTPs, 3.5 mM MgCl2, 200 mM cada promotor, amortiguador de PCR PROMEGA, 1.52.0 U Taq DNApol PROMEGA). En todos los experimentos se incluyeron controles negativos para verificar la ausencia de contaminación. Se utilizó un termociclador Perkin-Elmer 480 con una desnaturalización inicial de 7 min a 80°C, seguida de 35 ciclos con el siguiente perfil: 15 seg a 95°C, 45 seg a 45°C, 90 seg a 72°C, y una extensión final de 5 min a 72°C. Los productos de la reacción se verificaron en geles de agarosa al 1% teñidos con bromuro de etidio (0.25 µg/L).
Digestión con enzimas de restricción (RFLP)
Los productos de PCR fueron digeridos con cinco enzimas de restricción (Hsp92II, SspI, CfoI, HaeIII y MseI) con secuencias de reconocimiento de cuatro y seis bases. Las enzimas se eligieron por ser polimórficas en secuencias ortólogas de tres organismos recolectados en Oaxaca que no se incluyen en el estudio. Se digirieron 5 µL del producto de PCR siguiendo las indicaciones del fabricante y los fragmentos fueron separados electroforéticamente en geles de agarosa al 2%. Para los productos digeridos con MseI se utilizaron geles de poliacrilamida (29:1) al 8%. Los geles fueron teñidos con bromuro de etidio (0.25 µg/L) y posteriormente fotografiados y digitalizados para su análisis cuantitativo. Los patrones de restricción obtenidos de las enzimas se combinaron en un haplotipo compuesto para cada organismo.
Análisis de datos
La localización de los sitios de restricción (mapeo de sitios) se hizo con la ayuda de secuencias de ADN de tres organismos de Oaxaca. Se estimaron distancias genéticas interhaplotípicas a partir de la presencia o ausencia de sitios de restricción de acuerdo con Nei y Miller (1990, ec. 4). Los valores de diversidad genética haplotípica y nucleotídica se estimaron de acuerdo con Nei (1987, ec. 8.4 y 10.14) y la divergencia nucleotídica se calculó basándose en la tasa de sustitución de nucleótidos por sitio por haplotipo según el modelo de Nei y Tajima (1981, ec. 22). La homogeneidad en la distribución de los haplotipos se probó utilizando el estadístico x2 y su significancia se estimó vía 1000 simulaciones Monte Carlo (Roff y Bentzen, 1989). Estos análisis se realizaron con el programa REAP 4.0 (McElroy et al., 1992). Se calcularon índices de fijación entre las regiones como FST (Wright, 1978) y su análogo molecular Φst, incorporando una matriz de distancias euclideanas interhaplotípicas (Excoffier et al., 1992). Finalmente, se realizó un análisis de variancia molecular (AMOVA, Excoffier et al., 1992) para estimar la variabilidad genética molecular explicada por distintos niveles jerárquicos de organización (e.g., dentro de poblaciones, entre poblaciones dentro de una región y entre regiones). Estos últimos análisis se realizaron con el programa Arlequín 2.0 (Schneider et al., 1999) en el que la significancia estadística de los índices se calculó mediante métodos exactos (Guo y Thompson, 1992). Se reconstruyó un árbol filogenético Neighbor-Joining (NJ, Saitou y Nei, 1987) con las distancias de Nei y Tajima con el programa Mega 2.1 (Kumar et al., 2001). La raíz del árbol se infirió utilizando el haplotipo compuesto generado de la secuencia ortóloga de L. campechanus (GenBank AF239677) como grupo de referencia (outgroup).
Resultados
Diversidad molecular
El tamaño del fragmento amplificado fue de 1350 pares de bases. Todas las enzimas fueron polimórficas y los sitios de restricción por enzima variaron de 2 en SspI hasta 22 en MseI, encontrándose un total de 41 patrones de restricción. Se observaron un total de 60 haplotipos compuestos entre los 100 organismos analizados (tabla 1), de los cuales 31 se presentaron en Sinaloa, 32 en Baja California Sur (BCS), y 9 en Jalisco. Los haplotipos dominantes (1 y 2 en tabla 1) se encontraron en el 26% de los organismos; en BCS predominó el número 2 mientras que en Sinaloa ambos fueron abundantes (tabla 1). Sinaloa y BCS presentaron casi el mismo número de haplotipos únicos, mientras que sólo 2 de los 60 haplotipos fueron encontrados en las tres localidades. Las diversidades medias haplotípica (0.966) y nucleotídica (3.23%) fueron elevadas, presentando Jalisco los valores más altos y Sinaloa los más bajos (tabla 2). El número limitado de muestras procedentes de Jalisco obliga a interpretar con cautela las estimaciones de diversidad de esta población.

Diferenciación genética
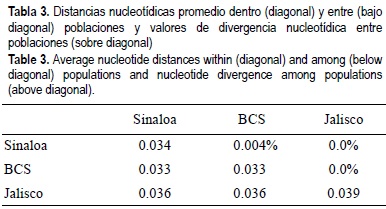
La divergencia entre poblaciones fue muy baja, alcanzando tan solo un máximo de 0.004% entre Sinaloa y BCS (tabla 3). Las distancias medias interhaplotípicas dentro de cada localidad promediaron 0.036, incluyendo a Jalisco, y fueron muy similares en Sinaloa y BCS (0.034 y 0.033, respectivamente), consecuentemente las distancias medias entre ambas localidades y Jalisco fueron iguales (0.036, tabla 3).
La prueba de homogeneidad de las frecuencias haplotípicas no fue significativa (x2 = 125.1, P > 0.25), por lo que no se rechazó la hipótesis nula de homogeneidad entre las poblaciones. El índice de fijación sugiere que sólo el 0.6% de la variabilidad genética haplotípica se encuentra distribuida entre las poblaciones y no es significativamente distinto de cero (FST = 0.0062, P = 0.140). Si bien la inclusión de la diferenciación molecular eleva dicha proporción a 1.9%, la diferenciación es marginalmente no significativa (Φst = 0.0194, P = 0.056). Cabe señalar que el tamaño de muestra de Jalisco limita el poder estadístico de las pruebas que la incluyen.
El AMOVA demostró que las fracciones de la variabilidad genética atribuibles a diferencias entre las dos poblaciones continentales y la peninsular, o a las poblaciones dentro de cada región, no son significativas (Φct = -0.052, P = 0.66; tabla 4a). Sin embargo, el índice Φst obtenido fue marginalmente significativo (Φst = 0.0116, P = 0.048). La exclusión de la muestra de Jalisco en el AMOVA corroboró la ausencia de diferenciación molecular entre los organismos de Sinaloa y BCS (Φst = 0.001, P = 0.36; tabla 4b), lo que confirma que la inclusión o exclusión de estos organismos (n = 10) no afecta el resultado principal de los análisis de estructura genética.
El árbol NJ construido con las distancias interhaplotípicas no muestra un patrón filogeográfico, al encontrarse los haplotipos de las tres localidades dispersos polifiléticamente en todos los linajes (fig. 1).

Discusión
Diversidad genética molecular
Los valores de diversidad haplotípica y nucleotídica mitocondrial de L. peru fueron altos. No obstante, puesto que diferentes regiones del ADNmt presentan distintas tasas de evolución (Randi, 2000), la comparación directa con otros resultados no es tan evidente. En principio, dichas comparaciones deberían de hacerse sobre la región de control ortóloga de otras especies. Además, la utilización de diferentes herramientas moleculares puede dar también diferentes valores de diversidad para una misma región del genoma. En el caso de los RFLPs, los valores de diversidad aumentan con el número y el tipo de enzimas utilizadas (Graves et al., 1992a), aunque esta relación no siempre se verifica ya que pueden encontrarse enzimas hipervariables como MseI para la región de control de L. peru. En general, la diversidad molecular del huachinango fue mayor que la observada en otros peces en RFLPs de toda la molécula del ADNmt (Graves et al., 1992a, 1992b; Grijalva-Chon et al., 1994; Gold et al., 1997) (tabla 5). Esto puede ser explicado por la diferencia entre la tasa de evolución media de todo el genoma mitocondrial y la de la región de control (Brown et al., 1993). Sin embargo, los valores de diversidad reportados en estudios en los que se ha utilizado todo el ADNmt tales como los de Gold et al. (1994), para tres especies de peces marinos, y los de Camper et al. (1993), para el huachinango del Golfo de México, son parecidos a los observados en ese trabajo. Cabe notar que, si bien la mayoría de los estudios en los que se ha utilizado exclusivamente la región control también han producido altas diversidades (e.g., Graves y McDowell, 1994; Chow et al., 1997; Tabata y Mizuta, 1997), en ellos se ha usado un mayor número de enzimas de restricción (8 a 13), por lo que los valores aquí reportados obtenidos con cinco enzimas pueden ser considerados excepcionalmente altos. En efecto, valores de magnitud comparable son más típicamente obtenidos a partir de secuenciación directa de toda la región de control (e.g., Rocha-Olivares et al., 2000).
Tanto la diversidad haplotípica como la nucleotídica están afectadas por eventos demográficos históricos y contemporáneos. Las distintas combinaciones de valores altos y bajos de ambas diversidades pueden ser indicativas de características de la historia de vida de las poblaciones. Altos valores en ambas diversidades, como las encontradas para L. peru, pueden ser atribuibles a un contacto secundario entre linajes alopátricos previamente diferenciados, o bien a una gran población estable con una larga historia evolutiva (Grant y Bowen, 1998). La alta diversidad nos permite suponer un tamaño poblacional efectivo considerable, ya que la variabilidad de la molécula de ADNmt es muy sensible a fenómenos como cuellos de botella, y en poblaciones de tamaño efectivo pequeño la variabilidad suele ser muy baja (Avise et al., 1984). La inferencia de un tamaño poblacional considerable es congruente con lo reportado por Espino-Barr et al. (1998) y Cruz-Romero et al. (2001, com. pers.) a partir de análisis cuantitativos de datos pesqueros.
Estructura poblacional
Para la mayoría de los peces pelágicos que han sido estudiados no se ha encontrado heterogeneidad entre poblaciones alopátricas (e.g., Graves et al., 1984; Graves y Dizon, 1989; Scoles y Graves, 1993; Grijalva-Chon et al., 1994; Toline y Baker, 1994). Esto se ha relacionado con una alta capacidad de dispersión (Graves, 1998). Si bien será necesario incrementar tanto el tamaño de algunas muestras como el número de poblaciones estudiadas para corroborar qué tan generalizables son nuestros hallazgos a lo largo de la costa del Pacífico mexicano, la ausencia de heterogeneidad encontrada en L. peru no concuerda con la observación de que adultos y juveniles son esencialmente sedentarios, sin extensas migraciones y que están típicamente asociados con sustratos específicos (Allen, 1985, 1987; Allen y Robertson, 1994; Díaz-Uribe y Elorduy-Garay, 1994). En el caso del huachinango del Golfo de México, que es ecológicamente similar, también se ha encontrado una homogeneidad genética (Camper et al., 1993; Gold et al., 1994, 1997). La homogeneidad observada puede ser explicada de dos maneras:
1. A pesar de que la falta de una estructura genética ha sido tomada como un resultado de la existencia de un flujo genético contemporáneo, el grado de diferenciación entre poblaciones no sólo depende del aislamiento reproductivo, sino también es influenciado por procesos evolutivos como la selección natural, la deriva génica, la tasa de mutación y el tiempo de aislamiento (Chakraborty y Leimar, 1987). A pesar de existir una alta variabilidad dentro de poblaciones reproductivamente aisladas, el establecimiento de divergencias suele ser complejo, porque la estructura social y geográfica así como el tamaño de la población reproductiva pueden afectar el tiempo de diversificación genética de los linajes, manteniendo la homogeneidad entre poblaciones aisladas por largo tiempo (Hoelzel, 1998). Por tanto, la falta de heterogeneidad entre las poblaciones puede deberse a un aislamiento muy reciente.
2. Más de un aspecto de la historia de vida de la especie promueve el flujo genético entre las poblaciones. Para organismos con estadios adultos y juveniles sedentarios, el transporte pasivo de huevos y larvas por advección puede ser un mecanismo importante para el flujo genético de la especie (Shaklee y Samollow, 1984).
Aunque diversos estudios genéticos poblacionales en especies con estadios larvarios pelágicos variables no han encontrado una relación clara, se ha propuesto que la estructura poblacional de los peces marinos se encuentra ligada a la duración del estadio larvario pelágico (PLD, por sus siglas en inglés), variando la heterogeneidad interpoblacional de manera inversa a la duración del PLD (Doherty et al., 1995). Aunque hay muy poca información sobre los estadios tempranos de L. peru, existen datos para la familia Lutjanidae (Leis, 1987). Las larvas meroplanctónicas de lutjánidos son poco comunes en relación con las de otros grupos. Esto no necesariamente implica la ausencia de un estadio meroplanctónico prolongado, sino que puede explicarse por el hecho de que los adultos son menos abundantes que otros peces en las comunidades en las que son depredadores. Además, a pesar de que la mayoría de las larvas se encuentran en la plataforma continental y en la misma área que los adultos, se han reportado larvas de huachinango del golfo a 160 km de la costa en el Golfo de México, por lo que considerando que el periodo larvario en la familia Lutjanidae es de entre 25 y 47 días, tal vez las larvas de L. peru pueden dispersarse grandes distancias con las corrientes adecuadas.
La Corriente de California puede llegar hasta el sur de Sinaloa durante parte de la primavera y el verano (Badan, 1997), coincidiendo con el primer (de febrero a abril) y parte del segundo (de junio a octubre) desove masivo de L. peru en algunas poblaciones continentales (Santamaría, 1998). La Corriente Costera de Costa Rica entra al sur del Golfo de California todo el año, pero recorre parte de la costa Pacífico de la península sólo durante el otoño y el invierno (Badan, 1997), coincidiendo con parte del segundo (de junio a octubre) desove masivo de L. peru (Santamaría, 1998). Si bien estas corrientes pueden ayudar al transporte pasivo de larvas y huevos tanto del continente a la península como en sentido contrario, la contribución de la migración activa de adultos al flujo genético entre poblaciones alopátricas no puede ser totalmente descartada y requiere de estudios adicionales.
Patrón filogeográfico
Aunque algunos linajes mitocondriales se encuentran dominados por haplotipos de alguna de las poblaciones (fig. 1), los bajos valores de divergencias interpoblacionales nos permiten suponer que las poblaciones no se encuentran aisladas y no presentan un alto potencial para ser heterogéneas en un corto periodo de tiempo evolutivo (poseen un bajo o nulo potencial evolutivo). Esto contrasta con lo reportado por Arnason et al. (1992), quienes a pesar de no haber encontrado heterogeneidad entre poblaciones de bacalao, sí observaron un linaje relacionado con la distribución geográfica, así como altos valores de distancia y divergencia nucleotídica. Por ello infirieron que la población de dicho linaje ha estado aislada por sólo un corto periodo de tiempo, pero presenta un alto potencial evolutivo.
Relación genotipo-fenotipo
La ausencia de diferenciación genética en las poblaciones estudiadas concuerda con similitudes fenotípicas en algunas características biológicas, como las tasas de crecimiento instantáneo reportadas por Cruz-Romero et al. (1991) en Colima (K = 0.15) y Rocha-Olivares (1998) en BCS (K = 0.13); aunque ambas difieren de la reportada por González-Ochoa (1997) en Jalisco (K = 0.091). Los picos reproductivos son más similares y se extienden de febrero a mayo y de julio a noviembre en Guerrero (Santamaría, 1998); de febrero a mayo y de julio a diciembre en BCS (Reyna-Trujillo, 1994); y de febrero a abril y de julio a noviembre en Colima (Cruz-Romero et al., 1991). No obstante, existen diferencias en las tallas máximas reportadas por Cruz-Romero et al. (1991) en Colima (60 cm), González-Ochoa (1997) en Jalisco (70 cm) y Rocha-Olivares (1998) en BCS (90 cm); así como en las tallas de primera madurez estimadas por Cruz-Romero et al. (1991) en Colima (25 a 30 cm), Santamaría (1998) en Guerrero (30 a 35 cm) y Rocha-Olivares (1998) en BCS (35 a 40 cm). Diferencias en poblaciones que presentan flujo genético son explicadas por una plasticidad fenotípica expresada en diferencias morfométricas, biológicas o demográficas, y que son inducidas por diferencias bióticas y abióticas en el hábitat (Allendorf et al. , 1987). Se obtendrá una mejor apreciación del grado de plasticidad fenotípica de la especie en la medida que se tenga una visión más completa de la estructura poblacional y se conozca mejor su biología a lo largo de su intervalo geográfico.
Agradecimientos
Agradecemos encarecidamente las facilidades brindadas para la obtención de muestras (H. Pérez-España, en Jalisco; H. Plascencia-González y A. van der Heiden, en Sinaloa; J.G. Díaz-Uribe y S. Ramírez-Luna, en BCS) y el acceso a equipo de laboratorio (J. Olmos-Soto, F.J. Márquez-Rocha y A. Licea-Navarro del CICESE, Ensenada, BC; J. de la Rosa-Vélez de la UABC, Ensenada, BC; R.D. Vetter, SWFSC/ NMFS, La Jolla EUA). Se aprecian las sugerencias de los revisores anónimos. La investigación fue financiada por CONACYT, proyecto I36064-V, y por CICESE. El trabajo se deriva de la tesis de licenciatura de J.R. Sandoval-Castillo (Mención Honorífica), egresado de la Universidad Autónoma de Baja California Sur, México.
Referencias
Allen, G.R. (1985). Snappers of the world: An annotated and illustrated catalogue of Lutjanid species known to date. FAO Species Catalog, 6. FAO Fish. Synop., 6(125): 208 pp. [ Links ]
Allen, G.R. (1987). Synopsis of the cicumtropical fish genus Lutjanus (Lutjanidae). In: J.J. Polovina and S. Ralston (eds.), Tropical Snappers and Groupers: Biology and Fisheries Management. Westview Press, Boulder, Colorado, pp. 33-87. [ Links ]
Allen, G.R. and Robertson, D.R. (1994). Fishes of the Tropical Eastern Pacific. Univ. Hawaii Press, Honolulu, 332 pp. [ Links ]
Allendorf, F.W., Ryman, N. and Utter, F.M. (1987). Genetics and fishery management: Past, present, and future. In: N. Ryman and F.M. Utter (eds.), Population Genetics and Fishery Management. Univ. Washington Press, Seattle, Washington, pp. 1-19. [ Links ]
Arnason, E., Palsson, S. and Arnason, A. (1992). Gene flow and lack of population differentiation in Atlantic cod (Gadus morhua) from Iceland and comparison of cod from Norway and Newfoundland. J. Fish Biol., 40: 751-770. [ Links ]
Avise, J.C. (1994). Molecular Markers, Natural History and Evolution. Chapman and Hall, New York, 511 pp. [ Links ]
Avise, J.C., Neigel, J.E. and Arnold, J. (1984). Demographic influences on mitochondrial DNA lineage survivorship in animal populations. J. Mol. Evol., 20(2): 99-105. [ Links ]
Badan, A. (1997). La Corriente Costera de Costa Rica en el Pacífico Mexicano. En: M.F. Lavín (ed.), Contribuciones a la Oceanografía Física en México, Monografía 3. Unión Geofísica Mexicana, Ensenada, Baja California, México, pp. 99-112. [ Links ]
Brown, J.R., Beckenbach, A.T. and Smith, M.J. (1993). Intraspecific DNA sequence variation of the mitochondrial control region of white sturgeon (Acipenser transmontanus). Mol. Biol. Evol., 10(2): 326-341. [ Links ]
Camper, J.D., Barber, R.C., Richardson, L.R. and Gold, J.R. (1993). Mitochondrial DNA variation among red snapper (Lutjanus campechanus) from the Gulf of Mexico. Mol. Mar. Biol. Biotechnol., 2(3): 154-161. [ Links ]
Chakraborty, R. and Leimar, O. (1987). Genetic variation within a subdivided population. In: N. Ryman and F.M. Utter (eds.), Population Genetics and Fishery Management. Univ. Washington Press, Seattle, Washington, pp. 89-120. [ Links ]
Chow, S., Okamoto, H., Uozumi, Y., Takeuchi, Y. and Takeyama, H. (1997). Genetic stock structure of the swordfish (Xiphias gladius) inferred by PCR-RFLP analysis of the mitochondrial DNA control region. Mar. Biol., 127(3): 359-367. [ Links ]
Cisneros-Mata, M.A., Montemayor-Lopez, G. and Roman-Rodríguez, M.J. (1995). Life-history and conservation of Totoaba macdonaldi. Conserv. Biol., 9(4): 806-814. [ Links ]
Cruz-Romero, M., Espino-Barr, E., Mimbela-López, J., García-Boa, A., Obregón-Alcaraz, L.F. y Girón-Botello, E. (1991). Biología reproductiva en tres especies del género Lutjanus en la costa de Colima, México. Secretaría de Pesca, INP, CRIP Manzanillo, Manzanillo, Colima, 118 pp. [ Links ]
D'Agrosa, C., Lennert-Cody, C.E. and Vidal, O. (2000). Vaquita bycatch in Mexico's artisanal gillnet fisheries: Driving a small population to extinction. Conserv. Biol., 14(4): 1110-1119. [ Links ]
Díaz-Uribe, J.G. y Elorduy-Garay, J.F. (1994). Análisis trofodinámico del huachinango (Lutjanus peru) en las bahías de La Paz y La Ventana, BCS, México. Tesis de maestria, CICESE, México, 57 pp. [ Links ]
Doherty, P.J., Planes, S. and Mather, P. (1995). Gene flow and larval duration in seven species of fish from the Great Barrier Reef. Ecology (Washington, DC), 76(8): 2373-2391. [ Links ]
Espino-Barr, E., Cruz-Romero, M. y García-Boa, A. (1998). Análisis de la captura y esfuerzo del huachinango Lutjanus peru en la costa de Colima, México (período 1983 a 1997). Memorias del VI Congreso de Ictiología. [ Links ]
Excoffier, L., Smouse, P.E. and Quattro, J.M. (1992). Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics, 131(2): 479-491. [ Links ]
Gold, J.R., Richardson, L.R., Furman, C. and Sun, F. (1994). Mitochondrial DNA diversity and population structure in marine fish species from the Gulf of Mexico. Can. J. Fish. Aquat. Sci., 51(Suppl. 1): 205-214. [ Links ]
Gold, J.R., Sun, F. and Richardson, L.R. (1997). Population structure of red snapper from the Gulf of Mexico as inferred from analysis of mitochondrial DNA. Trans. Am. Fish. Soc., 126(3): 386-396. [ Links ]
González-Ochoa, O.A. (1997). Edad y crecimiento de Lutjanus peru en la costa sur de Jalisco. Tesis de licenciatura, Universidad de Guadalajara, México, 80 pp. [ Links ]
Grant, W.S. and Bowen, B.W. (1998). Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation. J. Heredity, 89(5): 415-426. [ Links ]
Grant, W.S., García-Marin, J.L. and Utter, F.M. (1999). Defining population boundaries for fishery management. In: S. Mustafa (ed.), Genetics in Sustainable Fisheries Management. Fishing News Books, Blackwell Science, Oxford, UK, pp. 27-72. [ Links ]
Graves, J.E. (1998). Molecular insights into the population structures of cosmopolitan marine fishes. J. Heredity, 89(5): 427-137. [ Links ]
Graves, J.E. and Dizon, A.E. (1989). Mitochondrial DNA sequence similarity of Atlantic and Pacific albacore tuna (Thunnus alalunga). Can. J. Fish. Aquat. Sci., 46(5): 870-873. [ Links ]
Graves, J.E. and McDowell, J.R. (1994). Genetic analysis of striped marlin (Tetrapturus audax) population structure in the Pacific Ocean. Can. J. Fish. Aquat. Sci., 51(8): 1762-1768. [ Links ]
Graves, J.E., Ferris, S.D. and Dizon, A.E. (1984). Close genetic similarity of Atlantic and Pacific skipjack tuna (Katsuwonus pelamis) demonstrated with restriction endonuclease analysis of mitochondrial DNA. Mar. Biol., 79(3): 315-319. [ Links ]
Graves, J.E., McDowell, J.R., Beardsley, A.M. and Scoles, D.R. (1992a). Stock structure of the bluefish Pomatomus saltatrix along the mid-Atlantic coast. US Fish. Bull., 90(4): 703-710. [ Links ]
Graves, J.E., McDowell, J.R. and Jones, M.L. (1992b). A genetic analysis of weakfish Cynoscion regalis stock structure along the mid-Atlantic coast. US Fish. Bull., 90(3): 469-475. [ Links ]
Grijalva-Chon, J.M., Numachi, K., Sosa-Nishizaki, O. and De la Rosa-Vélez, J. (1994). Mitochondrial DNA analysis of North Pacific swordfish Xiphias gladius population structure. Mar. Ecol. Prog. Ser., 115(1-2): 15-19. [ Links ]
Guo, S. and Thompson, E. (1992). Performing the exact test of Hardy-Weinberg proportion for multiple alleles. Biometrics, 48: 361-372. [ Links ]
Hoelzel, A.R. (1998). Genetic structure of cetacean populations in sympatry, parapatry, and mixed assemblages: Implications for conservation policy. J. Heredity, 89(5): 451-458. [ Links ]
Kumar, S., Tamura, K., Jakobsen, I.B. and Nei, M. (2001). MEGA 2: Molecular Evolutionary Genetic Analysis software. Bioinformatics, 17(12): 1244-1245. [ Links ]
Leis, J.M. (1987). Review of the early life history of tropical groupers (Serranidae) and snappers (Lutjanidae). In: J.J. Polovina and S. Ralston (eds.), Tropical Snappers and Groupers: Biology and Fisheries Management. Westview Press, Boulder, Colorado, pp. 189-237. [ Links ]
McElroy, D., Moran, P., Bermingham, E. and Kornfield, I. (1992). The restriction enzyme analysis package (REAP). Heredity, 83: 157-158. [ Links ]
Nei, M., (1987). Molecular Evolutionary Genetics. Columbia University Press, New York. 512 pp. [ Links ]
Nei, M. and Miller, J. (1990). A simple method for estimating average number of nucleotide substitutions in and between populations from restriction data. Genetics, 125: 873-879. [ Links ]
Nei, M. and Tajima, F. (1981). DNA polymorphism detectable by restriction endonucleases. Genetics, 97: 145-163. [ Links ]
Palumbi, S.R. (1996). Nucleic acids II: The polymerase chain reaction. In: D.M. Hillis, C. Moritz and B.K. Mable (eds.), Molecular Systematics. 2nd ed. Sinauer Associates, Sunderland, Massachusetts, pp. 205-247. [ Links ]
Randi, E. (2000). Mitochondrial DNA. In: A.J. Baker (ed.), Molecular Methods in Ecology. Blackwell Science, Malden, pp. 136-167. [ Links ]
Reyna-Trujillo, M. (1994). Desarrollo gonádico y época de desove del huachinango (Lutjanus peru) en la bahía de La Paz, BCS, México. Tesis profesional, Universidad de Guadalajara, México, 73 pp. [ Links ]
Rocha-Olivares, A. (1998). Age, growth, mortality, and population characteristics of the Pacific red snapper, Lutjanus peru, off the southeast coast of Baja California, Mexico. US Fish. Bull., 96(3): 562-574. [ Links ]
Rocha-Olivares, A., Garber, N.M. and Stuck, K.C. (2000). High genetic diversity, large inter-oceanic divergence and historical demography of the striped mullet. J. Fish Biol., 57(5): 1134-1149. [ Links ]
Roff, D.A. and Bentzen, P. (1989). The statistical analysis of mitochondrial DNA polymorphisms: x2 and the problem of small samples. Mol. Biol. Evol., 6(5): 539-545. [ Links ]
Rosel, P.E. and Rojas-Bracho, L. (1999). Mitochondrial DNA variation in the critically endangered vaquita Phocoena sinus Norris and Macfarland, 1958. Mar. Mammal Sci., 15(4): 990-1003. [ Links ]
Saitou, N. and Nei, M. (1987). The neighbor-joining method: A new method for reconstructing phylogenetic trees. Mol. Biol. Evol., 4: 406-425. [ Links ]
Santamaría, A. (1998). Hábitos alimenticios y ciclo reproductivo del huachinango, Lutjanus peru (Nichols y Murphy, 1992) Pisces: Lutjanidae en Guerrero, México. Tesis de maestría, CICIMAR-IPN, México, 64 pp. [ Links ]
Schneider, S., Roessli, D. and Excoffier, L. (1999). Arlequin ver 2.0: A software for population genetic data analysis 2.0b1 edit. Genetics and Biometry Lab., Dept. of Anthropology, Univ. of Geneva, Switzerland. [ Links ]
Scoles, D.R. and Graves, J.E. (1993). Genetic analysis of the population structure of yellowfin tuna, Thunnus albacares, from the Pacific Ocean. US Fish. Bull., 91(4): 690-698. [ Links ]
Shaklee, J.B. and Samollow, P.B. (1984). Genetic variation and population structure in a deepwater snapper, Pristipomoides filamentosus, in the Hawaiian archipelago. US Fish. Bull., 82(4): 703-713. [ Links ]
Sinclair, M. (1988). Marine Population: An Essay on Population Regulation and Speciation. Univ. Washington, Seattle. 252 pp. [ Links ]
Tabata, K. and Mizuta, A. (1997). RFLP analysis of the mtDNA D-loop region in red sea bream Pagrus major population from four locations of western Japan. Fish. Sci., 63(2): 211-217. [ Links ]
Toline, C.A. and Baker, A.J. (1994). Genetic differentiation among populations of the northern redbelly dace (Phoxinus eos) in Ontario. Can. J. Fish. Aquat. Sci., 51(6): 1218-1228. [ Links ]
Waples, R.S. (1995). Evolutionarily significant units and the conservation of biological diversity under the Endangered Species Act. In: J.L. Nielsen (ed.), Evolution and the Aquatic Ecosystem: Defining Unique Units in Population Conservation. American Fisheries Soc. Symp. 17, Bethesda, Maryland, pp. 8-27. [ Links ]
Watson, W. and Brogan, M.W. (1996). Lutjanidae: Snappers. In: H.G. Moser (ed.), The Early Life Stages of Fishes in the California Current Region. CalCOFI Atlas 33. Allen Press, Lawrence, Kansas, pp. 977-989. [ Links ]
Wright, S. (1978). Evolution and the Genetics of Populations. Vol. 4. Variability within and among Natural Populations. Univ. Chicago Press, Chicago, 580 pp. [ Links ]














