Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Similares em
SciELO
Similares em
SciELO
Compartilhar
Revista mexicana de fitopatología
versão On-line ISSN 2007-8080versão impressa ISSN 0185-3309
Rev. mex. fitopatol vol.41 no.3 Texcoco Set. 2023 Epub 13-Out-2023
https://doi.org/10.18781/r.mex.fit.2305-4
Artículos científicos
Silenciamiento génico inducido por el vector EuMY-YP∆CHS4 durante la interacción Fusarium sp.-Nicotiana benthamiana
1 Unidad de Bioquímica y Biología Molecular de Plantas, Centro de Investigación Científica de Yucatán, Calle 43 No. 130 x 32 y 34, Chuburná de Hidalgo; CP 97205, Mérida, Yucatán, México;
2Escuela de Ciencias de la Salud- Universidad Marista de Mérida, Periférico Norte Tablaje Catastral 13941 Carretera Mérida-Progreso, C.P. 97300 Mérida, Yucatán, México;
3Unidad de Biotecnología, Centro de Investigación Científica de Yucatán, Calle 43 No. 130 x 32 y 34, Chuburná de Hidalgo; CP 97205, Mérida, Yucatán, México
4Unidad de Ciencias del Agua, Centro de Investigación Científica de Yucatán, A.C. Calle 8 No 39 SM 64 Mz 29 77500 Cancún, Quintana Roo, México;
5Investigadora por México-CONAHCYT, Red de Estudios Moleculares Avanzados, Instituto de Ecología, A.C. Carretera antigua a Coatepec 351, Col. El Haya, CP 91070, Xalapa, Veracruz, México;
En la literatura, se han propuesto métodos basados en el ARN de interferencia para el control de hongos patógenos en plantas, como el silenciamiento mediado por virus (VIGS). En este trabajo se evaluó la funcionalidad y la eficiencia del vector VIGS EuMV-YP∆CHS4, el cual contiene un fragmento de la secuencia del gen de la quitina sintasa 4 (CHS4) de Fusarium sp. y fue construido a partir del vector EuMV-YP:∆AV1. Se evaluó el vector VIGS en dos condiciones en plantas de Nicotiana benthamiana: plantas con inoculación previa con Fusarium sp. y en plantas con inoculación del hongo posterior a la inoculación del vector VIGS. Los resultados mostraron que la aplicación de EuMV-YP∆CHS4 en ambos tratamientos provocó un retardo en la caída de hojas durante los primeros días de la infección y una disminución significativa del daño en el tallo de las plantas infectadas, comparadas con el control infectado con el hongo. Además, se observó una reducción de hasta 30% en la expresión relativa del gen CHS4. Estos resultados sugieren que la aplicación de los vectores VIGS puede ser una estrategia eficiente para proteger a las plantas contra Fusarium sp.
Palabras clave: ARN de silenciamiento; Euphorbia mosaic virus-Yucatán península; entre reinos “cross-kingdom”
In the literature, RNA-based interference methods have been proposed for the control of pathogenic fungi in plants, such as virus-mediated silencing (VIGS). The objective of this work was to evaluate the functionality and efficiency of the VIGS EuMV-YP∆CHS4 vector, which contains a fragment of the Fusarium sp. chitin synthase 4 (CHS4) gene sequence, and was constructed from the EuMV-YP vector:∆AV1. The VIGS vector was evaluated under two conditions in Nicotiana benthamiana plants previously inoculated with Fusarium sp. and in plants with inoculation after the inoculation of the VIGS vector. The results showed that in both treatments with the EuMV-YP∆CHS4, a delay in leaf fall during the first days of infection and a decrease in the stem damage of almost one centimeter, compared to the control infected with the fungus. In addition, a reduction of up to 30% in the relative expression of the CHS4 gene was observed. These results suggest that the application of VIGS vectors may be efficient as a strategy to protect plants against Fusarium sp.
Keywords: Silencing RNA; Euphorbia mosaic virus-Yucatán peninsula; cross-kingdom
Las enfermedades ocasionadas en plantas por patógenos fúngicos son una de las amenazas principales en la seguridad alimentaria (Fernandez-San Millan et al., 2021; Delgado-Baquerizo et al.,2020). Se estima que los hongos son responsables de pérdidas de más del 65% en los cultivos en todo el mundo, las cuales año con año van en aumento debido a factores como el cambio en las prácticas agrícolas, el uso desmedido de fungicidas sintéticos, entre otros, lo que genera la aparición de cepas resistentes y la dificultad para controlar e identificar las infecciones fúngicas en etapas tempranas (Delgado-Baquerizo et al.,2020; Fernandez-San Millan et al., 2021; Sharma y Sharma, 2020).
Entre los principales hongos patógenos del suelo que afectan a las plantas se encuentran los hongos del género Fusarium, los cuales se caracterizan por ser altamente patogénicos y se han relacionado con diferentes enfermedades en más de 200 especies de cultivos a nivel mundial, ocasionando graves pérdidas económicas (Fernandez-San Millan et al., 2021; Sampaio et al., 2020; Summerell, 2019). Estos hongos se caracterizan por invadir el tejido vascular de las plantas e inducir síntomas como el oscurecimiento y la pudrición de la raíz y el tallo, el marchitamiento, el retraso del crecimiento y la defoliación, debido al bloqueo del xilema impidiendo el movimiento del agua y nutrientes en la planta, hasta provocar su muerte (Fernandez-San Millan et al., 2021; Sampaio et al., 2020). Además, el control de estos patógenos se ha dificultado principalmente por su capacidad de habitar en el interior de sus hospederos y por producir estructuras que les permiten permanecer en estado de latencia en el suelo cuando los hospederos están ausentes (Fernandez-San Millan et al., 2021; Panth et al., 2020).
Un enfoque alternativo para el control de plagas y enfermedades es el empleo de ARN de interferencia (ARNi), un mecanismo natural de las plantas, que emplean los ARNi producidos a partir de una infección por virus para generar una resistencia específica como defensa contra ese ataque viral (Baulcombe, 2004). Los ARNi se producen a través de un proceso conocido como silenciamiento génico post-transcripcional. Este mecanismo involucra el reconocimiento de intermediarios de ARN de doble cadena (ARNds) de la etapa de replicación de los virus, que son cortados en ARNs cortos de 21-26 nucleótidos por una enzima tipo RNasa III llamada Dicer. Estos ARN pequeños se incorporan a un complejo silenciador inducido por ARN (RISC, por sus siglas en inglés), que contiene una proteína tipo Argonauta (AGO), con actividad endonucleolítica para el corte del ARN objetivo (Baulcombe, 2004; Van Kammen, 1997; Zhu et al., 2019). Basados en este mecanismo natural, se desarrolló la herramienta denominada silenciamiento génico inducido por virus (VIGS, por sus siglas en inglés), que emplea vectores virales diseñados para transportar el fragmento del gen de interés que se quiere silenciar, lo cual permite el análisis de la función de genes en plantas (Baulcombe, 1999; Bekele et al., 2019) y el control de patógenos, al replicarse de manera sistémica junto con el genoma viral (Lange et al., 2013; Becker and Lange, 2010). El silenciamiento génico ha demostrado eficacia para reducir los síntomas producidos por diferentes especies de Fusarium en plantas de Arabidopsis thaliana, mediante el silenciamiento del gen lanosterol C14α-desmetilasa (Koch et al., 2013); en banano (Musa spp.), con el silenciamiento de los genes relacionados con el desarrollo de los conidios y en la producción de toxinas velvet y ftf (Ghag et al., 2014), en trigo (Triticum aestivum), silenciando el gen CHS3 de la familia de las quitina sintasas (Cheng et al.,2015); en plantas de cebada (Hordeum vulgare) mediante el silenciamiento de los genes AGO y DICER requeridos para el mecanismo de silenciamiento (Werner et al., 2020).
Otros posibles genes blancos para el control de hongos fitopatógenos mediante el silenciamiento génico son las quitinas sintasas (CHS), que son enzimas involucradas en la biosíntesis de quitina, un componente estructural importante de la pared celular fúngica (Roncero et al., 2003; Lenardon et al., 2010). La quitina y las CHS se encuentran ausentes en plantas y mamíferos, por lo que esta familia de genes ha sido considerada como un objetivo seguro y atractivo para el desarrollo de antifúngicos (Martín-Udíroz et al., 2004; Cheng et al., 2015). Los hongos fitopatógenos tienen múltiples genes de CHS y todos ellos presentan diferentes funciones en el desarrollo y en la virulencia (Lenardon et al., 2012; Geoghegan et al., 2017). Se ha reportado que la mutación de la quitina sintasa V puede provocar alteraciones como la disminución de la capacidad infectiva de Fusarium oxysporum en plantas de jitomate (Solanum lycopersicum) (Madrid et al., 2003), pero el silenciamiento de este mismo gen en Fusarium culmorum no está asociado con la reducción de los síntomas en plantas de maíz (Chen et al., 2016). Así mismo, en algunas especies de Fusarium, la evaluación de la CHS4 no ha sido posible mediante la generación de mutantes, pero la evaluación in silico ha reportado la posible existencia de uno o más genes de CHS4 que se ha relacionado con una alta identidad con el gen CHS7; sin embargo, análisis de la actividad de CHS4 en Saccharomyces cerevisiae, demostró que no presentan la misma localización celular ni la misma función en el desarrollo (Larson et al., 2011; Martín-Udíroz et al., 2004, Roncero et al., 2003). Sin embargo, en Penicillium chrysogenum se observó que la mutación de este gen reduce la formación de conidios y presenta hifas atrofiadas, provocando cambios en la producción de penicilina (Liu et al., 2013). Estos reportes demuestran que la función y características de cada quitina sintasa es diferente dependiendo de la especie de hongo y esto también varía de acuerdo con las interacciones que tenga el patógeno con la planta hospedera y su ambiente.
El silenciamiento génico entre organismos de diferentes reinos biológicos es posible mediante el intercambio bidireccional de moléculas, por ejemplo, entre el patógeno y la planta hospedera (Wang et al., 2016), a través del movimiento de moléculas de célula-célula, así como también se han observado diferentes mecanismos asociados al movimiento como el transporte mediante vesículas o proteínas receptoras (Majumdar et al., 2017; Weiberg et al., 2015). Además, se han reportado a patógenos fúngicos como Botrytis cinerea que producen ARNi para inhibir genes de la defensa de las plantas (Wang et al., 2017), pero también se ha inducido la producción de microARN fúngicos en las plantas para controlar a los patógenos, por ejemplo, se ha observado que plantas de algodón (Gossypium hirsutum) transformadas para expresar miR166 y miR159 logran el silenciamiento específico de genes de Verticillium dahliae y de esta manera se genera resistencia contra este patógeno (Zhang et al., 2016). Aunque es un método eficaz, implica la generación de organismos genéticamente modificados (OGM) (Cai et al., 2019).
Una alternativa para la generación de resistencia en las plantas, sin la necesidad de generar cultivos transgénicos, es la producción y transporte de ARNi de plantas a hongos empleando vectores virales que se pueden replicar en plantas (VIGS). Debido al movimiento sistémico de moléculas de ARNi de las plantas hacia los hongos, los modelos de silenciamiento por ARNi presentan una ventaja biotecnológica para su aplicación (Cai et al., 2018). El objetivo de este trabajo fue evaluar la funcionalidad y la eficiencia de un vector VIGS que contenga un fragmento del gen CHS4 de Fusarium sp. para controlar el proceso de infección de este hongo en Nicotiana benthamiana.
Materiales y métodos
Material vegetal. Se emplearon semillas de Nicotiana benthamiana, las cuales fueron germinadas en recipientes de unicel de 120 mL con mezcla fina especial Sunshine #3, el cual contiene peatmoss de fibra corta, cal dolomítica y vermiculita, a 25±2 °C, con fotoperiodo de 16/8 horas luz/oscuridad y fertilización semanal con 4 mL L-1 de Bayfolan® Forte (Bayer de México, S.A. de C.V. División CropScience).
Material fúngico. Se empleó el hongo Fusarium sp. cepa Inecol_BM-06, proporcionada por el Instituto de Ecología A. C (INECOL), el cual fue aislada del insecto ambrosial Xylosandrus morigerus en el parque ecológico Jaguaroundi, en Coatzacoalcos, Veracruz (Carreras-Villaseñor et al., 2022). La cepa se mantuvo en cajas de medio PDA (Potato Dextrose Agar, BD, Difco™, Franklin Lakes, NJ, USA), con resiembras cada 30 días a temperatura ambiente y también se conservó en glicerol al 20% a -80 °C de 6 a 24 meses.
Clonación del fragmento del gen de quitina sintasa 4 de Fusarium sp. El fragmento del gen CHS4 del hongo se amplificó a partir de ADN, usando los oligos CHS4F, 5’-CAAATTATCCTCATGTCGTTC-3’ y CHS4R 5’-TCAGGATCTTTCACCATGGC-3’. Los oligos amplifican una región de 204 pb, que comprende el dominio característico de la quitina sintasa 4 de hongos filamentosos, diseñados a partir de las secuencias de Fusarium graminearum (XM_011318750.1), F. oxysporum (XM_018376225.1) y F. verticillioides (XM_018888350.1), empleando la herramienta “Primer designing tool” del National Center for Biotechology Information (NCBI, por sus siglas en inglés).
Para la estandarización se realizaron pruebas de gradiente de temperatura de alineamiento, de concentración de templado y de concentración de oligos. Las condiciones de PCR óptimas fueron las siguientes: 1 ciclo a 95 °C por 1 min, 30 ciclos de 95 °C por 20 s, 58 °C por 30 s y 72 °C por 20 s y una extensión final a 72 °C por 5 min. El producto de PCR fue ligado en el vector de clonación pGEM-T Easy (Promega®, Madison, Wi, USA), siguiendo las instrucciones del proveedor. Posteriormente los productos de la ligación se utilizaron para transformar células competentes de Escherichia coli TOP10.
Construcción del vector VIGS EuMV-YP∆CHS4. Para la construcción del vector VIGS EuMV-YP∆CHS4 se empleó el vector VIGS pEuMV-YP:ΔAV1, diseñado a partir del hemidímero A del geminivirus bipartita Euphorbia mosaic virus-Yucatán península (EuMV-YP; DQ318937) (Villanueva-Alonzo et al., 2013). Se liberó el fragmento de 204 pb del gen CHS4 clonado en el vector pGEM:GCHS4 con las enzimas de restricción SphI y PstI y se ligó al VIGS pEuMV-YP:ΔAV1 que previamente había sido digerido con las mismas enzimas de restricción. La construcción obtenida fue enviada a secuenciar a la compañía Macrogen Inc (Seúl, Corea del Sur). El análisis de la secuencia se realizó en la plataforma BLASTn del NCBI. Las secuencias fueron editadas con los programas Mega 6 (Tamura et al., 2013) y Biedit (Hall, 1999) para eliminar la secuencia correspondiente al vector.
Inoculación de plantas de N. benthamiana con Fusarium sp. Se empleó un cultivo del hongo de 15-30 días de crecimiento en medio líquido PDB (Potato Dextrose Broth, BD Difco™, Franklin Lakes, NJ, USA), se colectó el micelio y se homogenizó en una licuadora durante 1 minuto a máxima velocidad. La concentración de fragmentos se ajustó a 106 fragmentos de micelio mL-1 y se mezcló con grenetina a una concentración final de 1% (v/v). La mezcla de fragmentos de micelio se aplicó sobre un corte de bisturí de aproximadamente 0.5 mm realizado previamente en el tallo, en el área entre la raíz y el primer peciolo de cada planta. A las plantas control sin inocular (SI) con Fusarium sp. se les colocó grenetina al 1% (v/v) en el punto de corte. Las plantas se mantuvieron en las condiciones de fotoperiodo descritas previamente, durante 14 días o hasta la muerte de la planta.
Silenciamiento promovido por el vector VIGS EuMV-YP∆CHS4 durante la interacción de Fusarium sp.-N. benthamiana. Para analizar el efecto de los tratamientos con el vector VIGS durante el proceso de infección, se observaron los síntomas de caída de hojas y el necrosamiento del tallo en plantas de N. benthamiana. Para ello se evaluaron dos grupos, en cada grupo se emplearon seis plantas de tres semanas de edad por tratamiento y por día de colecta: el primer grupo de plantas de N. benthamiana fueron inoculadas con el hongo cinco días antes de ser bombardeadas con microproyectiles de oro recubiertos con el ADN del vector VIGS (procedimiento descrito posteriormente) y se tomaron muestras de tallo a los 7 y 14 días posteriores al bombardeo (dpB) correspondiente a 12 y 19 días posterior a la inoculación (dpI) con Fusarium sp. Estas plantas se nombraron como E-CHS4 (7 dpB/12 dpI y 14 dpB/19 dpI, respectivamente). El segundo grupo de plantas de N. benthamiana, fueron bombardeadas primero con el vector VIGS y cinco días después del bombardeo fueron inoculadas con el hongo, con las condiciones descritas anteriormente. Se colectaron muestras de tallo a los 7 y 14 dpI. Estas plantas se nombraron como O-CHS4 (7 dpI/ 12 dpB y 14 dpI/ 19 dpB). Todos los experimentos se realizaron con 3 réplicas con 6 plantas por réplica, teniendo un total de 18 plantas por tratamiento/día de colecta en el curso temporal.
Para ambos grupos de plantas evaluadas, se bombardearon con microproyectiles de oro de 1 µm (BioRad, Hercules, California, USA) cubiertos con 10 µg de ADN de cada componente viral; para el componente A se emplearon 5 µg del vector VIGS EuMV-YP∆CHS4 y 5 µg del componente B de EuMV-YP del virus silvestre. El bombardeo se realizó a 2 cm de distancia de la planta y con 30 PSI de gas helio. Los controles empleados fueron plantas SI y plantas únicamente inoculadas con Fusarium sp (I). Se contabilizó el número de hojas pérdidas y la longitud del daño causado por el hongo durante la primera semana.
Análisis por PCR cuantitativo (qPCR) de la expresión relativa del gen CHS4 durante la interacción Fusarium sp. -N. benthamiana. Para evaluar la expresión relativa del gen CHS4, se realizó previamente la extracción del ARN de los tallos colectados, utilizando TRIzol™ Reagent (Invitrogen, Carlsband, CA, USA), siguiendo las indicaciones del proveedor. Posteriormente, para la síntesis de ADNc se emplearon 100-500 ng de ARN total y la SuperScript III Reverse Transcriptase (Invitrogen, Carlsband, CA, USA). Brevemente, se emplearon 200 ng de oligo dT (Invitrogen, Carlsband, CA, USA), se incubó por 5 min a 65 °C y luego en hielo por 1 min, se agregaron 4 µL de first-strand buffer, 1 µL de 0.1M DTT, 1 µL de RNAseOUT™, se incubó a 25 °C por 5 min, posteriormente a 50 °C por 60 min, finalmente se inactivó la reacción incubando a 70 °C por 15 min (modificado de Gerard et al., 1997). Luego se emplearon de 10-20 ng de ADNc para las reacciones de qPCR. Las reacciones se realizaron en el equipo de Applied Biosystems StepOne Real-Time PCR system (Applied Biosystems, Life Technologies, NY, USA) de 48 pozos y usando SYBR Green qPCR SuperMix-UDG (Invitrogen, Carlsband, CA, USA). Las condiciones de qPCR fueron las siguientes: un ciclo de 50 °C por 2 min y 95 °C por 5 min, 40 ciclos de 95 °C por 30s, 58 °C por 1min y 72 °C por 30 s. Los oligos utilizados fueron CHS4F, 5’-CAAATTATCCTCATGTCGTTC-3’ y CHS4R 5’-TCAGGATCTTTCACCATGGC-3’ mencionados anteriormente. Los resultados fueron normalizados con el gen de actina de Fusarium solani (XM_046282608.1). Los tratamientos empleados para evaluar la expresión fueron los siguientes: (1) Plantas sin inocular (2) Plantas inoculadas con el hongo, (3) Plantas inoculadas con el vector, (4) Plantas E-CHS4 7dpB/12dpI, (5) Plantas E-CHS14 dpB/19dpI, (6) Plantas O-CHS4 7dpI/12dpB y (7) Plantas 14dpI/19dpB. Las amplificaciones se realizaron por cuadruplicado y los niveles de expresión relativa se calcularon usando el método 2-ΔΔCT (Livak y Schmittgen, 2001).
Análisis estadístico. Los datos obtenidos de la pérdida de hojas y el tamaño de la lesión fueron analizados mediante la prueba de ANOVA y Tukey post hoc test con un nivel de significancia de p=0.05 usando el software RStudio Team (2022). Antes de la prueba de ANOVA, se usaron la prueba de Shapiro-Wilk para probar la normalidad de los datos y la prueba de Levene para la homogeneidad de las varianzas.
Resultados
Análisis del fragmento de CHS4 clonado. Los resultados obtenidos mostraron que el fragmento del gen clonado presentó un 95% de identidad con el gen de la quitina sintasa 4 de Fusarium vanettenii (XM_003053353.1), 95% de identidad con el gen CHS4 de F. facilforme (CP104050.1) y 94% de identidad con el gen CHS4 de una cepa de F. solani (CP090036.1). Al traducir a proteína el fragmento obtenido de 204 nucleótidos, se observó una secuencia de aminoácidos de 68 residuos (Figura 1), en el que se identificó el dominio DADT, que es uno de los dominios que caracteriza a la familia 4 de las quitinas sintasas, la más distribuida en hongos filamentosos (Pacheco-Arjona y Ramirez-Prado, 2014).

Figura 1 Secuencia de aminoácidos y nucleótidos del fragmento parcial de CHS4 de Fusarium sp. En el recuadro se muestra el dominio DADT, que caracteriza a la familia 4 de las quitinas sintasas de hongo.
Evaluación de la sintomatología de la inoculación de Fusarium en N. benthamiana . Se compararon los síntomas observados en los dos grupos de plantas. Los resultados mostraron que en el tratamiento E-CHS4 la pérdida promedio de hojas fue de 4 por planta durante la primera semana de la infección con Fusarium sp., mientras que para el tratamiento O-CHS4, la pérdida promedio de hojas fue de 3 por planta. En las plantas sanas SI, el promedio de hojas pérdidas fue de 2, esta caída natural fue consecuencia del daño mecánico realizado a la planta. Las plantas control de la infección con el hongo (I) el promedio de pérdida de hojas fue de 6. En el análisis estadístico se observó diferencia significativa en la caída de hojas entre los tratamientos E-CHS4, O-CHS4 y SI con el tratamiento I, pero no se observó diferencia significativa entre los tratamientos E-CHS4 y O-CHS4, así como tampoco se obtuvo diferencia significativa entre el tratamiento O-CHS4 con el control SI, pero si se observó diferencia significativa entre el tratamiento E-CHS4 con el control SI (Figura 2A). Otra variable analizada para establecer la eficiencia de los tratamientos con VIGS fue el tamaño de la lesión causada por el hongo en el tallo de las plantas. El tamaño promedio de las lesiones en los tallos de N. benthamiana con los tratamientos E-CHS4 y O-CHS4 fue de 2 cm, mientras que las plantas con el tratamiento I tuvieron lesiones con un promedio de 3 cm y en las plantas control SI presentaron un tamaño de lesión promedio de 1 cm, este debido al daño mecánico realizado con el bisturí. El análisis estadístico mostró que el promedio del tamaño de la lesión no tuvo diferencia significativa entre los tratamientos E-CHS4 y O-CHS4, pero ambos tratamientos mostraron una diferencia significativa con las plantas I, aquellas únicamente infectadas con Fusarium sp. (Figura 2B). Estos resultados sugieren que los tratamientos con el vector VIGS EuMV-YP∆CHS4 pueden atenuar los síntomas causados por Fusarium sp. en N. benthamia. Durante el curso del experimento se analizaron otros síntomas causados por el hongo como pérdida de turgencia, aumento del marchitamiento y la reducción del crecimiento de las plantas. En la Figura 3A línea 1, se observó que después de los 7dpB/12dpI, las plantas I presentaron síntomas como la pérdida de turgencia de las hojas superiores y el arrugamiento y marchitamiento de las mismas, mientras que las plantas con el tratamiento E-CHS4 mostraron características fisiológicas similares a las plantas control SI (Figura 3A, línea 1); sin embargo, se presentó arrugamiento en las hojas superiores y se observó una disminución del crecimiento en comparación con las plantas con el tratamiento control SI. También se observó el necrosamiento en el tallo (Figura 3A línea 2). El efecto en la atenuación de los síntomas no fue permanente, ya que a los 14 dpB/19 dpI (Figura 3B), las plantas con el tratamiento E-CHS4 mostraron síntomas de marchitamiento y arrugamiento de las hojas superiores, pérdida de turgencia de las hojas y necrosamiento del tallo (Figura 3B línea 1), específicamente el necrosamiento del tallo presentó características similares al observado en las plantas I (Figura 3B línea 2).

Figura 2 Número de hojas perdidas y tamaño promedio de lesión en cm, durante tratamiento E-CHS4 y O-CHS4 con el vector VIGS EuMV-YP∆CHS4. A. Número promedio de hojas perdidas en plantas de N. benthamiana con el tratamiento E-CHS4 y O-CHS4, plantas control sana (SI) y plantas inoculadas con Fusarium sp. (I). B. Tamaño promedio de lesión en cm en plantas con el tratamiento E-CHS4, O-CHS4, SI e I. Las letras arriba de la barra indican las diferencias significativas, con una prueba de análisis de Tukey’s con una p < 0.05. vector treatment 14 days post inoculation with Fusarium sp. I: inoculated control plants.

Figura 3 Análisis de síntomas en plantas de N. benthamiana inoculadas con Fusarium sp. y posteriormente inoculadas con el vector VIGS EuMV-YP∆CHS4 (tratamiento E-CHS4). A. Plantas de N. benthamiana a los 7dpB/12dpI y B. Plantas de N. benthamiana a los 14dpB/19 dpI; SI: plantas control sana, I: plantas inoculadas con Fusarium sp. y E-CHS4: plantas inoculadas con Fusarium sp. e inoculadas con el vector VIGS EuMV-YP∆CHS4. 1: Vista superior y 2: Vista lateral de las plantas.
En el tratamiento O-CHS4, se observó que las plantas a los 7 dpI/12 dpB mostraron menor nivel de síntomas en comparación con las plantas I y mostraron características similares a las plantas control SI (Figura 4A línea 1), solo se observó el daño en la base del tallo (Figura 4A línea 2). La disminución de los síntomas en este tratamiento no fue un efecto permanente, ya que a los 14dpI/19dpB (Figura 4B línea 1), los síntomas de marchitamiento y pérdida de turgencia de las hojas aumentó y fue similar a lo observado en las plantas control de infección con el hongo I. El daño de la base del tallo de las plantas con el tratamiento O-CHS4 fue similar a lo observado en las plantas I (Figura 4B línea 2). Estos resultados mostraron que el vector VIGS EuMV-YP∆CHS4 es capaz de retrasar los síntomas en la interacción Fusarium sp.-N. benthamiana, durante el establecimiento de la infección, pero este efecto es transitorio.

Figura 4 Análisis de síntomas en plantas de N. benthamiana inoculadas con el vector VIGS EuMV-YP∆CHS4 y posteriormente inoculadas con Fusarium sp. (tratamiento O-CHS4). A. Plantas a los 7dpI/12dpB. B. Plantas con 14 dpI y 19 dpB; SI: plantas control sana, I: plantas inoculadas con Fusarium sp y E-chs4: plantas inoculadas con Fusarium sp. e inoculadas con el vector VIGS EuMV-YP∆CHS4. 1: Vista superior y 2: Vista lateral de las plantas.
Expresión relativa del gen CHS4 de Fusarium sp. durante su interacción con plantas de N. benthamiana. En las plantas con el tratamiento E-CHS4 se observó que después de los 7 dpB la expresión del gen CHS4 se incrementó cuatro veces más comparado con el control I, en cambio a los 14 dpB con el vector VIGS EuMV-YP∆CHS4, la expresión del gen CHS4 disminuyó su expresión relativa un 20% con respecto al control infectado (Figura 5). En el caso de las plantas con el tratamiento O-CHS4 se observó una disminución en la expresión relativa de CHS4 después de los 7 dpI, del 30% con respecto a las plantas control infectadas, sin embargo, a los 14 dpI la expresión relativa del gen CHS4 aumentó 2.5 veces con respecto al control infectado (Figura 6). Estos resultados indican que el efecto observado en la sintomatología de las plantas E-CHS4 y O-CHS4 puede estar relacionado con el silenciamiento transitorio del gen CHS4, producto de la presencia del vector VIGS EuMV-YP∆CHS4 y su replicación en las plantas de N. benthamiana.
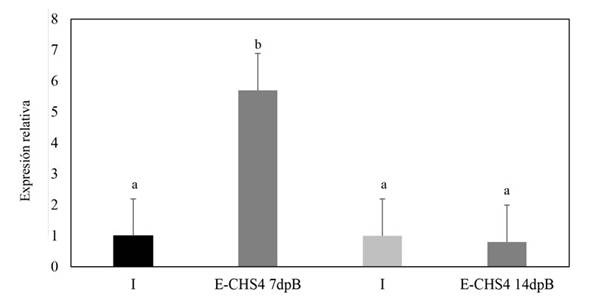
Figura 5 Cuantificación relativa mediante análisis qPCR del fragmento del gen CHS4 de Fusarium sp. en tallos de plantas con el tratamiento E-CHS4. Se empleó el gen de la actina de Fusarium para normalizar los datos. E-CHS4 7dpB: Expresión relativa de CHS4 en plantas inoculadas con Fusarium sp. a los siete días con el tratamiento E-CHS4; E-CHS4 14dpB: expresión relativa de CHS4 en plantas de N. benthamiana con Fusarium sp. a los 14 días con el tratamiento E-CHS4. I: Plantas control inoculadas.
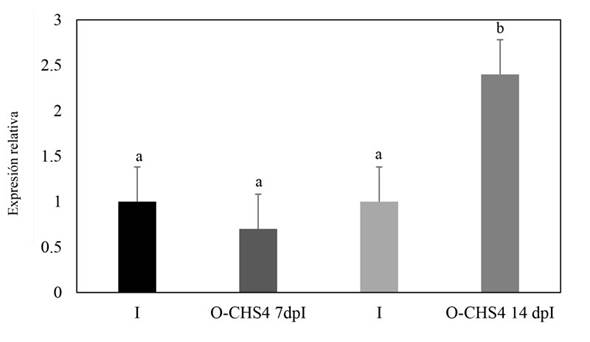
Figura 6 Cuantificación relativa mediante análisis qPCR del fragmento del gen CHS4 de Fusarium sp. en tallos de plantas con el tratamiento O-CHS4. Se empleó el gen de la actina de Fusarium para normalizar los datos. O-CHS4 7dpI: Expresión relativa de CHS4 en plantas de N. benthamiana con tratamiento con el vector VIGS EuMV-YP∆CHS4 siete días posterior a la inoculación con Fusarium sp.; O-CHS4 14 dpI: expresión relativa de CHS4 en plantas de N. benthamiana con tratamiento con el vector EuMV-YP∆CHS4 14 días posteriores a la inoculación con Fusarium sp. I: plantas control inoculadas.
Discusión
En la interacción planta-patógeno el intercambio bidireccional permite el establecimiento de una infección por parte del patógeno, así como la activación de la defensa del hospedero (Cai et al., 2019). En este estudio, se evaluó el efecto de la aplicación de un vector VIGS basado en el Euphorbia mosaic virus-Yucatán península (EuMV-YP), para inducir el silenciamiento del gen CHS4 de Fusarium sp. durante la interacción con N. benthamiana. Los resultados obtenidos demostraron la disminución de síntomas en las plantas infectadas con Fusarium sp. y bombardeadas con el vector VIGS EuMV-YP∆CHS4, sin la producción de plantas transgénicas estables o del uso de mutantes del hongo, que son los métodos empleados para el análisis de genes fúngicos.
Los sistemas VIGS son una herramienta desarrollada originalmente para el análisis génico en plantas, pero el desarrollo de nuevos protocolos y vectores virales han demostrado su utilidad para el análisis en hongos, como una estrategia directa y fácil de realizar, reduciendo costos y tiempo de trabajo (Tinoco et al., 2010; Mascia et al., 2014). La aplicación del vector VIGS EuMV-YP∆CHS4 disminuyó los síntomas característicos provocados por la infección con Fusarium sp. en plantas de N. benthamiana, como la caída de hojas y el tamaño de la lesión (Figura 2). Resultados similares se reportaron en trabajos en los que se aplica la tecnología HIGS (host inducing gene silencing), por ejemplo, en plantas de A. thaliana que expresaban un ARNds contra el gen de la citocromo P450 lanosterol C-14α-demetilasa (CYP5) de F. graminearum, las plantas infectadas con el hongo no presentaron síntomas de infección en comparación con el control infectado sin transformar con el ARNds a los tres días después de la inoculación, además, mostraron un porcentaje de área foliar infectada de 0.9%, en contraste con el 77% que presentó el control infectado (Koch et al., 2013). Por otro lado, en plantas de trigo que expresaban ARNds con estructura de horquilla para el gen de la quitina sintasa 3 (Chs3b) de F. graminearum, presentaron síntomas débiles de infección y una reducción en la longitud de la lesión de 8.5 mm a 6.7, 6.6, 5.7 y hasta 4.5 mm en todas las construcciones evaluadas para este gen (Cheng et al., 2015). En el caso de tecnologías de silenciamiento como SIGS (Spray-induced gene silencing), Höfle et al. (2020), mostraron que la aplicación de ARNds del gen CYP5 de diferentes longitudes en hojas de A. thaliana reducía los síntomas de infección por F. graminearum en hasta un 82% en comparación del control. Este resultado es comparable con lo observado en este trabajo empleando VIGS, donde se reportó una reducción del tamaño de la lesión de 3 cm presente en las plantas inoculadas unicamente con Fusarium sp. a 2 cm en las plantas que contenían el vector VIGS EuMV-YP∆CHS4. Con base en estos datos se sugiere que los mecanismos de silenciamiento presentarán diferentes respuestas y fenotipos, aún cuando se aplique a genes con actividades relacionadas o a la misma especie de hongo.
Si bien la técnica de HIGS se ha presentado como una estrategia para el estudio de la interacción hongo-planta, esta herramienta se realiza mediante la generación de plantas transgénicas capaces de producir ARNi (Nowara et al., 2010; Panwar et al., 2013; Cheng et al., 2015), lo que provoca que tenga poca aceptación para su uso por los consumidores y poca confiabilidad debido al desconocimiento de sus repercusiones ambientales (Cai et al., 2019; Wang et al., 2016), por lo que el empleo de los vectores VIGS es un mecanismo alternativo al sistema HIGS, como parte de la nueva generación de fungicidas basados en ARNs (Padilla-Roji et al., 2023).
Se ha reportado previamente la aplicación de VIGS para silenciar la expresión de genes de hongos, utilizando al Barley stripe mosaic virus (BSMV) como vehículo VIGS, para expresar ARNds dirigidos a genes asociados al haustorio de Puccinia triticina f. sp. tritici (Pst) (Yin et al., 2011). Estos resultados mostraron que el silenciamiento fue más eficiente en las células del haustorio que estaban en mayor contacto con las células del hospedero (Yin et al., 2011). Por otra parte, en plantas de trigo utilizando al BSMV como VIGS, se evaluaron ARNi contra tres genes de Puccinia triticina (Pt), el gen PtCYC1 (cyclophilin), PtMAPK1 (una proteína quinasa) y PtCNB (una unidad regulatoria de calcineurina), los cuales participan en la patogenicidad. En este estudio se observó que el silenciamiento de cualquiera de los tres genes blanco produjo una disminución en el tamaño de la lesión ocasionada durante el proceso infectivo (Panwar et al., 2013). Así mismo, el análisis del silenciamiento del efector PstCFEM de Pst, empleando BSMV en plantas de trigo, mostraron la reducción en la esporulación, lo que disminuyó significativamente la biomasa del hongo y el área de infección (Bai et al., 2022). En este trabajo, empleando al EuMV-YP como vector VIGS, se observó la disminución del daño foliar (caída de las hojas y arrugamiento), así como la disminución del tamaño de la lesión por la infección con Fusarium sp., como efecto de la presencia del vector VIGS EuMV-YP∆CHS4. Lo anterior nos sugiere que la aplicación de vectores VIGS son alternativas no transgénicas, novedosas y amigables para el medio ambiente, así como para el estudio y análisis de nuevas herramientas contra el combate de patógenos y de plagas de insectos de plantas (Coleman, 2016; Dinolfo et al., 2017; Martín-Udíroz et al., 2004), así mismo, muestra las posibilidades de aplicación de vectores VIGS basados en el EuMV-YP.
Además de las diferencias observadas en la sintomatología de las plantas infectadas con Fusarium sp. y tratadas con el vector VIGS EuMV-YP∆CHS4 en comparación con las plantas que solo fueron infectadas con Fusarium sp., también se observó el silenciamiento de hasta el 20% en la expresión del gen CHS4 en las plantas con el tratamiento E-CHS4 (Figura 5) y de 30% de silenciamiento en las plantas con el tratamiento O-CHS4 (Figura 6). Con todo, el silenciamiento de solo el gen de quitina sintasa 4 no fue suficiente para evitar el desarrollo de la infección del hongo y por lo tanto no permitió la supervivencia o recuperación de las plantas.
Este efecto de pérdida de silenciamiento puede estar relacionado con la multiplicidad de las quitinas sintasas, ya que se ha reportado que la perturbación en la síntesis de la pared puede desencadenar una respuesta compensatoria para garantizar la integridad de la pared celular de hongos, principalmente un aumento en la síntesis de quitina a través de otros miembros de las quitina sintasa (Kappel et al., 2020); pero también están involucrados procesos como la sobre-expresión de enzimas como las desacetilasas, que permiten la conversión de quitina a quitosano (Kappel et al., 2020). Además, se ha observado que la expresión de uno o más genes de quitina sintasa está relacionada con la etapa de desarrollo del hongo (Roncero et al., 2003; Kappel et al., 2020). Por ejemplo: mutantes de F. oxysporum en un solo gen quitina sintasa (1, 2 o 7) no mostraron diferencias en la morfología de las colonias o la producción de quitina comparadas con la cepa silvestre (Martín-Udíroz et al., 2004). Mientras que la eliminación del gen CHS5 en cepas de F. oxysporum mostraron hifas hinchadas e hipersensibles al H2O2 (Roncero et al., 2003), demostrando que la pérdida de actividad de un solo gen de quitina sintasa puede generar cambios estructurales importantes, mientras que en otras interacciones está perdida puede no influenciar en la virulencia o desarrollo del hongo: por ejemplo, en plantas de jitomate infectadas con una cepa de F. oxysporum con una mutante letal del gen CHS5, mostró una disminución en su capacidad para colonizar eficientemente el sistema vascular del jitomate, además de presentar menor tasa de crecimiento (Madrid et al., 2003), mientras que el análisis de cepas mutantes letales de los genes CHS1 y CHS2 de F. oxysporum no generaron efectos en el contenido de quitina ni en la esporulación, además de que fue imposible obtener una mutante viable del gen CHS3 (Martín-Udíroz et al., 2004). Por otro lado, Cheng et al. (2015), expresaron construcciones del CHS3 que silenciaron la expresión del gen en F. graminearum durante su interacción en plantas de trigo, lo que resultó en un 61% menos de espigas infectadas.
Por otro lado, la interrupción del gen CHS4 no provocó cambios en el fenotipo de B. cinerea, pero en especies como Aspergillus sp. y Neurospora crassa, donde se ha reportado como una enzima con actividad redundante, su eliminación no produjo cambios morfológicos aparentes en la pared del hongo, pero generó una disminución en la concentración de quitina de la pared (Din et al., 1996). En Saccharomyces cerevisiae, por el contrario, el producto del gen CHS4, es el responsable de la producción del 90% de la quitina de la pared celular y además está relacionado con el cambio en el ciclo sexual (Morcx et al., 2013). Así mismo, el silenciamineto del gen CHS4 de Penicillium chrysogenum, disminuyó 91% de la expresión del gen lo que resultó en la reducción del tamaño de la colonia y del número de conidios (Liu et al., 2013). Por lo contrario, en especies de hongos como F. oxysporum, el aislamiento de CHS4 no fue posible y solo mediante análisis in silico con datos de F. graminearum, se determinó la existencia de uno o más genes CHS4 en F. oxysporum, así como su relación con CHS7 (Martín-Udíroz et al., 2004). Por ello, el análisis de genes múltiples o genes difíciles de aislar mediante la tecnología VIGS evita la necesidad de eliminarlos completamente, lo que permite estudiar genes esenciales, cuya mutación es letal (Mascia et al., 2014). En este trabajo, mediante VIGS se observó el silenciamiento transitorio de CHS4 de Fusarium sp., lo que permite analizar su posible participación en el proceso de infección en N. benthamiana y sugiere la posible aplicación de vectores VIGS basados en EuMV-YP, para el análisis de otros genes pertenecientes a esta familia.
Con respecto a los resultados obtenidos en ambos tratamientos, E-CHS4 y O-CHS4, se observó una disminución de los síntomas en las plantas infectadas con el hongo, independientemente del momento de inoculación con el vector VIGS EuMV-YP∆CHS4. Previamente, Villanueva-Alonzo et al. (2013) y Luna-Rivero et al. (2016) demostraron que el vector pEuMV-YP:∆AV1 se mueve de manera sistémica y uniforme durante la infección, permitiendo la replicación del fragmento del gen a evaluar, por lo que el silenciamiento observado del gen CHS4 de Fusarium sp. puede deberse al movimiento de los ARNi del sitio de infección en N. benthamiana hacia toda la planta y a las células del hongo. Esta es una ventaja del silenciamiento mediado por VIGS sobre otros enfoques de ARNi en las plantas, donde los ARNi pueden moverse de manera sistémica desde el sitio de inoculación hacia todo el organismo (Mascia et al., 2014).
Diversos estudios han demostrado que el intercambio de ARNs y ARNi entre diferentes reinos durante la interacción planta-patógeno, es un proceso bidireccional que permite a los patógenos suprimir la defensa de las plantas, pero que también proporciona a las plantas resistencia a las enfermedades hasta en un 60% (Mapuranga et al., 2023). Al ser un proceso bidireccional entre patógeno y hospedero, es posible encontrar evidencia de estos intercambios, por ejemplo: Zhang et al. (2016), reportaron que al aislar el hongo Verticillium dhaliae de plantas de algodón, obtuvieron secuencias de microARN que eran compatibles principalmente con secuencias de microARN de algodón más que con el hongo, lo que sugiere que estos ARNs se transportaron del algodón al hongo durante el proceso de infección. En otro reporte se demostró la capacidad de las células fúngicas de B. cinerea para absorber exomas de plantas, a través de la exposición in vitro del hongo con exomas de A. thaliana marcados con TET8-GFP (un marcador específico de exomas, similar al observado en mamíferos). Se concluyó que después de dos horas los exomas se encontraban dentro de las células fúngicas y se mantuvieron hasta que éstas fueron lisadas con Tritón X-100 (Cai et al., 2018). Por otro lado, se ha reportado que la aspersión de ARNds sobre hojas de cebada inhibió cerca del 50% de la infección con F. graminearum mediante construcciones cuyo blanco fueron los genes AGO y DCL (genes que además de participar en el silenciamiento, en F. graminearum contribuyen en la producción de micotoxinas y en el desarrollo de la enfermedad en la planta); esto demuestra la capacidad de los patógenos fúngicos para absorber los ARN del ambiente (Werner et al., 2020).
Los resultados obtenidos mostraron que la tecnología VIGS permite el análisis de un gen durante la interacción planta-hongo, sin la necesidad de desarrollar protocolos para la transformación de hongos o la generación de plantas transgénicas, como sucede con metodologías como HIGS (Hua et al., 2018), así como evitar la dependencia a la obtención de cultivares resistentes o el riesgo de la inestabilidad que pueden presentan los ARNds en la metodología SIGS (Hua et al., 2018).
Conclusiones
El vector VIGS EuMV-YP∆CHS4, basado en el EuMV-YP, tuvo la capacidad de silenciar parcialmente al gen CHS4, durante el proceso infectivo de Fusarium sp. en plantas de N. benthamiana. El principal efecto del silenciamiento provocado por el vector VIGS EuMV-YP∆CHS4 fue la reducción de síntomas como menor caída de hojas, disminución del tamaño de la lesión en tallo, así como menor pérdida de turgencia en las plantas. La evaluación de este vector VIGS durante la interacción planta- patógeno, demuestra su potencial para estudiar la importancia de los genes en la viabilidad o patogenicidad de los hongos.
Agradecimientos
Este trabajo fue financiado por el proyecto FORDECYT “Generación de estrategias científico-tecnológicas con un enfoque multidisciplinario e interinstitucional para afrontar la amenaza que representan los complejos ambrosiales en los sectores agrícola y forestal de México” (#No. 292399). Agradecemos la beca de doctorado otorgada a Nidia Esther Hau Yama (Consejo Nacional de Ciencia y Tecnología “CONACYT”, CVU:694530). Agradecemos al IQI. Alberto Cortés Velázquez por su apoyo con el qPCR y al M.C Bartolomé Chí Manzanero (qepd) por su apoyo en el cuidado del hongo.
REFERENCIAS
Bai X, Peng H, Goher F, Islam MA, Xu S, Guo J, Kang Z and Guo J. 2022. A candidate effector protein PstCFEM1 contributes to virulence of stripe rust fungus and impairs wheat immunity. Stress Biology 2: 21. https://doi.org/10.1007/s44154-022-00042-5 [ Links ]
Baulcombe DC. 1999. Fast forward genetics based on virus-induced gene silencing. Current Opinion in Plant Biology 2(2): 109-113. https://doi.org/10.1016/S1369-5266(99)80022-3 [ Links ]
Baulcombe DC. 2004. RNA silencing in plants. Nature 431(1): 356-363. https://doi.org/10.1038/nature02874 [ Links ]
Becker A and Lange M. 2010. VIGS - genomics goes functional. Trends in Plant Science 15(1): 1-4. https://doi.org/10.1016/j.tplants.2009.09.002 [ Links ]
Bekele D, Tesfaye K and Fikre A. 2019. Applications of virus induced gene silencing (VIGS) in plant functional genomics studies. Journal of Plant Biochemistry & Physiology 7(1): 1-7. https://doi.org/10.4172/2329-9029.1000229 [ Links ]
Cai Q, He B, Weiberg A, Buck AH and Jin H. 2019. Small RNAs and extracellular vesicles: New mechanisms of cross-species communication and innovative tools for disease control. PLoS Pathogens 15(12): 1-13. https://doi.org/10.1371/journal.ppat.1008090 [ Links ]
Cai Q, Qiao L, Wang M, He B, Lin FM, Palmquist J, Huang SD and Jin H. 2018. Plants send small RNAs in extracellular vesicles to fungal pathogen to silence virulence genes. Science 360(6393): 1126-1129. https://doi.org/10.1126/ciencia.aar4142 [ Links ]
Carreras-Villaseñor N, Rodríguez-Haas JB, Martínez-Rodríguez LA, Pérez-Lira AJ, Ibarra-Laclette E, Villafán E, Castillo-Díaz AP, Ibarra-Juárez LA, Carrillo-Hernández ED and Sánchez-Rangel D. 2022. Characterization of two Fusarium solani species complex isolates from the ambrosia beetle Xylosandrus morigerus. Journal of Fungi 8(3): 231. https://doi.org/10.3390/jof8030231 [ Links ]
Chen W, Kastner C, Nowara D, Oliveira-Garcia E, Rutten T, Zhao Y, Deising HB, Kumlehn J and Schweizer P. 2016. Host-induced silencing of Fusarium culmorum genes protects wheat from infection. Journal of Experimental Botany 67(17): 4979-4991. https://doi.org/10.1093/jxb/erw263 [ Links ]
Cheng, W., Song, XS., Li, HP., Cao, LH., Sun, K., Qiu, XL., Xu, YB., Yang, P., Huang, T., Zhang, JB., Qu, B. and Liao, YC. 2015. Host-induced gene silencing of an essential chitin synthase gene confers durable resistance to Fusarium head blight and seedling blight in wheat. Plant Biotechnology Journal 13(9): 1335-1345. https://doi.org/10.1111/pbi.12352 [ Links ]
Coleman JJ. 2016. The Fusarium solani species complex: Ubiquitous pathogens of agricultural importance. Molecular Plant Pathology 17(2): 146-158. https://doi.org/10.1111/mpp.12289 [ Links ]
Delgado-Baquerizo M, Guerra CA, Cano-Díaz C, Egidi E, Wang JT, Eisenhauer N, Singh BK and Maestre FT. 2020. The proportion of soil-borne pathogens increases with warming at the global scale. Nature Climate Change 10(6): 550-554. https://doi.org/10.1038/s41558-020-0759-3 [ Links ]
Din AB, Specht CA, Robbins P.W and Yarden O. 1996. Chs-4, a class IV chitin synthase gene from Neurospora crassa. Molecular and General Genetics MGG 250(2): 214-222. https://doi.org/10.1007/BF02174181 [ Links ]
Dinolfo MI, Castañares E and Stenglein SA. 2017. Fusarium-plant interaction: state of the art - a review. Plant Protection Science 53(2): 61-70. https://doi.org/10.17221/182/2015-PPS [ Links ]
Fernandez-San Millan A, Larraya L, Farran I, Ancin M and Veramendi J. 2021. Successful biocontrol of major postharvest and soil-borne plant pathogenic fungi by antagonistic yeasts. Biological Control 160: 104683. https://doi.org/10.1016/j.biocontrol.2021.104683 [ Links ]
Geoghegan I, Steinberg G and Gurr S. 2017. The role of the fungal cell wall in the infection of plants. Trends in Microbiology 25(12): 957-967. https://doi.org/10.1016/j.tim.2017.05.015 [ Links ]
Gerard GF, Fox DK, Nathan M and D’Alessio JM. 1997. Reverse transcriptase. The use of cloned Moloney murine leukemia virus reverse transcriptase to synthesize DNA from RNA. Molecular Biotechnology 8(1):61-77. https://doi.org/10.1007/BF02762340 [ Links ]
Ghag SB, Shekhawat UKS, and Ganapathi TR. 2014. Host-induced post-transcriptional hairpin RNA-mediated gene silencing of vital fungal genes confers efficient resistance against Fusarium wilt in banana. Plant Biotechnology Journal 12(5): 541-553. https://doi.org/10.1111/pbi.12158 [ Links ]
Hall TA. 1999. BioEdit a user-friendly biological sequences alignment editor and analysis program for Windows 9598NT. Nucleic Acids Symposium Series 41: 95-98. https://www.academia.edu/2034992/BioEdit_a_user_friendly_biological_sequence_alignment_editor_and_analysis_program_for_Windows_95_98_NT [ Links ]
Höfle L, Biedenkopf D, Werner BT, Shrestha A, Jelonek L and Koch A. 2020. Study on the efficiency of dsRNAs with increasing length in RNA-based silencing of the Fusarium CYP51 genes. RNA biology 17(4): 463-473. https://doi.org/10.1080/15476286.2019.1700033 [ Links ]
Hua C, Zhao JH and Guo HS. 2018. Trans-kingdom RNA silencing in plant-fungal pathogen interactions. Molecular plant 11(2): 235-244. https://doi.org/10.1016/j.molp.2017.12.001 [ Links ]
Kappel L, Münsterkötter M, Sipos G, Rodriguez CE and Gruber S. 2020. Chitin and chitosan remodeling defines vegetative development and Trichoderma biocontrol. PLoS Pathogens 16(2): e1008320. https://doi.org/10.1371/journal.ppat.1008320 [ Links ]
Koch A, Kumar N, Weber L, Keller H, Imani J and Kogel KH. 2013. Host-induced gene silencing of cytochrome P450 lanosterol C14α-demethylase-encoding genes confers strong resistance to Fusarium species. Proceedings of the National Academy of Sciences of the United States of America 110(48): 19324-19329. https://doi.org/10.1073/pnas.1306373110 [ Links ]
Lange M, Yellina AL, Orashakova S and Becker A. 2013. Virus-induced gene silencing (VIGS) in plants: an overview of target species and the virus-derived vector systems. Pp: 1-14. In: Becker A(ed). Virus-Induced gene silencing: Methods and protocols; Methods in molecular biology, Humana Press. Gieben, Alemania. 975p. https://doi.org/10.1007/978-1-62703-278-0_1 [ Links ]
Larson TM, Kendra DF, Busman M and Brown DW. 2011. Fusarium verticillioides chitin synthases CHS5 and CHS7 are required for normal growth and pathogenicity. Current Genetics 57(3): 177-189. https://doi.org/10.1007/s00294-011-0334-6 [ Links ]
Lenardon MD, Munro CA and Gow NAR. 2010. Chitin synthesis and fungal pathogenesis. Current Opinion in Microbiology 13(4): 416-423. https://doi.org/10.1016/j.mib.2010.05.002 [ Links ]
Liu H, Wang P, Gong G, Wang L, Zhao G and Zheng Z. 2013. Morphology engineering of Penicillium chrysogenum by RNA silencing of chitin synthase gene. Biotechnology Letters 35(3): 423-429. https://doi.org/10.1007/s10529-012-1099-9 [ Links ]
Livak KJ and Schmittgen TD. 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods 25(4): 402-408. https://doi.org/10.1006/meth.2001.1262 [ Links ]
Luna-Rivero MS, Hernández-Zepeda C, Villanueva-Alonzo H, Minero-García Y, Castell-González SE and Moreno-Valenzuela OA. 2016. Expression of genes involved in the salicylic acid pathway in type h1 thioredoxin transiently silenced pepper plants during a begomovirus compatible interaction. Molecular Genetics and Genomics 291(2): 819-830. https://doi.org/10.1007/s00438-015-1148-x [ Links ]
Madrid MP, Di Pietro A and Roncero MIG. 2003. Class V chitin synthase determines pathogenesis in the vascular wilt fungus Fusarium oxysporum and mediates resistance to plant defence compounds. Molecular Microbiology 47(1): 257-266. https://doi.org/10.1046/j.1365-2958.2003.03299.x [ Links ]
Majumdar R, Rajasekaran K and Cary JW. 2017. RNA interference (RNAi) as a potential tool for control of mycotoxin contamination in crop plants: Concepts and considerations. Frontiers in Plant Science 8: 200. https://doi.org/10.3389/fpls.2017.00200 [ Links ]
Martín-Udíroz M, Madrid MP and Roncero MIG. 2004. Role of chitin synthase genes in Fusarium oxysporum. Microbiology 150(10): 3175-3187. https://doi.org/10.1099/mic.0.27236-0 [ Links ]
Mascia T, Nigro F, Abdallah A, Ferrara M, de Stradis A, Faedda R, Palukaitis P and Gallitelli D. 2014. Gene silencing and gene expression in phytopathogenic fungi using a plant virus vector. Proceedings of the National Academy of Sciences of the United States of America 111(11): 4291-4296. https://doi.org/10.1073/pnas.1315668111 [ Links ]
Mapuranga J, Chang J, Zhang L, Zhang N, Yang W. 2022. Fungal secondary metabolites and small RNAs enhance pathogenicity during plant-fungal pathogen interactions. Journal Fungi 20; 9(1):4. https://doi.org/10.3390/jof9010004 [ Links ]
Morcx S, Kunz C, Choquer M, Assie S, Blondet E, Simond-Côte E, Gajek K, Chapeland-Leclerc F, Expert D and Soulie MC. 2013. Disruption of Bcchs4, Bcchs6 or Bcchs7 chitin synthase genes in Botrytis cinerea and the essential role of class VI chitin synthase (Bcchs6). Fungal Genetics and Biology 52: 1-8. https://doi.org/10.1016/j.fgb.2012.11.011 [ Links ]
Nowara D, Gay A, Lacomme C, Shaw J, Ridout C, Douchkov D, Hensel G, Kumlehn J and Schweizer P. 2010. HIGS: Host-induced genen silencing in the oblígate biotrophic fungal pathogen Blumeria graminis. The Plant Cell 22(9): 3130-3141. https://doi.org/10.1105/tpc.110.077040 [ Links ]
Pacheco-Arjona JR and Ramirez-Prado JH. 2014. Large-scale phylogenetic classification of fungal chitin synthases and identification of a putative cell-wall metabolism gene cluster in aspergillus genomes. PLoS ONE 9(8): e104920. https://doi.org/10.1371/journal.pone.0104920 [ Links ]
Padilla-Roji I, Ruiz-Jiménez L, Bakhat N, Vielba-Fernández A, Pérez-García A and Fernández-Ortuño D. 2023. RNAi Technology: A new path for the research and management of obligate biotrophic phytopathogenic fungi. International Journal of Molecular Sciences 24(10): 9082. https://doi.org/10.3390/ijms24109082 [ Links ]
Panth M, Hassler SC and Baysal-Gurel F. 2020. Methods for management of soilborne diseases in crop production. Agriculture 10(1): 16. https://doi.org/10.3390/agriculture10010016 [ Links ]
Panwar V, McCallum B and Bakkeren G. 2013. Host-induced gene silencing of wheat leaf rust fungus Puccinia triticina pathogenicity genes mediated by the Barley stripe mosaic virus. Plant Molecular Biology 81(6): 595-608. https://doi.org/10.1007/s11103-013-0022-7 [ Links ]
Roncero MIG, Hera C, Ruiz-Rubio M, García-Maceira FI, Madrid MP, Caracuel Z, Calero F, Delgado-Jarana J, Roldán-Rodríguez R, Martínez-Rocha AL, Velasco C, Roa J, Martín-Urdiroz M, Córdoba D and Di Pietro A. 2003. Fusarium as a model for studying virulence in soilborne plant pathogens. Physiological and Molecular Plant Pathology 62(2): 87-98. https://doi.org/10.1016/S0885-5765(03)00043-2 [ Links ]
RStudio Team. 2022. RStudio: Integrated development environment for R. Rstudio, PBC, Boston, MA. http://www.rstudio.com/ [ Links ]
Sampaio AM, de Sousa Araújo S, Rubiales D and Patto MCV. 2020. Fusarium wilt management in legume crops. Agronomy 10(8): 1073. https://doi.org/10.3390/agronomy10081073 [ Links ]
Sharma IP and Sharma AK. 2020. Trichoderma-Fusarium Interactions: A Biocontrol Strategy to Manage Wilt pp. 167-185. In: Sharma, A. K and Sharma, P. (eds). Trichoderma: Host pathogen interactions and applications, Springer nature Singapore. Pantnagar, Uttarakhand, India. 331p. https://doi.org/10.1007/978-981-15-3321-1_9 [ Links ]
Summerell BA. 2019. Resolving Fusarium: Current status of the genus. Annual Review of Phytopathology 57(1): 323-339. https://doi.org/10.1146/annurev-phyto-082718-100204 [ Links ]
Tamura K, Stecher G, Peterson D, Filipski A and Kumar S. 2013. MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution 30(12): 2725-2729. https://doi.org/10.1093/molbev/mst197 [ Links ]
Tinoco MLP, Dias BBA, Dall’Astta RC, Pamphile JA and Aragão FJL. 2010. In vivo trans-specific gene silencing in fungal cells by in planta expression of a double-stranded RNA. BMC Biology 8(1):1-11. https://doi.org/10.1186/1741-7007-8-27 [ Links ]
Van Kammen A. 1997. Virus-induced gene silencing in infected and transgenic plants. Trends in Plant Science 2(11): 409-411. https://doi.org/10.1016/S1360-1385(97)01128-X [ Links ]
Villanueva-Alonzo HJ, Us-Camas RY, López-Ochoa LA, Robertson D, Guerra-Peraza O, Minero-García Y and Moreno-Valenzuela OA. 2013. A new virus-induced gene silencing vector based on Euphorbia mosaic virus-Yucatan peninsula for NPR1 silencing in Nicotiana benthamiana and Capsicum annuum var. Anaheim. Biotechnology Letters 35(5): 811-823. https://doi.org/10.1007/s10529-013-1146-1 [ Links ]
Wang M, Weiberg A, Dellota E, Yamane D and Jin H. 2017. Botrytis small RNA Bc-siR37 suppresses plant defense genes by cross-kingdom RNAi. RNA Biology 14(4): 421-428. https://doi.org/10.1080/15476286.2017.1291112 [ Links ]
Wang M, Weiberg A, Lin FM, Thomma BPHJ, Huang H Da and Jin H. 2016. Bidirectional cross-kingdom RNAi and fungal uptake of external RNAs confer plant protection. Nature Plants 2(10), 1-10. https://doi.org/10.1038/nplants.2016.151 [ Links ]
Weiberg A, Bellinger M and Jin H. 2015. Conversations between kingdoms: Small RNAs. Current Opinion in Biotechnology 32: 207-215. https://doi.org/10.1016/j.copbio.2014.12.025 [ Links ]
Werner BT, Gaffar FY, Schuemann J, Biedenkopf D and Koch AM. 2020. RNA-spray-mediated silencing of Fusarium graminearum AGO and DCL genes improve barley disease resistance. Frontiers in Plant Science 11:476. https://doi.org/10.3389/fpls.2020.00476 [ Links ]
Yin C, Jurgenson JE and Hulbert SH. 2011. Development of a Host-Induced RNAi system in the wheat stripe rust fungus Puccinia striiformis f. sp. tritici. Molecular Plant-Microbe Interactions 24(5): 554-561. https://doi.org/10.1094/MPMI-10-10-0229 [ Links ]
Zhang T, Zhao Y-L, Zhao J-H, Wang S, Jin Y, Chen Z-Q, Fang Y-Y, Hua C-L, Ding S-W and Guo HS. 2016. Cotton plants export microRNAs to inhibit virulence gene expression in a fungal pathogen. Nature Plants 2(10): 16153. https://doi.org/10.1038/nplants.2016.153 [ Links ]
Zhu C, Liu T, Chang YN and Duan CG. 2019. Small RNA functions as a trafficking effector in plant immunity. International Journal of Molecular Sciences 20(11): 2816. https://doi.org/10.3390/ijms20112816 [ Links ]
Recibido: 24 de Marzo de 2023; Aprobado: 13 de Agosto de 2023











 texto em
texto em 


